基于16S rDNA序列的2株氣單胞菌屬細菌的分子鑒定
譚鳳霞,葉 聰 (長江大學動物科學學院, 湖北 荊州 434025)
基于16S rDNA序列的2株氣單胞菌屬細菌的分子鑒定
譚鳳霞,葉 聰 (長江大學動物科學學院, 湖北 荊州 434025)
對分離自患病鯽魚的2株氣單胞菌的16S rDNA序列進行PCR擴增并測定其核酸序列,通過NCBI的BLAST查找同源序列,應用MegAlign采用Jotun Hein、ClustalV以及ClustalW 3種方法分別進行序列比對,并利用Mega4.0軟件采用鄰接法(N-J)、最小進化法(ME)、最大簡約法(MP)、非加權組平均法(UPGMA)構建系統發育樹。3種序列比對結果顯示,Aeromonassp.T1與Aeromonassp.T2序列具有較高的相似度,分別為97.5%(Jotun Hein)、96%(ClustalV)、96.4%(ClustalW);Aeromonassp.T1與Genebank登錄號為HM778461.1序列相似度最高,分別為97.1%(Jotun Hein)、93.8%(ClustalV)、94.5%(ClustalW);Aeromonassp.T2與同樣與Genebank登錄號為HM778461.1序列相似度最高,分別為97.7%(Jotun Hein)、95.3%(ClustalV)、96.8%(ClustalW)。4種方法構建的系統發育樹基本一致,可初步確立2株菌株的分類地位:Aeromonassp.T1、Aeromonassp.T2與登錄號為HM127065.1的菌株的聚為一類,親緣關系最近。
氣單胞菌(Aeromonas);16S rDNA;分子鑒定
氣單胞菌屬(Aeromonas)曾與弧菌屬(Vibrio)、原細菌屬(Protobacterium)和鄰單胞菌屬(Plesiomonas)被歸入弧菌科(Vibrionaseae)[1]。隨著現代分子遺傳學的發展,以往被歸類為弧菌科的氣單胞菌屬劃為一個獨立的科,即氣單胞菌科。到目前為止該項包括:嗜水氣單胞菌(Aeromonashydrophila)、豚鼠氣單胞菌(Aeromonascaviae)、溫和氣單胞菌(Aeromonassobria)、維隆氣單胞菌(Aeromonasveronii)、殺鮭氣單胞菌(Aeromonassalmonicida)、中間氣單胞菌(Aeromonasmedia)、嗜礦泉氣單胞菌(Aeromonaseucrenophila)、簡氏氣單胞菌(Aeromonasjandaei)、鰻魚氣單胞菌(Aeromonasichthiosmia)、舒氏氣單胞菌(Aermonasschubertii)、尺骨氣單胞菌(Aeromonastrota)、氣單胞菌群501、異常嗜糖氣單胞菌(Aeromonasallosaccharophila)和斑點氣單胞菌(Aeromonaspunctata)共14 個氣單胞菌表型種和16個基因種[1,2]。
細菌分類鑒定方法包括表型鑒定法和分子遺傳學鑒定法2大類,分成4個水平細菌形態和生理生化水平、細胞組分水平、蛋白質水平和核酸水平。表型鑒定法是對前3個水平的鑒定,包括常規鑒定法、數值分類鑒定法和化學分類鑒定法[3]。分子遺傳學鑒定法是核酸水平的鑒定,對細菌染色體或質粒DNA進行分析,包括G + C含量測定、核酸雜交、PCR 技術、16S rRNA 和16~23S rRNA序列分析、全基因組測序等。其中16S rRNA/rDNA 序列分析技術是分子生態學領域的一項重要技術,目前已被廣泛應用于微生物的分類、鑒定以及微生物的群落研究,由于該技術具有方便、準確、快捷等特點,在鑒定土壤樣品中的細菌[4]、動物內臟及腸道中的菌落[5]、野生菌株的歸屬[6]、致病菌的快速鑒定[7]等方面得到了廣泛的應用。
本研究對分離自患出血病鯽魚的腸道和腹水中的2株氣單胞菌屬細菌Aeromonassp.T1和Aeromonassp.T2進行16S rDNA基因的序列進行序列同源性和聚類分析,以鑒定其分類地位,為進一步應用提供科學依據。
1 材料與方法
1.1材料
1.1.1 菌株
氣單胞菌菌株Aeromonassp.T1和Aeromonassp.T2分離自患病鯽魚(Carassiusauratusgibelio)。
1.1.2 主要試劑
溴化乙錠(EB)、dNTP(10mmol/L)、溴酚藍、Tris堿、EDTA等購自Amresco公司;Taq酶購自Fermentas公司;DNA Mark、DNA 提取試劑盒購自TOYOBO公司;瓊脂糖購自Biowest公司;牛肉膏、胰蛋白胨、瓊脂粉、冰醋酸、NaCl等為國產分析純試劑。
1.2方法
1.2.1 細菌的培養
4℃斜面保存的菌株涂布于平板,28℃培養24h,用生理鹽水洗下菌苔制成菌懸液。
1.2.2 細菌基因組DNA的提取
按照DNA提取試劑盒的說明書提取基因組DNA,用1%瓊脂糖凝膠電泳檢測DNA的質量。
1.2.3 16S rDNA基因片段的擴增
采用16S rDNA細菌通用引物:8f: 5’-AGA GTT TGA TCC TGG CTC AG-3’;1492r: 5’-GGT TAC CTT GTT ACG ACT T-3’。聚合酶鏈反應(PCR)按以下次序,將各成分在0.5ml無菌PCR管內混合,總體積25μl:PCR buffer(10×,2.5μl)、MgCl2(25mmol/L,1.5μl)、dNTPs(2mmol/L,2.0μl)、上游和下游引物(10μmol/L,各0.5μl)、模板DNA(0.3~0.4μg)、Taq-DNA (1U/μl,0.5μl),最后補加滅菌ddH2O至25μl。 短暫離心(8000r/min,10s)混勻,每管加入30μl液體石蠟油覆蓋于反應混和液上,將樣品置于PCR合成儀中按以下條件進行PCR擴增反應:94℃預變性2min;94℃變性30s,50℃退火30s,72℃延伸1min,35個循環;72℃保溫8min。末輪循環后一直維持在4℃,擴增產物置于-20℃備用。
擴增得到的產物用1.5%瓊脂糖凝膠(含溴化乙錠1mg/L)在5V/cm的電壓下電泳。電泳結束后,用凝膠成像系統照相,觀察到所需產物的擴增帶后,送上海生工武漢測序部進行產物純化和正反測序。
1.2.4 數據分析
采用ContigExpress軟件進行序列拼接得到其16S rDNA基因全序列, BLAST相似序列,以MegAlign軟件比較這些序列的差異和相似度。用ClustalX軟件排列比對序列,用Mega4.0軟件分別進行、鄰接法(N-J)、最小進化法(ME)、最大簡約法(MP)、非加權組平均法(UPGMA)構建系統發育樹,其中自舉檢驗1000個副本。
2 結果與分析
2.1PCR產物擴增結果
按照1.2.2的方法提取得到各菌株的總DNA,電泳檢測無拖尾現象,表明質量較高(圖1),可直接用于PCR反應。以16S rDNA的通用引物(8f,1492r),擴增得到的片段大小為1500bp左右,符合預期大小,擴增特異性較高(圖2)。

圖1 DNA電泳圖 圖2 PCR擴增產物電泳圖
2.2目的基因測序結果
以通用引物作為測序引物,對PCR產物進行雙向測序,所得測序峰圖中大多數堿基峰清晰,雜峰較少,測序起始后的前40個左右堿基峰型相對雜亂,通過ContigExpress軟件進行雜峰修正和正反序列的拼接,得到2株氣單胞菌屬細菌的1500bp左右片段。Aeromonassp.T1和Aeromonassp.T2 DNA序列見表1。

2.3序列比對結果
以拼接后的各菌株16S rDNA序列在Genebank中進行BLASTA比對,搜索同源較近的序列,從中挑選出4個序列: Uncultured bacterium clone aaa64h02 (DQ817645.1)、Uncultured bacterium clone SINI708 (HM127056.1)、Uncultured bacterium clone JFR0501_aaa05c04 (HM778461.1)和Uncultured bacterium clone JFR0501_aaa03e08 (HM778618.1)。Megalign軟件比較序列得到的同源性結果如圖3~圖5。3種序列比對結果顯示,Aeromonassp.T1與Aeromonassp.T2序列具有較高的同源性,分別為97.5%(Jotun Hein)、96%(ClustalV)、96.4%(ClustalW);Aeromonassp.T1與Genebank登錄號為HM778461.1序列同源性最高,分別為97.1%(Jotun Hein)、93.8%(ClustalV)、94.5%(ClustalW);Aeromonassp.T2同樣與Genebank登錄號為HM778461.1序列同源性最高,分別為97.7%(Jotun Hein)、95.3%(ClustalV)、96.8%(ClustalW)。
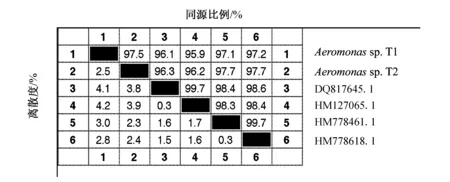
圖3 Jotun Hein法基因序列比較結果
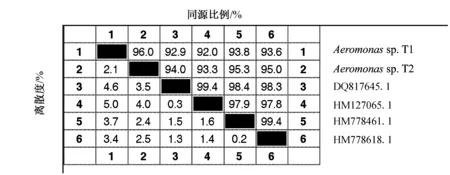
圖4 ClustalV法基因序列比較結果
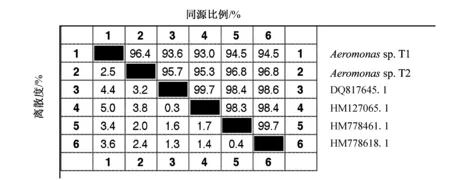
圖5 ClustalW法基因序列比較結果
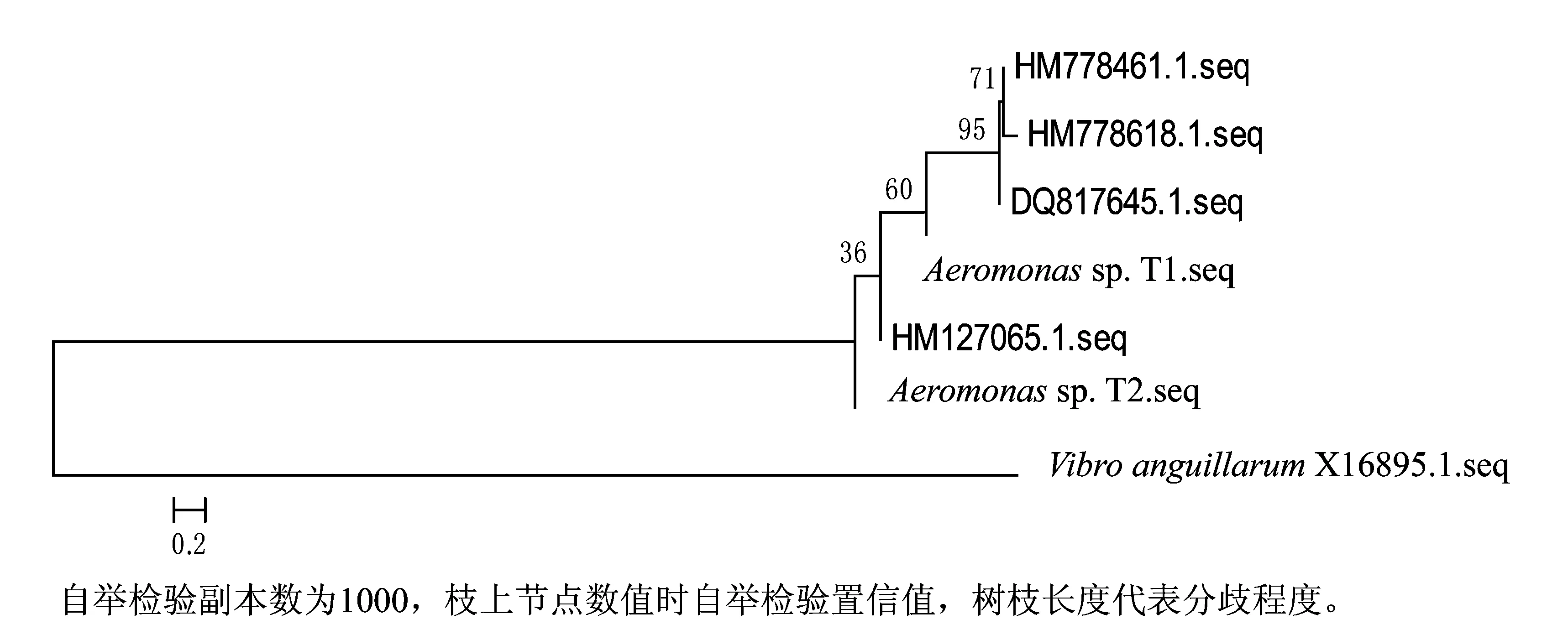
圖6 N-J法構建的系統發育樹
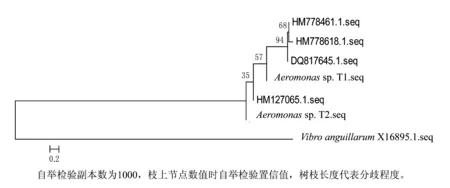
圖7 ME法構建的系統發育樹
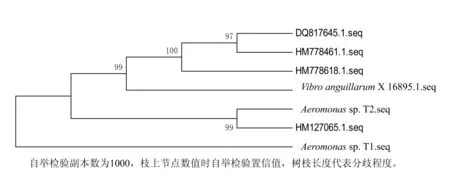
圖8 MP法構建的系統發育樹

圖9 UPGMA法構建的系統發育樹
2.4構建系統發育樹
以登錄號為X16895.1的鰻弧菌(Vibroanguiuarum)16S rDNA序列作為屬外序列,采用Mega4.0軟件以4種方法構建的系統發育樹基本相似(圖6~圖9)。初步確立兩株菌株的分類地位為,Aeromonassp.T1、Aeromonassp.T2與登錄號為HM127065.1的菌株的聚為一類,親緣關系最近。
3 討論
16S rDNA序列保守區和多變區間隔排列,選擇合適的區域作為分子標記,可以用于區分不同的分類等級。以多少相似性程度作為各分類等級的劃分標準應根據特定的研究對象來確定[8]。本研究中菌株16S rDNA的擴增、測序采用的是8f和1492r細菌通用引物,而其它菌株在擴增和測序時所采用的引物可能不盡相同,造成相應序列長度的差異。本研究數據分析中采用的是原始序列長度,未進行序列的人工切除,可能影響到序列比對的結果,在比對的序列中同源性100%的2對序列較少。另外本研究采用3種方法對分離株與BLAST序列進行了序列相似性的比對,結果分離株與其相似度最高的序列是一致的。而這3種序列排列方法有所不同,Jotun Hein法是根據已有的系統調整相關的序列進行比對,而其他2種方法不需要這個假設,ClustalW較ClustalV更進步,提供更準確的高度分化序列比對。因此,在本研究中應用Jotun Hein法得出的序列相似性較其他2種方法稍高。
對DNA序列進行系統發育分析的3個主要步驟是比對、建立取代模型和進化樹以及進化樹評估。目前建樹方法主要有3類,分別是距離法(包括最小進化法和鄰接法等)、最大簡約法(maximum parsimon,MP)和最大似然法(maximum likelihood,ML)。距離建樹法考察數據組中所有序列的兩兩比對結果,通過序列兩兩之間的差異決定進化樹的拓撲結構和樹枝長度;最大簡約法考察數據組中序列的多重比對結果,優化出的進化樹能夠利用最少的離散步驟去解釋多重比對中的堿基差異;最大似然法考察數據組中序列的多重比對結果,優化出擁有一定拓撲結構和樹枝長度的進化樹,這個進化樹能夠以最大的概率導致考察的多重比對結果。這3種建樹方法由于各自的局限性均可能導致系統誤差的產生[9],為了盡量避免誤差的影響,本研究同時采用了3類方法,通過比較它們所構建的系統進化樹,來綜合分析系統發生關系。可見4種方法建立的系統發育樹基本一致,本研究所構建的系統發育樹是可信的,各分離菌株的最近親緣關系菌株也是可信的。
本研究中BLAST查找同源性較高的條序列均為不可培養細菌克隆的序列,不可培養微生物的概念是1982年Colwell 實驗室提出的概念,他們發現將霍亂弧菌和大腸埃希菌(簡稱大腸桿菌)轉到不含營養物質的鹽水中,經常長時間的低溫保存,細菌會進入一種數量不減、有代謝活力、但在正常試驗室培養條件下不能生長產生菌落的狀態,稱為活的但不能培養(viable but nonculturable,VBNC)狀態。其后對多種屬細菌進行試驗證明,這種不可培養狀態是普遍存在的。自然界中也廣泛存在著這種活的但不可培養微生物,在各種生境中僅有小部分的微生物可用實驗室方法分離培養,而未被培養的種類卻代表了巨大的多樣性[10]。本研究中的2株氣單胞菌與GenBank中登錄號為HM127065.1的菌株的聚為一類,親緣關系最近。而HM127065.1的菌株為西藏湖泊中的1株不可培養細菌克隆(Uncultured bacterium clone SINI708),由此可見,細菌未被培養的種類可在特定的環境中被分離培養。
[1]楊東霞,曲險峰,楊暑伏.氣單胞菌分類及檢測技術的研究進展[J].中國獸藥雜志,2003,13(3):247-249.
[2] 胡靜儀,王金良.氣單胞菌屬分類與鑒定方法[J].國外醫學臨床生物化學與檢驗學分冊,2004,25(2): 22-23.
[3] 杜昕波,趙 耘,李偉杰.菌種保藏中的細菌鑒定方法[J].中國獸藥雜志,2009,43(3):50-53.
[4] 吳文龍,段淑蓉,李文均,等.3株抗灰霉病放線菌的分離和鑒定[J].云南大學學報,2004,26 (3):270-274.
[5] 蔣 燕,鮑寶龍,謝彩霞,等.16S rDNA文庫法分析草魚肝胰臟和膽汁細菌群落組成[J].上海海洋大學學報,2010,19(4):441-446.
[6] 張會敏,馮友軍.一株野生細菌的16S rDNA序列分析與系統發育樹的構建[J].生物信息學,2005,29(5): 1-5.
[7] 鄭桂麗,翟俊輝,唐學璽,等.16S rDNA寡核苷酸芯片鑒定致病菌的初步研究[J].軍事醫學科學院院刊,2005,29(1):13-18.
[8] Lee S K Y,Wang H Z,Law S H W,et al.Analysis of the 16S-23S rDNA intergenic spacers (IGSs)of marine vibrios for species-specific signature DNA sequences[J].Mairne Pollit Bullit,2002,44:412-420.
[9] Iguchi A,Ito H,Ueno M,et al.Molecular phylogeny of the deep-sea Buccinum species (Gastropoda:Buccinidae) around Japan:Inter-and intraspecific relationships inferred from mitochondrial 16S rRNA sequences[J].Science Direct,2007,44:1342-1345.
[10] 郭 瀟,雷曉凌.活的不可培養的細菌的研究進展[J].微生物學雜志,2010,30(3):82-86.
10.3969/j.issn.1673-1409.2011.07.118
Q939
A
1673-1409(2011)07-0257-06
2011-07-01
長江大學博士啟動基金(801200010110)。
譚鳳霞,女,博士,講師,主要從事微生物學研究。

