益生菌切達干酪成熟過程中細菌群落的多樣性分析
賈宏信,龔廣予,郭本恒,*
(1.光明乳業股份有限公司技術中心,乳業生物技術國家重點實驗室,上海 200436;2.上海海洋大學食品學院,上海 201300)
益生菌切達干酪成熟過程中細菌群落的多樣性分析
賈宏信1,2,龔廣予1,郭本恒1,*
(1.光明乳業股份有限公司技術中心,乳業生物技術國家重點實驗室,上海 200436;2.上海海洋大學食品學院,上海 201300)
采用選擇性培養基和聚合酶鏈式反應和變性梯度凝膠電泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)技術,研究益生菌切達干酪成熟過程中(6℃,180 d)細菌群落構成及益生菌(干酪乳桿菌LC2W)的存活情況。結果表明:SBM和MSE等選擇性培養基存在選擇專一性不強的缺點,不能客觀反映干酪內各種微生物的動態變化;隨著切達干酪成熟時間的增加,發酵劑嗜熱鏈球菌和乳酸乳球菌的數量明顯下降,而非發酵劑菌群乳桿菌的數量和主要種類呈上升趨勢;干酪成熟180 d后,干酪乳桿菌LC2W的存活量仍高于1×108CFU/g。切達干酪能作為干酪乳桿菌LC2W存活的良好載體;PCR-DGGE技術和選擇計數法聯用更加適合干酪細菌群落結構的分析。
切達干酪;干酪乳桿菌;聚合酶鏈式反應和變性梯度凝膠電泳;益生菌;細菌群落
干酪獨特的理化特性(相對高的pH值、低滴定酸度、強緩沖能力、高脂肪含量、低含氧量及致密的質構)使其成為了益生菌的良好載體[1],為益生菌干酪的研究提供良好前提。另外干酪內微生物由發酵劑菌群(主要是乳酸乳球菌、嗜熱鏈球菌、德氏乳桿菌和瑞士乳桿菌)和非發酵劑菌群(主要是嗜常溫乳桿菌、片球菌、丙酸菌、黏細菌、霉菌和酵母菌)組成[2]。干酪加工成熟過程中,先是發酵劑菌群占主導優勢,而后隨著成熟時間延長非發酵劑菌株的數量會顯著提高[3-4]。干酪內微生物這一獨特的組成和生長趨勢為益生菌干酪的研究提供了條件。從Dinakar等[5]研究益生菌切達干酪以來,益生菌干酪的研究迅速成為益生菌食品的研究熱點,到目前為止新鮮奶酪、軟質奶酪、半硬質奶酪、硬質奶酪和白鹵奶酪等幾乎所有類型的奶酪都已用 于益生菌干酪的研究[1],而涉及到的益生菌株主要為雙歧桿菌屬和乳桿菌屬[6]。
目前益生菌切達干酪的研究涉及到的益生菌有嗜酸乳桿菌、干酪乳桿菌、副干酪乳桿菌、鼠李糖乳桿菌和雙歧桿菌等,而益生菌存活情況的鑒定主要是選擇性培養基計數法[6],但是這一方法在進行菌相分析時存在同一屬很難分開的情況,為客觀地反映益生菌在某一基質內的數量,選擇合適的輔助方法就很重要。變性梯度凝膠電泳(denaturing gradient gel electrophoresis,DGGE)技術已廣泛應 用于海洋、湖泊、河流、昆蟲、動物腸道、環境和食品等各個領域內復雜微生物多樣性分析,能把大小相同、序列不同的DNA片段區分開來,并能實現區分同屬不同種的微生物[7]。本實驗以干酪乳桿菌LC2W(具有抗高血壓及免疫調節作用[8-9])作為切達干酪的附屬發酵劑制作益生菌切達干酪,并用傳統計數法和聚合酶鏈式反應和變性梯度凝膠電泳(polymerase chain reaction-DGGE,PCR-DGGE)技術聯用對乳桿菌LC2W在干酪內的存活情況和干酪的細菌群落構成進行分析。
1 材料與方法
1.1 材料與試劑
益生菌干酪乳桿菌LC2W、無抗生素牛奶 光明乳業股份有限公司;CHOOZTTTMRA021發酵劑、MARZYME 150MG凝乳酶 丹尼斯克添加劑(上海)有限公司;MRS固體培養基、M17固體培養基、SBM固體培養基、PCA固體培養基 英國Oxoid公司; MSE固體培養基 法國Biokar Diagnostics公司;2×Taq Plus PCR MasterMix 天根生化科技有限公司;引物合成和PCR擴增產物測序 上海立菲生物技術有限公司。其他試劑均為分析純。
1.2 儀器與設備
T50自動電位滴定儀、Inlab solids pro三合一pH計探頭 瑞士Mettler公司;厭氧培養箱 英國Ruskinn公司;隔水式恒溫培養箱 上海精宏實驗設備有限公司;高速低溫離心機 德國Eppendrof公司;變性梯度凝膠電泳儀、凝膠成像儀 美國Bio-Rad公司;Veriti 96 Well Thermal Cycler PCR儀 美國Life Technologies公司。
1.3 方法
1.3.1 干酪加工
益生菌切達干酪制作在光明乳業技術中心中試車間進行,取100L新鮮無抗牛奶進行標準化(蛋白、脂肪含量比值0.85)并進行巴氏殺菌(68℃,15 s),然后冷卻至31℃保溫加發酵劑(包含Lactococcus lactis subsp. lactis、Lactococcus lactis subsp. cremoris、Streptococcus thermophiles)和干酪乳桿菌LC2W,待乳熟化30 min(31℃)后加0.001%凝乳酶進行凝乳40 min左右,然后進行切割并熱燙(每5 min升高1℃)至38℃后排乳清、切達化、絞碎加2.5%無碘食鹽、入模壓榨成型并真空包裝,放入6℃成熟室成熟。
1.3.2 干酪內微生物計數實驗
取成熟時間為0、10、30、60、90、120、150、180 d的切達干酪進行菌相分析。將10 g干酪和90 mL 2g/100 mL檸檬酸鈉溶液混合,高速分散器勻漿2~4 min,然后用蛋白胨生理鹽水10倍梯度稀釋。取稀釋液200 μL進行涂布實驗,MRS、LM17、SBM、PCA和MSE培養基分別用于乳桿菌、乳球菌、糞腸球菌、總需氧菌和明串珠菌計數。培養條件:MRS和SBM平板厭氧培養(37℃,2 d);LM17和PCA平板非厭氧培養(31℃,2 d),MSE平板培養(28℃,5 d)。計數:取菌落數在30~300 CFU/平板的計數,并收集平板菌落至離心管,用于后續DGGE分析。
1.3.3 細菌總DNA的提取和擴增條件
參照Ercolini等[10]提取Stilton干酪總細菌DNA的方法提取,平板收集所得菌體DNA及干酪內細菌總DNA。PCR擴增總體系為50 μL:2×Taq Plus PCR MasterMix 25 μL,ddH2O 20 μL,10 μmol/L正、反向引物(R518和GC-F357)各2 μL,模板DNA 1 μL。PCR擴增條件為:95℃預變性5 min,95℃變性45 s,退火溫度從65℃起每個循環降1℃至56℃(退火40 s),72℃延伸30 s,56℃退火40 s,25個循環,72℃延伸30 s,最后72℃延伸10 min。擴增引物:R518(5’-ATTACCGCGGCTGCTGG-3’)和GC-F357(5’-CG CCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGG GGGCCTACGGGAGGCAGCAG-3’)。
1.3.4 DGGE分析
聚丙烯酰胺凝膠質量濃度8 g/100 mL,膠厚1 mm,丙烯酰胺-雙丙烯酰胺(質量比37.5∶1),尿素梯度40%~60%(100%變性劑含有7 mol/L尿素和40%甲酰胺)。電泳條件:電泳液1×TAE,60℃、75 V,電泳13 h。電泳結束后用EB染色20~30 min,凝膠成像儀拍照,然后割膠,把所割DNA放入加有50 μL無菌水的離心管里,4℃過夜,作為DNA模板。DGGE條帶測序:取上述DNA浸出液2 μL作為模板,2×Taq Plus PCR MasterMix 12.5 μL,ddH2O 8.5 μL,10 μmol/L引物F357和R518各1 μL進行PCR擴增,測序。PCR擴增條件:95℃預變性3 min,95℃變性45 s,56℃退火40 s,72℃延伸30 s,共25個循環,最后72℃延伸7 min。擴增引物:F357(5’-TACGGGAGGCAGCAG-3’)和R518(5’-ATTACCGCGGCTGCTGG-3’)。測序所得的16S rDNA 序列在線進行BLAST序列對比分析(http://www. ncbi.nlm.nih.gov)。
1.4 數據處理
顯著性分析采用SPSS17.0統計軟件單因素方差分析(analysis of variance,ANOVA)進行均值差異的顯著性分析,顯著水平P<0.05。
2 結果與分析
2.1 干酪內微生物平板計數結果
切達干酪內含的微生物具有多樣性和動態性,因此在不同成熟階段,干酪內不同種微生物的數量會有所變化。表1顯示益生菌干酪在不同成熟期內pH值和微生物的變化情況。切達干酪pH值的變化表現為從干酪壓榨后到成熟的前20 d在5.17左右,到第30天下降為5.11,而后隨著成熟時間的增加,其pH值一直下降,到成熟180 d時為4.88。pH值下降的因素主要有:乳酸菌利用殘余糖類進行代謝產生的乳酸;蛋白降解和脂質降解產生的酸性氨基酸及脂肪酸等;干酪成熟過程中微生物代謝消耗掉一部分自由水。
由表1可知,在干酪成熟180 d過程中,MRS培養基得到的乳桿菌數量一直高于1×108CFU/g,成熟前期乳桿菌數量低于成熟后期,這一點反映了乳桿菌在切達干酪內進行著代謝與生長。LM17培養基得到的乳球菌數量呈現逐漸降低的趨勢,不同時期相比存在顯著差異,干酪成熟90 d以前乳球菌數量高于2.65×109CFU/g,90 d后下降為108CFU/g左右。SBM培養基計數的糞腸球菌數量成熟前為1.38×104CFU/g,成熟時一直降低,到180 d時下降為5.33×102CFU/g。而PCA培養基計數的好氧細菌結果和MSE培養基計數的明串珠菌結果相似,都是在不同的成熟期內數量高于1×108CFU/g。
2.2 培養基菌落PCR-DGGE分析
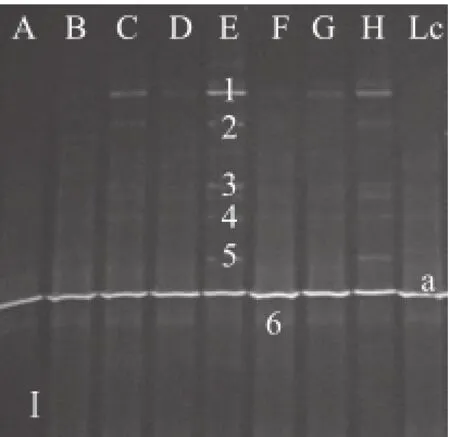
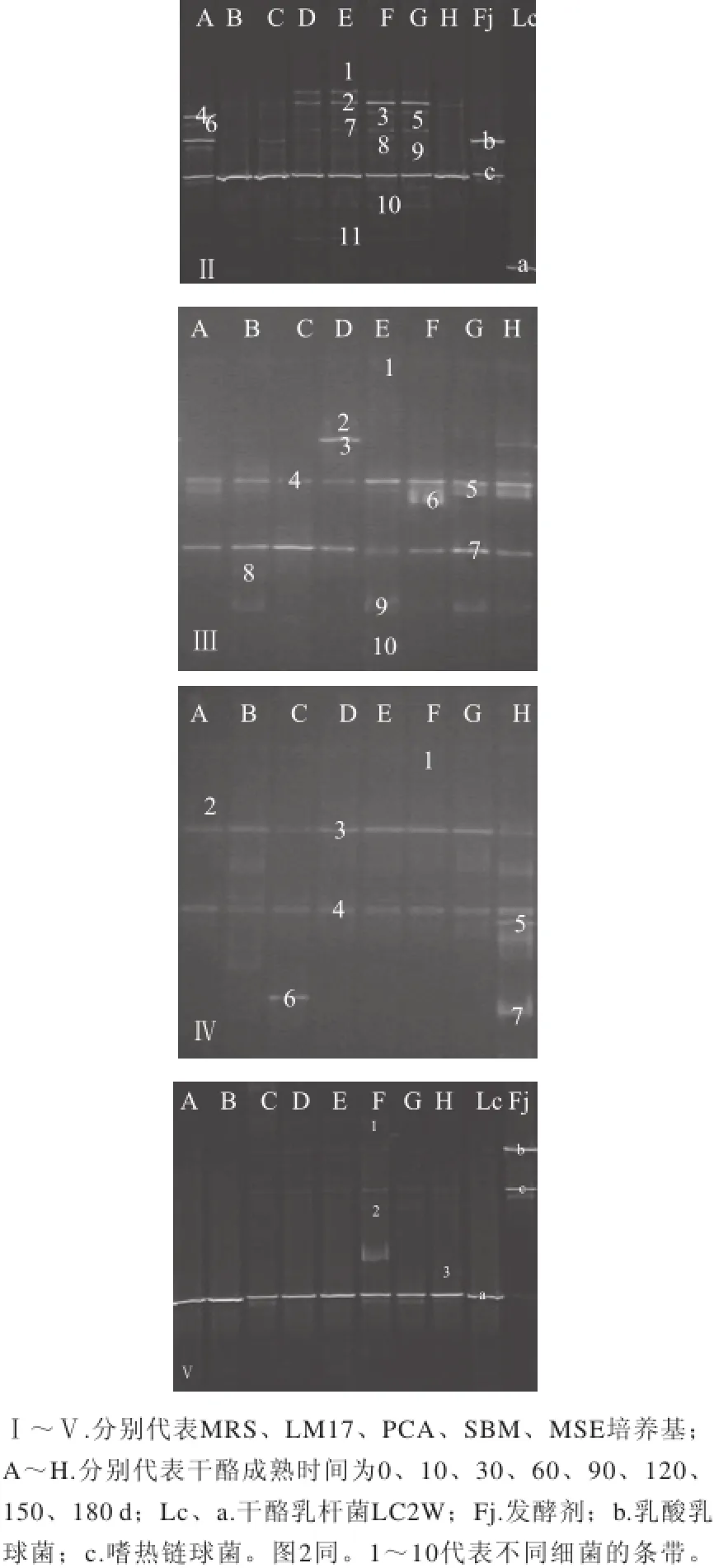
圖1 在不同成熟時間不同培養基收集所得細菌V3區DGGEE圖譜Fig.1 Bacterial DGGE profile of PCR amplicons of the V3 region of bulk cells from different media
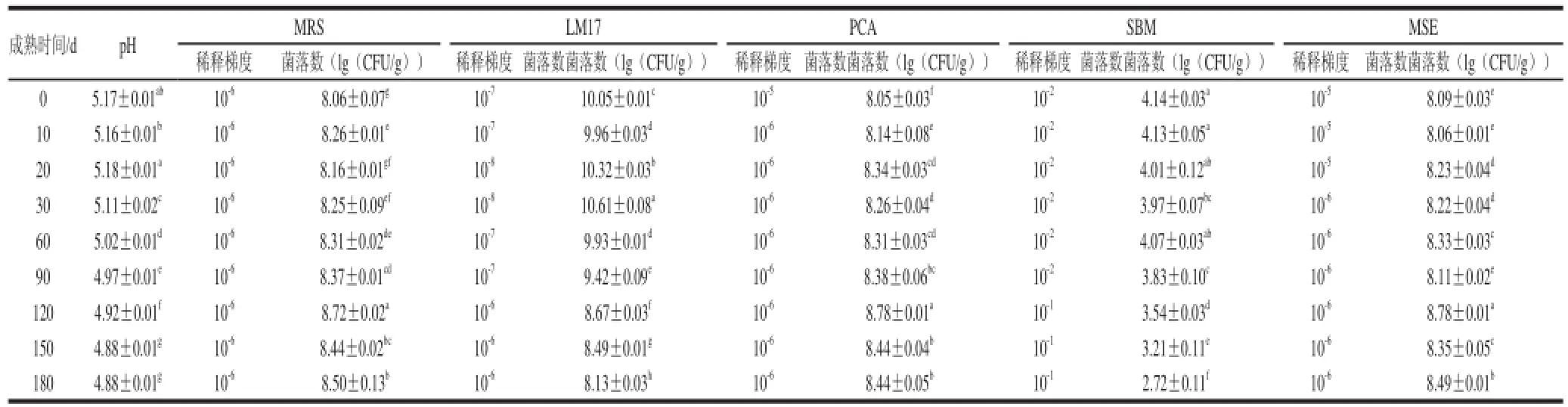
表1 切達干酪不同成熟期的pH值和主要細菌的數量Table 1 Mean pH, log bacterial counts (in lg(CFU/g)) and standard deviation (SD) of the main bacteria along ripening stages of cheddar cheese
培養基菌落PCR-DGGE分析結果如表2所示,MRS培養基上選擇生長的主要為乳桿菌,但也有鏈球菌的檢出;LM17培養基的選擇性相對較好,菌落分析結果主要為鏈球菌和乳球菌;SBM培養基不但有糞腸球菌,還有乳桿菌、乳球菌和鏈球菌等檢出,并且是鏈球菌居多;MSE培養基這一選擇生長明串珠菌的培養上反而沒有明串珠菌的檢出,檢出的主要為乳桿菌;PCA培養基上生長的微生物較復雜,有乳桿菌、乳球菌和鏈球菌。

表2 不同培養基計數得菌落16S rDNA V3區DGGE主要條帶測序結果Table 2 Sequencing of the 16S rDNA V3 region of DGGE bands of the bulk cells from different media
干酪在成熟過程中微生物的組成會有所變化。圖1-Ⅰ和表2的結果顯示,干酪乳桿菌LC2W一直是MRS培養基上的主要菌株,結合表1 MRS計數結果可以說明干酪乳桿菌LC2W在切達干酪內的存活率很高;另外隨著成熟時間的增加特別是到成熟30 d后,干酪中乳桿菌的種類有所增加。圖1-Ⅱ的結果顯示干酪內添加的發酵劑嗜熱鏈球菌在干酪成熟到180 d時數量很高,而乳酸乳球菌只有在干酪成熟0 d時的DNA條帶較亮,說明發酵劑乳酸乳球菌(圖1-Ⅱ-b)在干酪成熟前期會快速死亡。對圖1-Ⅱ中顯示的其他鏈球菌而言(圖1-Ⅱ,條帶1、2、7)只有在干酪成熟時間為60、90、120、150 d時才能檢出,說明這些菌的數量隨著干酪成熟時間的延長是動態變化的。圖1-Ⅲ結果和表2結果可以明確地表明PCA培養基上的菌株主要為鏈球菌(圖1-Ⅲ,條帶4)和乳桿菌(圖1-Ⅲ,條帶7),而且這兩條亮帶也說明在干酪成熟過程中這兩種菌的數量一直較高。圖1-Ⅳ結合表2明確顯示出SBM培養基上長出的主要為鏈球菌(圖1-Ⅳ,條帶3、5),糞腸球菌只在干酪未成熟時有檢出(圖1-Ⅳ,條帶2)。結合圖1-Ⅴ和表2再次明確了干酪乳桿菌LC2W在干酪內具有較高的存活率,也表明MSE培養基存在選擇性弱的缺點。發酵劑乳酸乳球菌(圖1-Ⅴ,條帶2)在成熟時間為60、90、120 d時在DGGE條帶中有顯現,在成熟時間為0、10、30、150 d和180 d時在DGGE條帶中無顯現。有研究表明干酪內乳球菌的活菌數和干酪的pH值存在一定的正相關關系[11]。干酪內乳酸乳球菌的這一變化,再結合圖1-Ⅱ和表1的數據可以得出干酪的pH值變化與乳酸乳球菌的變化相關,在干酪成熟前期pH值一直穩定,到成熟30 d以后pH值一直呈現下降趨勢,到成熟150 d后pH值又趨于穩定。
2.3 切達干酪菌相總DNA的PCR-DGGE分析

圖2 益生菌切達干酪不同成熟階段細菌V3區的DGGEE圖譜Fig.2 Bacterial DGGE profiles of PCR amplicons of the V3 region of ripened probiotic cheddar cheese during different ripening times
切達干酪在進行總DNA提取時由于面臨干酪內菌相復雜、菌種數量不同、干酪蛋白含量高等原因,會限制干酪內某些微生物DNA的提取效果,進而影響相應的PCR-DGGE效果。圖2顯示干酪內總DNA的DGGE圖譜,圖上主要顯示的3條亮帶分別代表菌株發酵劑嗜熱鏈球菌(條帶c),干酪乳桿菌LC2W(條帶a)以及鏈球菌(條帶1)。這一結果和計數得出的結果相一致,都表明嗜熱鏈球菌是切達干酪成熟過程中的優勢菌。這一結果也表明干酪乳桿菌LC2W的在成熟的過程中(6℃,180 d)的存活量一直處于較高水平。干酪內數量較低的植物乳桿菌、鼠李糖乳桿菌和副干酪乳桿菌等菌株(通過平板計數和表2結果得出)在干酪總DNA的PCR-DGGE圖譜上(圖2)沒有顯現,說明用PCR-DGGE技術直接分析干酪內菌相時只能分析出干酪內的優勢菌。
3 結論與討論
評價干酪微生態的方法主要有兩種:培養基依賴法(選擇性培養基法)和非培養基依賴法(分子生物學方法)。培養基依賴法一般只能確定到菌的屬,不能確定到菌所屬的種或是亞種,并且對于有些離開干酪特定微生態不能培養的微生物無法檢出。非培養基依賴法省去繁瑣的培養步驟,直接從干酪內取得微生物的DNA或RNA,進行全局的實時分析,一步就可以研究干酪內微生物多態性,而且極易實現分析到微生物的種型,可以實現快速、實時的監控干酪內微生物的種類和數量[12]。但是干酪內大分子物質(酪蛋白和脂質等)含量高,這些物質會和DNA提取時的洗滌劑、離液劑和螯合劑結合而影響總DNA的提取,進而使某些菌的檢出存在困難[13]。由表1、2和圖1結果顯示,MRS和LM17培養基在分析干酪內乳桿菌和乳球菌時相對準確,但是也存在一定的偏差,例如MRS培養基上有鏈球菌檢出。其他研究者也發現MRS培養基存在過度評價干酪內乳桿菌數量的問題,如葡萄球菌在MRS培養基上生長[10,14];LM17上檢出的鏈球菌(圖1-Ⅱ,條帶1、2、7)在發酵劑中沒檢出,說明這些菌很可能是原料奶本身含有的菌株,而圖1-Ⅱ顯示的乳酸乳球菌只有在干酪成熟0 d時有明顯的亮帶,說明在干酪成熟的前10 d內這一菌株的數量已發生急劇下降。這和郭本恒[15]介紹的切達干酪成熟過程中發酵劑乳球菌的變化一樣,干酪成熟前期(7 d左右)發酵劑乳球菌會因為自溶而快速減少。由圖1-Ⅰ和表1可以看到MRS培養基上乳桿菌的變化,在干酪成熟前期乳桿菌種類較少,而到后期種類明顯增加,說明隨著成熟時間的增加干酪本身具有的乳桿菌在干酪內處于生長狀態。
SBM培養基的選擇性對干酪內糞腸球菌的分析有所局限,在SBM培養基上檢出的主要是鏈球菌,而其選擇性分析的菌株糞腸球菌只在干酪成熟0 d時有檢出。這反映出SBM的對糞腸球菌的計數情況是嚴重失真的,沒有客觀地反映出干酪內糞腸球菌的數量。MSE本是選擇性培養明串珠菌的培養基,但是在MSE培養基上卻沒有明串珠菌的檢出,而檢出的主要為干酪乳桿菌LC2W,這說明MSE培養基的選擇性會受到乳桿菌的干擾。關于選擇性培養基選擇性不強的實例在其他文獻上也有報道,如Ercolini等[10]研究Stilton干酪菌相時發現,葡萄球菌能在LM17和MRS培養基上生長以及明串珠菌能在LM17上生長;Randazzo等[16]研究Sicilian干酪菌相時發現明串珠菌和腸球菌在LM17上有檢出,而MSE培養基卻無明串珠菌檢出。Randazzo等[17]分析Pecorino Crotonese干酪時也發現,LM17和MSE計數出來的菌數量都很高,但是通過對LM17和MSE上菌落進行進一步鑒定發現,LM17上長出的菌株屬于乳球菌的卻很少,MSE上長出的菌株屬于明串珠菌的更是少之又少。本實驗菌相分析結果和其他研究者[10-11,17-20]分析干酪菌相時的結果都證明單獨用培養基依賴法來評價干酪的菌相存在一定誤差性,而和非培養基依賴法結合起來分析干酪內菌相則更加真實和客觀。
乳球菌的數量(表1中LM17計數)在干酪不同成熟階段存在顯著性差異,PCR-DGGE分析發現發酵劑乳酸乳球菌僅在干酪成熟0d時存在,發酵劑嗜熱鏈球菌存在于整個成熟過程中,而非添加鏈球菌的數量隨著成熟時間的延長在升高(圖1-Ⅱ和圖2)。明串珠菌的MSE平板計數受乳桿菌、乳球菌和鏈球菌的影響較大,MSE平板計數一直高于1×108CFU/g,但MSE菌落的PCR-DGGE分析卻無明串珠菌的檢出,僅有鏈球菌、干酪乳桿菌、乳酸乳球菌和鼠李糖乳桿菌的檢出。MRS平板、PCA平板和SBM平板菌落的PCR-DGGE分析結果表明隨著切達干酪成熟時間的延長干酪內乳桿菌(如鼠李糖乳桿菌、副干酪乳桿菌等)的種類在增多(圖1-Ⅰ、Ⅲ、Ⅳ和表2)。培養基計數結果和PCR-DGGE分析結果表明:干酪乳桿菌LC2W在切達干酪成熟過程中(6℃,180 d)能保持較高的存活量(存活量高于1×108CFU/g),具有應用于益生菌切達干酪開發的潛力;培養基計數法和PCRDGGE結合起來分析切達干酪的菌相組成,可以更加真實和客觀的反映出干酪內微生物的動態變化。
[1] KARIMI R, MORTAZAVIAN A M, CRUZ A G. Viability of probiotic microorganisms in cheese during production and storage: a review[J]. Dairy Science & Technology, 2011, 91(3): 283-308.
[2] BERESFORD T P, FITZSIMONS N A, BRENNAN N L, et al. Recent advances in cheese microbiology[J]. International Dairy Journal, 2001, 11: 259-274.
[3] VOIGT D D, CHEVALIER F, QIAN M C, et al. Effect of highpressure treatment on microbiology, proteolysis, lipolysis and levels of flavour compounds in mature blue-veined cheese[J]. Innovative Food Science & Emerging Technologies, 2010, 11(1): 68-77.
[4] RANDAZZO C L, PITINO I, RIBBERA A, et al. Pecorino Crotonese cheese: study of bacterial population and flavour compounds[J]. Food Microbiology, 2010, 27(3): 363-374.
[5] DINAKAR P, MISTRY V V. Growth and viability of Bifidobacterium bifi dum in Cheddar cheese[J]. Journal of Dairy Science, 1994, 77: 2854-2864.
[6] KARIMI R, MORTAZAVIAN A M, AMIRI-RIGI A. Selective enumeration of probiotic microorganisms in cheese[J]. Food microbiology, 2012, 29(1): 1-9.
[7] ERCOLINI D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods, 2004, 56(3): 297-314.
[8] 吳正鈞. 干酪乳桿菌LC2W菌體的抗高血壓作用[J]. 天然產物研究與開發, 2011(2): 228-231; 257.
[9] 李瑞君, 郭本恒, 吳正鈞, 等. LC2W胞外多糖對大腸桿菌脂多糖誘導RAW264.7細胞因子表達的影響[J]. 中國藥理學通報, 2011, 27(3): 441-442.
[10] ERCOLINI D, HILL P J, DODD C E R. Bacterial community structure and location in Stilton cheese[J]. Applied and Environmental Microbiology, 2003, 69(6): 3540-3548.
[11] ALEGRIA A, ALVAREZ-MARTIN P, SACRISTAN N, et al. Diversity and evolution of the microbial populations during manufacture and ripening of casin, a traditional Spanish, starterfree cheese made from cow’s mi lk[J]. International Journal of Food Microbiology, 2009, 136(1): 44-51.
[12] JANY J L, BARBIER G. Culture-independent methods for identifying microbial communities in cheese[J]. Food Microbiology, 2008, 25(7): 839-848.
[13] BONAITI C, PARAYRE S, IRLINGER F. Novel extraction strategy of ribosomal RNA and genomic DNA from cheese for PCR-based investigations[J]. International Journal of Food Microbiology, 2006, 107(2): 171-179.
[14] DOLCI P, ALESSANDRIA V, ZEPPA G, et al. Microbiological characterization of artisanal Raschera PDO cheese: analysis of its indigenous lactic acid ba cteria[J]. Food Microbiology, 2008, 25(2): 392-399.
[15] 郭本恒. 干酪[M]. 北京: 化學工業出版社, 2004: 271-285.
[16] RANDAZZO C L, TORRIANI S, AKKERMANS A D L, et al. Diversity, dynamic s, and activity of bacterial communities during production of an Artisanal Sicilian cheese as evaluated by 16S rRNA analysis[J]. Applied and Environmental Microbiology, 2002, 68(4): 1882-1892.
[17] RANDAZZO C L, PITINO I, RIBBERA A, et al. Pecorino Crotonese cheese: study of bacterial popul ation and flavour compounds[J]. Food Microbiology, 2010, 27(3): 363-374.
[18] CARRARO L, MAIFRENI M, BARTOLOMEOLI I, et al. Comparison of culture-dependen t and -independent methods for bacterial community monitoring during Montasio cheese manufactu ring[J]. Research in Microbiology, 2011, 162(3): 231-239.
[19] MARTIN-PLATERO A M, MAQUEDA M, VALDIVIA E, et al. Polyphasic study of micr obial communities of two Spanish farmhouse goats’ milk cheeses from Sierra de Aracena[J]. Food Microbiology, 2009, 26(3): 294-304.
[20] DOLCI P, ALESSANDRIA V, RANTSIOU K, et al. Microbial diversity, dynamics and activity throughout manufacturing and ripening of Castelmagno PDO cheese[J]. International Journal of Food Microbiology, 2010, 143: 71-75.
Diversity of Bacterial Communities of Probiotic Cheddar Cheese during Ripening
JIA Hong-xin1,2, GONG Guang-yu1, GUO Ben-heng1,*
(1. State Key Laboratory of Dairy Biotechnology, Technology Center of Bright Dairy and Food Co. Ltd., Shanghai 200436, China; 2. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201300, China)
The survival of Lactobacillus casei LC2W and the bacterial communities in cheddar cheese were analyzed by using selective media and polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) method during the ripening period of 180 days at 6 ℃. Results demonstrated that SBM and MSE selective media could not objectively reflect the dynamic changes of various microorganisms in the cheese due to the poor selectivity. The population of the starters Streptococcus thermophilus and Lactococcus lactis decreased significantly, while the population and species of the non-starter Lactobacillus significantly increased during the ripening period. L. casei LC2W survived well in the cheese and retained its viability at > 1 × 108CFU/g even after 180 days ripening. It is concluded that cheddar cheese can be an effective vehicle for delivery of L. casei LC2W and that PCR-DGGE technology combined with traditional culturedependent methods can be more suitable for cheese microflora analysis.
cheddar cheese; Lactobacillus casei; polymerase chain reaction-denaturing gradient gel electrophoresis (PCRDGGE); probiotic; bacterial communities
TS201.3
A
1002-6630(2014)01-0145-05
10.7506/spkx1002-6630-201401028
2012-12-31
國家“973”計劃項目(2010CB735705)
賈宏信(1985—),男,碩士研究生,研究方向為食品生物技術。E-mail:jiahx0607@126.com
*通信作者:郭本恒(1963—),男,教授,博士,研究方向為乳業科學。E-mail:guobengheng@brightdairy.com

