基于粒子濾波算法的神經絲自動跟蹤
袁亮
Yuan Liang
新疆大學機械工程學院,新疆烏魯木齊市 830047
School of Mechanical Engineering, Xinjiang University, Urumqi, Xinjiang, 830047
1 介紹
生物細胞的功能主要取決于細胞內物質積極和定向運動1。這些物質非常多樣,包括細胞內蛋白質復合物,細胞骨架聚合物,核糖核蛋白顆粒,和膜細胞。由于熒光顯微鏡功能強大,可以對活細胞內移動結構直接觀察2,這就使得它成為我們研究這些物質的運動的強有力工具。為了提取所產生的圖像序列的動力學數據,我們采用實時或延時視頻錄像以在線或離線的方式跟蹤大量隨時間變化的運動目標。
神經細胞內運動的重要例子是軸突上的運動,這是沿神經細胞軸突上的亞細胞和分子結構運動3,4。軸突的一個有趣的特點是:他們是非常細長的圓柱形突起,通常直徑上只有幾個微米,但長度可達幾個毫米、厘米、甚至米。因此,軸突通常是將一個復雜的三維運動模型減少到一維模型,其中大部分沿軸突的長軸向前或向后移動,如圖1所示。

圖1 神經絲蛋白質在培養的老鼠皮質神經元軸突中運動的例子。
神經絲蛋白質在軸突上運動,測量直徑約10納米,長幾微米5,是軸突細胞骨架的主要組成部分。它對齊于平行軸突的長軸做間歇的雙向移動,這種移動是停頓時間長短不同的短時間向前或向后運動6,7。該微絲移動迅速平均速率約0.5微米/秒,但其整體運動速度卻要慢得多,因為他們要花大量的時間暫停7。由于運動的持續時間,以及移動和暫停狀態之間頻率的轉變都是高度可變的,因此其運動可視為隨機過程。為了分析運動動力性能,我們必須在連續幀視頻中跟蹤神經絲蛋白質。然而,目前這一過程完全由手動完成,這大大增加了勞動強度和人為錯誤影響8。因此,建立一個完全自動跟蹤方法是必要的,這樣可以提高跟蹤效率和消除人工操作引起的主觀性和潛在的可變性。
2 國內外研究概述
視頻跟蹤已應用于各種不同類型的細胞內運動,例如9,10,11。傳統的細胞內跟蹤方法分為兩個主要步驟:檢測和定位,對應。在檢測和定位的步驟中,目標的位置是通過局部圖像降噪和分割來確定的。關鍵是對應步驟,就是要在連續幀的視頻之間建立同一物體的對應關系。由于若干因素的影響,包括快速運動,物體變形,圖像噪聲,對象合并和分裂等等,這使得跟蹤細胞內運動的目標變得較為困難。Jaqaman等人12,13提出了一種跟蹤算法,被稱為線性分配法,這種方法可以在對應步驟中解決對象消失,合并和分裂的問題。Yang等人14基于Kalman濾波方法設計了可靠的跟蹤大規模密集反平行粒子運動的方法。然而,他們的方法對低信噪比圖像無法工作15,16。對于跟蹤有噪聲圖像序列中的物體,Smal17等人提出了一種使用粒子濾波的魯棒算法。他們的方法考慮到衍射極限對象的光學模糊,用一個點擴展函數模型計算觀測模型的可能性。然而,這種方法使用通用粒子濾波,增加了計算負擔。這是因為在通用粒子濾波算法中,為保證數據的可靠性,必須使用大量粒子,而每個觀察的粒子都需要一個二維的模型計算。當粒子的數量增加時,總的計算時間就會快速增加。
在本文中,我們提出了一種新的空間約束的粒子濾波跟蹤方法。這種方法最鮮明的特點是在神經絲蛋白質的跟蹤運動中利用軸突的幾何形狀。簡單地說,我們利用了軸突狹長的突起結構并且軸突上運動的物體必須在軸突內運動。因此,在空間上對先驗動態狀態能進行約束,從而明顯降低了在下一個視頻中可能性狀態分布的方差。這樣,在不影響跟蹤精度的情況下可以減少一些粒子的使用量,從而提高了跟蹤效率和準確性。
3 軸突約束的粒子濾波算法的實現
3.1 軸突路徑建模
正如圖1所示,培養的神經元軸突跟蹤軌跡為一條光滑曲線。為了建立這個模型的形狀,我們使用三次樣條插值獲得近似的分段多項式軸突路徑。利率曲線被節點分為多條線段,每條線段是由不同的多項式樣條插值函數建模而成。[u; v]是任何像素在圖像坐標系統的位置。對于給定節點的水平坐標u,每一段曲線段的垂直坐標v由三次樣條插值函數給定:

這里i代表節點下標(1,2, …, n),c0i,c1i,c2i和 c3i是每個部分的多項式系數并可以使用實際的像素坐標內曲線計算。這樣,假如曲線被分為n - 1段,4n個參數能被確定。為了獲得這4n個未知參數,要求有4n個條件:位置,連續性,一階導數的連續性,二階導數在節點兩邊界條件。一個自然樣條,即在其中的兩端不封閉,邊界條件是讓二階導數在兩個端點處等于零。圖2顯示仿照這種方式的軸突實例。由于神經絲蛋白質的運動受到軸突的限制,我們使用這個路徑定義軸突的中軸線。為了得到神經絲蛋白質路徑,我們在整個視頻序列中將最大像素強度投影到同一平面上(圖2b),然后我們手動地分出節點(圖2c)。

圖2 用三次樣條差值建立軸突模型。 (a)運動神經絲蛋白質,(b)是整個視頻序列每一圖像的最大強度投影,(c)是通過重疊在最大強度投影圖像上的三次樣條插值(白線)獲得的曲線擬合結果。
3.2 神經絲蛋白質狀態的建模
在跟蹤應用中,動態狀態的元素選擇依據是對象的運動特性。要跟蹤一個非對稱的移動物質,如神經絲蛋白質,所需的元素包括位置,速度和方向。為方便起見,我們將神經絲蛋白質的外觀模型設定為一個邊界框長度(l)和寬度(w)的矩形的方塊。那么此框就表示濾波算法中的粒子。然后,我們根據移動神經絲的長度和軸突的彎曲程度分別為每個視頻序列選擇邊界框的尺寸。對于直的軸突一個窄框是足夠的,但是對于有急彎的軸突,我們必須增加邊界框的寬度,來保證能在所有幀中采樣到完整的神經絲蛋白質。在時間t的邊界框的位置、速度和方向可以由動態定義,這里表示在圖像中坐標系方塊中心的位置,θt表示方塊的轉角是對應的速度。動態狀態可表示為如下形式:

這里Δt是兩禎之間的時間間隔。Ν表示正態分布,σu,σv和σθ分別是分布和θt的方差。我們這里使用正態分布是由于重要性密度函數具有高斯形式。在方程(2)中的均值和方差可以根據經驗來選擇。應該指出的是,我們除了位置和速度之外還使用轉角來確定的動態狀態,這與許多跟蹤方法不同。方向變量也許用于跟蹤軸突運輸中的球狀物質時不那么重要,但是當用于跟蹤細胞骨架聚合物如神經絲蛋白質時卻非常重要,因為這些物質是長的不對稱結構。
使用三個決定動態狀態的向量元素序列(u, v,和θ)的結果是產生大量的粒子,這將會降低粒子濾波算法的工作效率。但是,粒子的數量可以通過約束多個向量元素來降低。
3.3 方向約束建模
由于神經蛋白質必須在軸突路徑上運動,所以粒子的方向在某個特定位置上必須與軸突的位置保持一致。這也就是,在時刻t的方向θt應當是軸突方程v(ut)的切角方向。相應地,動態狀態模型方程(2)可以改寫為:

并且

我們稱含有方向約束的粒子濾波算法為方向約束粒子濾波(orientationally constrained particle filtering(OCPF)).
3.4 位置約束建模
除了對神經絲蛋白質方向進行約束外,軸突也限制了蛋白質位置。為了對位置約束建模,我們僅限粒子濾波算法中的粒子分布在三次樣條曲線擬合程序所定義的軸突上。考慮到三次樣條曲線插值的不準確,我們用一個寬度為2d?的很窄的條來代表軸突,這里窄帶的中心線就是軸突的三次樣條曲線(圖3所示)。d?是由曲線的擬合誤差決定的。假如在時刻t一個粒子到中心線的距離(dt)小于d?,這個粒子就被接受;如果dt>d?,神經經蛋白質在這個位置的可能性就為零,這個粒子就是被拒絕。這種運動神經絲蛋白質的位置約束就可以建模為:

現在整個先驗動態狀態方程就能改寫為:
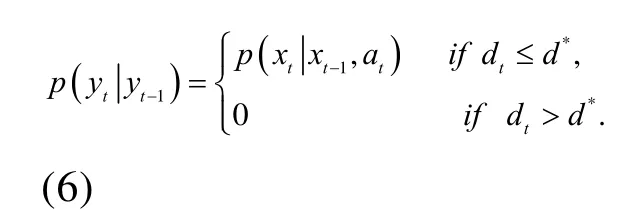
聯合位置約束方程(6) 和方向約束方程(3)和(4),我們用接受-拒絕機制來產生粒子,具體如下三個步驟:
Step 1: 根據(3)和(4)產生粒子tx′;
Step 2: 計算距離(td);
Step 3: 假如 dt<d*,接受這個粒子xt′為xt;否則,返回Step 1.
現在粒子可以從位置約束動態模型和方向約束動態模型中產生。我們稱這個方法為空間約束粒子濾波(spatially constrained particle filtering (SCPF)).
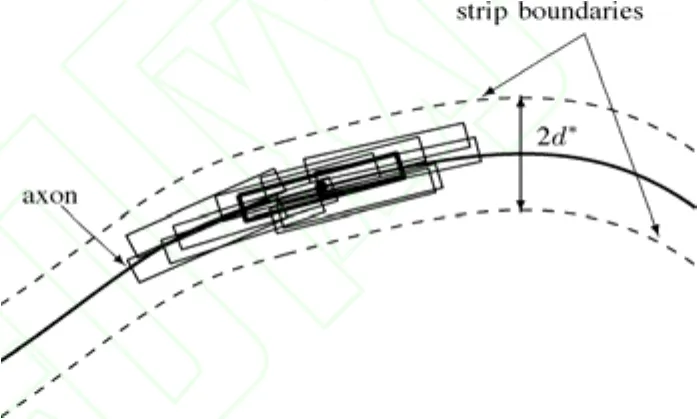
圖3 t時刻粒子在軸突邊界內的分布概略圖。實線代表三次樣條插值描述的軸突中心線,虛線代表軸突邊界。黑色矩形框代表在定義的軸突約束下的軸突內粒子的分布。粗黑色矩形框表示在這個最大似然估計例子下的粒子,它對應于神經絲蛋白質的位置。
4 實 驗
實驗設置如下:所有的實驗序列都是從小鼠大腦皮質分離的神經元中記錄下來的,該項工作由美國俄亥俄州立大學Anthony Brown教授實驗室中完成。從顯微鏡測得的原始圖像為512×512像素,倍率為0.131μm/像素。首先,我們將進行跟蹤初始化和參數描述,然后我們將介紹的跟蹤實驗的結果。
4.1 跟蹤算法初始化和參數
所有神經絲蛋白質都大約10納米寬。經驗上,我們認為用10個像素的(大約650納米)矩形框寬度w足夠在我們的圖像中包圍一個神經絲蛋白質。移動神經絲的長度是易變的,但平均是5-10微米長。這樣,我們設定矩形框的寬度為10個像素,長度隨跟蹤神經絲長度的不同而相應變化。從中心到兩邊的距離d?都選為5個像素。在水平和垂直方向的粒子分布的高斯噪聲方差(uσ和vσ)大致設定為25像素,粒子方向的方差(θσ)設定為0.5 rad。在觀測似然性的計算中,參數λ設定為20。這種跟蹤算法使用Python在2.1GHz的英特爾酷睿2雙核計算機上運行。為了計算神經絲蛋白質的實際運行速度,我們使用了MetaMorph軟件手動跟蹤神經絲蛋白質的運動。這個速度用作參考值來計算自動粒子跟蹤算法中的誤差。
4.2 實驗結果
我們的實驗包括通用粒子濾波跟蹤,方向約束粒子濾波跟蹤,空間約束粒子濾波跟蹤,并對其性能進行了評估和比較。圖4 (a) 和(b) 顯示出采用通用粒子濾波算法中的100和200個粒子的跟蹤結果。從圖中可以看出無論在位置還是在方向上都出現了大量的跟蹤誤差,即便200個粒子的跟蹤結果會比100個粒子略為好一些。這樣,100個和200個粒子的通用粒子濾波算法無法用于跟蹤這些錄像中的神經絲蛋白質。圖4 (c) 和(d) 顯示了采用50個和100個粒子的方向約束粒子濾波算法的跟蹤結果。正如我們在圖4 (c)中所看到的,50個粒子的方向約束粒子濾波算法在某些禎中仍然存在著跟蹤誤差。圖4(d) 顯示出了100個粒子的跟蹤精度有所提高,但仍然不能滿足所有禎。圖4 (e) 顯示了僅僅50個粒子的空間約束粒子濾波算法表現出了最佳的跟蹤結果。

圖4 三種不同粒子濾波算法跟蹤結果的比較。這些數據對應于視頻中的第42,50,52,55,61,和77幀。(a) 100個粒子的通用粒子濾波算法(GPF) 。(b) 200個粒子的通用粒子濾波算法(GPF) 。(c) 50個粒子的方向約束的粒子濾波算法(OCPF)。(d) 100個粒子的方向約束粒子濾波算法(OCPF) 。(e) 50個粒子的空間粒子濾波算法(SCPF) 。在(a),(b),(c),(d)和(e)上排表示粒子在跟蹤時的分布,下排表示跟蹤結果。

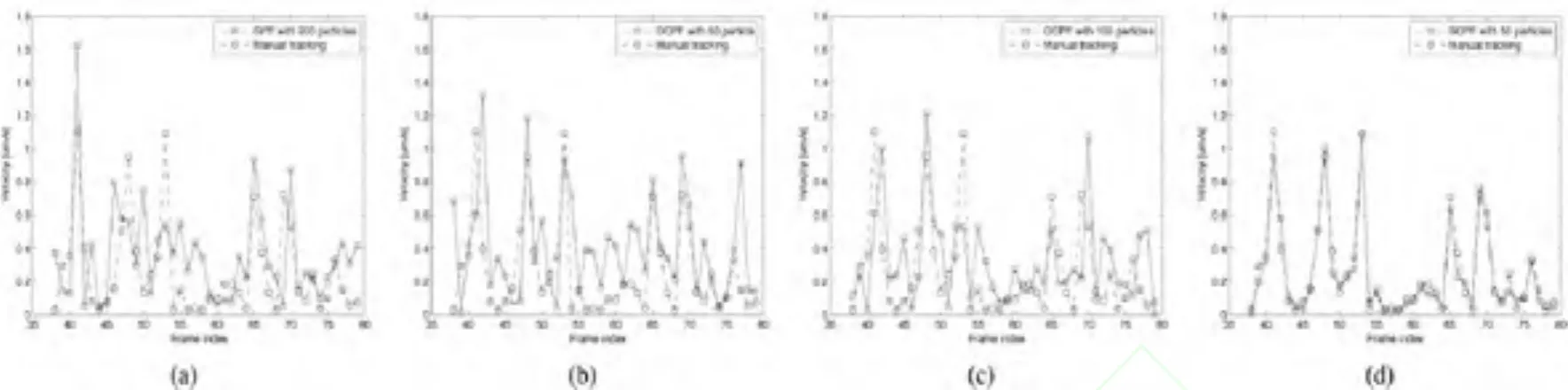
圖5 三種不同粒子濾波算法估計的神經絲蛋白質運動速度和實際的運動速度的比較。 (a)200個粒子的通用粒子濾波算法(GPF)。(b)50個粒子的方向約束的粒子濾波算法(OCPF)。(c)100個粒子的方向約束粒子濾波算法(OCPF)。(d)50個粒子的空間粒子濾波算法(SCPF)。注意:使用 GPF和OCPF獲得的估計速度和實際速度有相當大的差異,而使用SCPF獲得的估計速度與實際速度很相近。
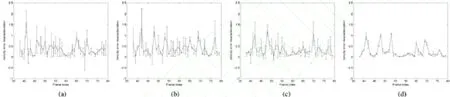
圖6 三種不同粒子濾波跟蹤算法跟蹤誤差的比較。(a)200個粒子的通用粒子濾波算法(GPF)。(b) 50個粒子的方向約束的粒子濾波算法(OCPF)。(c)100個粒子的方向約束粒子濾波算法(OCPF)。(d)50個粒子的空間粒子濾波算法(SCPF)。
為了進一步分析空間約束粒子濾波算法的跟蹤效果,每個跟蹤實驗我們使用通用粒子濾波算法、方向約束粒子濾波算法和空間粒子濾波算法分別重復執行了100次,并計算每個時間間隔神經絲蛋白質速度跟蹤誤差的平均值和標準偏差。跟蹤誤差定義為實際速度(通過有經驗專家手工跟蹤獲得)和估計速度(用粒子濾波算法法是計算有效粒子的數量(Neff),具體公式如下:自動跟蹤獲得)的比值。圖5顯示了用200個粒子的通用粒子濾波算法,50個和100個粒子的方向約束粒子濾波算法以及50個粒子的空間約束粒子濾波算法的跟蹤速度和手動跟蹤的速度相比較。圖6顯示出了相應的均值跟蹤誤差。200個粒子的通用粒子濾波在每一幀中都顯示出了大量的跟蹤誤差;50個和100個粒子的方向約束粒子濾波算法也不能得到令人滿意的跟蹤結果。空間約束粒子濾波算法產生的跟蹤誤差最小,產生的速度也最接近于手動跟蹤結果(如圖5(d)和圖6(d))。這些結果表明通用粒子濾波算法無法用于跟蹤延時視頻圖像序列中的運動神經絲蛋白質。跟蹤效果能通過方向約束來提高,最好是同時使用方向和位置這兩個約束。
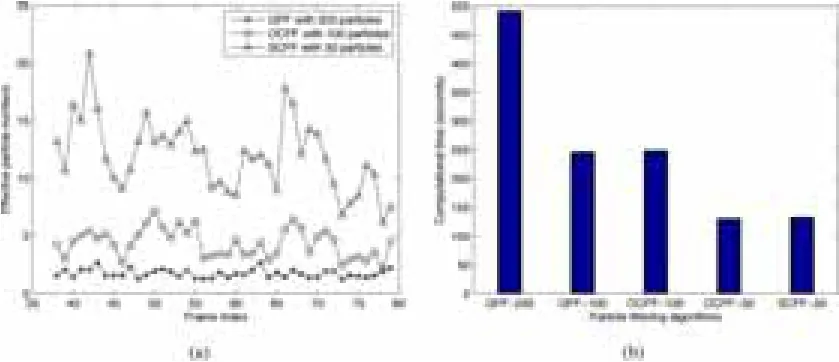
圖7 不同跟蹤算法的有效粒子數量和計算時間的比較。(a) 有效粒子數量。星號,空心圓和空心三角分別代表使用200個粒子的GPF,100個粒子的OCPF,和50個粒子的 SCPF所獲得的數據。(b) 計算時間。
為了進一步檢驗通用粒子濾波算法,方向約束粒子濾波算法和空間約束粒子濾波算法的運行效率,我們比較了他們的運行時間。在所有粒子濾波算法中,計算時間原則上由粒子的數量和根據觀測模型的粒子估計來決定。本論文中,我們在不同算法中使用了同一觀測模型,因此計算時間基本上與粒子使用的數量成正比。這樣50個粒子的空間約束粒子濾波算法的計算時間是100個粒子的方向約束粒子濾波算法計算時間的一半,是200個粒子的通用粒子濾波算法計算時間的四分之一(圖7(b))。因此,與通用粒子濾波算法和方向約束粒子濾波算法相比,空間約束粒子濾波算法對在軸突上跟蹤神經絲蛋白質運動更準確和效率更高。
5 結論
軸突神經絲蛋白質運動行為的非線性和不穩定性對自動跟蹤算法提出了挑戰。由于粒子濾波在處理非線性和不確定性運動的優越性能,它很自然的被用來解決這個問題。然而,由于通用粒子濾波必須使用大量的粒子來實現有效的跟蹤,這使得它的計算強度較高。在本文中,我們提出了一個新的空間約束的粒子濾波方法跟蹤神經絲蛋白質。這種方法利用神經絲蛋白質的運動范圍被限制在一個狹長的軸突內這一特點,對神經絲蛋白質可能的運動方向和位置進行了動力學限制。為了將這兩種約束用到粒子濾波算法中,我們利用三次樣條插值建立軸突路徑的模型,使粒子的位置限制在一個狹窄的路徑上,位置的方向限制在軸突路徑曲率的切線方向。同時使用這兩個約束增加了有效粒子數,從而減少了動態模型的不確定性。為了進行測試,我們比較了空間約束的粒子濾波與通用粒子濾波,以及其中只考慮方向約束的粒子濾波。結果表明,空間約束的粒子濾波方法在效率上大大高于通用或定向約束的粒子濾波方法。因此有必要采取位置和方向的兩個限制實現最優跟蹤效率。
1 D. Bray. Cell Movements: From Molecules to Motility [M]. 2nd ed. New York: Garland Science, 2000.
2 R. D. Goldman and D. L. Spector. Live Cell Imaging: A Laboratory Manual [M].2nd ed. New York: Cold Spring Harbor Laboratory, 2009.
3 A. Brown. Slow axonal transport, in New Encyclopedia of Neuroscience [M]. L. R.Squire, Ed. Oxford, U.K.: Academic, 2009,1–9.
4 A. Brown. Axonal transport of membranous and non-membranous cargoes:A unified perspective [J]. The Journal of Cell Biology, 2003, 160(6):817–821.
5 R. Perrot, R. Berges, A. Bocquet, and J.Eyer. Review of the multiple aspects of neurofilament functions, and their possible contribution to neurodegeneration [J].Cellular and Molecular Neurobiology, 2008,38(1): 27–65.
6 A. Brown. Slow axonal transport: Stop and go traffic in the axon [J]. Nature Reviews Molecullar Cell Biology, 2000,1(2): 153–156.
7 A. Brown. Axonal transport. In“Neuroscience in the 21st century”, ed. D.W.Pfaff [M], Springer, New York, 2013,255-308.
8 A. Brown and P. Jung. A critical reevaluation of the stationary axonalcytoskeleton hypothesis[J]. Cytoskeleton,2013, 70(1):1-11.
9 E. Meijering, I. Smal, and G. Danuser.Tracking in molecular bioimaging [J]. IEEE Signal Process Magzine, 2006, 23(3):46–53.
10 I. F. Sbalzarini and P. Koumoutsakos.Feature point tracking and trajectory analysis for video imaging in cell biology[J]. Journal of Structural Biology, 2005,151(2): 182–195.
11 J. Zhu and L. Yuan. Neurofilament tracking by detection in fluorescence microscopy images [C], in proceeding of international conference on image processing, 2013, 3123-3127.
12 K. Jaqaman, D. Loerke, M. Mettlen, H.Kuwata, S. Grinstein, S. Schmid, and G.Danuser. Robust single-particle tracking in live-cell time-lapse sequences [J]. Nature Methods, 2008, 5(8): 1212–1221.
13 K. Jaqaman and G. Danuser.Computational image analysis of cellular dynamics: A case study based on particle tracking [J]. Cold Spring Harb Protocal,2009, 2009(12): pdb.top65.
14 G. Yang, A. Matov, and G. Danuser.Reliable tracking of large scale dense antiparallel particle motion for fluorescence live cell imaging [C]. Proceding of IEEE Conference on Computer Vision and Pattern Recognition, San Diego, CA,2005: 9–17.
15 M. K. Cheezum,W. F.Walker, andW. H.Guilford. Quantitative comprison of algorithms for tracking single fluorescent particles [J]. Biophysical Jurnal, 2001, 81(4):2378–2388,.
16 B. C. Carter, G. T. Shubeita, and S.P.Gross. Tracking single particles:A user-friendly quantitative evaluation [J].Physical Biology, 2005, 2(1): 60–72,.
17 I. Smal,K.Draegestein, N.Galjart,W.Niessen, and E.Meijering. Particle filtering for multiple object tracking in dynamic fluorescence microscopy images:Application to microtubule growth analysis[J]. IEEE Transacitons on Medical Imaging,2008, 27(6): 789–803.

