PCR-DGGE技術檢測羊羔美酒大曲中細菌多樣性
李 艷,董振玲,李 佳,牟德華,*
(1.河北科技大學生物科學與工程學院,河北 石家莊 050018;2.河北省發酵工程技術研究中心,河北 石家莊 050018;3.河北一然生物科技有限公司,河北 石家莊 050800)
PCR-DGGE技術檢測羊羔美酒大曲中細菌多樣性
李 艷1,2,董振玲1,3,李 佳1,牟德華1,*
(1.河北科技大學生物科學與工程學院,河北 石家莊 050018;2.河北省發酵工程技術研究中心,河北 石家莊 050018;3.河北一然生物科技有限公司,河北 石家莊 050800)
目的:使用聚合酶鏈式反應-變性梯度凝膠電泳(polymerase chain reaction-denatured gradient gelelectrophoresis,PCR-DGGE)技術檢測羊羔美酒大曲中的細菌,探尋酒曲中細菌菌群的多樣性組成,研究羊羔美酒的風味特征。方法:對羊羔美酒大曲進行樣品處理、提取總DNA、PCR擴增、DGGE條帶的切膠與測序分析。結果:利用PCR-DGGE技術檢測到羊羔美酒大曲中包含魏斯氏菌屬(Weissella)、乳桿菌屬(Lactobacillus)、片球菌(Pediococcus)、芽孢乳桿菌(Sporolactobacillus)、葡萄球菌(Staphylococcus)、高溫放線菌屬(Thermoactinomyces)和4個不可培養的細菌種類。結論:羊羔美酒大曲中細菌菌群多樣性豐富,其中乳酸菌菌群是最大的細菌群系。
羊羔美酒大曲;細菌;聚合酶鏈式反應-變性梯度凝膠電泳
羊羔美酒是一種北方特色黃酒,其釀造所使用的酒曲是大曲。通常大曲的制作是以小麥為原料,經破碎加水壓制成磚塊裝的曲坯,自然接種微生物,在適宜的品溫、濕度等條件下自然培育而成[1-3]。大曲是釀制大曲酒的生香劑、糖化劑與發酵劑,含有豐富的微生物與酶系,其質量的優劣對酒的品質和風味有極大的影響,故有“曲是酒之骨”之稱[4-6]。獨特的大曲微生物群系組成是影響酒類香氣物質和種類繁多的風味物質的重要原因之一。羊羔美酒有著與眾不同的風味與口感,與大曲中獨特的微生物群系緊密相關。傳統的微生物培養方式可以得到菌株的純培養物,利于后續研究菌株的特性及其在酒類釀造中的作用,但是常規培養基與培養條件也會在微生物的分離研究中遺漏很多物種,況且大多數細菌很難培養或者不可培養。要想盡可能全面地了解羊羔美酒大曲中的細菌菌群的種類,應當尋求新的技術方法和手段。
聚合酶鏈式反應-變性梯度凝膠電泳(polymerase chain reaction-denatured gradient gelelectrophoresis,PCRDGGE)技術的原理是根據堿基序列上存在差異的不同DNA在雙鏈進行解鏈時需要的變性劑體積分數不同,一旦DNA雙鏈解鏈,其在聚丙烯酰胺凝膠中的電泳速度將急劇下降。因此,將PCR擴增得到長度相同的DNA片段加樣到含有變性劑梯度的聚丙烯酰胺凝膠中進行電泳,序列不同的DNA片段在其相應的變性劑體積分數條件下變性,發生解鏈行為,導致電泳速度的迅速下降,停留在與其相應的變性劑梯度位置,染色后即可在凝膠上呈現分散的條帶。自1979年Fischer和Lerman提出后,該技術經改進與完善,已經成為一項成熟穩定的現代分子生物學技術并得到廣泛應用[7-8]。
本研究以羊羔美酒釀造用酒曲為原料,以細菌菌群為研究對象,在非培養的方式下利用PCR-DGGE技術對酒曲中細菌直接進行分析檢測,為進一步研究羊羔美酒的風味物質形成和優化傳統發酵工藝提供參考依據。
1 材料與方法
1.1材料、試劑與引物
羊羔美酒大曲 河北味道府酒業有限責任公司。
配制溶液的各種試劑和生化試劑等均為分析純,引物F341(5’-CCTACGGGAGGCAGCAG-3’)、R534(5’-ATTACCGCGGCTGCTGG-3’)、F341-GC(5’-CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG GCC TAC GGG AGG CAG CAG-3’)上海生工生物工程技術服務有限公司。
1.2儀器與設備
PCR儀、凝膠成像分析儀、變性梯度凝膠電泳裝置美國Bio-Rad公司;DYY-8C型穩壓穩流電泳儀 北京六一公司。
1.3方法
1.3.1樣品的處理
稱取3 g均勻的酒曲樣品,加27 mL磷酸鹽緩沖液(phosphate buffered saline,PBS),200 r/min搖床30 min,旋渦振蕩5 min,4層紗布過濾,濾液經200 r/min離心5 min,取上清液,將上清液8 000 r/min離心3 min,得到沉淀,加入5 mL PBS,旋渦振蕩5 min,離心沉淀,重復洗滌2次[9-11]。將樣品置于-20℃保存備用。
1.3.2 DNA提取方法的選擇
羊羔美酒大曲中總基因組DNA的提取,共選擇3種不同的方法:1)化學裂解法:取樣品重懸于TE溶液,用TE溶液洗滌一次,除去上清液。加20 mg/mL的溶菌酶50μL,于37℃水浴60 min。向每管加入200μL裂解緩沖液,混合均勻。加入10μL蛋白酶K(10 mg/mL),55℃處理0.5 h,向每管加入66μL飽和的NaCl溶液,充分混合后,12 000 r/min離心10 min,然后除去蛋白質復合物及細胞壁等殘渣。離心后取上層清液,加入等體積的Tris飽和酚,14 000 r/min離心5 min,取上清液用等體積的氯仿-異戊醇(24∶1,V/V)溶液抽提2次,取出上清液,用預冷的1倍體積的冰異丙醇沉淀DNA,15 000 r/min離心10 min,棄去上清液。用400μL70%的乙醇洗滌沉淀2次。真空干燥后,用50μLTE緩沖液(10 mmol Tris-HCl,1 mmol EDTA,pH 8.0)溶解DNA,-20℃保存備用;2)CTAB法[12];3)氯化芐法[13]。
DNA樣品用0.8 g/100 mL瓊脂糖凝膠電泳檢測,Marker為λ-HindⅢ,電壓90 V,電泳時間60 min,拍照觀察。
1.3.3 PCR技術的選擇
1.3.3.1普通PCR
普通PCR反應體系(在0.2 mL離心管內配制50 ?L反應體系):10×buffer緩沖液5μL,dNTP 1μL,Taq酶(0.5 U/L)0.5μL,上、下游引物各1μL,模板DNA 2μL,補足水至50μL。
反應程序:95℃預變性7 min,94℃變性30 s,55℃退火1 min,72℃延伸30 s,35個循環,最后72℃延伸10 min。
1.3.3.2降落PCR
降落PCR:反應體系同普通PCR。
反應程序:95℃預變性7 min,94℃變性1 min,65℃退火1 min,每個循環降低0.5℃,經20個循環后,退火溫度降低至55℃,72℃延伸1 min;94℃變性1 min,55℃退火1 min,72℃延伸1 min,10個循環,最后72℃延伸10 min[14-15]。
PCR產物的檢測:吸取5μLPCR產物,通過1%瓊脂糖凝膠電泳檢測,80 V電壓50 min,經過EB染色15 min后,利用凝膠成像系統成像。
1.3.4 DGGE最佳實驗條件的研究
設置實驗變性劑體積分數為45%~60%[16-17],進行后續實驗的研究分析。實驗設計PCR產物加樣量為5、15、25、35、45μL,以尋求最佳PCR產物加樣量。
根據上述實驗確定的最佳PCR產物加樣量進行實驗,設計電泳時間為7、10、13、16 h,以尋求最佳電泳時間。
1.3.5條帶切膠回收、純化及測序
條帶的切膠回收:將DGGE電泳完畢的凝膠,置于凝膠成像儀上,選取目標條帶,使用無菌手術刀切割后放入滅過菌的1.5 mL離心管中,加入50μL的滅菌去離子水清洗2次,加入50μL的滅菌去離子水并于4℃靜置過夜。
取過夜儲存樣品液1μL作為PCR反應模板,進行PCR反應,反應程序與引物同1.3.3節。
目的條帶的純化:將上述PCR產物進行2次變性梯度凝膠電泳,重復切膠回收步驟,以使得目的條帶得到純化。直至目的條帶單一、無雜帶干擾為止。
目的條帶的測序:將純化完成的樣品進行2次PCR擴增,引物為F341(不帶GC夾子)與R534,反應程序與體系同1.3.3節。PCR產物經瓊脂糖電泳檢測合格后委托上海生工生物工程技術服務有限公司進行序列測定。測序結果在NCBI數據庫中進行BLAST比對,進行同源性分析。下載相關菌株序列信息,使用MEGA4軟件中的鄰接法構建系統樹圖,并以Bootstrap對進化樹進行1 000次可信度分析。
2 結果與分析
2.1 DNA提取方法的選擇
實驗選擇3種不同的DNA提取方法,得到的DNA經過0.8%瓊脂糖凝膠電泳分析后,電泳圖譜見圖1,羊羔美酒大曲中細菌總基因組DNA在23 kb左右;3種方法均可以對酒曲中細菌總DNA進行有效提取,但是從DNA的得率與DNA完整性來說,氯化芐法優于化學裂解法和CTAB法。圖譜中泳道前部有少量拖尾現象,可能是樣品中存在少量DNA降解或者是存在少量的RNA。從實驗結果可知,氯化芐方法提取的DNA完整性較好,可以滿足后續實驗的要求。
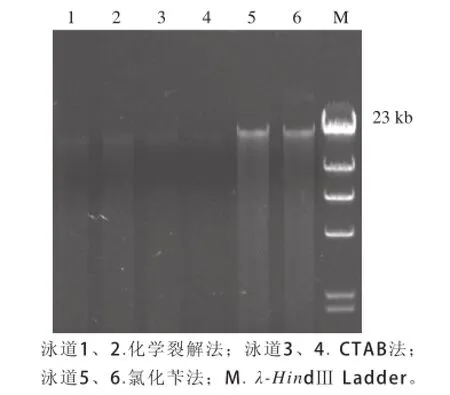
圖1 DNA電泳圖Fig.1 DNA electrophoretogram
2.2 PCR技術的選擇
本實驗采用普通PCR技術與降落PCR技術兩種方法對酒曲中宏基因組DNA進行擴增,將擴增產物進行瓊脂糖檢測,結果見圖2。
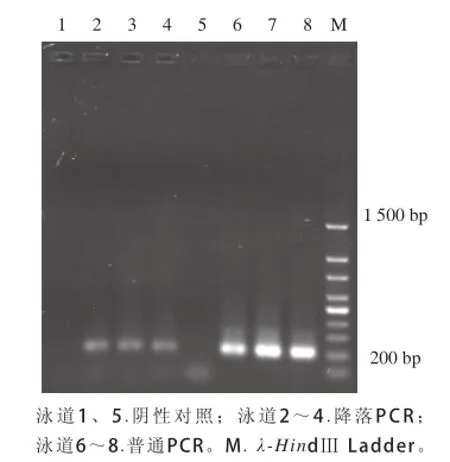
圖2 不同PCR方法擴增產物的圖譜Fig.2 Electrophoretogram of amplified products of the V3 region of 16S rDNA by different PCR techniques
由圖2可以看出,普通PCR技術與降落PCR技術都可以對酒曲樣品中總基因組DNA進行有效擴增,在200 bp處均有目的條帶。但是,普通PCR擴增產物的條帶有彌散現象,略有非特異性擴增現象,而降落PCR技術條帶無這兩種現象,且降落PCR技術本身可以減少非特異擴增,故本實驗選擇降落PCR技術進行后續的實驗。
2.3 DGGE最佳實驗條件的研究
從圖3可以看出,PCR產物加樣量5~35μL條帶越來越清晰明了,電泳圖譜效果也越來越好,但是35~45μL圖譜的清晰度沒什么差別,而背景色卻越來越嚴重,綜合考慮,PCR產物加樣量確定為35μL,并以此進行后續實驗。
從圖4可以看出,電泳時間較短時,泳道前方的條帶分離度很差,區分效果不好,而泳道后半部分的條帶分離度與區分度較好;隨著電泳時間的延長,泳道前方的條帶分離效果逐漸越來越好,但是后方的條帶卻逐漸在區分度上越來越差。雖然有文獻報道,由于在變性梯度電泳過程中PCR產物有了GC夾子,可以在遇到最佳變性劑體積分數時不完全解鏈,故此時不會繼續隨著電泳時間的延長而繼續移動,但從上述實驗結果可以看出,DNA序列解鏈后仍會繼續隨著電泳的進行而移動,只是移動速率會大大降低。綜合考慮,電泳時間為13 h時,電泳效果最佳。
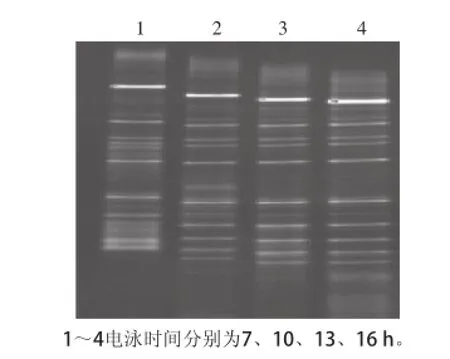
圖4 電泳時間的選擇Fig.4 Selection of electrophoresis time
2.4條帶切膠回收、純化及測序

圖5 條帶進切膠圖Fig.5 Electrophoresis of DNA bands of interest
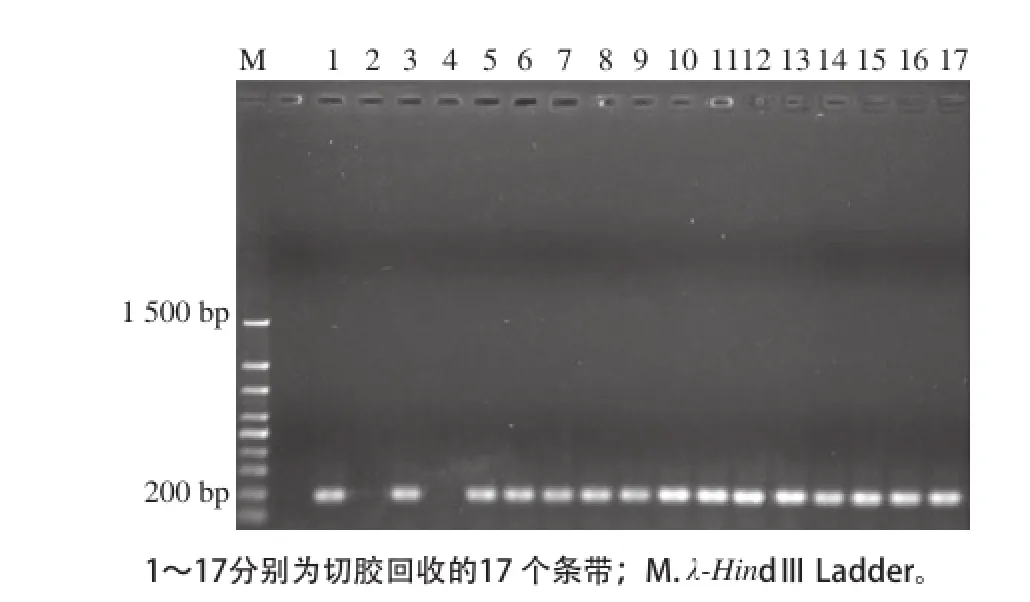
圖6 DGGE切膠條帶重新PCR電泳圖Fig.6 Electrophoresis of PCR-amplified products of target bands in DGGE profile
2.4.1條帶的切膠回收DGGE電泳完畢后,共選取了17個條帶進行了切膠回收,結果見圖5。將切膠回收的條帶使用F341與R534進行2次PCR擴增后,進行瓊脂糖電泳分析,結果見圖6。從圖6可以看出,除了條帶2與條帶4切膠回收失敗外,其他條帶均能滿足后續實驗的要求。條帶2、4失敗的原因可能是由于切膠回收產物的濃度太低,不能滿足PCR反應要求。
2.4.2電泳條帶純化
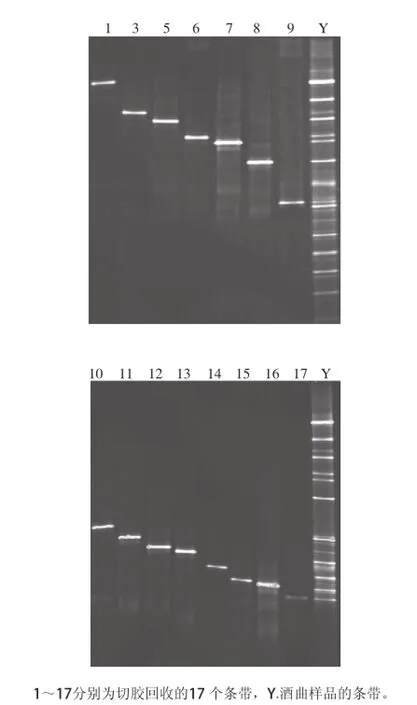
圖7 切膠條帶的純化結果Fig.7 Electrophoresis of purified bands
經過數次變性梯度凝膠純化后,回收條帶的DGGE圖譜(圖7)可以看出,使用DGGE凝膠可以對切膠條帶有很好的純化效果。
2.4.3測序
將純化后的條帶進行測序,測序所得基因序列結果登陸GenBank數據庫,進行BLAST相似性比對,測序結果見表1。
通過鄰接法構建的系統發育樹可以判斷細菌的同源關系,結果見圖8。在系統發育樹上同一分支的細菌同源關系最近,可以清楚地看出所分離細菌的分類情況及各自最相近的種屬名稱。

表1 條帶16S V3可變區序列分析Table 1 Sequence analysis of the bands based on 16S rDNA V3 region
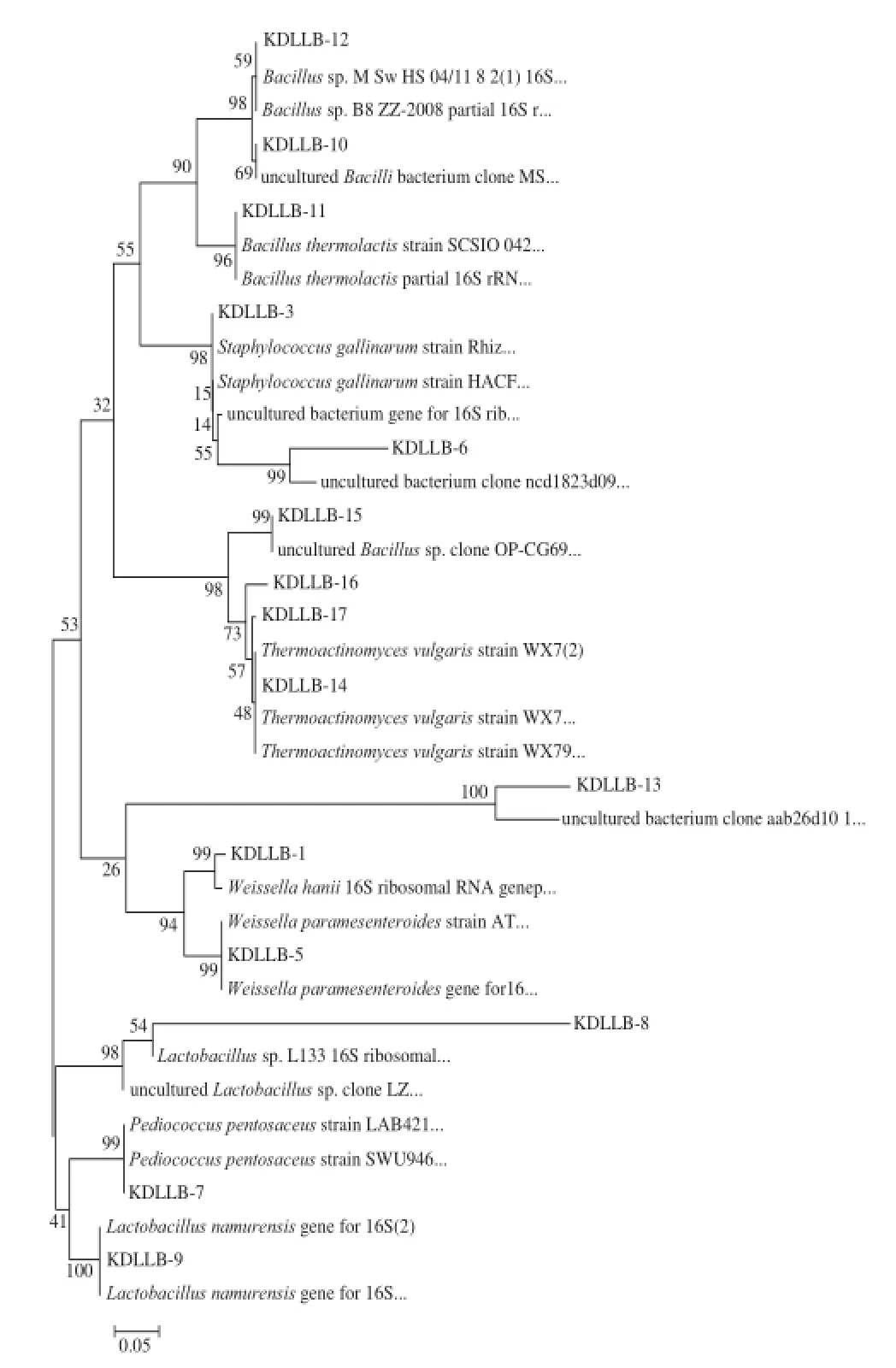
圖8 基于16S V3區序列和Neighbor-Joining法構建的系統發育樹Fig.8 Phylogenetic tree based on sequences of 16S rDNA V3 region and Neighbor-Jointing method
由圖8可以看出,所得序列與GenBank數據庫中16S rDNA序列的相似性在94%~100%之間。實驗結果中,與條帶1、5、7、8、9同源性最高的物種分別對應為Weissella hanii、Weissella paramesenteroides、Pediococcus pentosaceus、Lactobacillussp.、Lactobacillus namurensis,共涉及魏斯氏菌屬、乳桿菌屬和片球菌屬3個屬。乳酸菌對釀酒過程及最終產品的風味形成有突出貢獻,發酵初期產生大量乳酸,使發酵環境的pH值下降,可抑制有害菌繁殖;發酵中后期產生的乳酸是形成乳酸乙酯和其他香氣成分的提前物質,進而對增強酒的甜醇感和延長味感、增加酒的香氣柔和性等發揮作用[18]。可見,乳酸菌群系是羊羔美酒大曲中重要的產酸細菌菌群,推測其與羊羔美酒最終獨特的口感有一定相關性。條帶10、11、12、15同源性最高的物種分別對應為unculturedBacilli bacterium、Bacillus thermolactis、Bacillussp.、unculturedBacillussp.,都屬于芽孢桿菌屬細菌,趙佳[19]和李增勝[20]等研究表明,芽孢桿菌(如枯草芽孢桿菌)有水解淀粉、分解蛋白質的能力,其在酒類釀造過程中起著增香、產酸、增加酒體綿甜與清香有一定作用,所以必要的芽孢桿菌對釀造過程是有益的。而與高溫放線菌屬有較高同源性的條帶為14、16、17,最相似序列為普通高溫放線菌(Thermoactinomyces vulgaris)序列,可見其也在酒曲細菌菌群中占有一席之地。有研究[21-23]表明在芝麻香型白酒大曲中曾分離到普通高溫放線菌,并發現其具有嗜高溫酶、堿性磷酸鹽酶、酯酶、類脂酯酶活性等特性,而對其在酒曲和釀酒過程中的作用,尚需進一步研究。
3 結 論
本實驗建立了利用PCR-DGGE技術快速高效分析羊羔美酒大曲中細菌菌群構成的方法,并利用PCRDGGE技術分析了羊羔美酒大曲中細菌的多樣性。研究結果表明,在非培養方式下,檢測到羊羔美酒大曲中細菌物種資源信息非常豐富,從酒曲中直接進行總基因組DNA的提取,得到的物種里包含了魏斯氏菌屬(Weissella)、乳桿菌屬(Lactobacillus)、片球菌(Pediococcus)、芽孢乳桿菌(Sporolactobacillus)、葡萄球菌(S t a p h y l o c o c c u s)、高溫放線菌屬(Thermoactinomyces),除此之外,還發現了4個不可培養的微生物種類,分別為條帶6 uncultured bacterium、條帶10 unculturedBacilli bacterium、條帶13uncultured bacterium和條帶15 unculturedBacillussp.。由此可見,在羊羔美酒大曲中存在著部分不可培養的微生物。微生物的非培養鑒定技術結合PCR-DGGE分子生物學手段,非常有利于釀酒微生物資源的研究,尤其是對不可培養的物種。本實驗結果豐富了羊羔美酒大曲中微生物物種資源的信息,為進一步研究具有我國北方特色的地方名酒風味物質特征、改進傳統的配料和釀造工藝提供了可靠的參考依據。
[1]榮瑞金,李祖明.中國酒曲微生物研究進展[J].中國釀造, 2009, 28(6): 5-8.
[2]萬自然.大曲培養過程中微生物及酶的變化[J].釀酒科技, 2004(4): 25-26.
[3]傅金泉.中國酒曲的起源與發展史探討[J].中國釀造, 2010, 29(6): 180-185.
[4]惠豐立,褚學英,馮金榮,等.大曲中可培養霉菌多樣性的分子分析[J].食品與生物技術學報, 2007, 26(2): 76-79.
[5]敖宗華,陜小虎,沈才洪,等.我國濃香型大曲產業發展概況[J].釀酒科技, 2001(1): 78-81.
[6]傅金泉.中國酒曲技術的發展與展望[J].釀酒, 2002, 29(2): 7-9.
[7] NGUYEN T V, THUY M L, ANH T D. Microbial diversity of traditional vietnamese alcohol fermentation starters (banh men) as determined by PCR-mediated DGGE[J]. International Journal of Food Microbiology, 2008, 128(2): 268-273.
[8] KESMEN Z, YETIMAN A E, GULLUCE A, et al. Combination of culture-dependent and culture-independent molecular methods for the determination of lactic microbiota in sucuk[J]. International Journal of Food Microbiology, 2012, 153(3): 428-435.
[9]高亦豹.聚合鏈式反應-變性梯度電泳技術(PCR-DGGE)研究中國白酒大曲中微生物群落結構[D].無錫:江南大學, 2010.
[10]LüXucong, WENG Xing, ZHANG Wen, et al. Microbial diversity of traditional fermentation starters for Hong Qu glutinous rice wine as determined by PCR-mediated DGGE[J]. Food Control, 2012, 28(2): 426-434.
[11]閆亮珍,李曉然,全哲學,等.汾酒大曲和酒醅樣品DNA提取方法的優化[J].食品與發酵工業, 2011, 37(3): 32-36.
[12]譚映月,胡萍,謝和.應用PCR-DGGE技術分析醬香型白酒酒曲細菌多樣性[J].釀酒科技, 2012(10): 107-111.
[13]代道芳.基于宏基因組學技術的傳統發酵泡菜中乳酸菌多樣性研究[D].南寧:廣西大學, 2011.
[14] PEREIRA G V D M, MAGALH?ES-GUEDES K T, SCHWAN R F. rDNA-based DGGE analysis and electron microscopic observation of cocoa beans to monitor microbial diversity and distribution during the fermentation process[J]. Food Research International, 2013, 53(3): 482-486.
[15]施思,鄧宇,李波. DGGE法在盛夏習酒酒醅的微生物菌群結構解析中的應用[J].釀酒科技, 2010(3): 51-53.
[16] LI M Y, ZHOU G H, XU X L. Changes of bacterial diversity and main flora in chilled pork during storage using PCR-DGGE[J]. Food Microbiology, 2006, 23(7): 607-611.
[17] HU P, XU X L, ZHOU G H, et al. Study of theLactobacillus sakeiprotective effect towards spoilage bacteria in vacuum packed cooked ham analyzed by PCR-DGGE[J]. Meat Science, 2008, 8: 462-469.
[18]毛青鐘.論黃酒發酵過程酵母和乳酸桿菌協同作用關系[J].山東食品發酵, 2006(1): 29-31.
[19]趙佳,雷振河,呂利華,等.汾酒產酸細菌的分離鑒定及其產酸條件研究[J].食品與發酵工業, 2009, 35(12): 43-46.
[20]李增勝,任潤斌.清香型白酒發酵過程中酒酷中的主要微生物[J].釀酒, 2005, 32(5): 33-34.
[21]劉洋,趙婷,姚粟,等.一株芝麻香型白酒高溫大曲嗜熱放線菌的分離與鑒定[J].生物技術通報, 2012(12): 210-216.
[22]葛媛媛,姚粟,劉洋,等.芝麻香型白酒高溫大曲嗜熱細菌群落研究[J].食品與發酵工業, 2012, 38(11): 16-19.
[23]束瑩.古井貢酒大曲中微生物群落結構的鑒定及分析[D].哈爾濱:哈爾濱工業大學, 2013.
Bacterial Diversity of Daqu for Yanggaomeijiu as Determined by PCR-Mediated DGGE
LI Yan1,2, DONG Zhenling1,3, LI Jia1, MOU Dehua1,*
(1. College of Bioscience and Bioengineering, Hebei University of Science and Technology, Shijiazhuang 050018, China; 2. R&D Center for Fermentation Engineering of Hebei Province, Shijiazhuang 050018, China; 3. Hebei Inatural Biological Technology Co. Ltd., Shijiazhuang 050800, China)
Purpose: To detect and analyze the bacterial diversity of Yanggaomeijiu Daqu (traditional Chinese fermentation starter) by polymerase chain reaction-denatured gradient gelelectrophoresis (PCR-DGGE) for further understanding of the flavor characteristics of Yanggaomeijiu. Methods: Samples of Yanggaomeijiu were processed and then extracted to obtain total DNA for PCR amplification and the amplified DNA bands were excised from the gels for sequence analysis by DGGE. Results: By using PCR-DGGE, the genus ofWeissella,Lactobacillus,Pediococcus,Sporolactobacillus,Staphylococcus,Thermoactinomycesand 4 unculturablebacterial strains were identified in Yanggaomeijiu Daqu. Conclusions: This work shows that the bacterial diversity is abundant in Yanggaomeijiu Daqu among which, lactic acid bacteria constituted the largest group.
Yanggaomeijiu Daqu; bacteria; PCR-DGGE
TS261.1
A
1002-6630(2015)12-0142-06
10.7506/spkx1002-6630-201512027
2014-09-29
河北省石家莊市科技支撐計劃項目(101171271A;10117901A);河北省自然科學基金項目(C2011208028)
李艷(1958—),女,教授,本科,研究方向為飲料酒釀造、釀酒微生物。E-mail:lymdh5885@163.com
*通信作者:牟德華(1960—),男,教授,本科,研究方向為農產品加工。E-mail:dh_mou@163.com

