雞肝重和顏色的全基因組連鎖分析
孫艷發,趙桂蘋,劉冉冉,鄭麥青,劉 麗,李 鵬,文 杰*
(1.龍巖學院生命科學學院,龍巖 364012; 2.中國農業科學院北京畜牧獸醫研究所,北京 100193)
雞肝重和顏色的全基因組連鎖分析
孫艷發1,2,趙桂蘋2,劉冉冉2,鄭麥青2,劉 麗2,李 鵬2,文 杰2*
(1.龍巖學院生命科學學院,龍巖 364012; 2.中國農業科學院北京畜牧獸醫研究所,北京 100193)
旨在揭示雞肝重和顏色的遺傳基礎和分子機制,本研究以中國農業科學院(CAAS)雞F2資源群體和遺傳圖譜為基礎,測定300只F2代個體93日齡的肝重、肝率以及肝顏色亮度值(L*)、紅度值(a*)、黃度值(b*),進行這些性狀的全基因組連鎖分析。結果表明,本研究共檢測到影響肝重和顏色5個性狀的11個QTLs,解釋了這些性狀3.39%~6.43%的表型變異;在GGA5上22 cM、GGA12上56 cM和GGA15上1 cM共檢測到影響肝重和肝率3個QTLs;在6條染色體上共檢測到8個影響肝顏色的QTLs。其中GGA15上的2個QTLs,14 cM區間(16~30 cM)影響雞肝L*和b*;GGA1上的2個QTLs,6 cM區間(81~87 cM)影響肝顏色a*和b*。這些QTLs的發現為雞肝重和顏色的精細定位和分子標記輔助選擇奠定基礎。
雞;肝重;肝顏色;全基因連鎖分析
肝作為家禽代謝、免疫的重要器官,參與蛋白質、碳水化合物、脂肪代謝等各種生理生化反應[1]。肝的形態特征如重量和顏色,與其功能存在著密切的聯系。肝重與體重[2-3]、肥胖程度[4]等密切相關。家禽患脂肪肝綜合征[5]、日糧營養物質缺乏[6]以及禁食[7]等因素都會引起肝顏色改變。肝重和顏色也受遺傳因素的影響[8-9]。因此,揭示家禽肝重和顏色的遺傳基礎對于研究其形態和功能的關系具有重要的意義。研究人員將影響肝重的19個數量性狀基因位點(Quantitative trait loci,QTLs)分別定位在雞的14條染色體上,包括1~4、6~8、10、12、15、18、21、24和Z染色體[10-16]。影響肝率的10個QTLs分布在雞的8條染色體上,包括1、4~7、9、10和26號染色體[13]。D.Wright等使用白來航和紅色原雞F2雜交群體和兩種模型將影響肝顏色的QTL分別定位在雞4號染色體222和225 cM處[12]。上述研究使用微衛星標記通過連鎖分析或使用單基因關聯分析獲得影響雞肝重和顏色的QTLs,不能全面解析這些性狀的遺傳基礎。
因此,本研究以先前構建的中國農業科學院(CAAS)雞F2資源群體和遺傳連鎖圖譜為基礎,通過對雞肝重和顏色進行全基因組連鎖分析,鑒定影響它們的QTLs或功能基因,為進一步解析雞肝形態結構特征的分子機制奠定理論基礎。
1 材料與方法
1.1 試驗群體
本研究使用由中國農業科學院北京畜牧獸醫研究所家禽遺傳育種研究室建立的CAAS雞F2資源群體[17-19]進行肝重和顏色的全基因組連鎖分析。該群體親本為中國地方雞品種北京油雞和美國科寶公司白羽肉雞。北京油雞和科寶肉雞按照公母比1∶3,正反交配產生兩組F1代,每組F1代群體內避免半同胞橫交,產生F2代。CAAS雞F2群體各世代保持相同的營養水平和管理方式,飼養于中國農業科學院北京畜牧獸醫研究所北京油雞保種雞場[17-19]。
1.2 遺傳圖譜
本研究使用Y.Sun等構建的CASS雞F2遺傳連鎖圖譜[18]。三世代個體翅靜脈進行采血,ACD(檸檬酸鈉1.32%(M/V),檸檬酸0.48%(M/V)和葡萄糖1.47%(M/V))抗凝,血液樣本-20 ℃保存,用于基因組DNA的提取。使用常規的酚-氯仿法提取血液中的基因組DNA(gDNA),溶于TE溶液中,將質量合格的樣品稀釋至50~75 ng·μL-1,送加拿大DNA LandMarks公司,使用Illumina公司的雞60 K SNP分型芯片進行基因分型。SNP分型結果進行質量控制后,剩余42 585個SNPs用于遺傳圖譜的構建。遺傳連鎖圖譜使用CRI-MAP (2.503a,run in a 64-bit Unix system)軟件,利用CAAS雞F2資源群體中的6個正交家系中的19個半同胞家系(其中F0代有6只北京油雞公雞和12只科寶肉雞母雞,F1代有5只公雞和20只母雞,F2代包括148公雞和152只母雞)和42 585個SNPs進行構建,方法詳見參考文獻[19-20]。構建后遺傳連鎖圖譜總長度為3 040.8 cM,重組率為2.9 cM·Mb-1[21]。
1.3 性狀測定
本研究測定了CAAS資源群體F2代300只雞的肝表型性狀,包括公雞148只,母雞152只。93日齡時對F2代個體進行屠宰,取出肝,稱取肝重(Liver weight,LiW),并以胴體重為標準計算肝率(Percentage of liver weight to carcass weight,LiP)。肝表面顏色采用CIELAB體系進行測定。測定時,每個肝樣本選取2個區域,測定不同區域的亮度(L*)、紅色(a*)和黃色(b*)值,最后求平均值。
1.4 統計分析
使用SAS8.0統計軟件(SAS Institute Inc.,Cary,N.C.,USA)中的MEANS過程對性狀表型數據進行描述性統計分析,使用CORR過程進行性狀間的相關性分析。為了消除連鎖不平衡造成的樣本遺傳相關估計的偏差,利用Plink V1.07軟件[22]篩選出6 518個獨立的SNPs用于連鎖分析[21]。利用web基礎的GridQTL軟件[23]進行性狀和獨立SNPs標記全基因組連鎖分析。選用最小二乘回歸模型使用10 000次迭代運算檢測5%和1%染色體顯著性的QTL[24]。所用模型:
Y=μ+family+batch+sex+(bodyweight)+Caa+Cdd+e
其中,Y為性狀的表型值;μ為性狀的最小二乘均值;family、batch、sex為影響性狀的固定效應,即家系效應、批次效應和性別效應;bodyweight為影響性狀的協變量,分析肝重時,以胴體重為協變量;a和d為加性效應和顯性效應,Ca和Cd為加性效應和顯性效應系數;e為殘差效應。
2 結 果
2.1 表型性狀的描述性統計
肝重和顏色的描述性統計以及性狀間的相關性分析見表1和表2。由表1可知,肝重和肝顏色5個性狀的變異比較大,它們的變異系數為10.77%~32.46%。由表2可知,肝重和肝率之間呈高度正相關(相關系數r2=0.61,P<0.001),與肝表面顏色a*之間呈低度正相關(相關系數r2=0.13,P<0.05)。肝表面顏色L*與a*之間呈低度正相關(r2=0.17,P<0.01),與b*之間呈中度正相關(r2=0.57,P<0.001)。
表1 肝重和顏色的描述性統計
Table 1 Descriptive statistics of liver weight and color
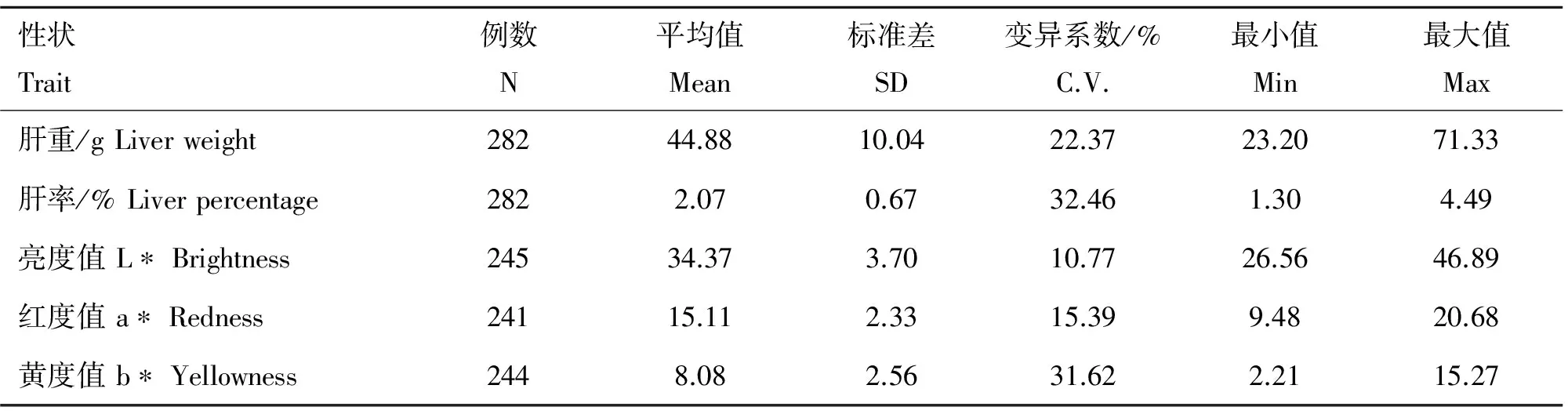
性狀Trait例數N平均值Mean標準差SD變異系數/%C.V.最小值Min最大值Max肝重/gLiverweight28244.8810.0422.3723.2071.33肝率/%Liverpercentage2822.070.6732.461.304.49亮度值L?Brightness24534.373.7010.7726.5646.89紅度值a?Redness24115.112.3315.399.4820.68黃度值b?Yellowness2448.082.5631.622.2115.27
表2 肝重和顏色性狀間的相關性
Table 2 Correlation between liver weight and color

肝重/gLiverweight肝率/%Liverpercentage亮度值L?Brightness紅度值a?Redness黃度值b?Yellowness肝重/gLiverweight1肝率/%Liverpercentage0.61???1亮度值L?Brightness-0.00-0.091紅度值a?Redness0.13?0.010.17??1黃度值b?Yellowness-0.120.000.57???0.071
*.P<0.05;**.P<0.01;***.P<0.001

圖1 雞5號染色體肝重和顏色的全基因組連鎖分析圖Fig.1 Plot of the genome-wide linkage analysis for the liver weight and color on GGA5 in chicken
2.2 雞肝重和顏色的全基因組連鎖分析
肝重和顏色的全基因組連鎖分析結果見表3和圖1(部分圖省略)。由表3可知,共檢測到影響肝重和顏色5個性狀的11個QTLs,這些QTLs解釋了3.39%~6.43%的表型變異。其中在3條染色體上共檢測到3個影響肝重的QTLs。影響肝重的QTLs位于雞5號染色體(GGA5)22 cM處(圖1),達到5%染色體顯著水平。在GGA12上56 cM處和GGA25上1 cM處,檢測到影響肝率的2個QTLs,分別達到5%和1%染色體顯著水平。
本研究在6條染色體上共檢測到8個影響肝顏色的QTLs。影響肝顏色L*的2個QTLs達到5%染色體水平顯著,分別位于GGA15和GGA28上30 cM和48 cM處。影響肝顏色a*的4個QTLs分別位于GGA1上87 cM、GGA5上125 cM(圖1)、GGA18上21 cM處和GGA26上4 cM處。其中GGA18上21 cM處的QTL達到了1%染色體顯著水平,解釋了5.09%的表型變異。影響肝b*的2個QTL分別位于GGA1和GGA15上81和16 cM處,分別達到了5%和1%染色體顯著水平,解釋了6.35%和5.13%的表型變異。
表3 肝重和顏色的全基因組連鎖分析
Table 3 Results of the genome-wide linkage analysis for the liver weight and color

性狀Trait染色體Chromosome遺傳距離/cMPosition置信區間/cMConfidenceintervalF值F?ratio肝重/gLiverweightGGA5223.0~128.07.17?肝率/%LiverpercentageGGA12568.0~62.05.88?肝率/%LiverpercentageGGA2510.0~58.07.36??亮度值L?BrightnessGGA15305.0~53.05.23?亮度值L?BrightnessGGA28481.0~48.05.13?紅度值a?RednessGGA18786.0~401.08.94?紅度值a?RednessGGA51257.0~130.06.84?紅度值a?RednessGGA182110.0~48.07.19??紅度值a?RednessGGA2641.0~47.05.91?黃度值b?YellownessGGA18169.0~437.08.93?黃度值b?YellownessGGA15162.0~42.07.32??性狀Trait染色體Chromosome加性效應Additiveeffect顯性效應DominanceeffectPV/%肝重/gLiverweightGGA5-2.99±0.841.58±1.254.37肝率/%LiverpercentageGGA12-0.09±0.060.26±0.083.46肝率/%LiverpercentageGGA25-0.16±0.060.26±0.094.48亮度值L?BrightnessGGA150.88±0.330.92±0.513.48亮度值L?BrightnessGGA28-0.75±0.32-1.08±0.503.39紅度值a?RednessGGA10.81±0.21-0.58±0.296.43紅度值a?RednessGGA50.69±0.20-0.28±0.304.83紅度值a?RednessGGA180.28±0.191.03±0.305.09紅度值a?RednessGGA26-0.42±0.20-0.78±0.314.08黃度值b?YellownessGGA10.83±0.24-0.87±0.346.35黃度值b?YellownessGGA150.81±0.22-0.42±0.355.13
*.5%染色體顯著水平;**.1%染色體顯著水平;PV.QTL解釋表型方差的百分比
*.5% chromosome-wide significance;**.1% chromosome-wide significance;PV.QTL explain the ratio of phenotypic variation
3 討 論
本研究中,CAAS雞F2資源群體中5個肝重和顏色性狀的變異比較大,變異系數為10.77%~31.62%,說明性狀處于分離狀態,有利于這些性狀的QTLs定位。此外,肝重和肝率、肝重與肝顏色a*、肝顏色L*與a*和b*存在從低度到高度的正相關,說明這些性狀間可能具有相同的遺傳基礎。
本研究共檢測到3個影響雞肝重的QTLs,其中1個影響肝重的QTL位于GGA5上22 cM處。通過AnimalQTLdb數據庫[25]檢索發現,GGA5上并沒有影響雞肝重的QTLs,但在5號染色體22 cM附近發現多個影響雞體重的QTLs[26]。通過檢索UCSC雞基因組數據庫發現,該QTL峰值處于人輸入蛋白7(Importin 7,IPO7)基因所在的基因組位置。IPO7基因編碼蛋白為大約20個潛在的Ran靶蛋白中的一員,該基因與人的高血壓和血壓密切相關[27],未見到該基因與肝重的功能研究。另外2個影響雞肝率的QTLs分別位于雞GGA12上56 cM處和GGA15上1 cM。在GGA12上25.9 cM處存在影響雞肝重的QTL[12],同時在44~65 cM范圍內存在影響雞體重的QTL[28-29]。可見,GGA12上發現的這個QTL為影響雞肝率的QTL。該QTL峰值位于人膜關聯的鳥苷酸激酶WW和PDZ結構域包含蛋白1異構體(Membrane-associated guanylate kinase WW and PDZ domain-containing protein 1 isoform,MAGI1)基因所在的基因組位置。該基因編碼蛋白在黏附連接的穩定性以及侵襲力和轉移抑制方法具有重要作用,其與肝癌的診斷密切相關[30]。本研究在GGA15上1 cM處發現影響雞肝率的QTL與5~49 cM較大的范圍內存在影響雞肝重和體重的QTLs[10,31]相距不遠。本研究檢測到的3個QTLs以及相關基因并未在先前的研究中有報告,它們可能是影響肝重和肝率的新的QTLs和候選基因。
先前的研究中只在4號染色體上發現了1個影響雞肝顏色的QTL[12]。本研究共發現 8個新的影響肝顏色的QTLs。其中在GGA15上14 cM區間(16~30 cM)存在影響肝顏色L*和b*的2個QTLs。由于這2個QTLs距離比較近,而且表型性狀間存在中等的正相關,因此,GGA15上14 cM區間為影響雞肝顏色重要的QTL。GGA1上6 cM區間(81~87 cM)存在影響肝顏色a*和b*的QTLs,這個QTL距離不遠(94~118 cM)存在影響雞肝重的QTL[12],由于肝重與肝顏色a*存在低度的正相關,所以這區間可能為影響雞肝顏色重要的QTL區間。GGA5上影響雞肝a*的QTL位于125 cM處,該QTL與影響肝率的QTL約7 cM[13],且位于影響雞體重的QTL內[32]。GGA18上21 cM處影響肝顏色a*的QTL也位于影響雞體重的QTLs內[29,33]。由于雞體重與肝重、肝重與a*存在正相關,這些QTLs可能也影響雞肝顏色。
4 結 論
本研究共檢測到影響肝重和顏色的5個性狀的11個QTLs,解釋了這些性狀3.39%~6.43%的表型變異;在GGA5上22 cM、GGA12上56 cM和GGA15上1 cM共檢測到影響肝重和肝率的3個QTLs;在6條染色體上共檢測到8個影響肝顏色的QTLs。其中GGA15上的2個QTLs,14 cM區間(16~30 cM)影響雞肝L*和b*;GGA1上的2個QTLs,6 cM區間(81~87 cM)影響肝顏色a*和b*。
[1] 岳 穎.不同基因型肉仔雞肝臟蛋白組學研究[D].北京:中國農業科學院,2013. YUE Y.Proteomic analysis on different genotype broilers liver[D].Beijing:Chinese Academy of Agricultural Sciences,2013.(in Chinese)
[2] VAUTHEY J N,ABDALLA E K,DOHERTY D A,et al.Body surface area and body weight predict total liver volume in Western adults[J].LiverTranspl,2002,8(3):233-240.
[3] TRUANT S,OBERLIN O,SERGENT G,et al.Remnant liver volume to body weight ratio>or=0.5%:a new cut-off to estimate postoperative risks after extended resection in noncirrhotic liver[J].JAmCollSurg,2007,204(1):22-33.
[4] 楊愛君,崔 雁,葉卉初,等.營養性肥胖動物模型的建立[J].臨床和實驗醫學雜志,2005,4(3):156-157. YANG A J,CUI Y,YE H C,et al.The establishment of the diet-induced obesity animal model[J].JournalofClinicalandExperimentalMedicine,2005,4(3):156-157.(in Chinese)
[5] 劉建國,陳 亮,王宗元,等.雞脂肪肝綜合癥病例主要內臟器官病理學觀察[J].廣東農業科學,2010,37(9):156-157. LIU J G,CHEN L,WANG Z Y,et al.Pathomorphology observation for fatty liver hemorrhagic syndrome in laying hens.[J].GuangdongAgriculturalSciences,2010,37(9):156-157.(in Chinese)
[6] 文 杰,王和民.日糧煙酸水平對產蛋雞生產性能及脂肪代謝的影響[J].營養學報,1993,15(2):200-206. WEN J,WANG H M.Effect of niacin on egg production and lipid metabolism of laying hens[J].ActaNutrimentaSinica,1993,15(2):200-206.(in Chinese)
[7] TRAMPEL D,SELL J,AHN D,et al.Preharvest feed withdrawal affects liver lipid and liver color in broiler chickens[J].PoultSci,2005,84(1):137-142.
[8] GJERDE B,GJEDREM T.Estimates of phenotypic and genetic parameters for carcass traits in Atlantic salmon and rainbow trout[J].Aquaculture,1984,36(1):97-110.
[9] DOUAIRE M,LE FUR N,EL KHADIR-MOUNIER C,et al.Identifying genes involved in the variability of genetic fatness in the growing chicken[J].PoultSci,1992,71(11):1911-1920.
[10] NAVARRO P,VISSCHER P,KNOTT S,et al.Mapping of quantitative trait loci affecting organ weights and blood variables in a broiler layer cross[J].BrPoulSci,2005,46(4):430-442.
[11] NONES K,LEDUR M,RUY D,et al.Mapping QTLs on chicken chromosome 1 for performance and carcass traits in a broiler × layer cross[J].AnimGenet,2006,37(2):95-100.
[12] WRIGHT D,KERJE S,LUNDSTR?M K,et al.Quantitative trait loci analysis of egg and meat production traits in a Red Jungle fowl × White Leghorn cross[J].AnimGenet,2006,37(6):529-534.
[13] ZHOU H,DEEB N,EVOCK-CLOVER C,et al.Genome-wide linkage analysis to identify chromosomal regions affecting phenotypic traits in the chicken.II.Body composition[J].PoultSci,2006,85(10):1712-1721.
[14] D′ANDRé HIRWA C,YAN W,WALLACE P,et al.Effects of the thyroid hormone responsive spot 14α gene on chicken growth and fat traits[J].PoultSci,2010,89(9):1981-1991.
[15] HAN R,WEI Y,KANG X,et al.Novel SNPs in the PRDM16 gene and their associations with performance traits in chickens[J].MolBiolRep,2012,39(3):3153-3160.
[16] LI S,CHEN W,KANG X,et al.Distinct tissue expression profiles of chicken Lpin1-α/β isoforms and the effect of the variation on muscle fiber traits[J].Gene,2013,515(2):281-290.
[17] 孫艷發,劉冉冉,鄭麥青,等.雞脛長和脛圍的全基因組關聯分析[J].畜牧獸醫學報,2013,44(3):358-365. SUN Y F,LIU R R,ZHENG M Q,et al.Genome-wide association study for shank length and shank girth in chicken[J].ActaVeterinariaetZootechnicaSinica,2013,44(3):358-365.(in Chinese)
[18] SUN Y,ZHAO G,LIU R,et al.The identification of 14 new genes for meat quality traits in chicken using a genome-wide association study[J].BMCGenomics,2013,14(1):458.
[19] GROENEN M A,WAHLBERG P,FOGLIO M,et al.A high-density SNP-based linkage map of the chicken genome reveals sequence features correlated with recombination rate[J].GenomeRes,2009,19(3):510-519.
[20] ELFERINK M G,VAN AS P,VEENENDAAL T,et al.Regional differences in recombination hotspots between two chicken populations[J].BMCGenet,2010,11:11.
[21] SUN Y,LIU R,ZHAO G,et al.Genome-wide linkage analysis and association study identifies loci for polydactyly in chickens[J].G3:GenesGenomesGenetics,2014,4(6):1167-1172.
[22] PURCELL S,NEALE B,TODD-BROWN K,et al.PLINK:a tool set for whole-genome association and population-based linkage analyses[J].AmJHumGenet,2007,81(3):559-575.
[23] SEATON G,HERNANDEZ J,GRUNCHEC J A,et al.GridQTL:a grid portal for QTL mapping of compute intensive datasets[C].Belo Horizonte Brazil:Proceedings of the 8th world congress on genetics applied to livestock production 2006,2006:13-18.
[24] CHURCHILL G A,DOERGE R W.Empirical threshold values for quantitative trait mapping[J].Genetics,1994,138(3):963-971.
[25] HU Z L,FRITZ E R,REECY J M.Animal QTLdb:a livestock QTL database tool set for positional QTL information mining and beyond[J].NucleicAcidsRes,2007,35(Database issue):604-609.
[26] WANG S,HU X,WANG Z,et al.Quantitative trait loci associated with body weight and abdominal fat traits on chicken chromosomes 3,5 and 7[J].GenetMolRes,2012,11(11):956-965.
[27] ADEYEMO A,GERRY N,CHEN G,et al.A genome-wide association study of hypertension and blood pressure in African Americans[J].PLoSGenet,2009,5(7):e1000564.
[28] MELISSA A,PATRICIA Y,DIANE E.Identification of quantitative trait loci associated with bone traits and body weight in an F2 resource population of chickens[J].GenetSelEvol,2005,37:677-698.
[29] AMBO M,MOURA A,LEDUR M,et al.Quantitative trait loci for performance traits in a broiler × layer cross[J].AnimGenet,2009,40(2):200-208.
[30] ZHANG G,LIU T,WANG Z.Down regulation of MAGI1 associates with poor prognosis of hepatocellular carcinoma[J].JInvestSurg,2012,25(2):93-99.
[31] PODISI B K,KNOTT S A,BURT D W,et al.Comparative analysis of quantitative trait loci for body weight,growth rate and growth curve parameters from 3 to 72 weeks of age in female chickens of a broiler-layer cross[J].BMCGenet,2013,14(1):22.
[32] JACOBSSON L,PARK H B,WAHLBERG P,et al.Many QTLs with minor additive effects are associated with a large difference in growth between two selection lines in chickens[J].GenetRes,2005,86(2):115-125.
[33] CARLBORG ?,KERJE S,SCHüTZ K,et al.A global search reveals epistatic interaction between QTL for early growth in the chicken[J].GenomeRes,2003,13(3):413-421.
(編輯 郭云雁)
Genome-wide Linkage Analysis for Liver Weight and Color in Chicken
SUN Yan-fa1,2,ZHAO Gui-ping2,LIU Ran-ran2,ZHENG Mai-qing2,LIU Li2,LI Peng2,WEN Jie2*
(1.CollegeofLifeScience,LongyanUniversity,Longyan364012,China;2.InstituteofAnimalScience,ChineseAcademyofAgriculturalSciences,Beijing100193,China)
To reveal the genetic basis and molecular mechanism of liver weight and color in chicken,5 traits,including liver weight,percentage of liver weight to carcass weight and liver color(lightness L*,redness a* and yellowness b*) were measured from 300 93-day-old F2 generation chickens from Chinese Academy of Agricultural Sciences(CAAS) chicken F2 resource population.Genome-wide linkage analysis for the liver weight and color traits were preformed on the basis of the CAAS chicken F2 resource population and genetic linkage map.The results of the present study showed that 11 quantitative trait loci(QTLs) for 5 traits were found,which explained 3.39%-6.43% of the phenotypic variation.A total of 3 QTLs on 3 chicken(Gallusgallus) chromosomes,at 22 cM on chicken chromosome 5(GGA5),56 cM on GGA12 and 1 cM on GGA15,for liver weight and the percentage of liver weight to carcass weight were identified.A total of 8 QTLs for liver color were found on 6 chicken chromosomes.Two interesting QTL regions for liver color were found,one region spanned 14 cM(16-30 cM) on GGA15 containing 2 QTLs for the chicken liver L* and b*,and the other region spanned 6 cM(81-87 cM) on GGA1 containing 2 QTLs for the chicken liver a* and b*.The present study provides the basis for further fine mapping and molecular marker-assisted selection for liver weight and color traits in chicken.
chicken;liver weight;liver color;genome-wide linkage analysis
10.11843/j.issn.0366-6964.2015.11.025
2015-04-01
農業產業技術體系項目(nycytx-42);龍巖學院博士科研啟動基金項目(LB2013008)
孫艷發(1981-),男,黑龍江人,博士,主要從事家禽營養與育種研究,E-mail:boysun2010@163.com
*通信作者:文 杰,研究員,主要從事家禽育種研究,E-mail:wenj@caas.net.cn
S831.2
A
0366-6964(2015)11-2104-07

