鈍頂螺旋藻FtsZ蛋白結構分析及同源建模
沈清清,田雪琪,常征
(文山學院 環境與資源學院,云南 文山 663099)
鈍頂螺旋藻FtsZ蛋白結構分析及同源建模
沈清清,田雪琪,常征
(文山學院 環境與資源學院,云南 文山 663099)
采用生物信息學的研究方法,應用一系列工具軟件分析研究鈍頂螺旋藻FtsZ蛋白,以認識鈍頂螺旋藻FtsZ蛋白一級和二級結構,并為進一步研究該蛋白空間結構和功能機制奠定理論基礎。獲得鈍頂螺旋藻FtsZ蛋白一級結構相關理化參數的詳細數據,預測蛋白二級結構,對蛋白的3D結構進行了同源建模、質量評價分析。研究結果表明鈍頂螺旋藻FtsZ蛋白是一種酸性蛋白,主要定位于細胞核區;蛋白二級結構主要由無規則卷曲和α-螺旋兩種結構組成;同源建模后得到的3D模型在空間構象上有較高的合理性,其中模型A有100%氨基酸殘基數落于Ramachandran圖的允許區,該模型含有14個α-螺旋結構,5個βα-β單位,2個β-折疊結構,1個β-發夾結構,16個β-轉角和1個γ-轉角。
蛋白;原核生物;同源建模
鈍頂螺旋藻(Αrthrospira platensis)是一種光合自養型藍藻,富含維生素、蛋白質和多糖等生物活性物質,具有較高的經濟價值。鈍頂螺旋藻是由許多柱狀單細胞構成的藻絲體,具有規則的螺旋狀或波浪卷曲狀的形態特征,但當環境條件或環境因子變化時,藻絲體的彎曲度、螺旋度和長度均有可能發生改變,甚至變成不可逆的直線形態,另外,有研究也證實鈍頂螺旋藻的藻絲體能自發的發生形態變異[1-2]。許多科研人員對螺旋藻形態轉變的機制進行了大量的假設和研究,但目前仍然沒有獲得肯定的答案。隨著FtsZ、MreB 和 Cres等原核生物細胞骨架蛋白的發現[3-4],許多科研人員逐漸把目光轉向了研究原核生物細胞骨架蛋白與螺旋藻藻絲體形態建成是否存在相關性,以期從這一角度挖掘出螺旋藻形態變異確切的答案。早在1980年,Lutkenhaus JF等[5]就已經發現細胞骨架蛋白FtsZ是調控原核生物細胞分裂的主要物質,與真核細胞微管蛋白具有許多類似的結構與功能。之后Mingorance J[6]和Owawa M[7]發現FtsZ能與GTP 結合裝配成FtsZ 原絲,這種FtsZ 原絲是中空的,能定位于細胞分裂的位點,在細胞分裂時裝配形成一個稱為Z環的環狀結構,細胞隔膜和新生壁的形成依賴于這一環狀結構。目前在C.crescentus和E.coli兩種原核生物中除了發現FtsZ環的存在與分裂位點新細胞壁的合成密切相關外,還與細胞兩極附近側壁合成有關,表明FtsZ在細胞壁的形態建成上起著非常重要的作用[8]。
2012年,鄒路陽等[1]研究了不同形態鈍頂螺旋藻藻絲體蛋白表達的情況,發現直線形和螺旋形藻絲體中原核骨架蛋白FtsZ的表達量相差較大,說明鈍頂螺旋藻的形態建成與FtsZ密切相關。這一發現對研究鈍頂螺旋藻形態變異的機制有著很大的促進作用,但目前有關鈍頂螺旋藻中FtsZ蛋白詳細的化學信息還未見報道,其晶體結構也尚未解析,生物大分子蛋白質的三級結構決定蛋白質的生物學功能[9],因此本研究旨在通過利用生物信息學的方法分析FtsZ蛋白的一、二級結構的理化性質,對其三級結構進行同源建模,為進一步研究FtsZ空間結構和功能機制奠定理論基礎。
1 序列來源
供項目研究的分析序列來源于美國生物信息中心(NCBI)的 Protein數據庫,序列信息:FtsZ (Αrthrospira platensis NIES-39),序列登錄號:BAI93200.1。
2 方法
2.1FtsZ蛋白的一、二級結構分析
2.1.1一級結構分析
將鈍頂螺旋藻FtsZ蛋白的氨基酸序列提交至瑞士ExPASy網站信息系統,利用該網站的工具ProtParam計算蛋白各方面的化學與物理參數值;利用亞細胞定位預測工具PSORT Prediction計算蛋白理論上的定位位點[10-11]。
2.1.2二級結構分析
將鈍頂螺旋藻FtsZ蛋白序列發送至法國里昂生物化學與蛋白研究所(IBCP)提供的服務器NPS@,用在線工具 SOPMA預測蛋白的二級結構,分析蛋白的局部結構元件[11]。
2.2蛋白的同源建模及評估
在瑞士生物信息學研究所網頁上運行在線工具SWISS-MODEL,利用同源建模法對提交的鈍頂螺旋藻FtsZ蛋白氨基酸序列進行計算,從PDB蛋白庫中選擇具顯著同源性的模板序列,構建出理想匹配值的3D模型,輸出相關建模參數;利用PROCHECK結構分析與驗證服務器對3D模型空間結構的可信度與合理性進行評估[10-11],輸出Ramachandran圖。
3 結果與分析
3.1FtsZ蛋白氨基酸組成及理化性質分析
鈍頂螺旋藻FtsZ蛋白分子式C1941H3157N565O625S5,由426個氨基酸組成,分子量是44.57 kD,含20種常見氨基酸,其含量見表1,其中丙氨酸(Ala)含量最高,為11.0%,其次是甘氨酸(Gly),含量為10.8%。酸性氨基酸殘基總數較堿性氨基酸總數多10,因此該蛋白屬酸性蛋白。脂肪族氨基酸指數為92.09,理論等電點PI為4.94,原子總數是6293,其疏水性平均值(GRAVY)為-0.171,N末端氨基酸為蛋氨酸(Met),蛋白的半衰期估算值為30 h。PSORT Prediction預測工具對FtsZ蛋白的亞細胞定位位點進行了預測,計算出的結果表明細胞核區是FtsZ蛋白的主要定位區域,其概率比較大,值為60%。
3.2FtsZ蛋白二級結構預測與分析
利用在線工具SOPMA預測分析鈍頂螺旋藻FtsZ蛋白的二級結構,結果如圖1所示,FtsZ蛋白二級結構中含39.91%的無規則卷曲(Random coil)結構,由170個氨基酸組成,33.80%的α-螺旋(Alpha helix)結構,由144個氨基酸組成,17.84%的擴展長鏈(Extended strand)結構,由76個氨基酸組成,8.45%的β-轉角結構,由36個氨基酸組成。以上數據表明無規則卷曲和α-螺旋是鈍頂螺旋藻FtsZ蛋白的主要成分。

表1 氨基酸組成及含量
3.3FtsZ蛋白的三級結構分析及同源建模
運行在線工具SWISS-MODEL,從PDB蛋白庫中篩選出具有顯著同源性的3個模板序列,PDB id分別為2vam.1、4dxd.1和4m8i.1,FtsZ蛋白的氨基酸序列與這3個模板的序列同源性分別為61.08%、59.31%和59.31%,同源建模后獲得的3D模型如圖2所示,對這3個3D模型結構的合理性與質量進行PROCHECK評估,輸出Ramachandran圖(見圖3),圖3中顯示模型A有100%的氨基酸殘基落在允許區,其中96.1%落在最適區,3.9%落在附加區;模型B有98.8%的氨基酸殘基落在允許區,其中92.6%落在最適區,6.2%落在附加區,剩余1.2%落在非允許區;模型C有99.6%的氨基酸殘基落在允許區,其中92.3%落在最適區,7.3%落在附加區,0.4%落在非允許區。以上結果表明通過SWISSMODEL構建的3個模型在空間結構上符合立體化學規則,合理性較高,各氨基酸殘基的落點滿足二面角的分布要求,其中模型A最能反映出FtsZ蛋白真實的空間構象,該模型含有14個α-螺旋結構,5個βα-β單位,2個β-折疊結構,1個β-發夾結構,16個β-轉角和1個γ-轉角。
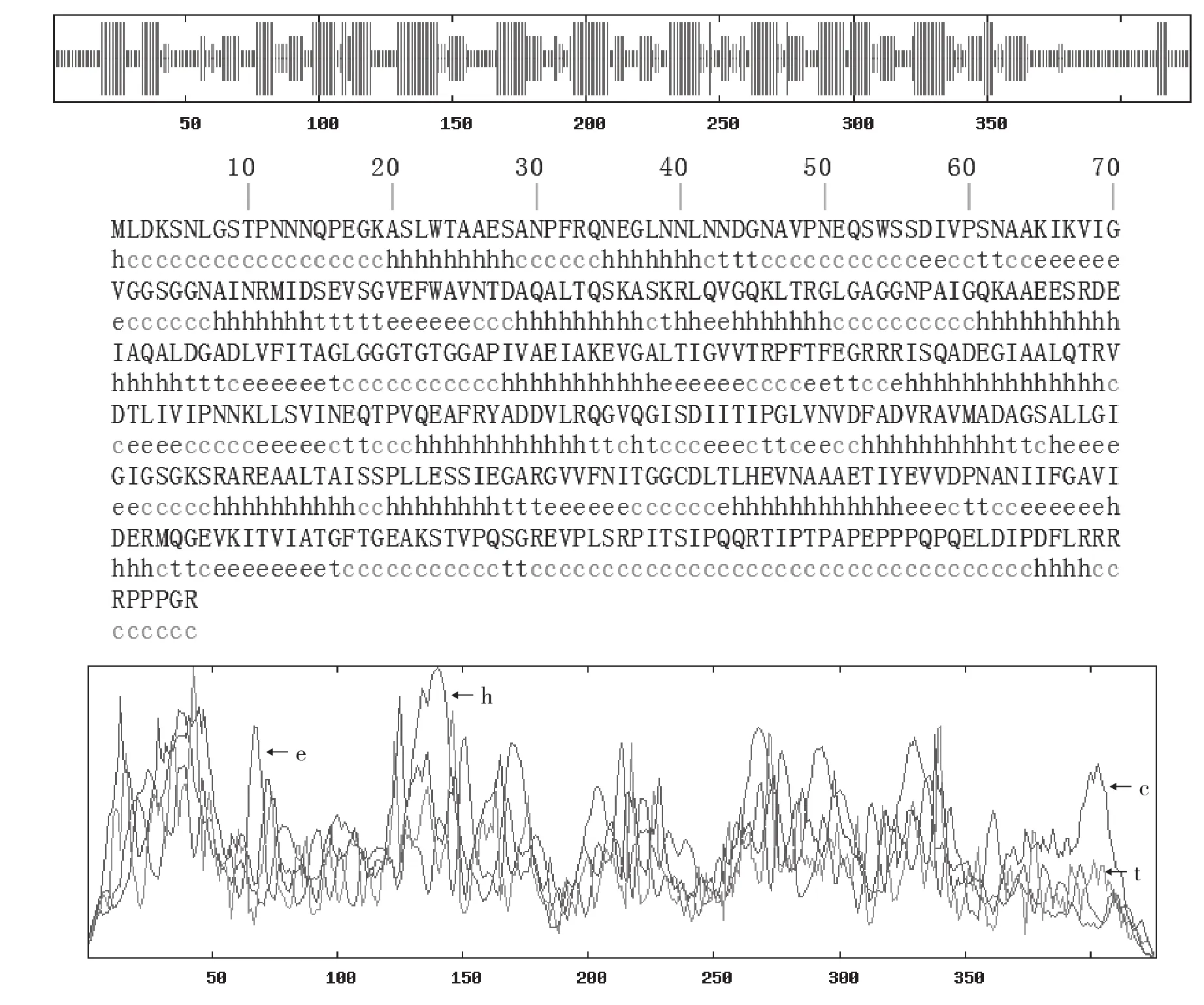
圖1 FtsZ二級結構分析
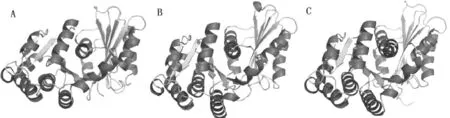
圖2 蛋白同源建模后的模型
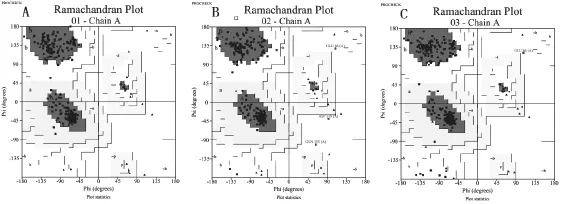
圖3 蛋白氨基酸殘基二面角分布圖(Ramachandran圖)
4 結論
1)鈍頂螺旋藻FtsZ蛋白由426個氨基酸組成,含20種氨基酸,其中丙氨酸(Ala)含量最高為11.0%,為酸性蛋白。PSORT的分析表明FtsZ蛋白主要定位于鈍頂螺旋藻的細胞核區,其概率值較高為60%。
2)鈍頂螺旋藻FtsZ蛋白二級結構預測結果表明無規則卷曲和α-螺旋是其主要結構,其比例分別為39.91%和33.80%。
3)SWISS-MODEL系統為建模選擇的3個模板與鈍頂螺旋藻FtsZ蛋白序列同源性均較高,均在59%以上,因此構建出的3D模型PROCHECK評價結果均屬高質量模型,其中模型A 有100%的氨基酸殘基落在允許區,具有較高利用價值。
[1] 鄒路陽,吳娟,曾群安,等. FtsZ在鈍頂螺旋藻形態建成中的作用[J]. 水生生物學報, 2012(5):898-904.
[2] 林重陽.鈍頂螺旋藻兩種藻絲體的差異膜蛋白分析及肌動蛋白類似物的初步研究[D]. 廈門:廈門大學, 2009.
[3] 張斌,東秀珠. FtsZ蛋白同源性分析在乳酸菌系統學研究中的應用[J]. 微生物學報, 2005(5):661-664.
[4] 陳曉燕,汪志平,楊靈勇. 原核細胞骨架蛋白的結構與功能[J]. 細胞生物學雜志, 2006(5):699-703.
[5] Lutkenhaus JF,Wolf-Watz H,Donachie WD. Organization of genes in the ftsA-envA region of the Escherichia coli genetic map and identification of a new fts locus (ftsZ)[J]. J Bacteriol, 1980(2):615-620.
[6] Mingorance J,Tamames J,Vicente M. Geonmic channelingin bacterial cell division [J]. Journal Molecular Recogintion, 2004(5): 481-487.
[7] Owawa M,Anderson DE,Erichson HP. Reconstitution of contractile FtsZ rings in liposomes [J]. Science,2008(5877): 792-794.
[8] Cabeen MT,Jacobs-Wagner C. Skin and bones: the bacterial cytoskeleton,cell wall,and cell morphogenesis[J]. The Journal of Cell Biology,2007(3):381-387.
[9] Reisler E,Egelman EH. Actin Structure and Function:What We Still Do Not Understand[J]. Journal of Biological Chemistry,2007(50):36133-36137.
[10] 沈清清,劉芳,陳紅惠. 維氏氣單胞菌MreB蛋白的結構分析及其系統發育學意義[J]. 微生物學通報, 2015(2):272-279.
[11] 沈清清,劉芳,趙芳,等. MreB蛋白與actin蛋白的同源建模和進化關系研究[J]. 文山學院學報,2015(3):31-37.
Structural Analysis and Homology Modeling of FtsZ Protein in Arthrospira Platensis
SHEN Qingqing, TIAN Xueqi, CHANG Zheng
(School of Environment and Resources, Wenshan University, Wenshan Yunnan 663099, China)
To understand the fi rst and secondary structures of Arthrospira Platensis FtsZ protein, and establish the foundation for further investigating its three-dimensional structure and function mechanism, the paper adopts biological information approaches and series of soft wares to study it. Online soft wares are used to analyze the primary structure and physicochemical properties of the protein, and its secondary structure is predicted. A threedimensional structure is established and a quality evaluation has been done. The research results show that FtsZ protein of Arthrospira Platensis is an acidic protein composed of 426 amino acids, which is mainly located in the cell nucleus. Secondary structure of the protein mainly consists of random coil and α-helices structures. The model built by homology modeling presents a reasonable spatial structure. Besides, Model A is high-quality model with 100% residual base of amino acid set in the allowed regions. The results of homology modeling reveal that the tertiary structure of the protein contains 14 α-helical structures, 5 beta-alpha-beta units structures, 2 β-folded structures, 1 beta bulge structures, 16 β-turns and 1 γ-turns.
Protein; prokaryote; homology modeling
TQ936.2
A
1674 - 9200(2015)06 - 0035 - 04
(責任編輯 張鐵)
2015 - 09 - 14
文山學院科研基金項目“真核與原核生物細胞骨架蛋白的同源建模比對分析及系統進化關系研究”(11WSY01)。
沈清清,文山學院環境與資源學院副教授,碩士。

