外周血單個核細胞hMLH1啟動子甲基化與類風濕關節炎發生發展的關系
甄艷鳳,劉興宇,周云濤,叢斌,付麗紅,李淑瑾,馬春玲,倪志宇,姚玉霞
外周血單個核細胞hMLH1啟動子甲基化與類風濕關節炎發生發展的關系
甄艷鳳,劉興宇,周云濤,叢斌,付麗紅,李淑瑾,馬春玲,倪志宇,姚玉霞
目的探討錯配修復基因-hMLH1啟動子甲基化及基因失活在類風濕關節炎(RA)發生發展中的作用。方法選取2006年4月—2007年4月河北醫科大學附屬第二醫院及第三醫院新診斷的RA患者33例為病例組,另選擇同期河北省血液中心提供的健康人群38例為對照組。取受試者外周血,以密度梯度離心法分離外周血單個核細胞。分別以RT-PCR法、蛋白印跡法檢測外周血單個核細胞hMLH1 mRNA、蛋白相對表達量。經亞硫酸氫鈉修飾后,分別設計hMLH1啟動子甲基化和非甲基化的DNA引物,檢測病例組和對照組患者hMLH1啟動子甲基化結果。結果病例組hMLH1 mRNA相對表達量為0.56(0.24,0.69),低于對照組的0.80(0.68,0.93),差異有統計學意義(Z=-3.98,P<0.05)。病例組hMLH1蛋白相對表達量為0.70(0.62,0.76),低于對照組的0.80(0.68,0.94),差異有統計學意義(Z=-2.23,P<0.05)。病例組hMLH1啟動子甲基化陽性率為90.9%(30/33),高于對照組的13.2%(5/38),差異有統計學意義(χ2=42.72,P<0.05)。hMLH1啟動子甲基化程度與mRNA、蛋白相對表達量呈負相關(rs=-0.866、-0.541,P<0.01)。類風濕因子陽性患者hMLH1 mRNA、蛋白相對表達量低于陰性患者,差異有統計學意義(P<0.05);兩者hMLH1啟動子甲基化陽性率比較,差異無統計學意義(P>0.05)。結論hMLH1啟動子甲基化可能是hMLH1基因失活的重要機制,hMLH1異常高甲基化可能參與了RA的發生發展。
類風濕,關節炎;DNA錯配修復;hMLH1;DNA甲基化
甄艷鳳,劉興宇,周云濤,等.外周血單個核細胞hMLH1啟動子甲基化與類風濕關節炎發生發展的關系[J].中國全科醫學,2016,19(7):808-812.[www.chinagp.net]
Zhen YF,Liu XY,Zhou YT,et al.hMLH1 promoter methylation from peripheral blood mononuclear cells of patients with newly diagnosed rheumatoid arthritis[J].Chinese General Practice,2016,19(7):808-812.
類風濕關節炎(RA)為全身性自身免疫性疾病,患病率為0.3%~0.4%[1],其具體發病機制尚不明確。DNA錯配修復(MMR)系統是重要的突變避免系統,通過校正正常DNA代謝中產生的堿基錯配及調節DNA損傷所誘導的凋亡來維持基因組的穩定性。MMR為細胞準確復制和增殖的必要手段,若MMR系統異常,細胞突變頻率可增加100倍以上[2]。hMLH1基因是維持MMR基因完整性和穩定性的重要基因,其易發生啟動子甲基化。hMLH1基因啟動子超甲基化普遍存在,并伴有hMLH1蛋白的低表達[3-4]。研究發現,由于MMR基因的缺失,導致結直腸癌、胃癌細胞中的微衛星不穩定性(MSI)[5-6]。RA患者也存在MSI,且其關節內MMR酶合成增加[7-8]。然而,RA患者hMLH1基因啟動子甲基化狀態如何,與RA發病是否有相關性,國內外尚罕見報道,為此,本研究對其進行了探討。
1 對象與方法
1.1 研究對象 選取2006年4月—2007年4月河北省第二醫院及第三醫院新診斷的RA患者33例為病例組,其中男8例,女25例;平均年齡(46±12)歲,均符合1987年美國風濕病協會RA診斷標準[9],并排除已接受藥物治療、非新診斷RA及合并其他疾病者。另選擇同期河北省血液中心提供的健康人群38例為對照組,其中男10例,女28例;平均年齡(45±10)歲。兩組性別、年齡比較,差異均無統計學意義(=0.04、 t年齡=0.54,P>0.05)。
1.2 方法
1.2.1 主要試劑 hMLH1鼠抗人單克隆抗體購自美國ZYMED公司,亞硫酸氫鈉和對苯二酚購自美國New Jersey公司,Sss-1酶購自美國NEB公司,UltraPureTM基因組DNA快速提取試劑盒、RT-PCR、MSP反應體系及TaqDNA聚合酶、引物購自北京賽百盛基因技術有限公司,淋巴細胞分離液購自上海恒信化學試劑有限公司,Tris飽和酚購自武漢博士德生物工程公司,蛋白酶K、RNA酶抑制劑、HEPES購自美國Promega公司。
1.2.2 密度梯度離心法分離外周血單個核細胞[10]取受試者外周血,4 h內從下層吸出全血2 ml,與2 ml 0.9%氯化鈉溶液充分吹打混勻,余全血-70℃備用;將充分吹打混勻的混合液用滴管逐滴平鋪于2 m l淋巴細胞分離液(比重=1.077 g/cm3)上,二者界面不能打散。4℃條件下密度梯度離心,離心加速度為265×g,2 000 r/min離心20 min;吸出白色膜狀的單個核細胞層于EP管中,即得單個核細胞。
1.2.3 RT-PCR法檢測外周血單個核細胞hMLH1 mRNA表達 Trizol一步法提取外周血單個核細胞RNA,cDNA合成反應體系20μl,其中5×RT buffer 4.0μl,dNTP 0.8 mmol/L,AMV(1 U/μl)2.0μl,模板RNA 10μl,隨機引物(100 pmol/μl)0.5μl。循環參數:30℃預變性10 min,42℃反轉錄1 h,95℃滅活反轉錄酶5 min,-20℃備用。hMLH1引物序列參考文獻[11],上游:5′-GTGCTGGCAATCAAGGGACCC-3′,下游:5′-CACGGTTGAGGCAATGGGTAG-3′,擴增產物215 bp。根據引物設計原則,應用Primer 5引物設計軟件設計內參β-actin引物序列,上游:5′-CATCCTGCGTCTGGACCT-3′,下游:5′-TCAGGAGCAATGATCTTG-3′,擴增產物480 bp。RT-PCR反應體系20μl,其中10×PCR buffer 2.0μl,dNTP 0.2 mmol/L,二氯化鎂(MgCl2)2.0 mmol/L,TaqE(1 U/μl)1.0μl,模板DNA 3.3μl,上下游引物(100 pmol/μl)各0.3μl。RT-PCR循環參數:95℃預變性5 min,94℃變性45 s,55℃(hMLH1)/57℃(β-actin)退火1 min,72℃延伸45 s,共35個循環,72℃繼續延伸5 min。取10μl PCR反應產物經1.5%瓊脂糖凝膠(含溴化乙錠)100 V恒壓電泳50 min后,紫外燈下觀察結果,拍照,掃描至計算機中。用Gel-pro凝膠分析軟件對電泳譜帶進行半定量分析,以hMLH1與β-actin凝膠譜帶的面積×熒光強度值的比值表示hMLH1 mRNA相對表達量。
1.2.4 蛋白印跡法檢測外周血單個核細胞hMLH1蛋白表達 提取外周血單個核細胞核蛋白,hMLH1抗體稀釋度為1∶150。陰性對照以PBS代替一抗,以β-actin為參照。將顯色條帶掃描至計算機中,用UVP凝膠圖像分析管理系統對結果進行半定量分析,以hMLH1與β-actin凝膠譜帶的面積×熒光強度值的比值表示hMLH1蛋白相對表達量。
1.2.5 hMLH1啟動子甲基化檢測[12]
1.2.5.1 亞硫酸氫鈉修飾 取2~5μg DNA去離子水定容至50μl,加入終濃度為0.2 mol/L的氫氧化鈉(NaOH)5.5μl,37℃水浴30 min;加30μl的10 mmol/L對苯二酚,3 mol/L的重亞硫酸鈉(pH=5.0)520μl,混勻后,其上覆蓋200μl液狀石蠟,50℃水浴18 h。按說明書使用基因組DNA快速提取試劑盒純化去鹽,50μl TE buffer洗脫DNA,加入終濃度為0.3 mol/L的NaOH 5.5μl,室溫5 min終止反應。加1/10體積的乙酸鈉和2倍體積的無水乙醇,-70℃保存1 h;離心加速度為1 879×g,14 000 r/min離心10 min;加入1/3體積70%乙醇溶液洗滌1次,再次離心獲得沉淀。所得DNA溶于20μl高壓去離子水中,-20℃保存或直接進行PCR擴增。
1.2.5.2 hMLH1啟動子甲基化分析 分別設計hMLH1啟動子甲基化和非甲基化的DNA引物[13],其中甲基化特異性引物序列上游:5′-TATATCGTTCGTAGTATTCGTGT-3′,下游:5′-TCCGACCCGAATAAACCCAA-3′,擴增產物153 bp;非甲基化特異性引物序列上游:5′-TTTTGATGTAGATGTTTTATTAGGGTTGT-3,下游:5′-ACCACCTCATCATAACTACCCACA-3′,擴增產物124 bp。反應體系25μl,其中10×PCR buffer 5.0μl,dNTP 0.2 mmol/L,MgCl22.0 mmol/L,TaqE(1 U/μl)2.0μl,亞硫酸氫鈉修飾的模板DNA 5μl,甲基化或非甲基化特異性上下游引物(100 pmol/μl)各0.5μl。循環參數:97℃預變性3 min,94℃變性45 s,44℃退火1 min,72℃延伸45 s,共35個循環,72℃繼續延伸5 min。用胎盤DNA經Sss-1酶催化,亞硫酸氫鈉處理后作為陽性對照PCR模板,陰性對照采用去離子水代替模板DNA進行PCR。擴增所得hMLH1甲基化和非甲基化產物,經6%非變性聚丙烯酰胺凝膠電泳進行分析。甲基化特異性引物擴增產物存在判定為甲基化陽性;甲基化特異性引物擴增產物不存在且非甲基化特異性引物擴增產物存在判定為甲基化陰性。
1.3 統計學方法 采用SPSS 11.5統計軟件進行統計學分析,正態分布的計量資料以(±s)表示,組間比較采用t檢驗;非正態分布的計量資料以〔M(P25,P75)〕表示,組間比較采用秩和檢驗;計數資料的分析采用χ2檢驗,采用Spearman等級相關分析hMLH1啟動子甲基化與hMLH1 mRNA、蛋白的相關性。以P<0.05為差異有統計學意義。
2 結果
2.1 外周血單個核細胞hMLH1 mRNA表達比較 病例組hMLH1 mRNA相對表達量為0.56(0.24,0.69),低于對照組的0.80(0.68,0.93),差異有統計學意義(Z=-3.98,P<0.05,見圖1)。
2.2 外周血單個核細胞hMLH1蛋白表達比較 病例組hMLH1蛋白相對表達量為0.70(0.62,0.76),低于對照組的0.80(0.68,0.94),差異有統計學意義(Z=-2.23,P<0.05,見圖2)。
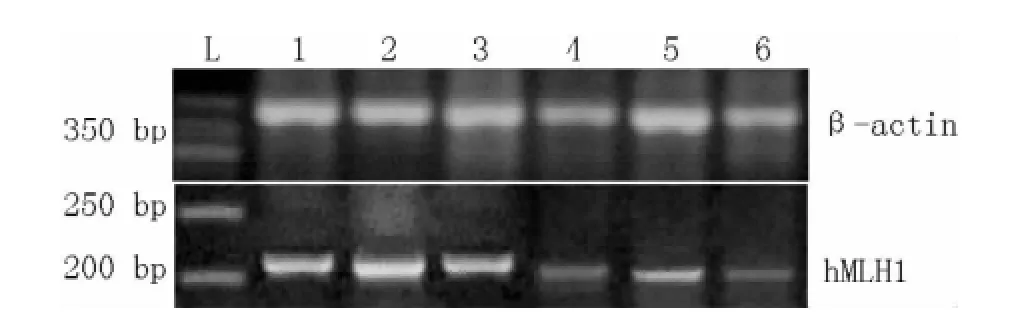
圖1 病例組和對照組hMLH1 mRNA凝膠譜帶Figure 1 hMLH1 mRNA gel bands from the case group and the control group
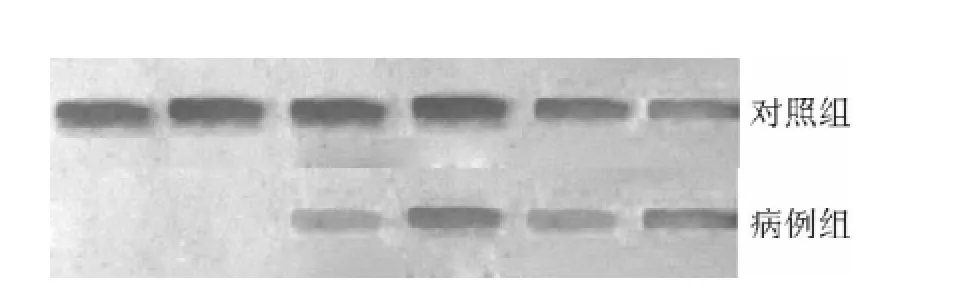
圖2 病例組和對照組hMLH1蛋白凝膠譜帶Figure 2 hMLH1 protein gel bands from the case group and the control group
2.3 hMLH1啟動子甲基化 病例組hMLH1啟動子甲基化陽性率為90.9%(30/33),高于對照組的13.2%(5/38),差異有統計學意義(χ2=42.72,P<0.05)。hMLH1啟動子甲基化程度與mRNA、蛋白相對表達量呈負相關(rs=-0.866、-0.541,P<0.01)。
2.4 病例組不同臨床特征患者hMLH1 mRNA、蛋白相對表達量及其啟動子甲基化陽性率比較 病例組不同性別、年齡、C反應蛋白(CRP)與紅細胞沉降率(ESR)水平患者hMLH1 mRNA、蛋白相對表達量及啟動子甲基化陽性率比較,差異均無統計學意義(P>0.05)。類風濕因子(RF)陽性患者hMLH1 mRNA、蛋白相對表達量低于陰性患者,差異有統計學意義(P<0.05);兩者hMLH1啟動子甲基化陽性率比較,差異無統計學意義(P>0.05,見表1)。
3 討論
證據顯示,類風濕性滑膜細胞慢性炎癥可能導致體細胞突變,使滑膜細胞變形形成攻擊性滑膜,侵略骨和軟骨,并從其上脫離[14]。而類風濕性滑膜細胞中存在的DNA損傷和突變[15],常伴有錯配修復酶變化及MSI現象[7-8]。
hMLH1位于染色體3p21-23,其基因組DNA全長約58 kb,含19個外顯子,cDNA有2 268 bp的開放閱讀框架,編碼蛋白含756個氨基酸[16]。hMLH1基因是錯配修復系統的重要組成成分,在錯配修復過程中具有識別錯配的重要功能,其功能缺陷使DNA復制時出現的錯配難以得到有效修復,常表現為MSI的發生[17-18]。

表1 病例組不同臨床特征患者hMLH1 mRNA、蛋白相對表達量及其啟動子甲基化陽性率比較Table 1 Comparison of hMLH1 mRNA,protein relative expression and promoter methylation positive rate among patients with different clinical features in the case group
本研究發現,RA患者外周血hMLH1啟動子甲基化陽性率為90.9%,明顯高于健康人群的13.2%。hMLH1啟動子的異常甲基化與hMLH1 mRNA及蛋白表達呈負相關,提示甲基化可能是導致hMLH1蛋白表達缺失的重要機制之一。當hMLH1啟動子發生異常甲基化時,在細胞傳遞信號的影響下轉錄激活復合物呈分散狀態,無法作用于DNA分子,導致轉錄失活,因此不能表達錯配修復蛋白,使其錯配修復基因功能喪失,最終導致RA的發生及發展。
hMLH1啟動子異常甲基化導致hMLH1蛋白表達減少或缺失,影響hMLH1錯配修復功能的發揮,可能導致單核苷酸多態位點的出現。已有研究顯示,RA患者存在MSI現象,hMLH1基因的功能缺陷使DNA復制時出現的錯配難以得到有效修復,常表現為MSI[7-8],而hMLH1表達的缺失與該基因啟動子胞嘧啶甲基化直接有關。推測hMLH1啟動子甲基化可能參與或影響RA的發生發展。本研究發現,RF陽性患者hMLH1 mRNA、蛋白表達均低于RF陰性患者,RF陽性與陰性患者間hMLH1基因啟動子甲基化陽性率無差異,提示hMLH1 mRNA、蛋白表達減少參與RA疾病過程,但未發現hMLH1啟動子甲基化與RA病情活動和嚴重程度存在相關性,hMLH1啟動子甲基化可能只是RA發病的早期事件,參與了RA疾病的發生。本研究樣本較少,也可能沒有足夠的證據證明hMLH1基因啟動子甲基化與RF的關系。
綜上所述,RA患者外周血hMLH1啟動子呈異常超甲基化狀態,其影響hMLH1轉錄和蛋白表達,可能參與了RA疾病的發生發展。
作者貢獻:甄艷鳳進行課題設計與實施、資料收集整理、撰寫論文、成文并對文章負責;付麗紅、李淑瑾、馬春玲、倪志宇、姚玉霞協助進行課題實施、評估、資料收集;劉興宇、周云濤、叢斌進行質量控制及審校。
本文無利益沖突。
[1]Wu ZB,Wang HK,Zheng CH,et al.Prevalence of rheumatoid arthritis in Chinese army force[J].Journal of the Fourth Military Medical University,2004,25(24):2190-2192.(in Chinese)吳振彪,王宏坤,鄭朝暉,等.我軍不同兵種在不同地區類風濕性關節炎患病的流行病學調查[J].第四軍醫大學學報,2004,25(24):2190-2192.
[2]Marinus MG.DNA mismatch repair[J].Ecosal Plus,2012,5(1).doi:10.1128/ecosalp lus.7.2.5.
[3]CoppedèF,Migheli F,Lopomo A,etal.Gene promotermethylation in colorectal cancer and healthy adjacent mucosa specimens:correlation with physiologicaland pathological characteristics,and with biomarkers of one-carbon metabolism[J].Epigenetics,2014,9(4):621-633.
[4]Sakata K,Tamura G,Endoh Y,et al.Hypermethylation of the hMLH1 gene promoter in solitary and multiple gastric cancers with microsatellite instability[J].Br J Cancer,2002,86(4):564-567.
[5]Woerner SM,Tosti E,Yuan YP,et al.Detection of coding microsatellite frameshiftmutations in DNA mismatch repair-deficient mouse intestinal tumors[J].Mol Carcinog,2015,54(11):1376-1386.
[6]Lee HJ,Jang YJ,Lee EJ,etal.The significance ofmismatch repair genes in gastric cancer[J].JCancer Res Ther,2013,9(1):80-83.
[7]Kullmann F,Widmann T,Kirner A,etal.Microsatellite analysis in rheumatoid arthritis synovial fibroblasts[J].Ann Rheum Dis,2000,59(5):386-389.
[8]Lee SH,Chang DK,Goel A,et al.Microsatellite instability and suppressed DNA repair enzyme expression in rheumatoid arthritis[J]. J Immunol,2003,170(4):2214-2220.
[9]Amett FC,Edworthy SM,Bloch DA,et al.The America rheumatoid association 1987 revised criteria for the classification of rhanmatoid arthdtis[J].Arthritis Rheum,1988,31(3):315-324.
[10]Deringer JR,Ely RJ,Monday SR,et al.Vbeta-dependent stimulation ofbovine and human T cells by hostspecific staphylococcal enterotoxins[J].Infect Immun,1997,65(10):4048-4054.
[11]Herman JG,Graff JR,Myohanen S,et al.Methylation-specific PCR:a novel PCR assay formethylation status of CpG islands[J]. Proc Natl Acad Sci USA,1996,93(18):9821-9826.
[12]Nagy G,Koncz A,Telarico T,et al.Central role of nitric oxide in the pathogenesis of rheumatoid arthritis and sysemic lupus erythematosus[J].Arthritis Res Ther,2010,12(3):210.
[13]Sakurai H,KohsakaM,Liu F,et al.Nitric oxide production and inducible nitricoxide synthase expression in inflammatory arthritides[J].JClin Invest,1995,96(5):2357.
[14]Mukai T,Gallant R,Ishida S,et al.SH3BP2 gain-of-function mutation exacerbates inflammation and bone loss in amurine collagen-induced arthritis model[J].PLoS One,2014,9(8):e105518.
[15]Viatte S,Plant D,Raychaudhuri S.Genetics and epigenetics of rheumatoid arthritis[J].Nat Rev Rheumatol,2013,9(3):141-153.
[16]Veigl ML,Kasturi L,Olechnowicz J,et al.Biallelic inactivation of hMLH1 by epigenetic gene silencing,a novel mechanism causing human MSI cancers[J].Proc Natl Acad Sci USA,1998,95(15):8698-8702.
[17]Kane MF,Loda M,Gaida GM,et al.Methylation of the hMLH1 promoter correlateswim lack of expression ofhMLH1 in sporadic colon tumors and mismatch repair defecfive human tumor cell lines[J]. Cancer Res,1997,57(5):808-811.
[18]Psofaki V,Kalogera C,Tzambouras N,et al.Promotermethylation status of hMLH1,MGMT,and CDKN2A/p16 in colorectal adenomas[J].World J Gastroenterol,2010,16(28):3553-3560.
hMLH1 Promoter Methylation From Peripheral Blood Mononuclear Cells of PatientsW ith New ly Diagnosed Rheumatoid Arthritis
ZHEN Yan-feng,LIU Xing-yu,ZHOU Yun-tao,et al.The Second Department of Endocrine,Tangshan Gongren Hospital,Tangshan 063000,China
ObjectiveTo explore promotermethylation and gene inactivation ofmismatch repair gene-hMLH1 in the genesis and developmentof rheumatoid arthritis(RA).MethodsA total of 33 RA patients diagnosed in the second and third hospital of HebeiMedical University from April2006 to April2007 were enrolled as the case group and at the corresponding period another 38 healthy persons from Hebei Province Blood Center were recruited as the control group.Peripheral blood mononuclear cells from subjects were separated by density gradient centrifugation.hMLH1 mRNA and protein relative expression of peripheral blood mononuclear cells were detected by RT-PCR and Western-blotting.DNA primers of promoter methylation and nonmethylation were designed respectively after sodium bisulfite modification.Promoter methylation of patients in two groups were detected.ResultshMLH1 mRNA relative expression of the case group was 0.56(0.24,0.69),lower than 0.80(0.68,0.93)of the control group(Z=-3.98,P<0.05).hMLH1 protein relative expression of case group was 0.70(0.62,0.76),lower than 0.80(0.68,0.94)of the control group(Z=-2.23,P<0.05).hMLH1 promotermethylation positive rate of the case group was 90.9%(30/33),higher than 13.2%(5/38)of the control group(χ2=42.72,P<0.05). hMLH1 promotermethylation was negatively correlated withmRNA and protein relative expression(rs=-0.866,-0.541,P<0.01).hMLH1mRNA and protein relative expression of patientswith positive rheumatoid factorswere lower than those of the patients with negative rheumatoid factors(P<0.05).The difference of hMLH1 promoter methylation positive rate between patients with positive and negative rheumatoid factors was not significant(P>0.05).ConclusionhMLH1 promoter methylation may be a crucial mechanism of hMLH1 gene inactivation.hMLH1 promoter abnormal hypermethylation may be involved in the occurrence and development of RA.
Rheumatoid,arthritis;DNA mismatch repair;hMLH1;DNA methylation
R 593.22
A
10.3969/j.issn.1007-9572.2016.07.015
2015-09-21;
2015-12-29)
(本文編輯:吳立波)
河北省自然科學基金資助項目(H2014105040)
063000河北省唐山市,唐山市工人醫院內分泌二科(甄艷鳳),神經外科(劉興宇),中心實驗室(周云濤);河北醫科大學法醫學系(叢斌,付麗紅,李淑瑾,馬春玲,倪志宇,姚玉霞)
叢斌,050017河北省石家莊市,河北醫科大學法醫學系;E-mail:jane791115@126.com

