Genbank中陸地棉表達序列標簽(EST)與基因組序列(GSS)的SNP特征
胡根海, 董 娜
(河南科技學院, 現代生物育種河南省協同創新中心, 河南 新鄉 453003)
Genbank中陸地棉表達序列標簽(EST)與基因組序列(GSS)的SNP特征
胡根海, 董 娜
(河南科技學院, 現代生物育種河南省協同創新中心, 河南 新鄉 453003)
陸地棉基因組精細定位需要更加豐富的分子標記,為了闡明單核苷酸多態性(SNP)標記資源開發利用的前景,對GenBank數據庫中陸地棉表達序列標簽(EST)和基因組序列(GSS)進行分析。下載GenBank數據庫中公布的陸地棉EST序列及GSS序列,利用DNAStar軟件進行疊連群構建及其候選SNP位點分析。結果表明:陸地棉EST序列307 414條及GSS序列242 015條,EST序列構建3 737個疊連群,序列累計10 477 241 bp。由4條及以上序列組成的疊連群累計序列長度為3 761 800 bp,發現候選SNP位點1 007 258個,疊連群平均出現1個SNP位點最低頻率為2.32%。GSS序列共構建1 517個疊連群,序列累計1 625 700 bp,發現SNP位點574 296個,疊連群平均出現1個SNP位點最低頻率為9.18%。陸地棉的EST和GSS均有頻率較高SNP位點,GSS出現SNP頻率高于EST序列,開發SNP引物3 254對。
陸地棉; 疊連群; SNP頻率; GenBank
我國現有的陸地棉品種來自47個基礎種質,品種間親緣關系近,分子標記多態性較低。據最新報道[1],陸地棉高密度連鎖圖譜共整合2 292個標記位點,覆蓋基因組5 115.16 cM,平均2.23 cM有1個標記,該標記數量和密度尚不能滿足陸地棉基因組精細定位的需要,高密度精細定位是進一步解析基因功能的基礎,棉花重要農藝性狀的基因組精細定位需要加大現有圖譜密度。新型分子標記SNP(Single Nucleotide Polymorphism)的出現,為進一步加密陸地棉圖譜提供可能。SNP是廣泛存在于植物基因組中的一類DNA高頻序列變異,是標記密度最高的一種遺傳多樣性標記,SNP比SSR(simple sequence repeats)標記具有更高的穩定性[2],在群體中符合孟德爾遺傳定律,可用于遺傳關聯分析[3],還可用于材料的基因分型[4-5]。SNP通常用DNA芯片自動化檢測[6-7],也可用簡化的EcoTILLING 技術[8]和普通PCR方法實現檢測[4,9]。目前,SNP已成為分子標記輔助育種的一種新型工具。但諸多作物SNP開發剛起步僅在荔枝[10]、花生[11]、菊花[12]、葡萄[13]、栽培番茄[3]和大白菜[14]等作物中進行開發。在陸地棉上利用數據庫開發EST(expressed sequence tag)-SSR標記已有報道[15],但開發SNP標記尚未見報道。已有記載顯示陸地棉SNP具有良好分型效果[16],筆者擬利用NCBI的GenBank數據庫中公布的陸地棉GSS(genomic survey sequence)和 EST序列數據,使用生物信息學軟件分析陸地棉SNP標記的潛在存在數量,對陸地棉SNP進行開發,以期為陸地棉基因組精細定位提供理論基礎。
1 材料與方法
1.1 數據材料
陸地棉EST和GSS序列下載于NCBI(美國國家生物信息中心)的GenBank數據庫,下載序列以FASTA格式保存,下載時間為2015年5月5號。
1.2 序列拼接形成疊連群
下載序列解壓后,采用EST-trimmer和cross-match程序清理下載序列中的短序列和載體序列,屏蔽低復雜度序列,處理后序列用DNAStar軟件包的子程序SeqMan進行疊連群分析。
1.3 SNP位點分析及引物開發
使用SeqMan軟件找出候選SNP,SNP出現頻率=候選SNP的數目/疊連群長度×100%;根據候選SNP位點序列,使用 Primer Premier 5.0設計陸地棉SNP引物,基本檢索條件:疊連群長度≥500 bp,組成序列條數≥10;篩選引物標準:長度18~24 bp,GC含量40%~60%,退火溫度(Tm) 40~60℃,不產生引物二聚體、發夾結構和引物錯配,引物中無 6個連續堿基出現,PCR擴增產物長度為200~500 bp。
2 結果與分析
2.1 陸地棉EST與GSS序列的疊連群構成及SNP出現頻率
在GenBank數據庫中總計下載307 414條陸地棉EST序列,通過剔除短序列和載體,屏蔽低復雜度序列,獲得有用序列255 002條,疊連群構建后,有9 002條序列未組裝成疊連群,序列組裝共使用246 000條序列,構建3 737個疊連群。疊連群最長達3 912 bp,其SNP候選位點 2 972個,出現SNP頻率75.97%,最短1個疊連群長度僅 142 bp,無候選SNP位點存在。疊連群累計長度3 761 800 bp,鑒定候選SNP位點1 007 258個,SNP位點出現的平均頻率為26.77%。從疊連群的長度與數量組成看,較長(≥2000 bp)53條,較短(≤499 bp)90條,疊連群長度主要集中在500~1 500 bp,約占86.76%,其SNP出現頻率隨疊連群長度增加也在增加,但陸地棉的SNP位點分布在EST中不均勻。
最長疊連群長度為10 498 bp,有7 345個SNP候選位點,疊連群SNP出現頻率69.97%;最短1個疊連群長度僅220 bp,無候選SNP位點存在。由表1可知,不同長度疊連群的數目表現很大差異,疊連群越長其數目越少,統計疊連群累計長度3 761 800 bp,鑒定出候選SNP位點574 296個,SNP平均出現頻率為44.57%。從疊連群的長度與數量看,疊連群長度主要集中在500~2 000 bp,約占91.67%,其SNP出現頻率隨疊連群長度增加而增加,但分析陸地棉的SNP位點在GSS疊連群中分布并不均勻。
2.2 陸地棉 EST 與 GSS序列疊連群組成條數及SNP 出現頻率
對3 737條疊連群進行構成疊連群序列條數分析顯示,僅有2條序列構成的疊連群高達1 801個,由3條序列構成疊連群有752條,這些疊連群因組成序列低于4條,故未檢測到SNP。由表2可見,4~6條序列構成的疊連群,SNP出現頻率為2.32%,隨著組成疊連群的序列條數增加疊連群中出現SNP的頻率也在增加,最高疊連群是2個由112條、119條序列構成疊連群,其出現SNP頻率均值為46.36%。
表1 陸地棉EST與GSS序列的疊連群構成與SNP出現頻率
Table 1 Contigs constitute and SNP frequency of EST and GSS sequences fromG.hirsutum
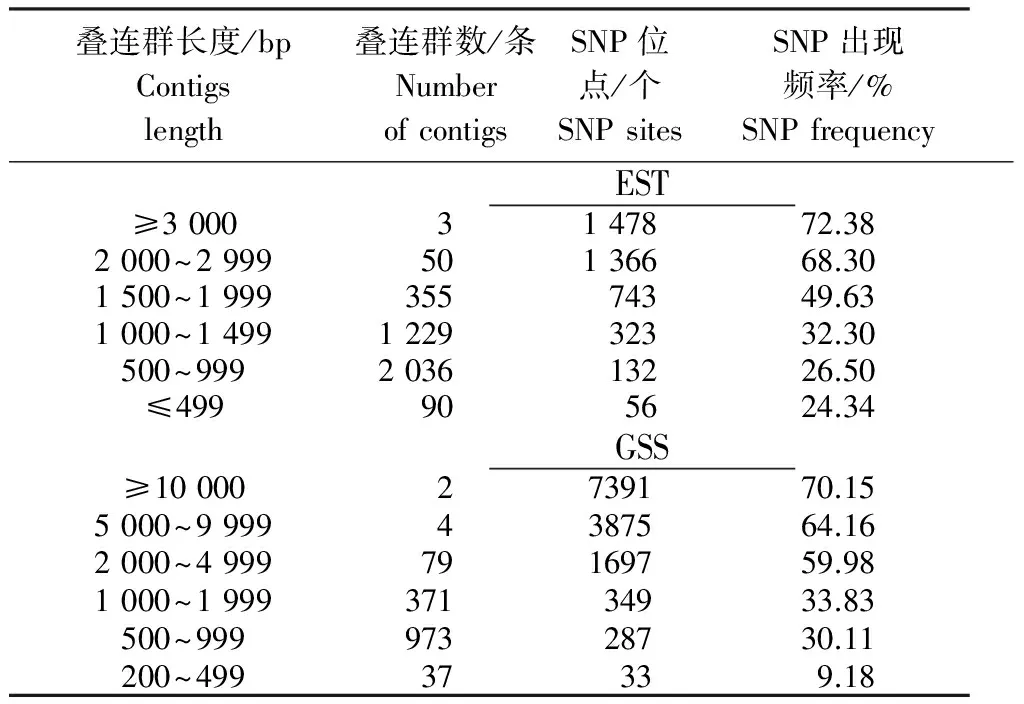
疊連群長度/bpContigslength疊連群數/條NumberofcontigsSNP位點/個SNPsitesSNP出現頻率/%SNPfrequencyEST≥30003147872.382000~299950136668.301500~199935574349.631000~1499122932332.30500~999203613226.50≤499905624.34GSS≥100002739170.155000~99994387564.162000~499979169759.981000~199937134933.83500~99997328730.11200~49937339.18
表2 陸地棉 EST序列組成疊連群的序列條數及候選SNP 出現頻率
Table 2 Number of sequences of contigs and candidate SNP frequency of EST sequences fromG.hirsutum
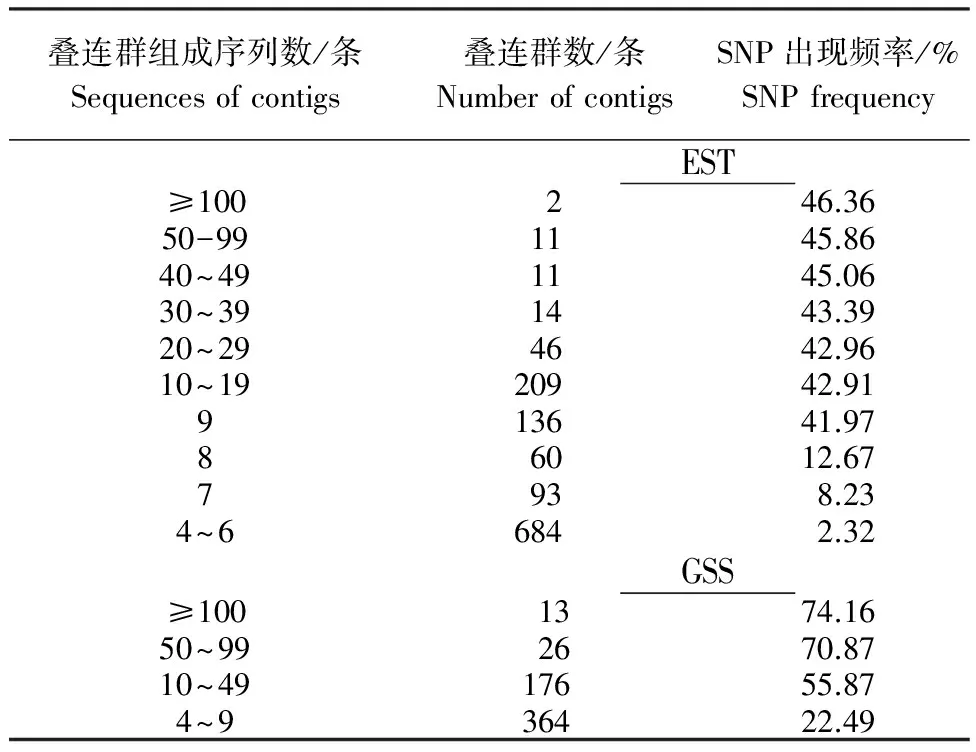
疊連群組成序列數/條Sequencesofcontigs疊連群數/條NumberofcontigsSNP出現頻率/%SNPfrequencyEST≥100246.3650-991145.8640~491145.0630~391443.3920~294642.9610~1920942.91913641.9786012.677938.234~66842.32GSS≥1001374.1650~992670.8710~4917655.874~936422.49
在GenBank數據庫中總計下載242 015條陸地棉GSS序列,通過剔除短序列和載體,屏蔽低復雜度序列,最后獲得有用序列24 144條,其中11 393條序列未組裝成疊連群,序列組裝共使用11 234條序列,構建1 517個疊連群。組成疊連群序列最多的有656條序列。由表2可知,在4條以上序列組成疊連群中,主要有4~49條序列組成,隨著組成疊連群的序列數目增加,其SNP位點數和SNP出現頻率也在增加。但在計算統計分析時發現,隨著組成疊連群序列的減少,出現SNP位點數極少或無位點的頻率也在增加。
2.3 陸地棉SNP 引物的開發
疊連群組成序列≥10條的共有508個疊連群,依據SNP位點兩側序列和引物篩選的基本原則,保守設計即可得到3 254對引物,表明陸地棉的SNP可設計的引物數量非常多,該標記在育種利用中有較大挖掘潛力。
3 結論與討論
單核苷酸多態性在植物基因組中廣泛存在,較高的多態性使材料的遺傳多樣性變得容易區分,也讓生產上種子純度鑒定變得簡單[14],SNP是單核苷酸變異,可能發生在基因的內含子區、表達調控區和基因功能區,因此部分SNP位點可能和基因的功能直接相關。EST是測序獲得的短cDNA 序列,是完整基因的一部分。因此,基于EST的SNP更有可能與功能基因及農藝性狀相關聯。
本研究中分析307 414條陸地棉EST序列,平均每43.1 bp EST序列中出現1個SNP位點,SNP位點出現頻率約為2.32%,比菊花[12]EST的SNP平均出現頻率(0.15%)略高,但遠遠低于對吳茱萸SOD基因分析得到的SNP 位點出現頻率(20%)[17]。這種結果的差異可能因為目前菊花EST數據偏少,很多SNP位點未被發現;陸地棉EST發現SNP位點少于吳茱萸的SOD基因,可能因EST一般為短cDNA 序列,僅完整基因的一小部分,而吳茱萸SOD基因分析是完整基因。從陸地棉GSS數據中獲得574 296個候選SNP位點,SNP的出現頻率最低為9.18%,平均每10.89 bp出現1個候選SNP位點。已有結果顯示,在水稻基因組序列中,平均每89 bp出現1個SNP位點[18];在花生栽培種基因組DNA的測序數據中,平均每27.86 bp出現1個SNP位點[11];玉米基因組DNA中平均每61 bp序列有1個SNP[19];試驗數據顯示陸地棉GSS序列的SNP出現頻率高于水稻、花生和玉米,比較4個物種可見物種間差別較大,這可能是物種不同引起。比較陸地棉EST和GSS構建的各個疊連群中含有的SNP數量,可見構成疊連群的序列條數越多,出現的SNP位點數量也越多,與Duran C等[20]在大麥EST序列的研究結果基本一致。EST序列和GSS序列出現SNP的頻率不均勻,與前人在菊花[12]上研究結果一致。
比較由EST和GSS獲得結果可知,利用GSS序列檢測到其有更多的SNP位點和更高出現頻率,這可能是因為GSS序列是基因組序列,含有非編碼區,這些區域的突變不會直接導致植物體變異,具有更低選擇壓和更高突變頻率。陸地棉基因組序列候選SNP出現頻率較高還可能與其為異源四倍體起源有關,這與唐月異[11]研究作物染色體倍數越大SNP頻率越高一致。本研究首次對陸地棉的GSS和EST序列進行了SNP標記出現頻率及可能數量進行分析并開發了3 254對SNP檢測引物, SNP變異比SSR豐富,是更有前景的新一代標記。
[1] Yuzhen Shi,Wentan Li,Aiguo Li,et al.Constructing a high density linkage map for Gossypium hirsutum×Gossypium barbadense and identifying QTLs for lint percentage[J].Journal of Integrative Plant Biology,2015,57(5):450-467.
[2] 李 雪,田紅麗,王鳳格,等.SSR和SNP兩種標記技術在玉米品種真實性鑒定中的比較分析[J].分子植物育種,2014,12(5):1000-1004.
[3] 郝轉芳,蘇治軍,李 亮,等.基于SNP標記的關聯分析在玉米耐旱研究中的應用[J].作物雜志,2009(6):1-7.
[4] 李亞玲,李景富,康立功,等.番茄Mi-1基因的SNP分型[J].東北農業大學學報,2010,41(10):36-42.
[5] 姚遠颋,謝青軒,杜培粉,等.利用SNP分型技術鑒別油菜FLC1與FLC3基因的AC組基因型[J].分子植物育種,2009,7(2):330-334.
[6] Olivier M.SNP genotyping using invader technology[J].Mutation Res,2005,573(1/2):103-110.
[7] 曹廷杰,謝菁忠,吳秋紅,等.河南省近年審定小麥品種基于系譜和SNP標記的遺傳多樣性分析[J].作物學報,2015,41(2):197-206.
[8] 滕 斌,李澤福,羅志祥,等.利用EcoTILLING簡化技術進行水稻基因型鑒定及單核苷酸多態性(SNP)檢測[J].農業生物技術學報,2012,20(3):254-260.
[9] Michaels S D,Amasino R M.A robust method for detecting single-nucleotide changes as polymorphic markers by PCR[J].Plant J.,1998,14(3):381-385.
[10] 孫清明,李永忠,向 旭,等.利用SNP和EST-SSR分子標記鑒定荔枝新種質御金球[J].分子植物育種,2013,11(3):403-414.
[11] 唐月異,張建成,王秀貞,等.GenBank中花生栽培種基因組DNA及EST序列的SNP分析[J].花生學報,2010,39(2):21-23.
[12] 萬志兵,王言言,陳 黎.菊花EST-SNP的發掘與特征分析[J].東北林業大學學報,2013,41(5):84-86,90.
[13] 李 猛,郭大龍,劉崇懷,等.葡萄EST-SNP位點的信息與特征[J].浙江大學學報:農業與生命科學版,2012,38(3):263-270.
[14] 趙 新,王 永,蘭青闊,等.基于復合EST-SSR標記的大白菜種子純度鑒定及SNP位點獲取[J].中國蔬菜,2013(14):31-38.
[15] 李華盛,范術麗,沈法富.從棉花ESTs數據庫中篩選微衛星標記的初步研究[J].棉花學報,2005,17(4):211-216.
[16] 鄭煒佳,曲延英,謝元元,等.ARMS-PCR對棉花SNP分型的研究[J].新疆農業科學,2013,50(12):2182-2188.
[17] 吳 波,高 丹,潘超美,等.吳茱萸SOD基因片段克隆和SNP分析[J].江西農業大學學報,2011,33(6):1206-1211.
[18] Nasu S,Suzuki J, Ohta R,et a1.Search for and analysis of single nucleotide polymorphisms(SNPs) in rice(Oryza sativa,Oryza rufipogon)and establishment of SNP markers[J].DNA Res.,2002,9:163-171.
[19] Ching A,Caldwell K S,Jung M,et a1.SNP frequency,haplotype structure and linkage disequilibrium in elite maize inbred lines[J].BMC Genet,2002,3:19.[20] Duran C,Appleby N,Vardy M,et a1.Single nucleotide polymorphism discovery in badey using auto SNPdb[J].Plant Biotechnology Journal,2009,7:326-333.
(責任編輯: 劉忠麗)
SNP Discovery in GSS and ESTs ofGossypiumhirsutumfrom GenBank
HU Genhai, DONG Na
(HenanCollaborativeInnovationCenterofModernBiologicalBreeding,HenanInstituteofScienceandTechnology,Xinxiang,Henan453003,China)
In order to explore the SNP marker resources development prospects, which can provide abundant number of molecular markers for cotton genome fine positioning. 242 015 GSS sequences and 307 414 ESTs ofG.hirsutumdownloaded from GenBank in NCB1 were analyzed. 3 737 contigs were assembled from the EST sequences by DNAStar, and the length of the total contigs was 1 047 7241 bp. The length of the tota1 contigs with≥4 ESTs was 3 761 800 bp, from which 1 007 258 candidate SNPs were identified directly, and the average frequency of SNPs was 2.32%. 1 517 contigs were assembled from the GSS, and the length of the total contigs was 1 625 700 bp. From which 574 296 candidate SNPs were identified directly, and the average frequency of SNPs was 9.18%. Conclusion: The upland cotton EST and GSS all had higher frequency of SNP loci, SNP frequency of GSS was higher than that of ESTs, it was more valuable that SNP loci of GSS was utilized. 3 254 pairs of SNP primers were designed by Primer Premier 5.0 following the basic criteria.
Gossypiumhirsutum; contigs; SNP frequency; GenBank
2015-08-28; 2016-03-24修回
國家自然科學基金項目“棉花早熟不早衰基因型的根系形態和生理機制研究”(31271648);河南省科技攻關計劃項目“轉基因耐鹽堿陸地棉新材料創制”(142102110126)
胡根海(1966-),男,副教授,博士,從事棉花分子育種研究。E-mail: hgh1013@126.com
1001-3601(2016)04-0150-0021-03
S562; Q946.2
A

