山羊MSH4和MSH5基因的cDNA克隆、序列分析及組織表達
鄭杰 劉霜 羅斌 胡亮 楊珂?zhèn)?字向東
(西南民族大學國家民委動物科學重點實驗室,成都 610041)
山羊MSH4和MSH5基因的cDNA克隆、序列分析及組織表達
鄭杰 劉霜 羅斌 胡亮 楊珂?zhèn)?字向東
(西南民族大學國家民委動物科學重點實驗室,成都 610041)
以低繁藏山羊和高繁金堂黑山羊為研究對象,分別提取處于發(fā)情期的5只藏山羊和5只金堂黑山羊的卵巢、子宮、輸卵管、垂體的總RNA,并通過RT-PCR技術(shù)對MSH4、MSH5基因cDNA進行克隆、序列分析,以Real-time PCR技術(shù)對其進行組織表達研究。結(jié)果表明,藏山羊和金堂黑山羊MSH4基因編碼區(qū)均長2 499 bp,編碼832個氨基酸,兩品種基因編碼區(qū)有5處堿基不同,并導致3處氨基酸的差異;MSH5基因編碼區(qū)均長2 496 bp,編碼831個氨基酸,兩品種基因編碼區(qū)有9處堿基不同,并導致5處氨基酸的差異。藏山羊MSH4基因編碼區(qū)核苷酸序列與金堂黑山羊、山羊、綿羊、牛、馬、小鼠、褐家鼠、人的同源性分別為:99.8%、99.8%、99.4%、98.1%、94.4%、85.1%、84.7%和93.5%;藏山羊MSH5基因編碼區(qū)核苷酸序列與金堂黑山羊、山羊、牛、家犬、小鼠、褐家鼠、人的同源性分別為:99.6%、99.6%、97.3%、88.0%、85.8%、85.3%和90.2%。MSH4和MSH5基因mRNA在兩個山羊品種的卵巢、子宮、輸卵管、垂體中均有表達,但兩品種間無顯著性差異(P>0.05)。說明MSH4和MSH5基因在動物進化中比較保守,與山羊多羔性狀的相關(guān)性有待進一步研究。
藏山羊;金堂黑山羊;Msh4;Msh5;克隆;定量PCR
藏山羊主要分布在青藏高原牧區(qū),其繁殖性能較低,多數(shù)一年產(chǎn)一羔[1]。金堂黑山羊產(chǎn)于四川省成都市金堂縣,是經(jīng)過長期自然選擇和人工重點選育的多胎地方山羊品種,其繁殖性能高、產(chǎn)羔率高(一胎2-3羔)[2]。山羊的產(chǎn)羔率是一個經(jīng)濟價值巨大的數(shù)量性狀,其遺傳力低,只有0.05-0.15[3],表明采用傳統(tǒng)的育種方法難以在較短時間內(nèi)提高山羊的繁殖力。國內(nèi)外研究者從生殖激素基因、綿羊多胎基因等方面開展了山羊多胎機制研究,但目前對山羊排卵數(shù)性狀形成的生理學和遺傳學機理還不清楚[4,5]。減數(shù)分裂是一種高度保守的有性生殖細胞分裂方式,是生殖細胞成熟的重要階段,為染色體數(shù)目的恒定以及生物體的變異奠定了物質(zhì)基礎(chǔ)。減數(shù)分裂的完成需要依賴一系列重要復雜的過程,任何一個階段出現(xiàn)問題都會導致生殖細胞異常,使機體出現(xiàn)生殖障礙。若人的同源染色體在減數(shù)分裂時未分離,會導致不孕和先天缺陷(如唐氏綜合癥)[6]。錯配修復蛋白除了參與DNA的錯配修復、細胞周期的調(diào)控外,還可以影響生殖細胞減數(shù)分裂過程中DNA重組的忠實性和效率[7],其缺陷可導致哺乳動物的不孕。對于減數(shù)分裂和錯配修復蛋白之間關(guān)系的研究是從MSH4、MSH5基因的研究開始的,現(xiàn)已確定參與減數(shù)分裂的錯配修復蛋白包括:MLH1、MLH3、MSH4、MSH5和PMS2[8]。MSH4和MSH5是MutS同源蛋白DNA錯配修復蛋白家族中的2個主要成員,二者可以形成異源二聚體,形成的異源二聚體在雙鏈DNA分子上形成“夾子”型結(jié)構(gòu),稱之為Holliday連接體,參與減數(shù)分裂過程中的重組過程,并在減數(shù)分裂Ⅰ期促進染色體互換,可影響減數(shù)分裂中交換位點的數(shù)量與分布[9-14]。MSH5蛋白是雄性和雌性小鼠減數(shù)分裂過程中染色體配對和聯(lián)會所必需的,參與DNA損傷應答以及在免疫球蛋白多樣性的形成中起重要作用,具有識別并特異性結(jié)合錯配堿基的特殊功能[15-18]。研究發(fā)現(xiàn),Msh4和/或Msh5基因被敲除后會造成小鼠減數(shù)分裂Ⅰ前期染色體聯(lián)會失敗,使得小鼠卵巢功能退化,最終導致其不育[18-22]。現(xiàn)階段,根據(jù)Shenker等[23]和Kazma等[24]研究團隊進行全基因組關(guān)聯(lián)研究,結(jié)果均發(fā)現(xiàn),MSH5可能是肺癌的一個重要相關(guān)基因。
目前,有關(guān)MSH4和MSH5基因在人、小鼠、酵母等物種的研究中均有報道,但在對山羊中的研究尚未見報道。本研究通過對單胎山羊品種藏山羊與多胎山羊品種金堂黑山羊MSH4和MSH5基因的cDNA克隆、序列分析、組織表達差異分析,探索MSH4和MSH5基因在山羊卵母細胞減數(shù)分裂中可能發(fā)揮的作用,以期為研究山羊多胎機制提供理論依據(jù)。
1 材料與方法
1.1 材料
1.1.1 實驗動物及樣品采集 分別選取處于同一發(fā)情周期的健康藏山羊、金堂黑山羊各5只作為研究對象。藏山羊選自四川省理縣,連續(xù)3胎產(chǎn)單羔的母羊;金堂黑山羊選自四川省金堂縣,連續(xù)3胎產(chǎn)3羔的母羊。對選取的10只母羊進行同期發(fā)情處理:將孕酮陰道栓(CIDR)放入山羊陰道內(nèi),經(jīng)11 d后取出,并在取出CIDR前1天肌肉注射氯前列醇鈉(Merck Animal Health,Canada)2 mL,在取出CIDR后40 h左右將山羊進行屠宰,屠宰后立即采集垂體、輸卵管、卵巢、子宮等組織放入凍存管中,并做好標記投入到液氮罐中保存?zhèn)溆谩?/p>
1.1.2 主要試劑 反轉(zhuǎn)錄試劑盒購自Fermentas(MBI)公司;DNA Marker DL2000、2×Taq PCR MasterMix、感受態(tài)細胞均購自上海天根生物科技有限公司;DNA凝膠回收試劑盒購自愛思進生物技術(shù)(杭州)有限公司;氨芐青霉素、克隆載體pMD19-T Vector 購自大連寶生物工程有限公司;E.coli DH5α感受態(tài)細胞購自康迪生物技術(shù)有限公司;SsoAdvancedTMSYBR?Green Supermix、八連管、蓋子均購自伯樂公司;引物合成與測序由成都擎科梓熙生物技術(shù)有限公司完成。
1.2 方法
1.2.1 總RNA的提取及cDNA的合成 在同一條件下,采用Trizol法分別提取藏山羊、金堂黑山羊4種組織(垂體、輸卵管、卵巢、子宮)的總RNA。使用反轉(zhuǎn)錄試劑盒Thermo Scientific RevertAid First Strand cDNA Synthesis Kit進行cDNA的合成。
1.2.2 引物設(shè)計與PCR擴增目的片段 根據(jù)NCBI上已發(fā)表的MSH4、MSH5的基因序列,利用引物設(shè)計軟件Primer Premier 5設(shè)計克隆引物,引物序列見表1。以合成的卵巢cDNA第一鏈為模板,擴增MSH4、MSH5基因,PCR擴增程序:95℃預變性4 min;95℃變性45 s,MSH4、MSH5退火溫度分別為55℃/40 s、53.2℃/40 s,72℃延伸120 s,30個循環(huán);最后72℃延伸5 min;4℃保存。
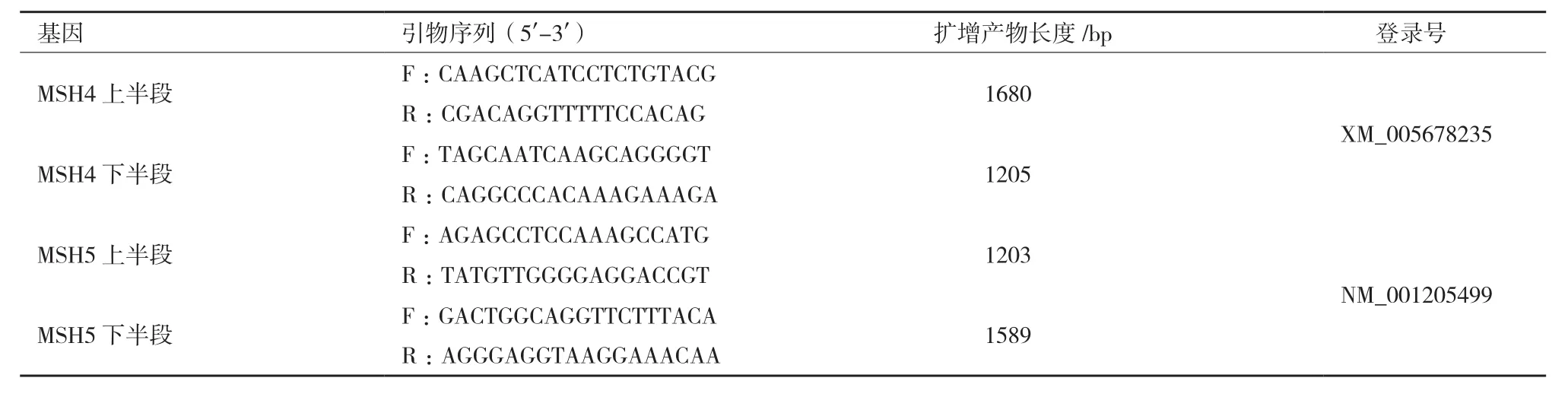
表1 克隆引物序列
1.2.3 目的基因的克隆與測序 用1%瓊脂糖凝膠電泳對PCR產(chǎn)物進行檢測,并用愛思進DNA凝膠回收試劑盒對其進行回收純化。將回收產(chǎn)物與pMD19-T Vector于16℃連接過夜,再將連接產(chǎn)物轉(zhuǎn)化宿主菌E.coli DH5α感受態(tài)細胞,并涂布于含有氨芐青霉素的LB固體培養(yǎng)基上,37℃恒溫培養(yǎng)12 h。挑選白色單克隆菌落于含有芐青霉素的LB液體培養(yǎng)基中,37℃震蕩培養(yǎng)12 h,再對菌液做PCR鑒定,產(chǎn)物經(jīng)1%瓊脂糖凝膠電泳檢測后,挑選藏山羊、金堂黑山羊的陽性克隆菌液各2 mL送至成都擎科梓熙生物技術(shù)有限公司測序。
1.2.4 山羊MSH4、MSH5基因的生物信息學分析 通過DNAman、DNAstar等生物分析軟件對基因序列進行同源性比對,利用生物軟件MEGA 5.1構(gòu)建核苷酸系統(tǒng)進化樹。
1.2.5 實時熒光定量PCR 根據(jù)已克隆出的藏山羊和金堂黑山羊MSH4、MSH5的序列設(shè)計基因的定量引物,引物序列及擴增長度見表2。
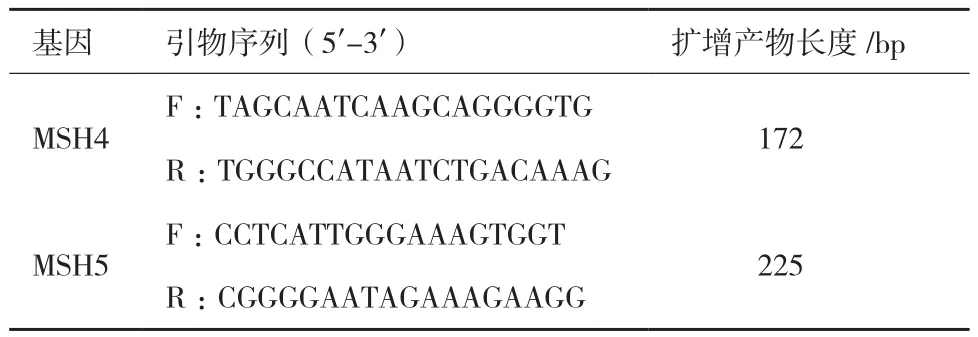
表2 定量引物序列
對藏山羊、金堂黑山羊的垂體,輸卵管,卵巢,子宮中的MSH4、MSH5基因及內(nèi)參β-actin基因mRNA表達量進行實時熒光定量PCR檢測。反應體系為10 μL:上下游引物各0.8 μL,cDNA模板0.5 μL,ddH2O 2.9 μL,SsoAdvancedTMSYBR?Green Supermix 5 μL。反應條件為:95℃預變性3 min;95℃變性10 s,MSH4、MSH5退火溫度分別為61℃/20 s、60℃/20 s,65℃延伸5 s,40個循環(huán);65℃延伸5 min;4℃保存。每個樣品設(shè)置3個重復,并設(shè)置陰性對照。實驗結(jié)果采用Pfaffl法[25]分析MSH4、MSH5基因分別在兩種山羊不同組織中的相對表達量,計算公式為:
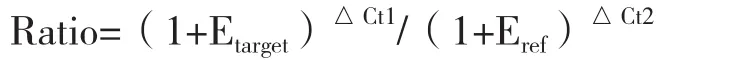
其中,Etarget表示目的基因的擴增效率;Eref表示內(nèi)參基因的擴增效率;△Ct1表示對照樣本中目的基因的CT值與實驗樣本中目的基因CT值之差,△Ct2表示對照樣本中內(nèi)參基因的CT值與實驗樣本中內(nèi)參基因的CT值之差。
2 結(jié)果
2.1 藏山羊和金堂黑山羊MSH4、MSH5基因PCR擴增結(jié)果
吸取藏山羊和金堂黑山羊MSH4、MSH5基因PCR擴增產(chǎn)物各5 μL,進行電泳檢測。經(jīng)1%瓊脂糖凝膠電泳檢測條帶后,結(jié)果(圖1和圖2)顯示,其中MSH4上半段、下半段和MSH5上半段、下半段條帶與預期大小基本一致,且目的條帶明亮清晰,可用于后續(xù)實驗。
2.2 藏山羊和金堂黑山羊MSH4、MSH5基因核苷酸序列比對
獲得的藏山羊、金堂黑山羊的MSH4和MSH5基因測序已提交到NCBI中(MSH4登錄號分別為KR864751和KR864752,MSH5登錄號分別為KR864753和KR864754)。藏山羊和金堂黑山羊MSH4基因編碼區(qū)均長2 499 bp,編碼832個氨基酸,兩品種基因編碼區(qū)有5處堿基不同,并導致了3處氨基酸的差異。藏山羊和金堂黑山羊MSH5基因編碼區(qū)均長2 496 bp,編碼831個氨基酸,兩品種基因編碼區(qū)有9處堿基不同,并導致了5處氨基酸的差異(表3)。
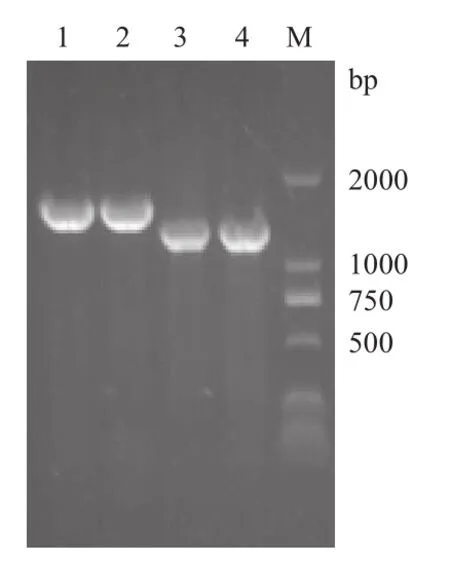
圖1 MSH4基因PCR產(chǎn)物電泳檢測
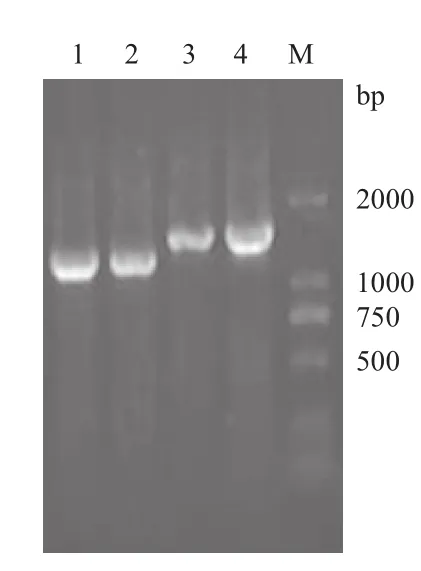
圖2 MSH5基因PCR產(chǎn)物電泳檢測

表3 藏山羊和金堂黑山羊MSH4、MSH5基因序列差異
2.3 MSH4、MSH5基因編碼區(qū)核苷酸序列同源性比對
通過DNAstar分析藏山羊和金堂黑山羊與其他物種MSH4、MSH5基因編碼區(qū)核苷酸序列同源性,結(jié)果顯示,藏山羊MSH4基因編碼區(qū)核苷酸序列與金堂黑山羊、山羊(XM_005678235)、綿羊(XM_004002088)、牛(NM_001206255)、馬(XM_ 001918273)、小鼠(AF178957)、褐家鼠(NM_001-106477)和人(HSU89293)的同源性分別為:99.8%、99.8%、99.4%、98.1%、94.4%、85.1%、84.7%和93.5%;藏山羊MSH5基因編碼區(qū)核苷酸序列與金堂黑山羊、山羊(XM_005696572)、牛(NM_ 001205-499)、家犬(XM_005627746)、小鼠(AF-107352)、褐家鼠(NM_212536)和人(AF070071)的同源性分別為:99.6%、99.6%、97.3%、88.0%、85.8%、85.3%和90.2%。
2.4 MSH4、MSH5基因系統(tǒng)發(fā)生樹的構(gòu)建
利用Clustal X、MAGA5.1對相應物種的MSH4、MSH5基因編碼區(qū)進行多序列對比,并構(gòu)建基因Neighbor-Joining(NJ)系統(tǒng)發(fā)生樹(圖3和圖4)。圖3顯示,藏山羊首先與金堂黑山羊、山羊聚為一類,再依次與綿羊、牛、馬、人聚為一類,最后與小鼠、褐家鼠聚為一類。圖4顯示,藏山羊首先與山羊聚為一類,再依次與金堂黑山羊、牛、家犬、人聚為一類,最后與小鼠、褐家鼠聚為一類。系統(tǒng)發(fā)生樹的構(gòu)建結(jié)果與核苷酸同源性比對結(jié)果一致,與哺乳動物進化程度相符合。
2.5 目的基因組織表達差異分析
本實驗通過實時熒光定量PCR技術(shù),以β-actin基因為參照,檢測MSH4、MSH5基因在單胎品種藏山羊和多胎品種金堂黑山羊不同組織間mRNA表達量,結(jié)果表明:MSH4和MSH5的mRNA在山羊卵巢、子宮、輸卵管、垂體中均有表達,但兩個品種間mRNA表達量無顯著性差異(P>0.05,圖5和圖6)。
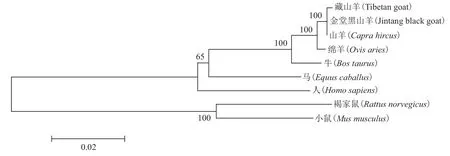
圖3 MSH4基因的核苷酸系統(tǒng)發(fā)生樹
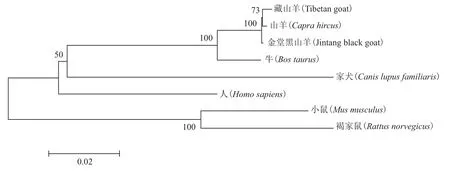
圖4 MSH5基因的核苷酸系統(tǒng)發(fā)生樹
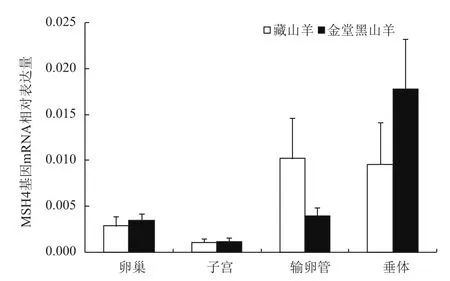
圖5 MSH4基因在藏山羊和金堂黑山羊不同組織中的相對表達量(±s)
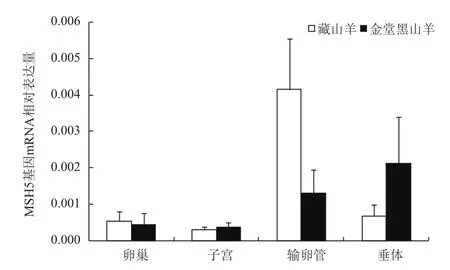
圖6 MSH5基因在藏山羊和金堂黑山羊不同組織中的相對表達量(±s)
3 討論
山羊的產(chǎn)羔率是一個具有巨大經(jīng)濟價值的數(shù)量性狀,提高山羊的產(chǎn)羔率已成為提高養(yǎng)羊業(yè)經(jīng)濟效益的重要途徑。山羊的產(chǎn)羔率主要取決于一個發(fā)情周期的排卵數(shù),所以研究者對山羊開展了大量有關(guān)生殖激素基因、綿羊多胎基因等方面的研究,以期揭示其排卵數(shù)性狀形成的分子調(diào)控機制,但未取得突破性進展[3,26]。減數(shù)分裂是生物體有性生殖的基礎(chǔ),是生物遺傳、進化和多樣性的重要保證,受精和減數(shù)分裂一同確保了物種世代染色體數(shù)目的穩(wěn)定不變。研究報道,在生殖細胞減數(shù)分裂過程中,若減數(shù)分裂相關(guān)基因遭到破壞,則后期卵巢內(nèi)生殖細胞迅速凋亡,機體出現(xiàn)生殖障礙[20],雌性動物主要表現(xiàn)為卵巢功能早衰,雄性動物主要表現(xiàn)為少精、無精等生精缺陷。錯配修復蛋白主要參與了減數(shù)分裂粗線期的細胞同源重組,在一定程度上保證生殖細胞在減數(shù)分裂過程中的高效率與準確性。MSH4、MSH5是MutS同源蛋白DNA錯配修復蛋白中的兩個重要成員,二者以MSH4-MSH5異源二聚體的形式通過重組參與減數(shù)分裂,并且MSH4、MSH5蛋白的表達大部分在睪丸跟卵巢,說明它們對生殖細胞的發(fā)育與減數(shù)分裂有直接作用[27-29]。Ross-Macdonald等[30]報道,敲除MSH4基因的小鼠的精子與卵母細胞出現(xiàn)了受損的聯(lián)會復合體結(jié)構(gòu)以及染色體錯配現(xiàn)象。MSH4和/或MSH5基因敲除的小鼠生長發(fā)育正常,但生殖器官出現(xiàn)異常,雄性小鼠表現(xiàn)為睪丸縮小,雌性小鼠則表現(xiàn)為卵巢退化,最終導致不育[19-21]。研究結(jié)果表明MSH4、MSH5基因在小鼠的減數(shù)分裂Ⅰ期同源染色體配對中起到至關(guān)重要的作用[20,31]。
本實驗選取連續(xù)3胎產(chǎn)單羔的藏山羊和連續(xù)3胎產(chǎn)3羔的金堂黑山羊為研究對象,克隆得到各自的MSH4、MSH5基因序列。藏山羊MSH4基因編碼區(qū)序列與金堂黑山羊有5處堿基不同,導致3處氨基酸的差異;MSH5基因編碼區(qū)序列有9處堿基不同,導致5處氨基酸的差異。藏山羊與金堂黑山羊MSH4、MSH5氨基酸序列的差異是否會引起生殖細胞減數(shù)分裂的變化,進而使得山羊排卵數(shù)發(fā)生改變而影響山羊的繁殖性狀還有待進一步研究。藏山羊和金堂黑山羊MSH4、MSH5基因編碼區(qū)序列與牛、小鼠、人等都具有較高的同源性,說明MSH4和MSH5基因在哺乳動物進化中均具有較高的保守性。根據(jù)MSH4和MSH5基因編碼區(qū)核苷酸序列構(gòu)建的分子系統(tǒng)發(fā)生樹,藏山羊、金堂黑山羊跟山羊親緣關(guān)系最近,各分支置信度高,表明系統(tǒng)發(fā)生樹具有較高的可信度。MSH4、MSH5的mRNA分別在藏山羊和金堂黑山羊的卵巢、子宮、輸卵管和垂體中均有表達,但兩個品種各組織間mRNA表達量均無顯著性差異(P>0.05),說明兩個基因在山羊卵巢、子宮等組織中發(fā)揮著重要的作用,但是這兩個基因mRNA表達量的差異可能不是引起排卵數(shù)差異的主要原因。
4 結(jié)論
采用RT-PCR技術(shù),首次克隆了藏山羊、金堂黑山羊MSH4和MSH5基因,兩個山羊品種的MSH4基因編碼區(qū)序列有5處堿基不同,導致3處氨基酸的差異;MSH5基因編碼區(qū)序列有9處堿基不同,導致5處氨基酸的差異。MSH4和MSH5基因在兩個山羊品種的卵巢、子宮、輸卵管和垂體中均有表達,但品種間mRNA表達量無顯著性差異。
[1] 王杰, 王永, 歐陽熙, 等. 藏山羊研究[J]. 中國畜牧雜志,1993, 29(1):10-13.
[2] 許德貴, 張家明, 唐詩軍, 等. 金堂黑山羊生產(chǎn)性能研究[J].四川畜牧獸醫(yī), 2006, 33(11):25-26.
[3] Notter DR. Genetic improvement of reproductive efficiency of sheep and goats[J]. Anim Reprod Sci, 2012, 130(3-4):147-151.
[4] 胡亮, 龍石太, 吳憲紅, 等. 川中黑山羊OPN基因cDNA克隆及生物信息學分析[J]. 江蘇農(nóng)業(yè)科學, 2014, 42(5):34-36.
[5] 龍石太, 胡亮, 吳憲紅, 等. 川中黑山羊BMP2、BMP4基因cDNA克隆及生物信息學分析[J]. 江蘇農(nóng)業(yè)科學, 2014, 42(3):19-23.
[6] Nagaoka SI, Hassold TJ, Hunt PA. Human aneuploidy:mechanisms and new insights into an age-old problem[J]. Nat Rev Genet,2012, 13:493-504.
[7] Schroering AG, Edelbrock MA, Richards TJ, et al. The cell cycle and DNA mismatch repair[J]. Exper Cell Res, 2007, 313(2):292-304.
[8] Jirieny J. The multifaceted mismatch-repair system[J]. Nat Rev Mol Cell Biol, 2006, 7(5):335-346.
[9] Eisen JA. A phylogenomic study of the MutS family of proteins[J]. Nucleic Acids Res, 1998, 26(18):4291-4300.
[10] Fishel R, Wilson T. MutS homologs in mammalian cells[J]. Curr Opin Genet Dev, 1997, 7(1):105-113.
[11] Karen VM, Cassandra J, Yashna T, et al. Separable crossoverpromoting and crossover-constraining aspects of Zip1 activity during budding yeast meiosis[J]. PLoS Genet, 2015, 11(6):e1005335.
[12] 駱驊. 黃牛和犏牛睪丸組織中減數(shù)分裂同源重組基因表達、克隆與啟動子區(qū)甲基化分析[D]. 南京:南京農(nóng)業(yè)大學,2013.
[13] 祿婷婷. 錯配修復蛋白基因多態(tài)性與少精、無精癥的相關(guān)研究[D]. 長沙:中南大學, 2009.
[14] Ramaswamy R, Narayanaswamy S, Koodali TN. Structural insights into saccharomyces cerevisiae Msh4-Msh5 complex function using homology modeling[J]. PLoS One, 2013, 8(11):e78753.
[15] Snowden T, Acharya S, Butz C, et al. hMSH4-hMSH5 recognizes Holliday Junctions and forms a meiosis-specific sliding clamp that embraces homologous chromosomes[J]. Mol Cell, 2004, 15(3):437-451.
[16] 祿婷婷, 徐克前. 一組多功能的錯配修復蛋白[J]. 生命的化學, 2008, 165(6):696-700.
[17] Wu XL, Xu KQ, Her CT. The Role of muS homologues MSH4 and MSH5 in DNA metabolism and damage response[M]// Kusic-Tisma J. DNA replication and related cellular processes. Rijeka,Croatia:In Tech Press, 2001:87-110.
[18] Her C, Zhao N, Wu X, et al. MutS homologues hMSH4 and hMSH5:diverse functional implications in humans[J]. Front Biosci, 2007, 12(3):905-911.
[19] Edelmann W, Cohen PE, Kneitz B, et al. Mammalian MutS homologue 5 is required for chromosome pairing in meiosis[J]. Nature Genetics, 1999, 21:123-127.
[20] Kneitz B, Cohen PE, Avdievich E, et al. MutS homolog 4 localization to meiotic chromosomes is required for chromosome pairing during meiosis in male and female mice[J]. Genes Dev,2000, 14(9):1085-1097.
[21] De Vries SS, Baart EB, Dekker M, et al. Mouse MutS-like protein Msh5 is required for proper chromosome synapsis in male and female meiosis[J]. Genes Dev, 1999, 13(5):523-531.
[22] Ehrenstein MR, Neuberger MS. Deficiency in Msh2 affects the efficiency and local sequence specificity of immunoglobulin classswitch recombination:parallels with somatic hypermutation[J]. EMBO J, 1999, 18(12):3484-3490.
[23] Shenker NS, Polidoro S, van Veldhoven K, et al. Epigenome-wide association study in the European Prospective Investigation into Cancer and Nutrition(EPIC-Turin)identifies novel genetic loci associated with smoking[J]. Hum Mol Genet, 2013, 22(5):843-851.
[24] Kazma R, Babron MC, Gaborieau V, et al. Lung cancer and DNA repair genes:multilevel association analysis from the International Lung Cancer Consortium[J]. Carcinogenesis, 2012, 33(5):1059-1064.
[25] Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Res, 2001, 29(9):e45.
[26] 胡亮, 字向東, 盧建遠, 等. 多胎和單胎山羊品種Bcl-2和Bax基因的克隆及組織表達研究[J]. 畜牧與獸醫(yī), 2015, 47(1):28-32.
[27] Bocker T, Barusevicius A, Snowden T, et al. hMSH5:a human MutS homologue that forms a novel heterodimer with hMSH4 and is expressed during spermatogenesis[J]. Cancer Res, 1999, 59(4):816-822.
[28] Her C, Doggett NA. Cloning, structural characterization, and chromosomal localization of the humanorthologue of Saccharomyces cerevisiae MSH5 gene[J]. Genomics, 1998, 52(1):50-61.
[29] Moens PB, Kolas NK, Tarsounas M, et al. The time course and chromosomal localization of recombination-related proteins at meiosis in the mouse are compatible with models that can resolve the early DNA-DNA interactions without reciprocal recombination[J]. J Cell Sci, 2002, 115(8):1611-1622.
[30] Ross-Macdonald P, Roeder GS. Mutation of a meiosis-specific MutS homolog decreases crossing over but not mismatch correction[J]. Cell, 1994, 79(6):1069-1080.
[31] Her C, Wu X, Griswold MD, et al. Human MutS homologue MSH4 physically interacts with von Hippel-Lindau tumor suppressorbinding protein 1[J]. Cancer Res, 2003, 63(4):865-872.
(責任編輯 馬鑫)
cDNA Cloning,Sequence Analysis and Tissue Expression of Gene MSH4 and MSH5 in Goats
ZHENG Jie LIU Shuang LUO Bin HU Liang YANG Ke-wei ZI Xiang-dong
(The Key Laboratory of Animal Science of State Ethnic Affairs Commission,Southwest University for Nationalities,Chengdu 610041)
The total RNA of ovary,endometrium,oviduct and pituitary extracted from 5 Tibetan goats and 5 Jintang black goats in the period of estrus were used to clone and analyze cDNA sequences of gene MSH4 and MSH5 by RT-PCR,and their expression levels in different tissues were determined by real-time PCR. The result showed that the coding region of gene MSH4 from Tibetan goat and Jintang black goat were 2 499 bp,encoding 832 amino acids;there were 5 base variations between two breeds,leading to 3 differences of amino acid. The coding region of gene MSH5 from Tibetan goat and Jintang black goat were 2 496 bp,encoding 831 amino acids;there were 9 base variations between two breeds,leading to 5 differences in amino acid. The nucleotide sequences of coding region of gene MSH4 from Tibetan goat were 99.8%,99.8%,99.4%,98.1%,94.4%,85.1%,84.7% and 93.5% homology with that of Jintang black goat,Capra hircus,Ovis aries,Bos taurus,Equus caballus,Mus musculus,Rattus norvegicus,and Homo sapiesn,respectively. The nucleotide sequences of coding region of gene MSH5 from Tibetan goat were 99.6%,99.6%,97.3%,88.0%,85.8%,85.3% and 90.2% homology with that of Jintang black goat,C. hircus,B. taurus,Canis lupus familiaris,M. musculus,R. norvegicus,and H. sapiens,respectively. The mRNA of gene MSH4 and MSH5 were all expressed in ovary,endometrium,oviduct and pituitary of two breeds of goat,but there was no significant difference between two breeds(P>0.05). The results reveal that both gene MSH4 and MSH5 are conservative in the course of animal evolution,and the role of their effect on prolificacy of goats needs to be further studied.
Tibetan goat;Jintang black goat;MSH4;MSH5;cloning;qPCR
10.13560/j.cnki.biotech.bull.1985.2016.03.016
2015-06-29
四川省應用基礎(chǔ)項目(2013JY0043),西南民族大學研究生創(chuàng)新型科研項目(CX2015SZ088)
鄭杰,男,碩士研究生,研究方向:動物遺傳育種與繁殖;E-mail:zhengjie201505@163.com
字向東,男,教授,研究方向:動物遺傳育種與繁殖;E-mail:zixd@sina.com

