普通小麥幾丁質酶基因的克隆與表達分析
陳冬陽,楊明明,高 翔,2,張龍龍,郭江岸,李 萬
(1.西北農林科技大學農學院,陜西楊凌 712100; 2.陜西省小麥新品種培育工程研究中心,陜西楊凌 712100)
?
普通小麥幾丁質酶基因的克隆與表達分析
陳冬陽1,楊明明1,高 翔1,2,張龍龍1,郭江岸1,李 萬1
(1.西北農林科技大學農學院,陜西楊凌 712100; 2.陜西省小麥新品種培育工程研究中心,陜西楊凌 712100)
幾丁質酶是植物重要的防衛因子之一,與植物抗病性密切相關。為深入了解幾丁質酶基因表達及功能,先利用引物ChiF/ChiR從10份具有不同抗病性的小麥中克隆出幾丁質酶基因后對其進行序列分析及原核表達分析,再利用熒光定量PCR(qRT-PCR)技術分析小麥根、莖和葉不同組織中幾丁質酶基因的表達情況,并選取在小麥根、莖和葉中表達量最高的幾丁質酶基因進行真菌性病害的抗性反應試驗。結果表明,從10份具有不同抗病性的小麥中克隆得到5條長度約960 bp的幾丁質酶基因序列,其中1條序列與已公布的 Chi-3基因序列(序列登錄號為KJ507387)一致,其余序列命名為 Cht1~Cht4(GenBank登錄號為KR049247~KR049250)。序列分析表明,克隆所得序列無內含子,編碼的幾丁質酶屬于ClassⅠb類型,也屬于糖苷水解酶第19家族,是胞外分泌蛋白且兼具溶菌酶活性。SDS-PAGE分析表明,重組質粒pE-Chi能在BL21(DE3)中表達一個33 kDa左右的特異蛋白。qRT-PCR分析表明,這5種幾丁質酶基因在小麥根、莖和葉中均有表達,但在不同器官中表達量差異較大,其中,葉中表達量最高,根中次之,莖中最低;同時,這5種幾丁質酶基因在同一器官中表達量大小具體表現為 Cht4> Cht2> Cht3> Cht1> Chi-3。選取相對表達量最高的 Cht4基因驗證其對真菌性病害的抗性反應,結果表明,在條銹菌脅迫下, Cht4基因在抗病材料92R137中的表達量顯著高于感病材料銘賢169,并且均高于同等條件下接ddH2O的表達量。本研究擴增到的幾丁質酶基因在小麥中組成型表達,說明其參與了小麥的正常生長發育過程; Cht4基因的表達受條銹菌誘導,推測其可能參與了小麥葉部抗條銹菌的防御反應。
小麥;幾丁質酶;原核表達;組織表達;功能分析
由真菌引起的小麥病害嚴重影響小麥的產量和品質。而幾丁質酶(EC 3.2.1.14)能夠水解菌絲生長初期細胞壁中的幾丁質,對真菌具有廣譜抗性[1-3]。目前,已在水稻、小麥、大麥和棉花等100多種植物中檢測到幾丁質酶活性[4]。正常情況下,植物體內幾丁質酶的含量很低,甚至不表達,但當受到病原菌侵染、病毒感染或其他非生物因素誘導時,植物體內的幾丁質酶活性會迅速提高[5-6]。油菜[7]、菜豆[8]、擬南芥[9]、煙草[10]、棉花[11]和甘蔗[12]經真菌誘導后,體內的幾丁質酶大量表達,并且酶活性也有所提高[13]。體外抑菌實驗證明,不同的幾丁質酶經過純化后對綠色木霉菌[14]、立枯絲核菌[15-16]、灰霉病菌[17]、葉霉病菌[18]、尖孢鐮刀菌[19]、赤霉菌[16]、黃萎菌[16]和赤星菌[16]等20多種病原真菌的孢子萌發和菌絲生長具有抑制作用。轉基因研究表明,成功表達異源幾丁質酶基因的煙草[20]、油菜[20-21]、西瓜[22]、黑楊[23]、小麥[24-25]和水稻[26-27]對真菌的抗性明顯高于對照植物。說明幾丁質酶基因是植物重要的防衛基因,在植物生長發育、抗逆和防御反應中發揮重要作用[3,28]。因此,利用幾丁質酶基因培育抗病新品種是植物抗病性研究的熱點之一。但目前基因工程中可被利用的幾丁質酶基因仍很稀少[6,29-30],幾丁質酶基因在不同小麥品種中是否存在突變,及該基因在不同組織中表達量尚不明確。本研究從不同小麥品種中克隆幾丁質酶基因,并利用實時熒光定量的方法研究其在小麥根、莖、葉等不同部位的表達差異,以及受條銹菌誘導后的表達量變化,驗證其對真菌性病害的抗性,以期發掘小麥中的有效抗條銹病基因,為小麥的抗病分子育種提供基礎材料。
1 材料與方法
1.1供試材料
供試材料共計10份,包括陜627、陜715、西農528、西農538、西農556、西農558、西農583、小偃22、銘賢169(感條銹病)和92R137(抗條銹病),除銘賢169和92R137種子由西北農林科技大學韓德俊課題組提供外,其他材料均由本實驗室保存。
1.2幾丁質酶基因的克隆
所有供試材料播種于直徑10 cm的花盆中,24 ℃下光照16 h/黑暗8 h培養2周,收集葉片,利用微量CTAB法[31]提取基因組DNA。根據NCBI中已公布的中國春幾丁質酶基因(KJ507385)的開放閱讀框,設計包含上游起始密碼子和下游終止密碼子的引物ChiF/ChiR(ChiF:5′-ATGAGAGGAGTTGTGGTGG-3′; ChiR:5′-CTATGCGAACGGCCTCT-3′),并交由金斯瑞生物技術有限公司合成。以基因組DNA為模板,利用引物ChiF/ChiR進行PCR擴增。PCR擴增體系:2×TaqMaster Mix(康為世紀公司)12.5 μL,10 μmol·L-1上下游引物各1 μL,DNA模板100 ng,加ddH2O至25 μL。PCR擴增程序:94 ℃預變性5 min;94 ℃變性50 s,61 ℃退火50 s,72 ℃延伸90 s,30個循環;72 ℃延伸10 min。回收目的片段,連接克隆載體pEASY-T1,轉化入感受態細胞Trans-T1,挑選陽性單克隆送金斯瑞生物技術有限公司測序。
1.3生物信息學分析
利用NCBI中BLAST在線軟件進行基因序列比對分析;利用NCBI中ORF Finder預測序列開放閱讀框;利用DNAMAN 7.0進行基因序列翻譯及蛋白序列比對;利用MEGA 5.0構建系統進化樹;利用SignalP 4.1 Server預測氨基酸序列信號肽;利用ProtParam預測蛋白質理化性質;利用ProtScale分析蛋白質親疏水性;利用Prosite預測氨基酸序列基序;利用NCBI中CDD在線預測氨基酸序列保守結構域;利用NPS@網站預測蛋白質二級結構;利用SWISS-MODEL軟件預測蛋白質三級結構。
1.4幾丁質酶基因原核表達載體的構建與誘導表達
以1.2中得到的陽性單克隆為模板,利用ChiF1/ChiR(ChiF1:5′-GAGCAATGCGGCT CGC-3′)進行PCR擴增,反應體系同1.2。回收目的片段,連接表達載體pEASY-E1,構建重組表達載體pE-Chi。轉化入感受態細胞Trans-T1,挑選陽性單克隆,以ChiF1/T7 terminator primer為引物進行正反向檢驗并測序。將插入正確的質粒轉化感受態細胞BL21(DE3),37 ℃、200 r·min-1過夜培養,然后將菌液以1∶100的體積比重新接種新鮮LB培養基(含100 μg·mL-1Amp),28 ℃、200 r·min-1震蕩培養至OD≈0.8時,取1 mL菌液作為對照,在剩余菌液中加入IPTG至終濃度1 mmol·L-1,繼續誘導12 h。收集表達蛋白,處理樣品并進行12% SDS-PAGE電泳檢測。
1.5幾丁質酶基因的熒光定量PCR分析
田間采集西農538花后10 d的根、莖和葉,經液氮快速冷凍后于-80 ℃保存備用。將銘賢169和92R137播種于直徑10 cm的花盆中,待其第一葉充分展開后,參照康振生課題組的方法進行條銹病菌的接種[32],分別采集接菌和ddH2O處理后0、6、12、24、48和72 h的小麥葉片,經液氮快速冷凍后于-80 ℃保存備用。
利用植物總RNA提取試劑盒(Bioteke公司)提取上述-80 ℃保存組織的總RNA。采用Nondrop2000及電泳檢測RNA的濃度及質量。按照Roche公司提供的反轉錄試劑盒說明,取1 μg總RNA進行cDNA第一鏈的合成,于-20 ℃保存備用。
參照克隆得到的基因組序列,利用Primer Premier 5.0設計各基因特異的熒光定量PCR(Quantitative real-time PCR,qRT-PCR)引物(表1),擴增片段為75~200 bp,退火溫度設定在58~62 ℃。以上述各組織cDNA為模板,以小麥肌動蛋白基因β-actin(AB181991.1)為內參,進行qRT-PCR分析。反應體系10 μL,包含Fast Start Essential DNA Green Master 5 μL,5 μmol·L-1混合引物1 μL,cDNA模板4 μL。擴增程序:95 ℃ 10 min;95 ℃ 10 s,62 ℃ 15 s,72 ℃ 20 s,35個循環;65~95 ℃緩慢上升,其中,每0.5 ℃讀取一次熒光信號,通過溶解曲線鑒定擴增產物。每個樣品設置3個重復。采用2-ΔΔCt法分析實驗數據,確定基因的相對表達量[33]。
2 結果與分析
2.1小麥幾丁質酶基因的克隆及核酸序列分析
分別以10個小麥品種的基因組DNA為模板,利用引物ChiF/ChiR進行PCR擴增。擴增產物經1%瓊脂糖凝膠電泳檢測后,得到約為1 000 bp的單一條帶,將目的條帶切膠回收、克隆和測序后,得到5條不同序列。
利用NCBI在線BLAST工具對所得序列進行同源性搜索,結果表明,所有序列與已公布的幾丁質酶基因序列相似度較高,達99%以上,其中,1條序列與胡周波等[34]分離得到的 Chi-3基因序列(序列登錄號為KJ507387)完全相同,初步斷定本研究擴增得到的序列均為幾丁質酶基因。將4條新基因序列命名為 Cht1~ Cht4,并將其提交至GenBank數據庫,得到的序列登錄號為 KR049247~KR049250。進一步比對發現,這四條序列在185~225 bp之間有兩個堿基插入/缺失突變區(圖1),這兩個區域的變化堿基數是三的整數倍,因此只能造成個別氨基酸的插入/缺失,不會造成移碼突變。除此之外的其他位點也存在多個堿基差異。

表1 qRT-PCR分析所用引物Table 1 Primers in qRT-PCR analysis

圖1 幾丁質酶核酸序列主要差異區域Fig.1 Major differences in chitinase DNA sequences
2.2推導的蛋白序列分析
2.2.1理化性質分析
ORF Finder在線預測可知,新擴增到的4條序列均有完整的開放閱讀框,無內含子。SignalP Server預測可知,4條蛋白序列均含有由前20個氨基酸組成的信號肽,并且屬于分泌蛋白。ProtParam預測可知,4個蛋白均屬于穩定蛋白。ProtScale分析可知,除了信號肽及150~200位置的氨基酸外,疏水性區域和親水性區域分布比較均勻,但是親水性氨基酸含量高于疏水性氨基酸,因此4個蛋白屬于親水性蛋白。此外,根據酶切位點的不同,這4條幾丁質酶屬于內切酶,水解位點為同聚物內部的β-1,4-糖苷鍵。
利用Prosite分析幾丁質酶氨基酸序列的模體結構,發現擴增得到的4條幾丁質酶序列N端具有20個氨基酸組成的信號肽,富含8個半胱氨酸的幾丁質結合區(20~61 aa)、可變區和保守的催化區(VSFKTALWFWM),缺少C端延伸區,編碼蛋白應歸屬于ClassⅠb型幾丁質酶;結合NCBI中CDD在線預測得到編碼蛋白屬于糖苷水解酶第19家族,因此,推測這4個編碼蛋白具有幾丁質酶和溶菌酶的雙功能活性。
2.2.2結構分析
利用NPS@網站預測幾丁質酶蛋白序列中的α-螺旋、β-折疊、β-轉角、無規卷曲等二級結構,結果表明,無規則卷曲比例最高,達到62.50%左右;延伸鏈為19.69%左右;α-螺旋最少,約占17.81%;不含其他二級結構。
在SWISS-MODEL網頁提交幾丁質酶基因的氨基酸序列,在蛋白結構庫中搜索比對模板,得到2dkv.1.A序列與KR049250氨基酸序列的21到320殘基一致性達74.07%,具備同源建模的條件。從而得到KR049250等4個幾丁質酶蛋白的三維結構模型(圖2),該蛋白由一個雙葉α+β折疊模體組成[35]。
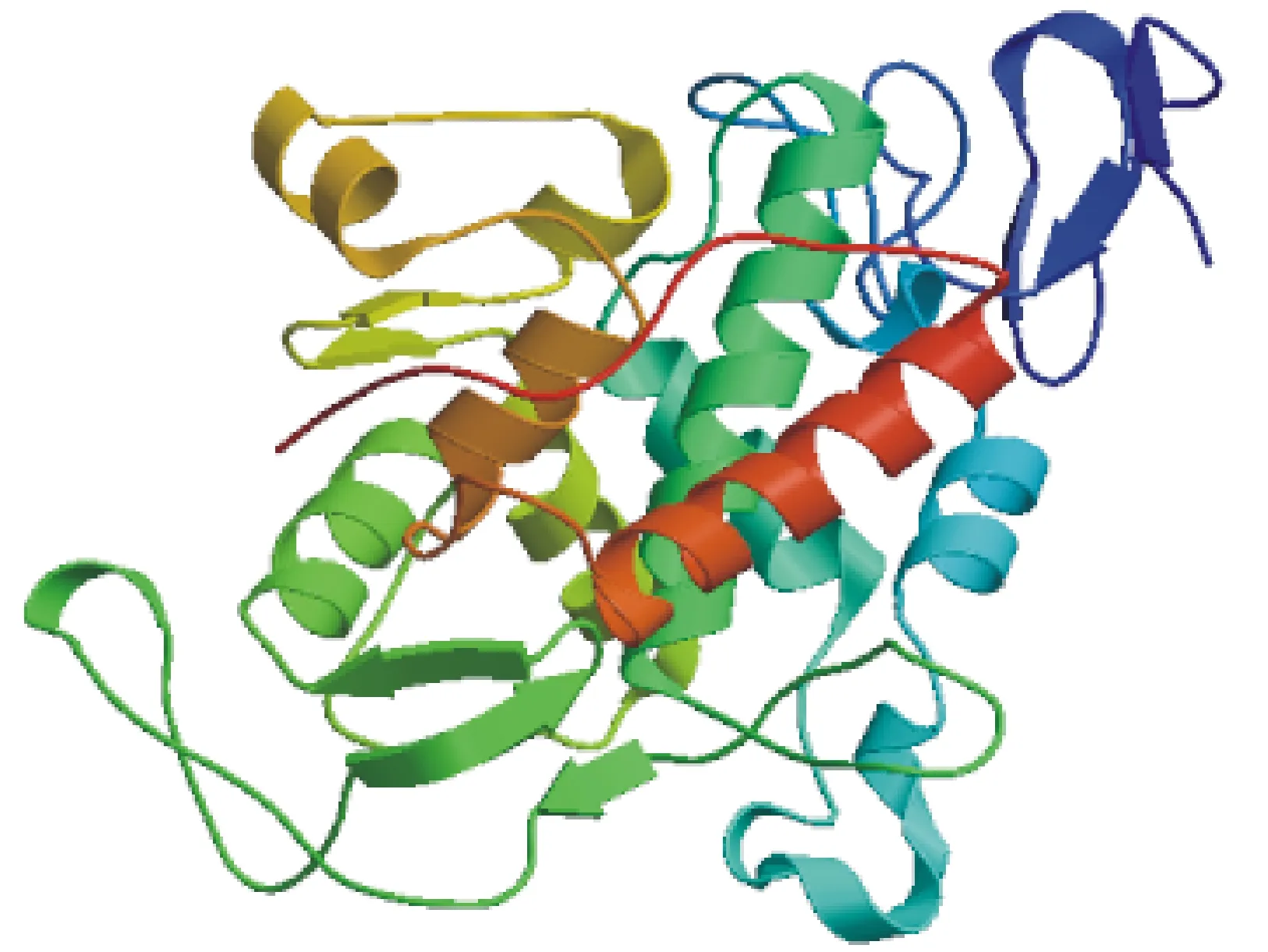
圖2 幾丁質酶蛋白序列的三級結構預測Fig.2 3D structure prediction of chitinase protein sequence
2.3系統進化分析
利用DNAMAN 7.0進行基因序列翻譯及氨基酸序列比對,結果(圖3)顯示,克隆得到的4條氨基酸序列相似度極高,主要依據2個位點出現的插入缺失進行區分。此外,KR049250氨基酸序列與小麥(CAA53626.1)的同源性為96.57%,與黑麥(AAG53610.1)的同源性為47.04%,與水稻(AAM08773.1)、擬南芥(AAM14810.1)、苦瓜(AAM18075.1)、大蕁麻(P11218.3)和陸地棉(AAP80801.1)的同源性分別為12.35%、37.58%、10.89%、38.64%和30.79%。
用MEGA 5.0將獲得的4條蛋白序列與其他類型的幾丁質酶蛋白序列構建系統進化樹,結果(圖4)顯示,本研究得到的蛋白與ClassⅠ型幾丁質酶親緣關系最近,應歸屬于此類;此外與ClassⅡ類幾丁質酶親緣關系相對較近,但與其他種類的幾丁質酶親緣關系較遠。

實線框示信號肽位置;圓點框示區分4個幾丁質酶基因的位點;短線框示保守催化位點
Bold wireframe indicates signal peptide; Dot frame indicates the sites to distinguish 4 chitinases; Short-term frame indicates catalytic
圖3幾丁質酶基因編碼氨基酸的序列比對分析
Fig.3Alignment of the deduced amino acid sequences of chitinase

“★”代表本研究中4條幾丁質酶基因推導得到的蛋白
“★” indicates the deduced proteins of 4 chitinase genes obtained in this study
圖4幾丁質酶氨基酸序列的系統進化樹分析
Fig.4Phylogenetic analysis of chitinase amino acid sequences
2.4小麥幾丁質酶基因的原核表達
將去掉信號肽的核酸序列連接至載體pEASY-E1,構建重組表達載體pE-Chi,并在感受態細胞BL21(DE3)中誘導表達。結果(圖5)發現,28 ℃、200 r·min-1震蕩培養至OD≈0.8時加入IPTG至終濃度1 mmol·L-1,表達效率最高,得到分子量約為33 kDa大小的特征條帶,與推測得到的蛋白分子量大小一致,而作為對照未誘導的菌株未表達此蛋白,說明小麥幾丁質酶基因在大腸桿菌中得到正確表達。
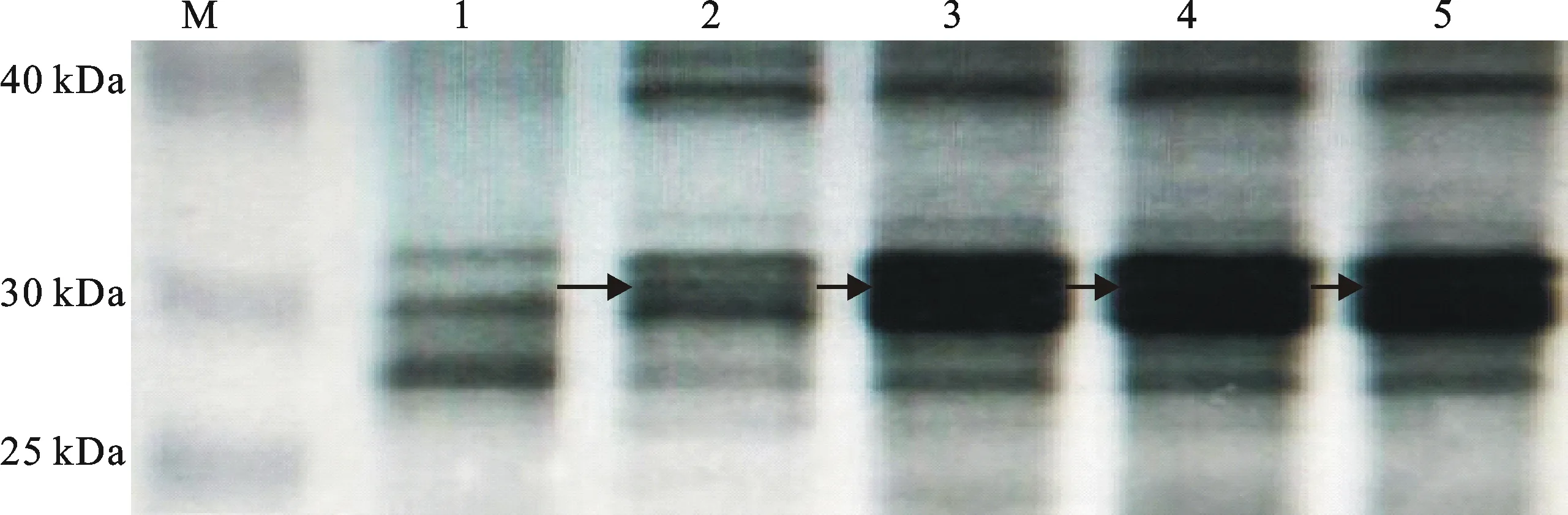
M:Blue Plus Protein Marker;1:未誘導的重組質粒;2~5:誘導的重組質粒KR049247、KR049248、KR049249和KR049250
M:Blue Plus Protein Marker; 1:Protein of recombinant plasmid without induction; 2-5:Protein of recombinant plasmid KR049247,KR049248,KR049249 and KR049250 after induction
圖5誘導表達產物的SDS-PAGE分析
Fig.5SDS-PAGE analysis of protein expression
2.5小麥幾丁質酶基因的表達特性分析
2.5.1小麥幾丁質酶基因的組織表達特性
以小麥花后10 d的根、莖和葉cDNA為模板,利用qRT-PCR技術對 Cht1~Cht4和 Chi-3基因的表達特性進行了分析。結果(圖6、圖7)顯示,在以上3種組織中均能檢測到這5種基因的轉錄表達,但各基因在不同組織中的表達量存在較大差異,其中,葉中表達量最高,根中次之,莖中最低。同時也可發現,不同基因在同一組織中的表達量大小具有一定的規律,具體表現為 Cht4> Cht2> Cht3> Cht1> Chi-3。
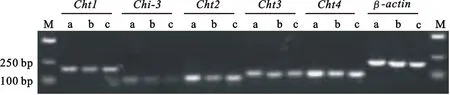
M:D2000 DNA marker; a、b和c分別代表一次重復
M:D2000 DNA marker; a,b and c represent three replicates,respectively
圖6qRT-PCR產物檢測
Fig.6Detection of qRT-PCR products
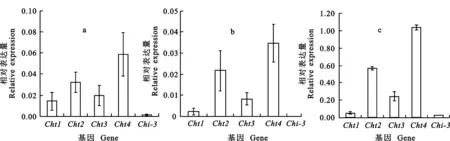
圖7 小麥根(a)、莖(b)和葉(c)中幾丁質酶基因的表達分析Fig.7 Expression analysis of chitinase in wheat′s root (a),stem (b) and leaf (c)
2.5.2小麥幾丁質酶基因在條銹菌侵染后的表達特性
以感病品種接ddH2O后0 h的基因表達量為對照,研究在正常情況下表達量最高的 Cht4基因在條銹菌誘導下的表達量變化。結果(圖8)顯示,92R137(抗病品種)和銘賢169(感病品種)在接種ddH2O后葉片中的 Cht4基因表達量變化不大;而在條銹菌誘導條件下兩者的 Cht4基因表達量均呈現先上升后下降的變化趨勢,不過銘賢169中變化沒有92R137明顯。在銹菌處理后葉片中的 Cht4基因表達量迅速升高,6 h時達到峰值,此時92R137中 Cht4基因表達量是對照的12.5倍,而銘賢169中 Cht4基因的表達量僅是對照的6.33倍。隨著誘導時間的延長,幾丁質酶基因表達量逐漸下降,至脅迫72 h后92R137和銘賢169葉片中 Cht4基因的表達量分別是對照的3.23倍和2.75倍,仍然維持較高水平。
3 討 論
隨著對幾丁質酶生化性質、作用機理及表達調控的深入研究,利用其培育抗病新品種成為植物抗真菌病害研究的熱點之一[6]。本研究在10個不同小麥品種中均克隆到幾丁質酶基因,為進一步分析小麥幾丁質酶對病原真菌的抑制作用以及植物抗病基因工程提供了材料。對序列進行分析表明,本研究得到的幾丁質酶基因兼具溶菌酶活性,并且無內含子。而有研究[36]表明,當發生外界脅迫或mRNA剪切功能障礙時,對于幾丁質酶基因不含內含子或含內含子很少的植物體仍能快速合成幾丁質酶,不會影響其正常生理功能。
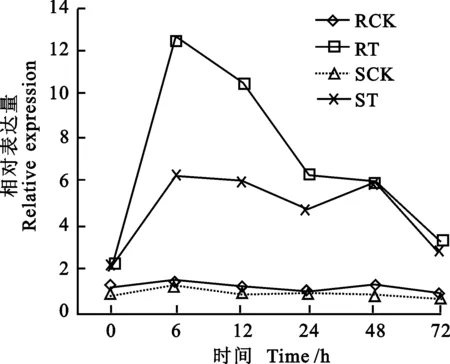
RCK:92R137接種ddH2O; RT:92R137接種條銹菌; SCK:銘賢169接種ddH2O; ST:銘賢169接種條銹菌
RCK:92R137 induced by ddH2O; RT:92R137 induced byPucciniastrilformis;SCK:Mingxian 169 induced by ddH2O; ST:Mingxian 169 induced byPucciniastrilformis
圖8條銹菌處理后 Cht4基因的表達分析
Fig.8Expression analysis of Cht4 gene induced byPucciniastrilformis
本研究獲得的幾丁質酶具有幾丁質結合區、可變交聯區和催化區,而不含C端延伸區,屬于ClassⅠb類型。亞細胞定位預測該幾丁質酶將分泌于胞外,與已知的ClassⅠb類型幾丁質酶分泌于胞外的研究[37-38]一致。很多病原菌初期侵染發生在寄主植物的細胞間隙,胞外的ClassⅡ類幾丁質酶通過分解病原真菌的幾丁質,產生信號分子,促成包括幾丁質酶在內的病程相關蛋白的大量表達[4],但ClassⅡ類幾丁質酶幾乎沒有抑菌作用,而抑菌效果最好的Ia類幾丁質酶大部分定位于液泡[39]。因而本研究得到的幾丁質酶既能夠迅速的對病原真菌的侵染做出反應,又能充分發揮其抑菌作用,增強植物抗病性。此外,本研究中幾丁質酶基因在大腸桿菌中的成功表達,有利于其體外活性的分析及蛋白結構的研究,對幾丁質酶在工業上的發展也有促進作用[34]。
幾丁質酶主要分布于植物的根、莖、葉、花、種子以及愈傷組織中[40-42],但不同組織中的含量不同[4]。如番茄中1種幾丁質酶在花柱中含量較高,而葉中含量很低[43]; Ghachi3和 Ghachi4基因在棉花的根、莖、葉、花和種子中均有表達,但在花和韌皮部中表達量較高,而在木質部和子房中表達量較低[11]; ScChiⅦ1基因在甘蔗芽中表達量最高[12]。本研究中幾丁質酶基因在根、莖、葉中均存在轉錄表達,說明這些基因為組成型表達,可能參與了小麥正常生長發育過程。所有基因均在葉中的表達量最高,根中其次,莖中最低,推測這些幾丁質酶基因更多的參與對小麥葉部病害的防御反應。不同組織中 Cht4基因的表達量均為最高,推測其在該組同工酶中發揮作用時貢獻最大。而對于是否是由于185~225 bp間的兩個堿基插入/缺失突變區,造成這幾種幾丁質酶含量的顯著差異還需要進一步探究。該實驗結果與 Wch1基因在中國春莖中表達,而在葉和根中未表達結果[44]不一致,可能是因為抗性不同的小麥品種中幾丁質酶含量有所差異,并且植物體在接種條銹菌后會影響未經接種部位幾丁質酶基因的表達。
抗性不同的品種在受到病原真菌侵染后,抗病品種中幾丁質酶含量高,而感病品種中表達量低或不表達[45-46]。不同抗性的小麥品種在受到葉銹菌[47]、腥黑穗病菌[48]、條銹菌[49]侵染后,抗病小麥中幾丁質酶基因的表達活性明顯高于感病品種。本研究中, Cht4基因在不同抗性品種以及同一抗性品種不同時間的表達水平具有明顯差異。受條銹菌誘導后, Cht4基因在92R137和銘賢169中的表達量較對照都有所升高,推測該基因能夠受條銹菌誘導表達,是小麥抵抗條銹菌侵染的非特異性抗性基因。該結果與中國春近等基因系接種條銹菌后,不親和組合中小麥葉片 Wch1基因表達上調結果一致,可見,條銹病菌能誘導不同的幾丁質酶基因表達,間接證明了幾丁質酶在抗條銹病中的作用。接種條銹病菌后, Cht4基因在銘賢169中的變化比92R137中緩慢、表達量低,可能是由于銘賢169啟動防御反應的信號轉導比92R137慢,使得其體內的幾丁質酶表達量相對較少,給病原真菌侵染提供可能,造成其表現為感病。此外,在本研究中 Cht4基因在92R137和銘賢169接種條銹菌后均出現上調表達,并且反應時間較 Wch1基因短,而 Wch1基因只在親和組合中受條銹菌誘導,推測 Cht4基因抑制病原菌生長更加有效。但是該幾丁質酶基因在抗病機制中的貢獻究竟有多少尚不清楚,還有待于通過基因沉默、過表達等技術進一步深入研究。
[1]夏 偉,張 紅,顏艷偉,等.棘孢木霉L4對立枯絲核菌的拮抗機制 [J].植物保護學報,2010,37(5):477-478.
Xia W,Zhang H,Yan Y W,etal.Rivalry mechanisms ofTrichodermaasperellumaL4 toRhizoctoniasolani[J].JournalofPlantProtection,2010,37(5):477-478.
[2]吳志剛,朱旭芬.幾丁質酶的分子生物學特性及其在轉基因植物中的應用 [J].生命科學,2002,14(2):117-121.
Wu Z G,Zhu X F.Molecular biology characterization and application in transgenic plant for chitinase [J].ChineseBulletinofLifeSciences,2002,14(2):117-121.
[3]Emani C,Garcia J M,Lopata F E,etal.Enhanced fungal resistance in transgenic cotton expressing an endochitinase gene fromTrichodermavirens[J].PlantBiotechnologyJournal,2003,1(5):321-336.
[4]歐陽石文,趙開軍,馮蘭香,等.植物幾丁質酶的研究進展 [J].生物工程進展,2001,21(4):30-34.
Ouyang S W,Zhao K J,Feng L X,etal.Advances in research of plant chitinases [J].ProgressinBiotechnology,2001,21(4):30-34.
[5]Linthorst H J,Van Loon L C.Pathogenesis-related proteins of plants [J].CriticalReviewsinPlantSciences,1991,10(2):123-150.
[6]陳愛葵,莊文宋,陳冬梅.植物幾丁質酶及其基因工程研究進展 [J].廣東教育學院學報,2010,30(3):47-52.
Chen A K,Zhuang W S,Chen D M.Advances in the research on genetic engineering of plant chitinase [J].JournalofGuangdongEducationInstitute,2010,30(3):47-52.
[7]Rasmussen U,Bojsen K,Collinge D B.Cloning and characterization of a pathogen-induced chitinase inBrassicanapus[J].PlantMolecularBiology,1992,20(2):277-287.
[8]Roby D,Broglie K,Cressman R,etal.Activation of a bean chitinase promoter in transgenic tobacco plants by phytopathogenic fungi [J].ThePlantCell,1990,2(10):999-1007.
[9]Samac D A,Hironaka C M,Yallaly P E,etal.Isolation and characterization of the genes encoding basis and acidic chitinase inArabidopsisthaliana[J].PlantPhysiology,1990,93(3):907-914.
[10]Fukuda Y,Ohme M,Shinshi H.Gene structure and expression of a tobacco endochitinase gene in suspension cultured tobacco cells [J].PlantMolecularBiology,1990,16(1):1-10.
[11]楊郁文,張保龍,倪萬潮,等.兩個棉花幾丁質酶基因的克隆與表達分析 [J].棉花學報,2008,20(2):88-93.
Yang Y W,Zhang B L,Ni W C,etal.Molecular cloning and expression analysis of two chitinase in upland cotton [J].CottonScience,2008,20(2):88-93.
[12]王珊珊,蘇亞春,楊玉婷,等.甘蔗幾丁質酶基因ScChiⅦ1 的克隆與表達分析 [J].熱帶作物學報,2014,35(2):289-298.
Wang S S,Su Y C,Yang Y T,etal.Molecular cloning and expression analysis of chitinase geneScChiⅦ1 in sugarcane [J].ChineseJournalofTropicalCrops,2014,35(2):289-298.
[13]梁美霞,李景富,許向陽,等.幾丁質酶基因在植物抗真菌病基因工程中的應用 [J].黑龍江農業科學,2004(5):29-31.
Liang M X,Li J F,Xu X Y,etal.Chitinases and its application in anti-fungal gene engineering [J].HeilongjiangAgriculturalSciences,2004(5):29-31.
[14]Schlumbaum A,Mauch F,Voegeli U,etal.Plant chitinases are potent inhibitors of fungal growth [J].Nature,1986,324(6095):365-367.
[15]朱 慧,王云霞,胡白石,等.產酶溶桿菌OH11菌株幾丁質酶基因的克隆與表達 [J].南京農業大學學報,2008,31(1):47-50.
Zhu H,Wang Y X,Hu B S,etal.Cloning and expression of a chitinase gene fromLysobacterenzymogenesstrain OH11 [J].JournalofNanjingAgriculturalUniversity,2008,31(1):47-50.
[16]劉寶業,梁國魯,張增艷.水稻幾丁質酶的原核表達、復性及體外抑茵活性的研究 [J].核農學報,2009,23(3):369-374.
Liu B Y,Liang G L,Zhang Z Y.Prokaryotic expression,refolding and antifugal activity of a rice chitinaseinvitro[J].JournalofNuclearAgriculturalSciences,2009,23(3):369-374.
[17]Huang C J,Wang T K,Chung S C,etal.Identification of an antifungal chitinase from a potential biocontrol agent,bacilluscereus28-9 [J].BiochemistryandMolecularBiology,2005,8(1):82-88.
[18]Lee K Y,Heo K R,Choi K H,etal.Characterization of a chitinase gene exhibiting antifungal activity from a biocontrol bacteriumBacilluslicheniformisN1 [J].PlantPathology,2009,25(4):344-351.
[19]仲 健,朱 沙,張 琦.葡萄幾丁質酶基因在畢赤酵母中的表達及其生物活性研究 [J].河南農業科學,2009(5):90-93.
Zhong J,Zhu S,Zhang Q.Expression and functional analysis of grape chitinase inPichiapastorisGS115 [J].JournalofHenanAgriculturalSciences,2009(5):90-93.
[20]Broglie K,Chet I,Holliday M,etal.Transgenic plants with enhanced resistance to the fungal pathogenRhizoctoniasolani[J].Science,1991,254(5035):1194-1197.
[21]藍海燕,張麗華,劉桂珍,等.導入β-1,3-葡聚糖酶及幾丁質酶基因的轉基因可育油菜及其抗菌核病的研究 [J].生物工程學報,2000,16(2):142-146.
Lan H Y,Zhang L H,Liu G Z,etal.Studies on transgenic oilseed rape (Brassicanapus) plants transformed with β-1,3-glucanase and chitinase genes and its resistance toSclerotiniasclerotiorium[J].CriticalReviewsinPlantSciences,2000,16(2):142-146.
[22]王果萍,王景雪,孫 毅,等.幾丁質酶基因導入西瓜植株及其抗病性鑒定研究 [J].植物遺傳資源學報,2003,4(2):104-109.
Wang G P,Wang J X,Sun Y,etal.Transformation of watermelon plants with chintinase gene and evaluation forFusariumwilt resistance [J].JournalofPlantGeneticResources,2003,4(2):104-109.
[23]孟 亮,李紅雙,金德敏,等.轉幾丁質酶基因黑楊的獲得 [J].生物技術通報,2004(3):48-51.
Meng L,Li H S,Jin D M,etal.Transformation ofPopulusdeltoidswith CH5B gene [J].BiotechnologyBulletin,2004(3):48-51.
[24]牛吉山,鄭 磊,洪德峰,等.轉水稻幾丁質酶基因小麥的獲得和白粉病抗性鑒定 [J].河南農業大學學報,2008,5(42):483-486.
Niu J S,Zheng L,Hong D F,etal.Obtaining of transgenic wheat of rice chitinase gene and the powdery mildew assessment [J].JournalofHenanAgriculturalUniversity,2008,5(42):483-486.
[25]葉興國,程紅梅,徐惠君,等.轉幾丁質酶和β-1,3-葡聚糖酶雙價基因小麥的獲得和鑒定 [J].作物學報,2005,31(5):583-586.
Ye X G,Cheng H M,Xu H J,etal.Development of transgenic wheat plants with chitinase and β-1,3-glucosanase genes and their resistance toFusariumHeadBlight[J].ActaAgronomicaSinica,2005,31(5):583-586.
[26]Nishizawa Y,Nishio Z,Nakazono K,etal.Enhanced resistance to blast (Magnaporthegrisea) in transgenicjaponicarice by constitutive expression of rice chitinase [J].TheoreticalandAppliedGenetics,1999,99(3-4):383-390.
[27]袁紅旭,許新萍,張建中,等.轉幾丁質酶基因( RC24)水稻中大2號抗紋枯病特性研究 [J].中國水稻科學,2004,18(1):39-42.
Yuan H X,Xu X P,Zhang J Z,etal.Characters of resistance to rice sheath blight of zhongda 2,a transgenic rice line modified by chitinase gene(RC24) [J].ChineseJournalofRiceScience,2004,18(1):39-42.
[28]Vellicce G R,Díaz Ricci J C,Hernández L,etal.Enhanced resistance toBotrytiscinereamediated by the transgenic expression of the chitinase gene ch5B in strawberry [J].TransgenicResearch,2006,15(1):57-68.
[29]高麗美,李永鋒,徐子勤.水稻堿性幾丁質酶基因表達載體pCAMBIA 1301- RC24的構建 [J].山西師范大學學報:自然科學版,2009,23(3):82-86.
Gao L M,Li Y F,Xu Z Q.Construction of the expression vector pCAMBIA 1301- RC24 carrying chitinase gene [J].JournalofShanxiNormalUniversity:NaturalScienceEdition,2009,23(3):82-86.
[30]王步云,李 聰,王涌鑫,等.紫花苜蓿幾丁質酶基因MsChiⅣ克隆及序列分析 [J].分子植物育種,2009,7(2):347-354.
Wang B Y,Li C,Wang Y X,etal.Cloning and characterization ofMsChiⅣ gene fromMedicagosativaL.[J].MolecularPlantBreeding,2009,7(2):347-354.
[31]Murray M G,Thompson W F.Rapid isolation of high molecular weight plant DNA [J].NucleicAcidsResearch,1980,8(19):4321-4326.
[32]康振生,李振岐.洛夫林10常溫致病新菌系的發現 [J].西北農學院學報:自然科學版,1984,12(4):18-28.
Kang Z S,Li Z Q.Discovery of a normal type new pathogenic strain to Lovrin 10 [J].JournalofNorthwestAgricultureCollege:NaturalScienceEdition,1984,12(4):18-28.
[33]Livak K J,Schmittgenb T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct method [J].Methods,2001,25(4):402-408.
[34]胡周波,高 翔,陳其皎,等.普通小麥中國春Chi基因的克隆、染色體定位及原核表達 [J].西北農業學報,2014,23(8):25-33.
Hu Z B,Gao X,Chen Q J,etal.Cloning,chromosomal localization and prokaryotic expression ofChigenes from wheat cultivar Chinese Spring(TriticumaestivumL.) [J].ActaAgriculturaeBoreali-occidentalisSinica,2014,23(8):25-33.
[35]Hart P J,Pfluger H D,Monzingo A F,etal.The refined crystal structure of an endochitinase fromHordeumvulgareL.seeds at 1.8A resolution [J].JournalofMolecularBiology,1995,248:402-413.
[36]藍海燕,陳正華.幾丁質酶及其研究進展 [J].生命科學研究,1998,2(3):163-171.
Lan H Y,Chen Z H.Advances in plant chitinase research [J].LifeScienceResearch,1998,2(3):163-171.
[37]Shinshi H,Neuhaus J M,Ryals J,etal.Structure of a tobacco endochitinase gene:evidence that different chitinase genes can arise by transposition of sequences encoding a cysteine-rich domain [J].PlantMolecularBiology,1990,14(3):357-368.
[38]Kunze I,Nilsson C,Adler K,etal.Correct targeting of a vacuolar tobacco chitinase inSaccharomycescerevisiae-post-translational modifications are dependent on the host strain[J].BiochimicaetBiophysicaActa:GeneStructureandExpression,1998,1395(3):329-344
[39]Legrand M,Kauffmann S,Geoffroy P,etal.Biological function of pathogenesis-related proteins:Four tobacco pathogenesis-related proteins are chitinases [J].ProceedingsoftheNationalAcademyofScienceoftheUnitedStatesofAmerica,1987,84(19):6750-6754.
[40]陳崇順,徐鳳彩,李明啟.21科41種(變種)植物葉片幾丁質酶系的研究 [J].植物資源與環境,1993,2(4):28-33.
Chen C S,Xu F C,Li M Q.Studies on the chitinase system in leaves from 41 plant species (var.) related 21 families [J].JournalofPlantResourcesandEnvironment,1993,2(4):28-33.
[41]Flach J,Pilet P E,Jolles P.What’s new in chitinase research [J].CellularandMolecularLifeSciences,1992,48(8):701-716.
[42]趙光潔,王志英,趙述禹.幾丁質酶及其在轉基因植株內活力的測定及定位 [J].山西林業科技,2009,38(1):40-43.
Zhao G J,Wang Z Y,Zhao S Y.Introduction of chitinase,determination and location of its activity in transgenic plant [J].ShanxiForestryScienceandTechnology,2009,38(1):40-43.
[43]Leah R,Skriver K,Knudsen S,etal.Identification of an enhancer/silencer sequence direction the aleurone-specific expression of a barley chitinase gene [J].PlantJournal,1994,6(4):579-789.
[44]Liao Y C,Kreuzaler F,Fischer R,etal.Characterization of a wheat Class Ib chitinase gene differentially induced in isogenic lines by infection withPucciniagraminis[J].PlantScience,1994,103(2):177-187.
[45]李和平,鐘永旺,趙純森,等.小麥幾丁質酶基因 Wch2的克隆與表達分析 [J].植物生理與分子生物學學報,2003,29(4):347-352.
Li H P,Zhong Y W,Zhao C S,etal.Cloning and expression analysis of a wheat chitinase gene Wch2 [J].JournalofPlantPhysiologyandMolecularBiology,2003,29(4):347-352.
[46]Hietala A M,Kvaalen H,Schmidt A,etal.Temporal and spatial profiles of chitinase expression by Norway spruce in response to bark colonization byHeterobasidionannosum[J].AppliedandEnvironmentalMicrobiology,2004,70(7):3948-3953.
[47]Anguelova-Merhar V S,Westhuizen V D,Pretorius Z A.β-1,3-glucanase and chitinase activities and the resistance response of wheat to leaf rust [J].JournalofPhytopathology,2001,149(7-8):381-384.
[48]Lu Z X,Gaudet D,Puchalski B,etal.Inducers of resistance reduce common bunt infection in wheat seedlings while differentially regulating defence-gene expression [J].PhysiologicalandMolecularPlantPathology,2005,67(3):138-148.
[49]Mohammadi M,Roohparvar R,Torabi M.Induced chitinase activity in resistant wheat leaves inoculated with an incompatible race ofPucciniastriiformisf.sp.tritici,the causal agent of yellow rust disease [J].Mycopathologia,2002,154(3):119-126.
Cloning and Expression Analysis of Chitinase Genes in Common Wheat (TriticumaestivumL.)
CHEN Dongyang1,YANG Mingming1,GAO Xiang1,2,ZHANG Longlong1,GUO Jiang’an1,LI Wan1
(1.College of Agronomy,Northwest A&F University,Yangling,Shaanxi 712100,China;2.New Varieties Cultivation of Wheat Engineering Research Centre of Shaanxi Province,Yangling,Shaanxi 712100,China)
Chitinase,an important defensive factor in plants,is closely related to plant disease resistance. In order to understand the expression and function of chitinase genes,by the using of primer ChiF/ChiR,chitinase genes were cloned by PCR from 10 wheat cultivars with different resistance to disease in this study. After that,these sequences were analyzed by different biology software,and were expressed inE.coli.The expression of these genes in root,stem and leaf were analyzed by quantitative real-time PCR. The highest expression of chitinase gene in these tissues was selected to test the resistance with fungal diseases. The result showed that 5 chitinase gene sequences (960±3 bp in length) were cloned from 10 wheat cultivars with different resistance to disease. Of them,one sequence is consistent with the published Chi-3 gene sequence with the accession number KJ507387,and the others were named Cht1 to Cht4 with the accession numbers of KR049247 to KR049250,respectively.Sequence analysis showed that the chitinase genes in this research have no intron,encoding chitinase belonging to ClassⅠb type. The chitinase are the members of glycoside hydrolase family 19,which are extracellular secreted protein and have lysozyme activity. The results of SDS-PAGE showed that the recombinant plasmid pE-Chicould express a 33 kDa protein after induction in BL21(DE3).The results of quantitative real-time PCR showed that these five kinds of chitinase genes were expressed in wheat roots,stems and leaves. However,the amount of expression in different organs are quite different. The content of each chitinase gene in this experiment is highest in leaf,followed by root,and is minimum in stem. Meanwhile,the expression of these five kinds of chitinase genes in the same tissue is ranked as: Cht4> Cht2> Cht3> Cht1> Chi-3. Therefore, Cht4 gene,which has the maximum content in the same organ,was selected to validate its resistance to fungal diseases. The results showed that under the stripe rust stress,the expression of Cht4 gene in 92R137 (resistant material) was significantly higher than in Mingxian 169 (susceptible material),and both of them were higher than control. In this study,the chitinase genes are constitutively expressed in wheat,indicating their participation in the regular growth and development of wheat. The expression of Cht4 gene is induced by stripe rust,suggesting that it may be involved in the defense reaction for stripe rust in wheat leaves.
Wheat; Chitinase; Prokaryotic expression; Gene expression; Function analysis
時間:2016-05-10
2015-12-10
2016-01-20
“十二五”農村領域國家科技計劃項目(2011AA100501);國家現代農業產業技術體系建設專項(CARS-3-2-47)
E-mail:chendongyang1990@163.com
高 翔(E-mail:gx@nwsuaf.edu.cn)
S512.1;S330
A
1009-1041(2016)05-0539-10
網絡出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20160510.1623.002.html

