貝達葡萄酒中酵母菌的分離鑒定
謝俊云,姚 笛,郭 瑜,佐兆杭,張 微
(黑龍江八一農墾大學食品學院,黑龍江大慶 163319)
貝達葡萄酒中酵母菌的分離鑒定
謝俊云,姚 笛,郭 瑜,佐兆杭,張 微
(黑龍江八一農墾大學食品學院,黑龍江大慶 163319)
為了獲得一株葡萄酒發酵后期的主導菌,試驗對貝達葡萄酒中的酵母菌進行分離純化,經菌落形態鑒定,并進行酵母分離株26S rRNA的PCR擴增,進一步通過基因序列分析進行鑒定。結果顯示,貝達葡萄酒的酵母菌菌落總數為3.4×107CFU/mL,分離的酵母菌菌株Y3,Y5,Y13均為釀酒酵母。因此,貝達葡萄酒發酵后期的優勢菌即為釀酒酵母。
貝達葡萄酒;酵母菌;鑒定
葡萄酒是目前世界上產量最大、普及最廣的單糖釀造酒,是用葡萄果實或葡萄汁經過發酵釀制而成的酒精飲料[1]。葡萄酒具有養顏護膚、抗癌、抗衰老、抗輻射等功效,能夠增進食欲、消除疲勞[2]。葡萄酒的發酵是多個酵母菌種及菌株互作生長和發展變化的復雜體系[3]。在葡萄酒發酵過程中,酵母菌的種群構成及變化對葡萄酒的風味和品質有重要影響,尤其是天然酵母的存在,對葡萄酒特色的形成極為重要。一般情況下,在自然發酵過程中,畢赤酵母屬、假絲酵母屬、梅奇酵母屬等酵母菌可啟動發酵,并在發酵中期逐步死亡和減少。釀酒酵母使發酵繼續,直至完成[4]。
利用發酵中后期的優勢菌作為菌種,發酵生產葡萄酒已成為葡萄酒生產的關鍵步驟。因此,篩選和鑒定葡萄酒發酵中后期的主導菌對于葡萄酒的生產具有重要意義。所以,尋找能夠適應當地葡萄原料特性,且有利于形成地域葡萄酒風格的新酵母菌株已成為研究的焦點[5-6]。目前,序列的同源性分析已被廣泛用于酵母種屬水平的鑒定等[7]。本研究擬對大慶市葡萄產區的貝達葡萄酒中的酵母菌進行分離純化,并運用分子生物學技術對酵母分離株進行鑒定,鑒定出的酵母分離株旨在為葡萄酒生產菌種資源的進一步利用奠定基礎。
1 材料和方法
1.1 試驗材料
1.1.1 原料
貝達葡萄酒,由黑龍江八一農墾大學食品加工實驗室自釀。
1.1.2 主要試劑和培養基
纖維素酶、溶菌酶、瓊脂糖、膠回收試劑盒、dNTP和TaqDNA聚合酶,購自大連Takara公司;YPD瓊脂培養基、麥芽汁瓊脂培養基等,青島海博公司產品;氯化鈉、飽和酚、乙醇、氯仿、異戊醇、異丙醇、EDTA和Tris等,均為國產分析純。
1.1.3 引物
根據Genebank發表的酵母菌26S rRNA基因序列,利用Primer 5設計引物,引物序列如下,F:5'-G GCTTTAAGTGAGTTGATTG-3',R:5'-TTGCCAGCATC CTTGACTTA-3',引物由上海生工生物工程有限公司合成。
1.1.4 培養基
YPD培養基按照說明書配制,121℃高壓蒸汽滅菌20 min。
1.1.5 主要儀器與設備
EDC-810型PCR擴增儀,東勝創新生物科技有限公司產品;JY04S型凝膠成像系統,北京君意東方電泳設備有限公司產品;XD-30A型生物顯微鏡,寧波舜宇儀器有限公司產品;DRP-9082型電熱恒溫培養箱,上海森信實驗儀器有限公司產品;YX-280D型壓力蒸汽滅菌器,合肥華泰醫療設備有限公司產品;NW10UF型水純化系統,HealForce產品;TGL-16B型離心機,上海安亭科學儀器廠制造產品。
1.2 方法
1.2.1 葡萄酒中酵母菌總數的測定
YPD培養基冷卻至50~60℃時,加入青霉素G鈉和鏈霉素溶液,以防細菌的生長。取25 mL葡萄酒樣品于含225 mL無菌生理鹽水的三角瓶中,然后將葡萄酒樣品進行10倍系列稀釋,取1×10-4~1× 10-6的樣品液1 mL傾注接種,傾入YPD培養基,每個稀釋度做2個平行,待凝固后28℃培養5 d后進行菌落計數。
1.2.2 葡萄酒中酵母菌的分離
取上述葡萄酒樣品的稀釋液于滅菌平皿中,傾入YPD培養基,置28℃下恒溫箱中培養,每24 h觀察1次,對培養基上長出的疑似酵母菌菌落進行純化培養,鏡檢為純種后接入YPD固體培養基斜面,于4℃下低溫保存。生長的酵母菌菌種用阿拉伯數字Y1,Y2,Y3,Y4……依次編號。
1.2.3 酵母菌分離株的形態學鑒定
對純化培養的酵母菌單菌落進行形態學觀察,然后取分離得到的單菌落進行染色,酵母菌采用番紅進行染色。在載玻片上滴1滴蒸餾水,用接種環取一環菌體,均勻涂布于載玻片上,用火焰進行干燥固定,番紅染色1~2 min,經水洗、干燥,顯微鏡觀察微生物個體形態。
1.2.4 酵母菌分離株的分子鑒定
(1)葡萄酒中酵母菌分離株的DNA提取。葡萄酒中酵母菌基因組DNA的提取,按《分子生物學手冊》中常規方法的改進方法進行。
具體如下:取葡萄酒中酵母分離株的單個菌落接入YPD液體培養基中,以轉速200 r/min,溫度28℃培養過夜,取適量菌體,加入少量DNA提取緩沖液(100 mmol/L EDTA,100 mmol/L Tris,1.5 mol/L NaCl,pH值8.0) 于1.5 mL離心管中,加入纖維素酶和溶菌酶(終質量濃度分別為6,1 mg/mL),混合均勻后于37℃下水浴1.5~2 h。加入20%SDS100 μL,于65℃下水浴30~60 min,以轉速12 000 r/min離心10 min。取上清液加入等體積酚、氯仿和異戊醇混合液(體積比為25∶24∶1)并混勻,以轉速12 000 r/min離心10 min。取上清液并加入0.6倍體積的異丙醇,顛倒混勻,-20℃放置20 min,以轉速12 000 r/min離心10 min。棄上清液,用70%乙醇洗滌,以轉速12 000 r/min離心10 min。干燥后用50 μL ddH2O溶解,DNA樣品于-20℃下保存。
(2)26S rRNA基因的PCR擴增。以26S rRNA的F,R引物擴增提取的葡萄酒中酵母分離株的基因組DNA,PCR反應體系為10×2.5 μL,dNTP 2.0 μL,上、下游引物各1 μL,DNA模板1 μL,Taq酶0.2 μL,加ddH2O至25 μL。PCR擴增條件為94℃5 min,94℃45 s,54℃50 s,72℃60 s,做30個循環,于72℃下延伸10 min,PCR產物進行瓊脂糖凝膠電泳分析。
(3)目的片段的回收與純化。PCR產物進行2%瓊脂糖凝膠電泳,溴化乙錠(EB)染色。紫外燈下分別切下含有目的片段的凝膠塊,置于1.5 mL EP管中。利用DNA純化回收試劑盒對目的片段進行回收和純化,具體方法按照操作說明書進行。
(4)26S rRNA基因的序列分析。將已純化的PCR產物送至上海生工生物工程有限公司進行測序,得到菌株PCR擴增片段的基因序列。在GenBank核酸序列數據庫中進行同源序列搜索(BLAST search),比較分離株與已知微生物相應序列的同源性,確定分離株的分屬。
2 結果與分析
2.1 貝達葡萄酒中酵母菌的計數
對貝達葡萄酒樣品進行酵母菌的菌落總數測定,測得的酵母菌菌落總數為3.4×107CFU/mL。
2.2 酵母菌的形態學觀察結果
酵母菌的菌落特征為圓形、大而厚,菌落表面光滑、濕潤、黏稠,與培養基結合不緊密,容易挑起,菌落質地均勻,正反面和邊緣、中央部位的顏色都很均一,菌落為乳白色。
分離株Y3的菌落形態見圖1,分離株Y3的鏡檢形態見圖2。
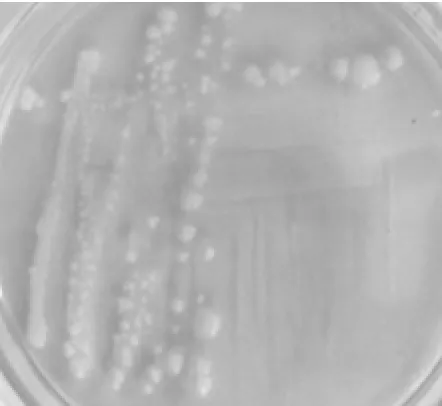
圖1 分離株Y3的菌落形態
酵母菌細胞呈球形、橢圓形,生殖方式為芽殖,無假菌絲,無色素;其他酵母菌分離株的菌落形態和鏡檢形態均與Y3相似。
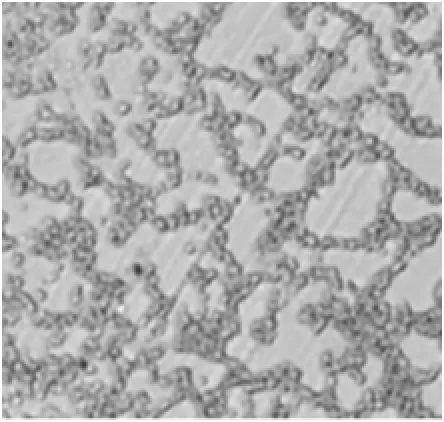
圖2 分離株Y3的鏡檢形態
2.3 葡萄酒中酵母菌的PCR擴增結果
對分離的3株酵母菌Y3,Y5,Y13進行了26S rRNA基因的PCR擴增。
酵母分離株的PCR擴增效果見圖3。
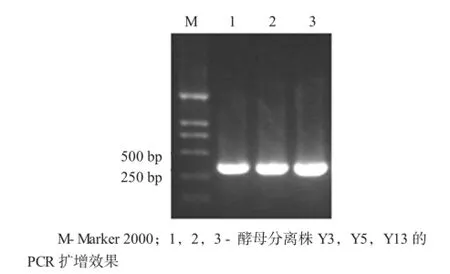
圖3 酵母分離株的PCR擴增效果
由圖3可知,獲得了與預期大小相符的PCR擴增產物,片段大小為300~400 bp。
2.4 PCR擴增產物的回收與純化
利用DNA純化回收試劑盒對PCR擴增片段進行回收和純化。
酵母分離株的PCR擴增產物純化結果見圖4。
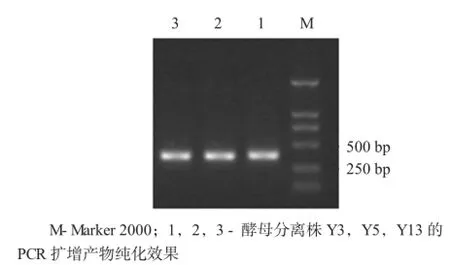
圖4 酵母分離株的PCR擴增產物純化結果
由圖4可知,獲得的目標產物與預期大小相符,片段大小為300~400 bp,無引物二聚體。
2.5 酵母分離株的26S rRNA序列分析
對酵母分離株的26S rDNA基因序列進行BLAST
同源性比較,代表菌株Y3的26SrRNA基因序列分析。酵母菌分離株Y3的序列分析結果見圖5。

圖5 酵母菌分離株Y3的序列分析結果
由圖5可知,酵母菌分離株Y3與釀酒酵母菌株的同源性為99%以上,可將Y3鑒定為釀酒酵母;Y5和Y13的序列分析結果與Y3相同,均為釀酒酵母。因此,說明貝達葡萄酒發酵后期的優勢菌即為釀酒酵母。
3 結論
葡萄酒的生產需要微生物的參與,在此過程中發生了一系列復雜的生物化學變化,使制品具有特定的風味和營養。酵母是葡萄酒發酵中后期的主導菌。本試驗對貝達葡萄酒中的酵母菌進行分離純化,通過形態學觀察和分子生物學方法對酵母分離株進行鑒定。結果顯示,貝達葡萄酒的酵母菌菌落總數為3.4×107CFU/mL,分離的酵母菌菌株Y3,Y5,Y13均為釀酒酵母,因此貝達葡萄酒發酵后期的優勢菌即為釀酒酵母。
[1]翟蘅,杜金華,管雪強,等.釀酒葡萄栽培及加工技術 [M].北京:中國農業出版社,2005:147-151.
[2]顧沛雯,張軍翔.葡萄酒酵母檢測方法研究 [J].葡萄釀酒,2005(3):38-40.
[3]蘇龍,劉樹文,何玲,等.東北山葡萄酒自然發酵酵母菌群的研究 [J].食品與生物技術學報,2007,26(3):110-115.
[4]劉慧.現代發酵微生物試驗技術 [M].北京:化學工業出版社,2005:60-67.
[5]劉小珍,張漢堯.昆明葡萄酒相關酵母菌的分離與鑒定 [J].西北農林科技大學學報(自然科學版),2014,42(5):78-81.
[6]朱麗霞,李紅梅,郭東起,等.新疆慕薩萊思自然發酵過程中酵母菌表型多樣性及優勢菌分析 [J].食品科學,2012,33(7):142-147.
[7]Dlauchy D,Tornai-Lehoczki J,Peter G.Restriction enzyme analysis of PCR amplified rDNA as a taxonomic tool in yeast identification[J].Systematic and Applied Microbiology,1999,22(3):445-453.◇
Separation and Identification of Yeast in Beida Grape Wine
XIE Junyun,YAO Di,GUO Yu,ZUO Zhaohang,ZHANG Wei
(College of Food Science,Heilongjiang Bayi Agricultural University,Daqing,Heilongjiang 163319,China)
In order to obtain a dominant yeast strains at later stage of grape wine fermentation,isolation and purification of yeast in beida grape wine is performed and colony morphology is identified.Then PCR amplification of 26S rRNA of yeast isolates is conducted,further identification by gene sequence analysis.Results show that yeast colony number in beida grape wine is 3.4×107CFU/mL,the yeast isolated strains Y3,Y5,Y13 are saccharomyces cerevisiae.Therefore,the advantage yeast in grape wine later fermentation is saccharomyces cerevisiae.
beida grape wine;yeast;identification
TS262.61
A
10.16693/j.cnki.1671-9646(X).2016.01.006
2015-11-12
謝俊云(1993— ),男,本科,研究方向為食品加工。

