長野芽孢桿菌普魯蘭酶的同源建模及三維結構分析
韋旭欽,李曉明,廖東慶,黃日波
(1. 南寧邦爾克生物技術有限責任公司,南寧 530003;2. 廣西大學生命科學與技術學院,南寧 530004;3. 廣西科學院,南寧 530007)
長野芽孢桿菌普魯蘭酶的同源建模及三維結構分析
韋旭欽1*,李曉明1,廖東慶1,黃日波2,3
(1. 南寧邦爾克生物技術有限責任公司,南寧 530003;2. 廣西大學生命科學與技術學院,南寧 530004;3. 廣西科學院,南寧 530007)
普魯蘭酶(Pullulanase)是脫支酶,因其能水解葡聚糖的α-1,6-糖苷鍵而有不同的工業應用潛力。本研究通過同源建模和分子對接的方法對長野芽孢桿菌(Bacillusnaganoensis) 普魯蘭酶進行建模及其三維結構分析,表明該酶由CBM41-X45a-X25-X45b-CBM48-GH13_14多結構域組成,酶蛋白中心形成其催化區,催化區的Asp619、Glu648和 Asp733三個殘基構成酶的催化三聯體。同時,通過柔性對接研究了酶與底物分子相互作用的關系,并預測構成酶的活性中心相關氨基酸殘基,為進一步改良酶的特性提供重要的理論依據。
長野芽孢桿菌;普魯蘭酶;同源建模;結構分析
普魯蘭酶(Pullulanase,EC 3.2.1.41)是一類水解α-1,6-糖苷鍵脫支酶,因其能專一性水解普魯蘭糖而得名,該酶可以水解支鏈淀粉的α-1,6-糖苷鍵而將淀粉分解為葡萄糖、麥芽糖、果糖等食用甜味劑;在以纖維素和淀粉質為原料的生物加工過程中,葡萄糖淀粉酶類水解直鏈的α-1,4-糖苷鍵,普魯蘭酶專門水解支鏈α-1,6-糖苷鍵,使支鏈淀粉型多糖的分支鏈脫離主鏈,形成一系列鏈長短不一的直鏈淀粉,可見普魯蘭酶和葡萄糖淀粉酶配合使用,可以顯著提高淀粉類原料的利用率,因此,普魯蘭酶在食品、紡織和生物能源等領域有重要的用途[1]。
不同來源的普魯蘭酶由于其活性低和穩定性差等特點,限制了該類酶的工業化應用。研究發現,長野芽孢桿菌普魯蘭酶(Bacillusnaganoensispullulanase,BnPulB)的最適反應溫度為62.5 ℃,在pH為4.5~5.0范圍內保持90%的酶活性,這些酶學特性與淀粉糖化過程較高的溫度(55~65 ℃)和微酸性環境(pH 4.5~5.5)相符合,表明長野芽孢桿菌普魯蘭酶有更大的工業應用潛力[2]。
同源建模法(Homology model)是從蛋白質的氨基酸序列出發預測其三維模型的常用方法,為研究未知結構蛋白質的功能提供基礎,如果目的蛋白與模板蛋白的序列同一性大于60%,同源建模的結果將接近實驗測試的結果[3]。為了進一步提高酶的活性和穩定性等特性,本研究通過對長野芽孢桿菌普魯蘭酶進行同源建模及其結構分析,并預測其活性部位和功能位點,為改良酶的性能提供理論參考。
1 材料與方法
1.1 序列來源
長野芽孢桿菌普魯蘭酶BnPulB的蛋白質序列來源于NCBI,該酶是由926個氨基酸殘基組成的多肽,其GenBank登陸號為AEV53626,本研究所采用的6個普魯蘭酶序列及其晶體結構數據(見表1)取自蛋白質數據庫(Protein Data Bank, PDB)[4],序列分析利用DiAlige服務器(http://www.genomatix.de/)進行[5]。
1.2 同源建模與結構分析
將BnPulB蛋白質序列提交到在線服務器SWISS-MODEL(http://swissmodel.expasy.org/)進行自動模式搜索和同源建模[6],用PyMol生物軟件對其模型進行結構分析和作圖。
1.3底物對接和活性位點分析
用Autodock 4.2生物軟件對普魯蘭酶BnPulB與底物進行柔性對接及其活性位點分析[7]。
2 結果與分析
2.1BnPulB的同源建模
通過在線服務器SWISS-MODEL自動模式搜索(各參數為默認值),發現BnPulB與普魯蘭酶家族的有不同程度的序列同一性(見表1,各酶的PDB編號分別為2WAN、3WDI、2E9B、2YA0、2YOC和2FHC,aa表示蛋白序列的氨基酸殘基個數),其中它與嗜酸普魯蘭芽孢桿菌普魯蘭酶(Bacillusacidopullulyticuspullulanase,BaPul13A)蛋白質序列同一性達64%,以BaPul13A三維結構(PDB:2WAN,1.65?)為模板[8],對BnPulB進行同源建模,得到N端和C端分別缺失108和8個氨基酸的并由其Pro109~Gln918區間氨基酸肽段組成的分子量更小的結構,其QMEAN值僅為-0.01,加上BnPulB蛋白質序列與模板BaPul13A的同一性很高,可見BnPulB同源建模的結果接近實驗測試的結果。
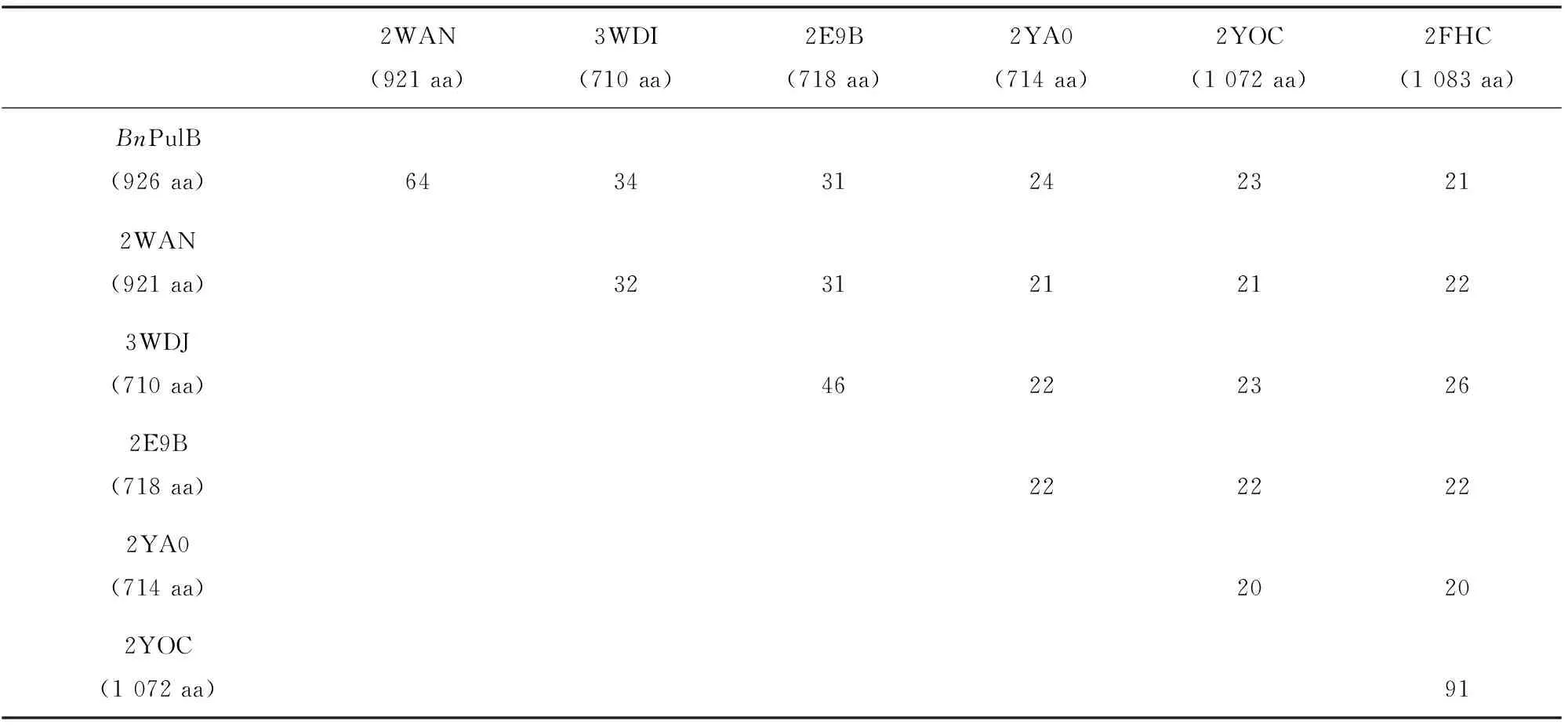
表1 BnPulB與6個有晶體結構的普魯蘭酶序列同一性Table 1 Pairwise sequence identity of BnPulB and 6 known crystal structure pullulanases %
2.2 BnPulB結構分析
將BnPulB模型與BaPul13A的進行整體結構比對分析,發現這兩個酶各殘基的Ca原子空間坐標幾乎完全重疊,其總體RMSD 值僅為0.050?,表明BnPulB結構與BaPul13A的結構非常相似,兩者的整體結構幾乎重疊在一起(見圖 1)。
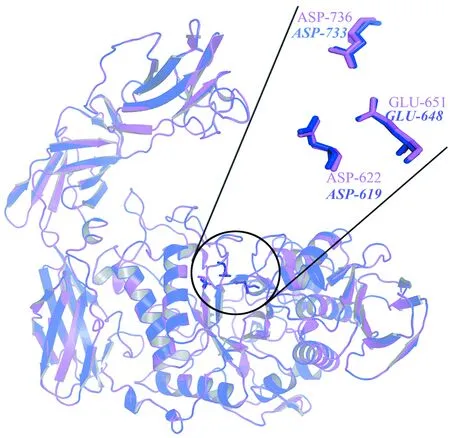
圖 1 普魯蘭酶BnPulB與BaPul13A結構重疊分析*Fig.1 Structural superposition of pullulanase BnPulB and BaPul13A
*注:圖中BnPulB的結構為藍色,BaPul13A 結構(PDB編號:2WAN)為粉紅色,BaPul13A催化三聯體為Asp622、Glu651 和 Asp736三個氨基酸殘基,對應BnPulB的分別為Asp619、Glu648 和 Asp733.
彩圖見電子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第1期DOI:10.3969/j.issn.1672-5565.2017.01.201606002)。
將BnPulB模型與表1中所述的6種普魯蘭酶同時進行結構比對(見圖2),由于它們的序列長度和序列本身存在較大差異(見表1),它們的整體結構并不完全重疊(見圖2(a)),但它們的活性中心的結構(底物結合口袋)卻非常相似(見圖2(b)),其氨基酸殘基高度保守,都由空間坐標幾乎重疊的Asp、Glu和Asp三個殘基構成催化三聯體(見圖2(c)),表明不同來源的普魯蘭酶在漫長的家族進化過程中存在未知的可變性,它們整體的序列同一性可能很低,但其催化區是高度保守的,符合蛋白質生物進化的基本規律,可見長野芽孢桿菌普魯蘭酶的催化區同樣具有普魯蘭酶家族共同的結構特點。
已有研究表明,BaPul13A是由Asp622、Glu651和Asp736三個氨基酸殘基構成其催化三聯體,在CAZY GH13糖苷酶家族中,這三個位于催化區的氨基酸殘基非常保守,在酶的催化過程中,Asp622充當進攻底物的親核基團,Glu651作為廣義的酸堿催化劑,Asp736對酶-底物復合物的過度態起到穩定性的作用[8]。通過結構比對分析,可以預測,構成對應的長野芽孢桿菌BnPulB催化三聯體是由Asp619、Glu648和Asp733三個氨基酸殘基構成(如圖1和圖2(c)所示),這三個殘基與表1所述的6種同類普魯蘭酶催化三聯體的空間坐標大致重疊(見圖2(c)),因此這三個殘基在酶的催化過程中可能起到與BaPul13A催化三聯體相似的關鍵作用。

圖 2 BnPulB與6種普魯蘭酶的結構比對分析*Fig.2 Structural superposition of BnPulB with 6 known pullulanases
注:圖中BnPulB (藍色)、2WAN(粉紅色)、3WDI (紅色)、2E9B (青色)、2YA0 (黃色)、2YOC (紅色)、2FHC (灰色)。
彩圖見電子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第1期DOI:10.3969/j.issn.1672-5565.2017.01.201606002)。
BaPul13A是由921個氨基酸殘基組成CBM41-X45a-X25-X45b-CBM48-GH13_14多結構域的蛋白,其中X45a-X25-X45b 的功能未知,CBM48為糖原結合結構域,催化結構域GH13_14屬于 CAZY GH13家族,其N端1~100氨基酸為功能未明的結構域CBM41,CBM41高度無序而無法模擬其結構[7]。Chen等[9]最新研究表明,刪除BaPul13A酶的CBM41結構域,能有效地提高該酶的可溶表達和分泌水平,突變酶的活力是野生酶的2.9倍,并且其作用的溫度和pH與野生酶的相當,由此可見CBM41結構域是酶分子上冗余的元件,刪除該結構域的突變酶更適合工業化應用。因此,可以推斷,長野芽孢桿菌普魯蘭酶BnPulB對應的N端1~108氨基酸可能是酶分子上冗余的序列,刪除這個結構可能得到分子量更小的結構更穩定的突變酶。
Wang等[10]近期研究發現,刪除長野芽孢桿菌BnPulB酶N端前106個氨基酸殘基,或C端9個,或C端36個殘基的突變體,突變酶的催化溫度和pH沒有改變,突變酶對底物親和力和催化效率有不同程度的改觀,與本研究預測的結果基本相符。
通過結構比對分析,BnPulB各結構域在酶分子上的氨基酸區間分布如圖3(a)所示,各結構域在酶空間結構的分布如圖3(b)所示,其中X45a、X25和X45b結構域遠離酶的催化區(GH13_14結構域),它們可能有助于結合更大分子的底物,與酶的結構穩定性有關;CBM48是糖原結構域,它和催化區的GH13_14結構域鄰近,這兩個結構域的結合維持了這個脫支酶的基本功能。BnPulB的催化三聯體位于GH13_14結構域,底物位于該結構域的深溝中,GH13_14結構域與酶的催化作用密切相關,改變這個結構域的相關氨基酸可能會明顯影響酶的活性和催化穩定性。Chen等[11]將BaPul13A酶位于GH13_14結構域的3個氨基酸殘基同時突變(E518I-S662R-Q706P),突變體酶在60 ℃的半衰期是野生型的11倍,最適反應溫度也從60 ℃提升到65 ℃。由于與BaPul13A酶的空間結構非常相似,若將BnPulB空間位置對應的氨基酸殘基(E515、A659和L703)進行相應的突變,也可能得到相應性能更穩定的突變酶。這種通過比對結構類似的(關鍵位點的氨基酸殘基存在差別),并經過突變其相應關鍵位點氨基酸殘基來影響酶功能的方法(類比差別法),將為酶的分子改良開辟一條新的途徑。
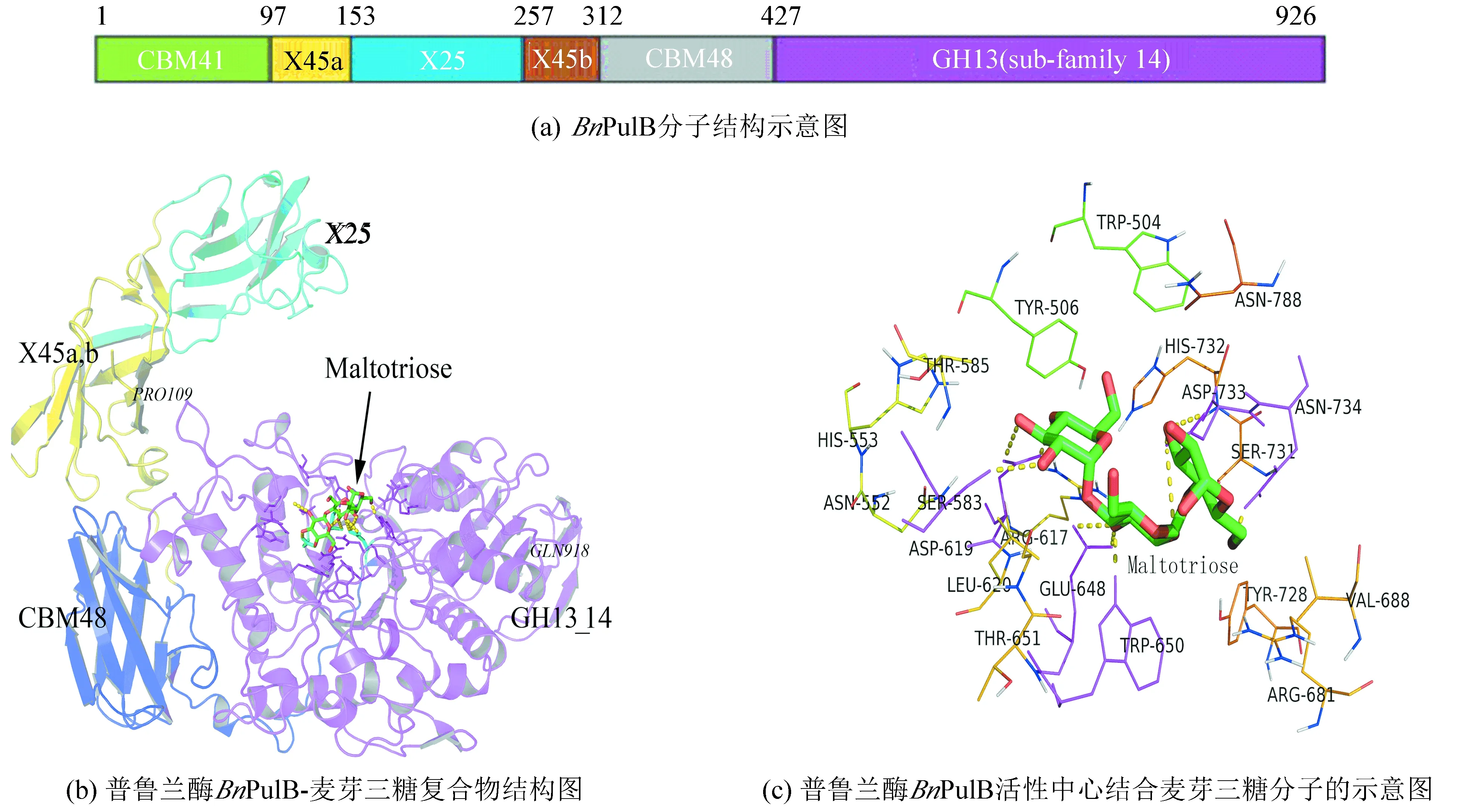
圖 3 長野芽孢桿菌普魯蘭酶(BnPulB)的三維結構圖*Fig.3 3-D structure of the Bacillus naganoensis pullulanase(BnPulB)
注:圖(b)的BnPulB結構用卡通圖形顯示,結構域X45為黃色,X25的為青色,CBM48為藍色,GH13_14的為粉紅色,麥芽三糖分子為棍棒模型;圖(c)活性中心鄰近氨基酸殘基以繩形顯示,麥芽三糖(棍棒模型)與氨基酸殘基(粉紅色)形成的氫鍵以虛線顯示。
*彩圖見電子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第1期DOI:10.3969/j.issn.1672-5565.2017.01.201606002)。
2.3底物對接
用軟件Autodock 4.2將底物與酶分子進行對接,將BnPulB的催化三聯體Asp619、Glu648和Asp733設定為柔性殘基,經分析得到這三個氨基酸殘基的中心坐標,將Gridbox中心設定為X:39.21,Y:37.82,Z:-29.32,將Gridbox大小設定為30×52×32(單位?),格點間距為默認值0.375 ?,對接過程選擇Lamarckian genetic algorithm (LGA)遺傳算法,其他條件均為默認值。由于普魯蘭糖是由α-1,4-糖苷鍵連接的麥芽三糖重復單位經α-1,6-糖苷鍵聚合而成的分子量達2 萬~200 萬之間直鏈狀多糖,本研究以構成普魯蘭糖單元的麥芽三糖為配體,麥芽三糖分子從厭氧芽孢桿菌 LM18-11(Anoxybacillussp. LM18-11)的同類普魯蘭酶晶體結構(PDB:3WDI)中提取[12]。
分子對接結果見圖3(b),底物分子深埋在酶分子的GH13_14結構域空穴中,底物結合口袋由多個氨基酸殘基組成,其結合自由能為-2.75 kcal/mol,抑制劑常數Ki=9.58 mM,底物分子的成功對接,為研究酶與底物的相互作用,研究酶的活性中心及其分子改良奠定了理論基礎。
2.4 活性中心分析
根據底物對接的結果,表明構成BnPulB的底物結合區和催化區的結構非常復雜,如圖3(c)所示,由Trp504、Try506、Asn552、His553、Ser583、Thr585、Arg617、Asp619、Leu620、Glu648、Trp650、Thr651、Arg681、Val688、Tyr728、Ser731、His732、Asp733、Asn734和Asn788 20個氨基酸殘基組成酶的活性中心,其中催化區直接與麥芽三糖形成的氫鍵的有Ser583、Asp619、Glu648、Trp650、Asp733和Asn734 6個關鍵氨基酸殘基,其催化三聯體由Asp619、Glu648和Asp733 3個氨基酸殘基組成,這3個殘基在同類酶中高度保守(見圖2(c)),直接突變這些位點,可能使酶失去活性,因此,可以考慮對構成其活性中心的其他非形成氫鍵的殘基著手,進行適當的突變來優化其底物結合口袋和催化區的結構,從而提高酶的催化效率;也可以通過研究與活性中心鄰近(5~15 ?)的氨基酸殘基來優化酶的構象或微環境來改善酶的特性[13]。
3 結論與討論
本研究對長野芽孢桿菌普魯蘭酶BnPulB進行同源建模,構建了BnPulB蛋白的三維結構。通過理論模擬表明,該普魯蘭酶具有CBM41-X45a-X25-X45b-CBM48-GH13_14多結構域的結構,其催化區位于GH13_14結構域,它的催化三聯體由Asp619、Glu648和Asp733三個氨基酸殘基組成,符合已知同類普魯蘭酶結構的相關特性;酶與底物對接及其活性中心的預測和分析表明,普魯蘭酶家族的底物結合口袋非常相似,BnPulB與底物形成氫鍵的氨基酸殘基為Ser583、Asp619、Glu648、Trp650、Asp733和Asn734,突變這些殘基可能使酶失去活性,所以在分子改良時,可考慮對BnPulB活性中心沒有形成氫鍵的殘基著手來改良酶的特性。本文的研究方法具有操作簡單和直觀易懂等特點,理論成果對酶的分子改良有一定的參考意義,其不足之處是在同類酶非保守區或非同源性蛋白的預測可能存在較大偏差,其研究方法和理論成果還有待更多的實驗驗證。
References)
[1]GUZMAN-MALDONADO H, PAREDES-LOPEZ O. Amylolytic enzymes and products derived from starch: A review [J].Critical Reviews in Food Science and Nutrition,1995,35(5): 373-403.DOI: 10.1080/10408399509527706.
[2]嚴偉, 聶堯, 徐巖. 長野芽孢桿菌(Bacillusnaganoensis)普魯蘭酶在大腸桿菌中的活性表達與分泌調控 [J]. 微生物學報,2013,53(2): 145-153.
YAN Wei,NIE Yao,XU Yan. Expression and secretion regulation ofBacillusnaganoensispullulanase in recombinantEscherichiacoli[J].Acta Microbiologica Sinica,2013,53(2): 145-153.
[3]KACZANOWSKI S, ZIELENKIEWICZ P. Why similar protein sequences encode similar three-dimensional structures? [J].Theoretical Chemistry Accounts,2010(125): 543-550.DOI: 10.1007/s00214-009-0656-3.
[4]BERMAN H M, WESTBROOK J, FENG Z, et al. The protein data bank [J]. Nucleic Acids Research,2000,28(1): 235-242.
[5]MORGENSTERN B, FRECH K, DRESS A, et al. Dialign: Finding local similarities by multiple sequence alignment [J].Bioinformatics,1998,14(3): 290-294.DOI: 10.1093/bioinformatics/14.3.290.
[6]SCHWEDE T, KOPP J, GUEX N, et al. Swiss-model: An automated protein homology-modeling server [J].Nucleic Acids Research,2003,31(13): 3381-3385.DOI: 10.1093/nar/gkg520.
[7]MORRIS G M, HUEY R, LINDSTROM W, et al. Autodock4 and autodocktools4: Automated docking with selective receptor flexibility [J].Journal of Computational Chemistry,2009,30(16): 2785-2791.DOI: 10.1002/jcc.21256.
[8]TURKENBURG J P, BRZOZOWSKI A M, SVENDSEN A, et al. Structure of a pullulanase fromBacillusacidopullulyticus[J].Proteins Structure Function &Bioinformatics,2009,76(2): 516-519.DOI: 10.1002/prot.22416.
[9]CHEN A N, SUN Y, ZHANG W, et al. Downsizing a pullulanase to a small molecule with improved soluble expression and secretion efficiency inEscherichiacoli[J].Microbial Cell Factories,2016,15(9):1-10.DOI: 10.1186/s12934-015-0403-5.
[10]WANG X Y, NIE Y, MU X Q, et al. Disorder prediction-based construct optimization improves activity and catalytic efficiency ofBacillusnaganoensispullulanase [J].Scientific Reports,2016,6(24574):1-9.DOI: 10.1038/srep24574.
[11]CHEN A N, LI Y M, NIE J Q, et al. Protein engineering ofBacillusacidopullulyticuspullulanase for enhanced thermostability using in silico data driven rational design methods [J].Enzyme and Microbial Technology,2015(78): 74-83.DOI: 10.1016/j.enzmictec.2015.06.013.
[12]XU J Y, REN F F, HUANG C H, et al. Functional and structural studies of pullulanase fromAnoxybacillussp. LM18-11[J]. Proteins Structure Function &Bioinformatics,2014,82(9): 1685-1693.DOI: 10.1002/prot.24498.
[13]MORLEY K L, KAZLAUSKAS R J. Improving enzyme properties: When are closer mutations better? [J]. Trends in Biotechnology,2005,23(5): 231-237.DOI: 10.1016/j.tibtech.2005.03.005.
Homology modeling and three-dimensional structure analysis ofBacillusnaganoensispullulanase
WEI Xuqin1*, LI Xiaoming1, LIAO Dongqing1, HUANG Ribo2,3
(1.GuangxiNanningBiocloneBiotechnologyCo.,Ltd.,Nanning530003,China;2.CollegeofLifeScienceandTechnology,GuangxiUniversity,Nanning530004,China;3.GuangxiAcademyofSciences,Nanning530007,China)
Pullulanase is a debranching enzyme that specifically hydrolyzes the α-1,6-glycosidic bonds in complex carbohydrates and has great potential in various industries. Here, we investigate the structural characteristics of theBacillusnaganoensispullulanase (BnPulB) by homology modeling and molecule docking. Results show that theBnPulB structure comprises the CBM41-X45a-X25-X45b-CBM48-GH13_14 multi-domain architecture and the central region of the protein forms the catalytic domain, and the highly conserved Asp619, Glu648, and Asp733 residues are identified to be catalytic triad. In addition, flexible docking studies of the enzyme-substrate system show the interactions betweenBnPulB and its substrate, maltotriose, and some conserved residues locate in the active centre that participate in ligand binding site are predicted. This study may provide important information for the design of new pullulanase with novel properties.
Bacillusnaganoensis;Pullulanase; Homology modeling; Structure analysis
2016-06-18 ;
2016-09-08.
南寧市科技型中小企業技術創新基金資助項目 (20146371)。
10.3969/j.issn.1672-5565.2017.01.201606002
Q514
A
1672-5565(2017)01-027-06
*通信作者:韋旭欽,男,理學博士,高級工程師,研究方向:微生物發酵與分子酶工程;E-mail:xqweigxu@163.com.

