大葉石上蓮ISSR—PCR反應體系的建立與優(yōu)化
傅瓊+王英強
摘 要 為建立并優(yōu)化大葉石上蓮(Oreocharis benthamii)ISSR-PCR反應體系,采用改良的CTAB法提取大葉石上蓮葉片DNA,利用正交試驗設計方法,從Mg2+、dNTP、引物、Taq DNA聚合酶以及模板DNA 5因素4水平,對大葉石上蓮 ISSR-PCR 反應體系進行優(yōu)化,確立了適用于大葉石上蓮的擴增多態(tài)性高、穩(wěn)定性強、條帶清晰的ISSR最佳反應體系:2.0 μL的10×PCR 緩沖液,2.0 mmol/L Mg2+,0.25 mmol/L dNTP,0.7 μmol/L 引物,2.0 U Taq DNA聚合酶,30 ng DNA模板(20 μL反應體系)。在此基礎上,從100條引物中篩選出13條ISSR擴增引物,其中5條引物最為合適并已確定其最佳退火溫度。
關鍵詞 大葉石上蓮 ;ISSR ;正交設計 ;反應體系 ;引物篩選
中圖分類號 S339.1 文獻標識碼 A Doi:10.12008/j.issn.1009-2196.2017.05.006
Optimization and Establishment of ISSR-PCR Reaction System for
Oreocharis benthamii
FU Qiong WANG Yingqiang
(Guangdong Key Laboratory of Biotechnology for Plant Development, College of Life Sciences,
South China Normal University, Guangzhou, Guangdong 510631)
Abstract An attempt was made to establish and optimize a stable ISSR-PCR reaction system for Oreocharis benthamii. The leaf DNA of O. benthamii was extracted by using the modified CTAB protocol. An orthogonal design was arranged to optimize ISSR-PCR amplification system with five factors (Mg2+, dNTP, primer, Taq DNA polymerase and DNA template) at four levels, based on which a satisfactory ISSR-PCR reaction system for O. benthamii with high polymorphism, desirable repeatability and clear band patterns was established. This system was a total volume of 20 μL ISSR-PCR system that contained 2.0 μL 10×PCR buffer, 2.0 mmol/L Mg2+, 0.25 mmol/L dNTP, 0.7 μmol/L primer, 2.0 U Taq DNA polymerase, and 30 ng DNA template. With this reaction system 13 ISSR primers were screened from 100 primers, of which five primers were found optimum and their annealing temperatures were proposed.
Keywords Oreocharis benthamii ; ISSR ; orthogonal design ; reaction system ; primers screening
大葉石上蓮(Oreocharis benthamii)是苦苣苔科馬鈴苣苔屬(Oreocharis)的一種多年生草本植物,為我國華南地區(qū)特有物種,一般生長在石灰?guī)r密林下陰濕處,生境相對特殊,其分布范圍以廣東、廣西的華南地區(qū)最為集中[1-2]。苦苣苔科植物為我國南方卡斯特生境的代表性植物類群,其生境十分脆弱[3]。長期以來,大面積的天然林被砍伐,資源被過度開發(fā)利用,環(huán)境問題日益突出等使植物賴以生存的環(huán)境遭到嚴重破壞,而對生境要求極為苛刻的大葉石上蓮來說,生境一旦被破壞即難以恢復。目前苦苣苔科的大多數種類處于瀕危狀態(tài),而生境明顯退化是該科植物瀕危的主要原因[4-5]。以往關于大葉石上蓮的研究主要集中在CYC花對稱基因表達[6]、繁育系統(tǒng)[7]、系統(tǒng)學[8-9]等方面。然而隨著分子生物學技術的發(fā)展,利用以PCR為基礎的分子標記,加快并深入對大葉石上蓮的遺傳多樣性、遺傳結構等研究和保護工作是當務之急。
ISSR(簡單序列重復區(qū)間多態(tài)性,Inter Simple Sequence Repeat)分子標記是Zietkeiwitcz[10]等在1994年對微衛(wèi)星技術發(fā)展起來的一種分子標記,可用來檢測兩段SSR之間一段短DNA序列的多態(tài)性。相對于AFLP[11]、RAPD[12]、SSR[10]等分子標記,該技術雖然為顯性標記,不能區(qū)分純合體和雜合體,但是其多態(tài)性高、操作簡便、成本低。目前該技術已被廣泛應用于遺傳多樣性檢測[13-14]、種質資源鑒定[15-16]、遺傳圖譜構建[17]等方面研究。本文根據正交試驗設計方法,從5因素(Mg2+、dNTP、引物、Taq DNA聚合酶以及模板DNA)4水平對大葉石上蓮ISSR-PCR反應體系進行優(yōu)化,以期獲得大葉石上蓮的最佳ISSR擴增反應,為下一步大葉石上蓮種質資源的遺傳多樣性研究提供可靠的技術支持。
1 材料與方法
1.1 材料
實驗所需材料采自廣東、廣西等地的大葉石上蓮。每個樣品選取植株中幼嫩葉片(1~2片),清洗并迅速放入茶葉袋,加硅膠快速干燥,封口保存并放入-70℃超低溫冰箱備用。采集標本放置華南師范大學生命科學學院標本館。
用于ISSR-PCR反應的PCR Buffer、Taq DNA聚合酶、dNTP、Mg2+均購自 TaKaRa公司。標準分子量(Marker)DL15 000和DL100購于廣東東盛生物科技有限公司。所用引物采用加拿大哥倫比亞大學設計公布的引物序列,由上海生工生物工程公司合成。經初步篩選,引物UBC841[(GA)8Y*C;*Y代表G/C]作為此次正交試驗的引物。GoldViewTM核酸染料購自北京賽百盛基因技術有限公司。PCR反應在美國BIO-RAD公司溫度梯度PCR循環(huán)儀上進行。葉片研磨過程在冷凍混合球磨儀MM400(德國Retsch公司)中進行。
1.2 方法
1.2.1 總DNA的提取
根據鄒喻蘋[18]的CTAB法,并加以改進,提取大葉石上蓮基因組DNA,利用紫外可見分光光度計(ALLSHENG: Nano-100)檢測 DNA的純度和濃度。用0.8%瓊脂糖凝膠電泳檢測DNA質量,并將其濃度稀釋至25 ng/μL,置于-20℃儲存?zhèn)溆谩?/p>
改良CTAB法:(1)研磨后加預處理液(2% β-巰基乙醇;6% PVP;洗液(0.2 mol/L Tris-HCl pH 8.0,50 mmol/L EDTA,pH=8.0,0.25 mol/L NaCl)震蕩混勻,放置-20℃ 10 min,12 000 r/min,4℃。(2)取出研磨珠;(3)CTAB提取液(2%的CTAB,pH 8.0的100 mmol/L Tris-HCl,20 mmol/L EDTA,1.4 mol/L NaCl,2% β-巰基乙醇)。
1.2.2 PCR擴增
基于已有的研究[19],本文確定的擴增程序為:94℃預變性5 min,94℃變性30 s,46~60℃退火45 s,72℃延伸90 s,共40個循環(huán),后72℃延伸10 min,4℃低溫保存。1.8%瓊脂糖凝膠電泳檢測擴增產物。
1.2.3 正交試驗設計
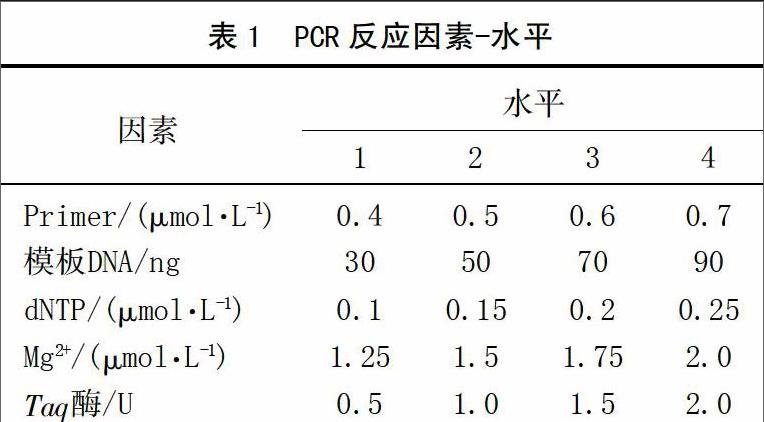
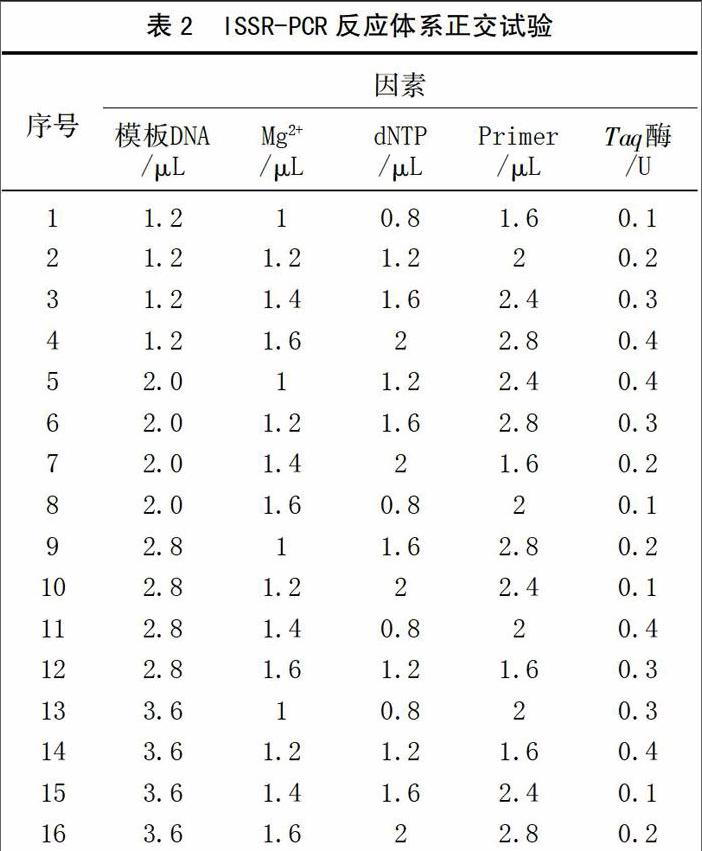
根據邵果園[19]大巖桐反應體系,本文設計L16(45)正交試驗因素及水平,其中5個因素和4個試驗水平見表1,具體試驗組合見表2。ISSR試驗總體積采用20 μL反應體系,每個反應管內加有1 × PCR Buffer,并用無菌水補至20 μL。將19個處理[另3管為陰性對照(不加模板)]各試驗2次。引物UBC 841的退火溫度根據其Tm值最初確定為56.3℃。參照何正文[20]等的方法,依據擴增條帶的敏感性、特異性及穩(wěn)定性即條帶的強弱及雜帶的多少做1~16分計分,分數越高則表明敏感性、特異性和穩(wěn)定性越好,2次重復分別統(tǒng)計。
1.2.4 引物篩選及最佳退火溫度的確定
從5個地理距離相距較遠的居群中分別選取2個DNA模板,利用優(yōu)化的反應體系從100條ISSR引物中進行擴增篩選。再根據PCR儀器自動形成的12個退火溫度梯度(46、46.4、47.2、48.3、49.9、52、54.3、56.3、57.8、58.9、59.7、60℃),從而最終確定每條引物的最佳退火溫度。
1.2.5 體系穩(wěn)定性檢測
根據1.2.4篩選得到的條帶清晰、穩(wěn)定性強、多態(tài)性高的引物,隨機選擇其中的引物和不同居群的樣品進行ISSR-PCR擴增,檢測優(yōu)化體系在不同居群的穩(wěn)定性。
2 結果與分析
2.1 DNA濃度與純度檢測
采用改進的CTAB法提取的大葉石上蓮DNA OD值A260 nm/A230 nm介于1.80~2.05,表明所提取的DNA純度較好(表3)。并且從瓊脂糖凝膠電泳結果也能夠看出,改良CTAB法所提取的DNA條帶更加明亮,并具有單一的、較完整的高分子量條帶,且加樣孔沒有殘留的雜質(圖1)。無論是在DNA純度還是濃度上,改進的CTAB法比傳統(tǒng)的CTAB法效果更佳,可用于后續(xù)ISSR-PCR反應。
2.2 PCR反應體系的優(yōu)化結果
大葉石上蓮ISSR-PCR正交試驗的電泳結果(圖2)表明,處理3、4、5、11、12、13、14、15擴增條帶較多。其中以處理4、11、12的條帶更為清晰,主帶更加明顯。因此,這3個處理在大葉石上蓮ISSR-PCR擴增反應中效果較好。
參照何正文[20]的方法,對擴增結果依次計分:7、7;7、4;14、14;16、16;12、10;6、11;5、5;7、7;2、5;2、2;14、14;14、14;10、10;12、12;11、11;2、5。根據分數求出每個因素在同一水平下的試驗值之和的平均值(ki),并求出同一因素不同水平間平均值的極差(R)(表4)。極差R越大,代表影響因素對反應體系的影響情況越顯著。由表4可知,Taq酶濃度對大葉石上蓮ISSR-PCR反應的影響最大,其次為Mg2+和dNTP,DNA 模板和引物的作用較小。5個因素的最佳水平并未在處理組合中出現,但分值最高的4號處理與其較接近,并且4號處理的3個較大影響因素的濃度,與最佳水平相近,考慮到節(jié)約DNA模板,最終確定大葉石上蓮的最佳反應體系為4號處理,即在20 μL反應體系中包括2.0 μL的10×PCR緩沖液,2.0 mmol/L Mg2+,0.25 mmol/L dNTP,0.7 μmol/L引物,2.0 U Taq DNA聚合酶,DNA模板30 ng。
2.3 引物篩選及退火溫度的確定
根據最佳反應體系,從100條ISSR引物中共篩選出13條引物。其中5條引物具有更強的重復性、穩(wěn)定性和條帶清晰性,并對其進行梯度溫度退火實驗,確定最佳退火溫度(表5)。對引物UBC 879的退火溫度進行梯度篩選,結果(圖3)表明,退火溫度低于49.9℃時,亮度低,且主帶有彌散;隨著49.9℃退火溫度的升高,引物與模板結合差,擴增條帶少且模糊,主條帶不明顯。當退火溫度為49.9℃時,條帶清晰,主條帶明顯、亮度高且穩(wěn)定性強。所以,引物UBC 879的最佳退火溫度為50℃。
2.4 最優(yōu)體系的檢驗
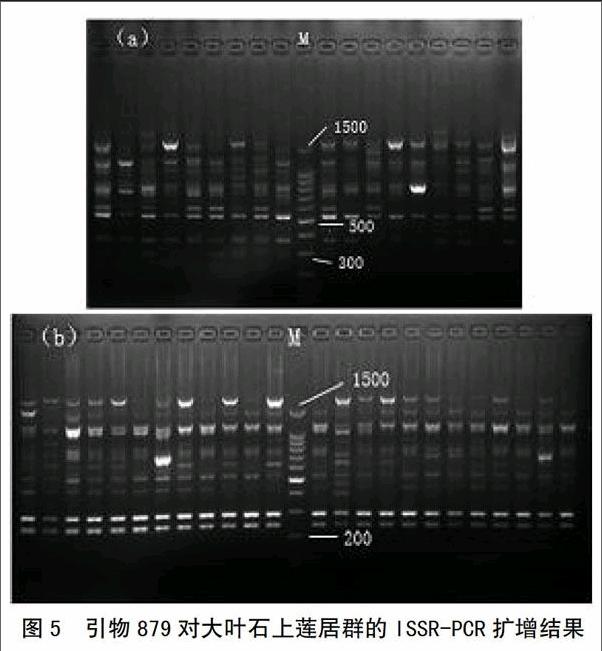
利用最佳的反應體系對大葉石上蓮2個居群(共42個個體)進行ISSR-PCR擴增,結果顯示該擴增反應體系穩(wěn)定性強、條帶清楚、多態(tài)性高,因而此反應體系可適用于大葉石上蓮的遺傳多樣性分析(圖4、5)。
3 結論與討論
DNA提取是進行分子生物學研究的重要步驟,而高質量、高濃度的DNA不僅有利于下游實驗進行,而且可以減少DNA的浪費[21-22]。有研究表明,當A260/A280小于1.8時,DNA中可能有蛋白質污染,當A260/A280大于2.0時,DNA中可能有RNA污染[23],而當A260/A230低于2.0時,DNA中可能有碳水化合物、鹽、酚類等污染[24]。從大葉石上蓮提取的DNA純度(A260/A280、A260/A230)和濃度來看(表3,圖1),傳統(tǒng)CTAB法提取效果均低于改良CTAB法。這可能是大葉石上蓮葉片本身含有較高的多糖、多酚等物質,而利用傳統(tǒng)CTAB法提取大葉石上蓮DNA,其多糖、多酚不能去除干凈,這些殘留的物質與DNA 共沉淀,形成粘稠的膠狀物,難以溶解以及產生褐變[21]。但利用改進的CTAB法提取大葉石上蓮DNA,在細胞膜破碎前的研磨過程中加入少量的PVP,并在預處理液和CTAB中加入適量的β-巰基乙醇,這種做法可有效地抑制多酚的氧化反應,防止褐化,同時能夠有效的去除多糖,并且還具有較高的DNA濃度。在加入CTAB之前將研磨鋼珠取出,可有效避免因機械碰撞而導致DNA斷裂的后果。
ISSR-PCR擴增反應容易受多重因素的影響,例如:Taq DNA聚合酶、Mg2+、dNTP、引物退火溫度等[25-26]。許多研究表明,影響ISSR-PCR反應的最大因素是Taq DNA聚合酶[27]和Mg2+[28],本研究結果與其具有一致性。在PCR反應過程中,如果Taq酶用量過高,將會導致擴增條帶彌散,積累大量的非特異性條帶,并且也提高了實驗成本[27]。適合的Mg2+濃度也非常重要,低的Mg2+濃度會降低Taq酶的活性,從而使反應產物減少。并且Mg2+還能與反應中其他因素結合,影響PCR的效應及特異性[28]。然而在本研究中,dNTP也表現出較大的影響性,由于dNTP是PCR的原料,其濃度太高產生錯誤摻入,濃度太低產率太低[18]。本研究中模板DNA的用量對PCR擴增的影響較小,可能是因為本研究所選擇的濃度范圍恰好在可影響區(qū)間內,但是其純度對擴增效果具有較大的作用。當模板DNA純度較高時,DNA用量少且能擴增出較好的結果,而純度較低時,DNA樣品中的多糖、多酚及蛋白質等都會干擾擴增反應。除了以上因素,引物的退火溫度對擴增反應的影響也相當重要,不同的引物在同一物種內具有不同的最佳退火溫度,而同一引物在不同的物種中也具有不同的最佳退火溫度。
綜上所述,一個適宜的ISSR-PCR擴增反應體系受到多重方面的影響。本研究結果表明,通過正交設計試驗方法優(yōu)化得出的大葉石上蓮ISSR-PCR擴增反應體系具有較高的穩(wěn)定性,可用于今后大葉石上蓮種質資源遺傳多樣性研究。
參考文獻
[1] 王文采,潘開玉,張志耘. 中國植物志:第69卷[M]. 北京:科學出版社,1990:165-166.
[2] 李振宇,王印政. 中國苦苣苔科植物[M]. 鄭州:河南科學技術出版社,2004:45.
[3] 吳征鎰,孫 航,周浙昆,等. 中國植物區(qū)系中的特有性及其起源和分化[J]. 云南植物研究,2005,27(6):577-604.
[4] Wang H, Zhang B, Cheng Y, et al. Genetic diversity of the endangered Chinese endemic herb Dayaoshania cotinifolia(Gesneriaceae)revealed by simple sequence repeat (SSR) markers[J]. Biochemical Systematics and Ecology, 2013, 48(2): 51-57.
[5] 楊文光,儲嘉琳,張耀廣,等. 中國苦苣苔科植物瀕危狀況評估分析[J]. 河南農業(yè)大學學報,2014,48(6):476-456.
[6] Zhi-Yan D U, Wang Y Z. Significance of RT-PCR expression patterns of CYC-like genes in Oreocharis benthamii (Gesneriaceae) [J]. Journal of Systematics and Evolution, 2008, 46(1): 23-31.
[7] 郭艷峰. 后蕊苣苔屬繁育系統(tǒng)的研究[D]. 廣州:華南師范大學,2011.
[8] Moeller M, Middleton D, Nishii K, et al. A new delineation for Oreocharis incorporating an additional ten genera of Chinese Gesneriaceae[J]. Phytotaxa, 2011, 23(1): 1-36.
[9] 馮翠元. 藥用植物馬鈴苣苔屬和近緣類群的系統(tǒng)學研究[D]. 鄭州:河南農業(yè)大學,2015.
[10] Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification.[J]. Genomics, 1994, 20(2): 176-183.
[11] Vos P, Hogers R, Bleeker M, et al. AFLP: a new technique for DNA fingerprinting.[J]. Nucleic Acids Research, 1995, 23(21): 4 407-4 414.
[12] Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification.[J]. Genomics, 1994, 20(2): 176-183.
[13] Lu P L, Mitsuko Y, Morden C W. Population Genetics of the Endemic Hawaiian Species Chrysodracon hawaiiensis and Chrysodracon auwahiensis (Asparagaceae): Insights from RAPD and ISSR Variation[J]. International Journal of Molecular Sciences, 2016, 17(8): 1 341.
[14] Wang L, Liu J, Zhang W, et al. Genetic diversity and population structure in Elephantopus scaber (Asteraceae) from South China as revealed by ISSR markers [J]. Plant Biosystems, 2006, 140(3): 273-279.
[15] Tsumura Y, Ohba K, Strauss S H. Diversity and inheritance of inter simple sequence repeat polymorphisms in douglas-fir (Pseudotsuga menziesii) and sugi(Cryptomeria japonica) [J]. Theoretical and Applied Genetics. Theoretische und Angewandte Genetik, 1996, 92(1): 40-45.
[16] Wolfe A D, Xiang Q Y, Kephart S R. Diploid hybrid speciation in Penstemon (Scrophulariaceae) [J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(9): 5 112-5 113.
[17] 姜 帆,高慧穎,陳秀萍,等. 龍眼ISSR-PCR反應體系的優(yōu)化及指紋圖譜初探[J]. 福建農業(yè)學報,2007,22(3):256-260.
[18] 鄒喻萍,葛 頌,王曉東. 系統(tǒng)與進化植物學中的分子標記[M]. 北京:科學出版社,2011:16-41.
[19] 邵果園,陸方方. 遠緣植物試管嫁接及ISSR分析[J]. 浙江林學院學報,2010,27(4):630-634.
[20] 何正文,劉運生,陳立華,等. 正交設計直觀分析法優(yōu)化PCR條件[J]. 湖南醫(yī)科大學學報,1998,23(4):403-404.
[21] 黃曉丹,張云貴,應鐵進. 高質量植物基因組DNA的提取[J]. 植物生理學通訊,2006,42(2):311-314.
[22] 高洪曉,楊 凱,劉建斌. 3種植物DNA提取法中多糖類物質去除效果的研究[J]. 北京農學院學報,2011,26(1):70-72.
[23] 李榮華,夏巖石,劉順枝,等. 改進的CTAB提取植物DNA方法[J]. 實驗室研究與探討,2009,28(9):14-16.
[24] 張 宇,黃國弟,唐志鵬,等. 芒果總DNA提取方法比較分析[J]. 經濟林研究,2014,32(2):62-65.
[25] 陳美霞,朱文寧,孫 煥,等. 石斛屬植物ISSR擴增體系的建立與優(yōu)化[J]. 中國農學通報,2014,30(10):177-181.
[26] 李喜鳳,邱天寶,張紅梅,等. 蒲公英ISSR-PCR反應體系及擴增程序的建立與優(yōu)化[J]. 2012,18(16):119-122.
[27] 陳大霞,李隆云,魯 成,等. 黃連ISSR反應條件優(yōu)化的研究[J]. 植物研究,2007,27(1):77-81.
[28] 代紅艷,張志宏,周傳生,等. 山楂ISSR分析體系的建立和優(yōu)化[J]. 果樹學報,2007,24(3):313-318.

