膀胱癌相關(guān)lncRNA及其共表達(dá)mRNA的初步篩選與功能預(yù)測
舒靜,黃夢鴿,田強(qiáng),姜源,陳俊霞
(1西南醫(yī)科大學(xué)附屬醫(yī)院,四川瀘州 646000;2重慶醫(yī)科大學(xué))
膀胱癌相關(guān)lncRNA及其共表達(dá)mRNA的初步篩選與功能預(yù)測
舒靜1,黃夢鴿1,田強(qiáng)1,姜源1,陳俊霞2
(1西南醫(yī)科大學(xué)附屬醫(yī)院,四川瀘州 646000;2重慶醫(yī)科大學(xué))
目的 篩選出在膀胱癌組織中特異性表達(dá)的長鏈非編碼RNA(lncRNA)及其共表達(dá)mRNA,并進(jìn)行初步驗(yàn)證及功能預(yù)測。方法 采用lncRNA v4.0芯片篩選4對膀胱癌和癌旁組織中l(wèi)ncRNA及其共表達(dá)mRNA的差異表達(dá)譜,通過聚類分析比較二者表達(dá)差異;采用實(shí)時熒光定量PCR(qRT-PCR)法對5個異常調(diào)節(jié)的lncRNA(RP11-359E19.2、AL928768.3、AC002519.6、RP11-79H23.3、AK021804)和4個共表達(dá)的mRNA(HRAS、VEGFA、ITGB1、DNMT3B)進(jìn)行驗(yàn)證。同時,對lncRNA共表達(dá)的mRNA進(jìn)行GO及KEGG pathway富集分析。結(jié)果 差異表達(dá)的lncRNA共4 155條,其中2 045條高表達(dá)、2 110條低表達(dá);與lncRNA共表達(dá)的mRNA共4 416條,其中2 472條高表達(dá)、1 944條低表達(dá)。|FC|≥10的lncRNA 345條,包括127條高表達(dá)和218條低表達(dá)。RP11-436F21.1和H19是上調(diào)最明顯的lncRNA。|FC|≥10的共表達(dá)mRNA有75條,其中57條上調(diào)、18條下調(diào)。與癌旁組織相比,膀胱癌組織5種lncRNA中RP11-359E19.2表達(dá)上調(diào),AL928768.3、AC002519.6、RP11-79H23.3及AK021804表達(dá)下調(diào)(P均<0.05);4種共表達(dá)的mRNA中HRAS、VEGFA、ITGB1和DNMT3B表達(dá)均上調(diào)(P均<0.05);qRT-PCR結(jié)果與芯片結(jié)果一致。GO分析顯示,上調(diào)的共表達(dá)mRNA主要參與細(xì)胞代謝和有絲分裂的生物學(xué)過程,下調(diào)的共表達(dá)mRNAs主要參與免疫系統(tǒng)的刺激反應(yīng)和免疫應(yīng)答;KEGG pathway富集分析顯示,10個通路與上調(diào)或下調(diào)的共表達(dá)mRNA有關(guān),其中在上調(diào)的共表達(dá)mRNA中,p53信號通路和膀胱癌的富集度最高,在下調(diào)的共表達(dá)mRNA中細(xì)胞因子-因子受體相互作用富集度最高。結(jié)論 成功篩選出膀胱癌特異表達(dá)的lncRNA及其共表達(dá)mRNA,這些特異表達(dá)的lncRNA及共表達(dá)mRNA在膀胱癌的發(fā)生、發(fā)展和轉(zhuǎn)移中發(fā)揮重要的生物學(xué)作用,有望成為膀胱癌新的標(biāo)志物。
膀胱癌;長鏈非編碼RNA;lncRNA芯片;信息學(xué)分析
近年來膀胱癌發(fā)病率有逐年升高之勢。膀胱癌術(shù)后易復(fù)發(fā)和轉(zhuǎn)移,患者預(yù)后極差[1]。迄今腫瘤發(fā)生、發(fā)展及轉(zhuǎn)移的根本原因和機(jī)制還不完全清楚,部分關(guān)鍵腫瘤標(biāo)志物尚未被發(fā)現(xiàn)。長鏈非編碼RNA(lncRNA)是一組長度>200 nt且不具有編碼蛋白功能的RNA轉(zhuǎn)錄本,可在表觀遺傳水平、轉(zhuǎn)錄水平和轉(zhuǎn)錄后水平調(diào)控基因的表達(dá),廣泛參與機(jī)體的生理和病理過程[2]。越來越多的研究顯示,lncRNA表達(dá)與腫瘤的發(fā)生、發(fā)展密切相關(guān)[3~7]。但目前關(guān)于膀胱癌患者血漿中特異性lncRNA新的標(biāo)志物的篩選與鑒定少見報(bào)道。本研究篩選并驗(yàn)證了膀胱癌組織中特異性lncRNA及其共表達(dá)的mRNA,并初步分析、預(yù)測了其功能,旨在為膀胱癌的臨床診斷、治療、判斷預(yù)后及分子機(jī)制研究提供幫助。
1 實(shí)驗(yàn)材料
本研究所用的樣本來自重慶醫(yī)科大學(xué)附屬第一醫(yī)院,收集未經(jīng)化療和放療的膀胱癌和癌旁組織30例份,樣本均經(jīng)病理證實(shí),并在手術(shù)切除后立即液氮保存。本實(shí)驗(yàn)所有樣本的采集、處理通過重慶醫(yī)科大學(xué)倫理委員會批準(zhǔn),并征得患者和(或)家屬知情同意。
2 方法與結(jié)果
2.1 lncRNA及其共表達(dá)mRNA表達(dá)譜建立及其特異表達(dá)的篩選 抽提4對膀胱癌與癌旁組織總RNA,對抽提所得RNA進(jìn)行質(zhì)檢,合格的RNA按照Quick Amp Labeling kit (Agilent Technologies, palo Alto, USA)標(biāo)記方法對RNA進(jìn)行擴(kuò)增、轉(zhuǎn)錄生成熒光標(biāo)記的cRNA;標(biāo)記后樣品雜交于Arraystar公司的Human LncRNA Array v4.0芯片上,漂洗后由Agilent Scanner G2505C掃描儀進(jìn)行掃描;原始數(shù)據(jù)采集由Agilent Feature Extraction Software (version 11.0.1.1)軟件完成。應(yīng)用R Software Package進(jìn)行原始數(shù)據(jù)標(biāo)準(zhǔn)化與進(jìn)一步的數(shù)據(jù)分析。將膀胱癌組織與癌旁組織相比較,篩選出特異表達(dá)的lncRNA及其共表達(dá)mRNA(|FC| ≥ 2.0,且P< 0.05,F(xiàn)RD<0.05)。結(jié)果顯示,與癌旁組織相比,膀胱癌組織差異表達(dá)的lncRNAs共4 155條,其中2 045條高表達(dá)、2 110條低表達(dá);lncRNA共表達(dá)mRNA共4 416條,其中2 472條高表達(dá)、1 944條低表達(dá)。差異表達(dá)的lncRNA中|FC|≥10者345條,包括127條高表達(dá)和218條低表達(dá)。RP11-436F21.1 (FC: ~236)和H19(FC: ~30)是上調(diào)最明顯的lncRNA。|FC|≥10的共表達(dá)mRNA有75條,其中57條上調(diào)、18條下調(diào)。膀胱癌與癌旁組織lncRNA及其共表達(dá)mRNAs層次聚類圖見插頁Ⅱ圖1。
2.2 特異表達(dá)的LncRNA及其共表達(dá)mRNA的驗(yàn)證 采用實(shí)時熒光定量PCR(qRT-PCR)法進(jìn)一步驗(yàn)證表達(dá)差異的lncRNA和共表達(dá)mRNA的表達(dá)水平,我們從芯片結(jié)果中篩選出特異表達(dá)的5個lncRNA(RP11-359E19.2、AL928768.3、AC002519.6、RP11-79H23.3、AK021804)和4個共表達(dá)的mRNA(HRAS、VEGFA、ITGB1、DNMT3B)。按照TRIzol試劑盒說明書分別提取膀胱癌和癌旁組織中細(xì)胞總RNA,經(jīng)逆轉(zhuǎn)錄反應(yīng)和PCR反應(yīng)檢測mRNA表達(dá)水平。RP11-359E19.2上游引物: 5′TTTCCCTTCTCA-ACGACCTG3′;下游引物:5′GAGTAAAGACAAGCTGAACCCA3′;AL928768.3上游引物:5′AGATGCCGCCTCCCTATGT3′;下游引物:5′CACGGTCAGTGCTGTGCTAT3′;AC002519.6上游引物:5′TGCTCATTTCACTCCACCCA3′;下游引物:5′ATTTCCTTTGCCTTCTCGC3′;RP11-79H23.3上游引物:5′GCAA-GGAGAGTAATGCTGGA3′;下游引物:5′CAATGAGGATGAGAAGAGGTC3′;HRAS上游引物:5′CAGTACAGGGAGCAGATCAAACG3′ ;下游引物:5′TGAGCCTGCCGAGATTCCA3′;VEGFA上游引物:5′CATGCAGATTATGCGGATCAA3′ ;下游引物:5′GCATTCACATTTGTTGTGCTGTAG3′;ITGB1上游引物:5′GA-ATGCCTACTTCTGCACGATGT3′;下游引物:5′TGTTCCTTTGCTACGGTTGGTTA3′;DNMT3B上游引物:5′AAGAGTTGGGCATAAAGGTAGGA3′;下游引物:5′GGCTGGATTCACATTTGAGAGA3′;β-actin上游引物:5′CCTGTACGCCAACACAGTGC3′;下游引物:5′ATACTCCTGCTTGCTGATCC3′。采用SYBR? Pre-mix Ex TaqⅡ試劑盒,應(yīng)用實(shí)時定量PCR擴(kuò)增儀(ABI 7500)檢測每個擴(kuò)增循環(huán)后SYBR? Green熒光信號水平。反應(yīng)體系共10 μL:上下游引物各0.5 μL,cDNA 2 μL,ddH2O 2 μL,2×Master Mix 5 μL。反應(yīng)條件:95 ℃預(yù)變性10 min,95 ℃ 10 s,60 ℃ 1 min,共40個循環(huán)。收集熒光信號,建立熔解曲線,運(yùn)用熔解曲線檢測擴(kuò)增產(chǎn)物純度。采用相對定量2-ΔΔCt法對獲得的CT值進(jìn)行分析。
結(jié)果顯示,與癌旁組織相比,膀胱癌組織5種lncRNAs中RP11-359E19.2表達(dá)上調(diào),AL928768.3、AC002519.6、RP11-79H23.3及AK021804表達(dá)均下調(diào)(P均<0.05),見表1;4種共表達(dá)mRNA中HRAS、VEGFA、ITGB1和DNMT3B表達(dá)均上調(diào)(P均<0.05),見表2。qRT-PCR結(jié)果與芯片結(jié)果一致。

表1 膀胱癌組織與癌旁組織5種lncRNA表達(dá)
注:與癌旁組織比較,*P<0.05。
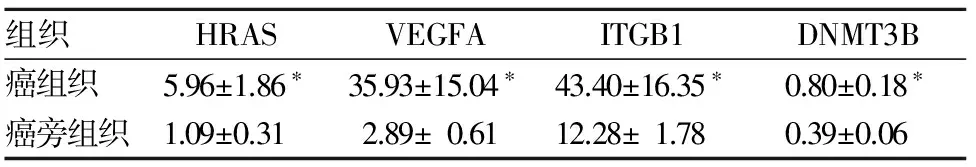
表2 膀胱癌組織與癌旁組織4種與lncRNA共表達(dá)的mRNA表達(dá)
注:與癌旁組織比較,*P<0.05。
2.3 lncRNA共表達(dá)mRNA的功能預(yù)測 采用GO和KEGG pathway富集分析對lncRNA共表達(dá)的mRNA進(jìn)行功能預(yù)測,推斷l(xiāng)ncRNA的功能。GO分析(http://www.geneontology.org)通過生物過程、細(xì)胞組分和分子功能三部分對基因或蛋白質(zhì)進(jìn)行相應(yīng)的注釋以及分類。對共表達(dá)mRNA進(jìn)行KEGG pathway分析,通過富集分?jǐn)?shù)顯示相關(guān)信號通路的重要性。GO分析結(jié)果顯示,上調(diào)的共表達(dá)mRNA主要參與細(xì)胞代謝和有絲分裂等生物學(xué)過程;下調(diào)的共表達(dá)mRNAs主要參與免疫系統(tǒng)的刺激反應(yīng)和免疫應(yīng)答。KEGG pathway富集分析顯示,10個通路與上調(diào)的共表達(dá)mRNA有關(guān),10個通路與下調(diào)的共表達(dá)mRNA有關(guān)。在上調(diào)的共表達(dá)mRNA中,p53信號通路和膀胱癌的富集度最高;在下調(diào)的共表達(dá)mRNAs中,細(xì)胞因子-因子受體相互作用富集度最高。采用Cluster 3.0程序完成hierarchical clustering分析,結(jié)果以TreeView 1.60程序可視化,見插頁Ⅱ圖2。
3 討論
lncRNA是在真核生物中新發(fā)現(xiàn)的一類長度>200個核苷酸,沒有長閱讀框架,但多具有mRNA結(jié)構(gòu)特征(帽式結(jié)構(gòu)和poly A尾巴)的RNA[8]。近年來研究表明,lncRNA參與了X染色體沉默、基因組印記以及染色質(zhì)修飾、轉(zhuǎn)錄、激活、剪切、轉(zhuǎn)錄干擾、表觀遺傳學(xué)調(diào)控、免疫監(jiān)控等多種重要的生物學(xué)過程,其轉(zhuǎn)錄和功能失調(diào)可能導(dǎo)致多種疾病的發(fā)生[9]。雖然近年來關(guān)于lncRNA的研究進(jìn)展迅猛,但絕大部分lncRNA的功能仍不明確,越來越多的研究表明,lncRNA作為癌基因和抑癌基因在腫瘤中異常表達(dá),且lncRNA的突變和異常調(diào)節(jié)與人類疾病密切相關(guān),lncRNA作為新的生物標(biāo)志物在輔助診斷、判斷治療效果中發(fā)揮重要作用[10]。
本研究膀胱癌組織特異表達(dá)lncRNA的篩選結(jié)果顯示,|FC|≥10的lncRNA有345條,包括127條高表達(dá)的和218條低表達(dá)。RP11-436F21.1是上調(diào)最明顯的lncRNA,而H19是在膀胱癌組織上調(diào)最明顯的lncRNA之一。研究發(fā)現(xiàn),膀胱癌組織H19表達(dá)水平不僅與分化程度、病理分期、復(fù)發(fā)轉(zhuǎn)移相關(guān)[11],還可通過上調(diào)P57的表達(dá),促進(jìn)膀胱癌腫瘤細(xì)胞侵襲與轉(zhuǎn)移、血管形成[12]。提示lncRNA在膀胱癌中的表達(dá)具有特異性,lncRNA的表達(dá)量可能成為潛在的膀胱癌診斷和靶向治療生物標(biāo)志。
為進(jìn)一步驗(yàn)證特異表達(dá)的lncRNA和共表達(dá)mRNA的表達(dá)水平,我們從芯片結(jié)果中篩選出特異表達(dá)的5個lncRNA和4個共表達(dá)mRNA。qRT-PCR結(jié)果顯示,與癌旁組織相比,膀胱癌組織5種lncRNA中RP11-359E19.2表達(dá)上調(diào),AL928768.3、AC002519.6、RP11-79H23.3及AK021804表達(dá)下調(diào);4種共表達(dá)mRNA中HRAS、VEGFA、ITGB1和DNMT3B表達(dá)均上調(diào)。qRT-PCR結(jié)果與芯片結(jié)果一致。
本研究篩選出特異表達(dá)lncRNA及其共表達(dá)mRNA的表達(dá)譜,通過GO和KEGG pathway富集分析其共表達(dá)mRNA的生物學(xué)功能,以推斷l(xiāng)ncRNA的功能。GO分析結(jié)果顯示,與癌旁組織相比,膀胱癌組織中上調(diào)的共表達(dá)mRNA主要參與細(xì)胞代謝和有絲分裂等生物學(xué)過程;下調(diào)的共表達(dá)mRNA主要參與免疫系統(tǒng)的刺激反應(yīng)和免疫應(yīng)答等,進(jìn)一步證實(shí)了免疫系統(tǒng)功能下降與腫瘤發(fā)生密切相關(guān)[13]。KEGG pathway富集分析結(jié)果顯示,與癌旁組織相比,10個顯著信號通路分別與上調(diào)的共表達(dá)mRNA和下調(diào)的共表達(dá)mRNA有關(guān)。其中在上調(diào)的共表達(dá)mRNA中,p53信號通路和膀胱癌的富集度最高,在下調(diào)的共表達(dá)mRNAs中,細(xì)胞因子-因子受體相互作用富集度最高。這些結(jié)果與Zhu等[14]研究結(jié)果一致,提示與lncRNAs共表達(dá)的mRNAs可能通過調(diào)節(jié)相關(guān)通路在膀胱癌的發(fā)生、發(fā)展中發(fā)揮著至關(guān)重要的作用。
綜上所述,本研究發(fā)現(xiàn)并篩選出膀胱癌特異表達(dá)的lncRNA及其共表達(dá)mRNA,這些差異表達(dá)的lncRNA及共表達(dá)mRNA在膀胱癌的發(fā)生、發(fā)展和轉(zhuǎn)移中發(fā)揮重要的生物學(xué)作用,有望成為膀胱癌新的標(biāo)志物,為進(jìn)一步研究膀胱癌的發(fā)生機(jī)制奠定基礎(chǔ)。
[1] Zhao F, Lin T, He W, et al. Knockdown of a novel lincRNA AATBC suppresses proliferation and induces apoptosis in bladder cancer[J]. Oncotarget, 2015,6(2):1064-1078.
[2] Chen Z, Luo Y, Yang W, et al. Comparison analysis of dysregulated lncRNA profile in mouse plasma and liver after hepatic ischemia/reperfusion injury[J]. PLoS One, 2015,10(7):e0133462.
[3] Huarte M, Rinn JL. Large non-coding RNAs: missing links in cancer[J]. Hum Mol Genet, 2010,19(R2):R152-161.
[4] Xie H, Ma H, Zhou D. Plasma HULC as a promising novel biomarker for the detection of hepatocellular carcinoma[J]. Biomed Res Int, 2013,2013:136106.
[5] Hessels D, Klein Gunnewiek JM, van Oort I, et al. DD3(PCA3)-based molecular urine analysis for the diagnosis of prostate cancer[J]. Eur Urol, 2003,44(1):8-15; discussion 15-16.
[6] Huang KC, Rao PH, Lau CC, et al. Relationship of XIST expression and responses of ovarian cancer to chemotherapy[J]. Mol Cancer Ther, 2002,1(10):769-776.
[7] Yang F, Zhang L, Huo XS, et al. Long noncoding RNA high expression in hepatocellular carcinoma facilitates tumor growth through enhancer of zeste homolog 2 in humans[J]. Hepatology, 2011,54(5):1679-1689.
[8] Hung T, Chang HY. Long noncoding RNA in genome regulation: prospects and mechanisms[J]. RNA Biol, 2010,7(5):582-585.
[9] Orom UA, Derrien T, Beringer M,et al. Long noncoding RNAs with enhancer-like function in human cells[J]. Cell, 2010,143(1):46-58.
[10] Esteller M. Non-coding RNAs in human disease[J]. Nat Rev Genet, 2011,12(12):861-874.
[11] Luo M, Li Z, Wang W,et al. Long non-coding RNA H19 increases bladder cancer metastasis by associating with EZH2 and inhibiting E-cadherin expression[J]. Cancer Lett, 2013,333(2):213-221.
[12] Bozdogan O, Atasoy P, Batislam E,et al. Significance of p57(Kip2) down-regulation in oncogenesis of bladder carcinoma: an immunohistochemical study[J]. Tumori, 2008,94(4):556-562.
[13] Bigley AB, Spielmann G, LaVoy EC, et al. Can exercise-related improvements in immunity influence cancer prevention and prognosis in the elderly[J]. Maturitas, 2013,76(1):51-56.
[14] Zhu YP, Bian XJ, Ye DW,et al. Long noncoding RNA expression signatures of bladder cancer revealed by microarray [J]. Oncol Lett, 2014,7(4):1197-1202.
·作者·編者·讀者·
關(guān)于醫(yī)學(xué)名詞與統(tǒng)計(jì)學(xué)符號的應(yīng)用說明
醫(yī)學(xué)名詞要以1989年及以后由全國自然科學(xué)名詞審定委員會審定、公布,科學(xué)出版社出版的《醫(yī)學(xué)名詞》和相關(guān)科學(xué)的名詞為準(zhǔn),暫未公布者仍以人民衛(wèi)生出版社出版的《英漢醫(yī)學(xué)詞匯》為準(zhǔn)。中文藥物名稱應(yīng)使用1995年藥典(法定藥物)或衛(wèi)生部藥典委員會編輯的《藥名詞匯》(非法定藥物)中的名稱,英文藥物名稱則采用國際非專利藥名,不用商品名。
Preliminary screening and functional prediction of long non-coding RNA and its co-expression mRNA in bladder carcinoma
SHUJing1,HUANGMengge,TIANQiang,JIANGYuan,CHENJunxia
(1TheAffiliatedHospitalofSouthwestMedicalUniversity,Luzhou646000,China)
ObjectiveTo screen out differentially expressed long non-coding RNAs (lncRNAs) and co-expression mRNAs and to perform the preliminary validation and functional prediction in bladder carcinoma.MethodsFour pairs of carcinoma and para-carcinoma tissues were used for microarray assay to screen out the differentially expressed lncRNAs and co-expression mRNAs. Hierarchical clustering was performed to show lncRNA and mRNA expression patterns among samples. Five lncRNAs (RP11-359E19.2, AL928768.3, AC002519.6, RP11-79H23.3, AK021804) and four co-expression mRNAs (HRAS, VEGFA, ITGB1, DNMT3B) were chosen for verification of the microarray results by quantitative real-time PCR. Gene Ontology (GO) and KEGG pathway were used to analyze the biological function of co-expression mRNA.ResultsA total of 4 155 lncRNAs and 4 416 co-expression mRNAs were detected to be differentially expressed, among which, 2 045 lncRNAs were up-regulated and and 2 110 were down-regulated, meanwhile, 2 472 co-expression mRNAs were up-regulated and and 1944 were down-regulated, respectively. Totally 345 lncRNAs displayed fold change ≥10, including 127 up-regulated lncRNAs and 218 down-regulated lncRNAs. RP11-436F21.1 and H19 were the most up-regulated lncRNAs. Overview of coding gene profile showed that 75 mRNAs had fold change ≥10 (up: 57; down: 18). Comapred with the para-carcinoma tissues, the expression of RP11-359E19.2 in 5 lncRNAs was up-regulated, whereas AL928768.3, AC002519.6, RP11-79H23.3, and AK021804 expression was down-regulated in the bladder carcinoma tissues (allP<0.05). Meanwhile, the expression of HRAS, VEGFA, ITGB1, and DNMT3B in four co-expression mRNAs was all up-regulated (allP<0.05). The result was consistent with the microarray assay. GO assay showed that the up-regulated co-expression mRNAs mainly participate in the biological processes, such as cellular metabolic and mitotic cell cycle. Meanwhile, the down-regulated co-expression mRNAs mainly participate in the immune system process of stimulus response and immune response. KEGG pathway assay showed that 10 pathways were associated with the up-regulated or down-regulated mRNAs. Among the co-expression mRNAs, the enrichment of P53 signal pathway and bladder cancer was the highest, whereas in the down-regulated co-expression mRNAs, the cytokine-factor receptor interaction was the highest.ConclusionsThe differentially expressed LncRNAs and co-expression mRNAs are found and screened out in the bladder cancer. The specially expressed lncRNAs and and mRNAs could play important roles in the pathogenesis and development of bladder carcinoma, which might be new markers of bladder cancer.
bladder carcinoma; long non-coding RNA; LncRNA chip; informatics analysis
國家自然科學(xué)基金資助項(xiàng)目(81672536)。
舒靜(1986-),女,碩士,技師,主要研究方向?yàn)槟[瘤相關(guān)基因的篩選及其功能預(yù)測。E-mail: shujing0723@163.com
10.3969/j.issn.1002-266X.2017.20.002
R737.14
A
1002-266X(2017)20-0005-04
2016-08-31)

