珊瑚菜Gl4CL基因克隆與生物信息學分析*
宋潔潔,羅紅梅,朱珣之,張玉,高婷**
(1.青島農業大學生命科學學院山東省高校植物重點實驗室青島266109;2.中國醫學科學院北京協和醫學院藥用植物研究所中草藥物質基礎與資源利用教育部重點實驗室北京100193;3.江蘇科技大學生物與化學工程學院鎮江212018)
珊瑚菜Gl4CL基因克隆與生物信息學分析*
宋潔潔1,羅紅梅2,朱珣之3,張玉1,高婷1**
(1.青島農業大學生命科學學院山東省高校植物重點實驗室青島266109;2.中國醫學科學院北京協和醫學院藥用植物研究所中草藥物質基礎與資源利用教育部重點實驗室北京100193;3.江蘇科技大學生物與化學工程學院鎮江212018)
目的:對珊瑚菜(Glehnia littoralis)4-香豆酸:輔酸A連接酶(4-Coumarate:Coenzyme A Ligase,Gl4CL)基因編碼區進行克隆及序列分析。方法:本研究在前期珊瑚菜高通量測序的基礎上,利用cDNA末端快速擴增(Rapid Amplification of cDNA Ends,RACE)方法對Gl4CL基因全長cDNA序列進行克隆。對Gl4CL蛋白進行理化性質、蛋白二級結構及三維結構預測分析。利用實時熒光定量PCR方法檢測Gl4CL基因在珊瑚菜的根、葉中的表達情況。結果:Gl4CL基因cDNA序列全長1 951 bp,編碼544個氨基酸,其中開放閱讀框1 635 bp,5′非編碼區153 bp,3′非編碼區163 bp。生物信息學分析表明,Gl4CL蛋白大小約為59.481 kDa,等電點為8.20。Gl4CL基因在珊瑚菜的根和葉均存在表達,且在根中表達量顯著高于葉。結論:本研究為進一步開展Gl4CL基因功能和遺傳調控研究奠定基礎,通過深入探討Gl4CL基因的表達調控與木質素、植株生長表型等關系,有望獲得抗病蟲害、抗逆性強的北沙參高產優質品系。
珊瑚菜4-香豆酸:輔酸A連接酶基因克隆生物信息學分析
珊瑚菜Glehnia littoralis Fr.Schmidt ex Miq.是傘形科Umbelliferae珊瑚菜屬Glehnia的多年生草本植物,是國家Ⅱ級重點保護野生植物,其干燥根入藥名為北沙參。北沙參是中國的傳統中藥,具有養陰清肺、益胃生津的功效,有較高藥用價值[1]。目前,臨床上主要用于治療肺熱燥咳、熱病傷津、勞嗽痰血等癥[2]。北沙參在中國分布面積較廣,以山東萊陽為地道產地[3],在日本、朝鮮和俄羅斯等國亦有產出[4]。在北沙參的農業生產中,銹病、病毒病、根結線蟲病及黑絨金龜子、大灰象甲、鉆心蟲、蚜蟲等病蟲害時常發生[5],病蟲害的侵害嚴重影響了北沙參的產量和品質,使其藥用價值和經濟價值受到較大影響。
木質素是存在于維管植物細胞壁中的一類苯丙烷類衍生物,在植物體內含量約15%-35%[6]。木質素可增強細胞壁硬度,提高植物抵御病原微生物侵害的能力[7]。基因工程調節植物木質素合成已成為國際上研究的熱點,對煙草、黃瓜、玉米、擬南芥、馬鈴薯、哈密瓜、棉花[8-14]等物種的研究表明:植物體內木質素含量的增加可使植物抗病性增強,對木質素合成途徑中關鍵酶基因的改造可改良植物木質素含量及組成,特異地改變植物的品質,提高植株產量,使其具有更高的經濟價值[15]。4-香豆酸:輔酶A連接酶(Gl4CL)是連接苯丙酸途徑與木質素合成途徑的關鍵限速酶,是合成木質素及其它苯丙烷類衍生物的代謝流向調控點。4CL催化4-香豆酸、咖啡酸、阿魏酸、5-羥基阿魏酸和芥子酸生成相應的CoA酯[16-18],再通過肉桂酰輔酶A還原酶和肉桂醇脫氫酶催化進入木質素生物合成途徑,最終促進木質素單體的合成。
4CL基因位于植物代謝網絡的關鍵位點,發揮著重要作用。目前,4CL基因已在苔鮮植物、裸子植物、單子葉植物、雙子葉植物如桂花Osmamthus fragrans、象草Pennisetum purpureum、毛白楊Populus tomentosa、碭山酥梨Pyrus bretschneideri、擬南芥Arabidopsis thali?ana、煙草Nicotiana tabacum[19-24]等眾多物種中均有文獻報道;4CL基因在上述物種中被克隆鑒定,其功能也被鑒定。但珊瑚菜Gl4CL基因研究還未見報道。本實驗在前期高通量測序(PRJNA387325)的基礎上,利用RACE方法對Gl4CL基因進行克隆,用生物信息學工具分析,同時利用熒光定量PCR的方法分析該基因的組織表達模式,為今后深入研究Gl4CL基因在珊瑚菜植株的具體功能及其對植株生長特性的影響奠定基礎。
1 材料與方法
1.1 主要儀器與試藥
SIM-F140AY65制冰機(日本三洋公司);Milli-Q超純水儀(美國Millipore公司);YXQG02高溫滅菌鍋(美國Zealway公司);HW-SY21-K水浴鍋(上海博迅實業公司);YY0027-90電熱恒溫培養箱(上海藍豹實驗設備公司);2720PCR儀(德國AppLied Biosystems公司);DYY-10C型電泳儀(美國Bio-rad公司);Tanon500凝膠成像分析系統(上海領城公司);Mx3000P實時熒光定量PCR儀(美國安捷倫Stratagene公司)。
SMARTer?RACE5′/3′Kit試劑盒(大連寶生物公司,批號:1505943A);TLAN Midi Purification Kit瓊脂糖凝膠DNA回收試劑盒(北京天根生物科技有限公司,批號:P4411);瓊脂糖(西班牙BIOWEST公司,批號:111860);SYBR?Premix Ex TapTMⅡ(TLi RNaseH Plus)定量試劑盒(大連寶生物公司,批號:AKA306)。
1.2 實驗材料
采集山東省萊陽市高格莊鎮新鮮的珊瑚菜,由青島農業大學基原鑒定專家辛華教授鑒定為傘形科珊瑚菜屬珊瑚菜Glehnia littoralis Fr.Schmidt ex Miq.的根和葉,清潔后放入液氮中速凍,將材料置于-80℃超低溫冰箱保存備用。
2 實驗方法
2.1 總RNA的提取
用Trizol法提取珊瑚菜根和葉的總RNA。經核酸測定儀和瓊脂糖凝膠電泳檢測產量和質量后,保存于-80℃冰箱中備用。
2.2 第1鏈cDNA的合成
RACE-Ready first-strand cDNA的合成方法參照SMARTer?RACE 5′/3′Kit User Manual。
2.3 RACE擴增
在轉錄組測序數據庫中選取表達量較高的一條Gl4CL基因,依據其部分cDNA序列(5′端完整,3′端不完整)為依據,利用Primer Premier 5.0軟件設計3′RACE特異性引物(3′GSP:ATTGGTTCTGGTGCTGCTCCGTTAG)。引物由青島擎科梓熙生物技術公司合成。以反轉錄成的3′-RACE-Ready first-strand cDNA為模板,以3′GSP和UPM為引物,利用Touchdown PCR程序擴增3′末端序列。反應體系參照SMARTer? RACE 5′/3′Kit User Manual。RACE程序為:第1步,94℃,30 s;72℃,1 min;5 cycles;第2步,94℃,30 s;70℃,30 s;72℃,1 min;5 cycles;第3步,94℃,30 s;68℃,30 s;72℃,1 min;25 cycles。
2.4 RACE產物的克隆和測序
PCR產物經1%瓊脂糖凝膠電泳檢測后,用瓊脂糖凝膠DNA回收試劑盒回收。回收產物與pMD18-T載體連接,連接產物轉化DH5α感受態細胞,隨機挑取6個抗性克隆,經菌落PCR檢驗后,將3個陽性克隆送至青島擎梓熙生物技術有限公司測序。將測序獲得序列與已知序列進行拼接,得到該基因的cDNA全長序列。
2.5 熒光定量PCR分析
獲得的Gl4CL基因序列設計特異性引物,以近緣物種當歸Actin[25]作為內參基因。以珊瑚菜根和葉總RNA為模板,反轉錄成cDNA,構建熒光定量PCR體系后置于熒光定量PCR儀中反應(采用三步法程序):第一步,95℃,10 min;第二步,95℃,30 s;55℃,30 s;72℃,20 s;40 cycles第三步,95℃,1 min;55℃,30 s;95℃,30 s。利用SPSS 16.0軟件分析熒光定量PCR數據,利用SigmaPLot 10.0軟件作圖。
2.6 生物信息分析
采用DNAMAN軟件將測序結果獲得的Gl4CL基因3′端序列與已知序列進行拼接并且分析氨基酸特性;利用NCBI提供的Open Reading Frame Finder分析基因的開放閱讀框;利用在線軟件NCBI提供的Con?served Domains數據庫對蛋白進行保守結構域分析;使用ExPASy的Protscale在線工具(http://web.expasy.org/ protscale/)分析蛋白親疏水性;利用NCBI提供的blast工具分析對蛋白相似;利用在線軟件Sompa分析蛋白的二級結構;利用SWISS-MODEL在線軟件(http:// swiss-model.expasy.org/interactive)模建蛋白的三級結構,并用PymoL1.3優化;利用MEGA5.0軟件構建系統進化樹。
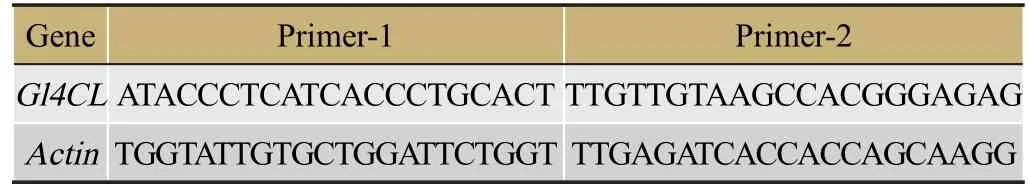
表1 熒光定量PCR引物
3 結果與分析
3.1 珊瑚菜Gl4CL基因的RACE擴增和序列分析
利用已知Gl4CL基因序列設計的引物3′GSP,通過RACE技術,克隆出912 bp的3′末端序列(圖1)。把測序結果與已知序列進行拼接,獲得1 951 bp的Gl4CL基因全長cDNA序列(圖2)。將該蛋白序列與NCBI上植物的同源序列進行比對。結果表明,其與水曲柳Frax?inus mandshurica(AHL44983)、丹參Salvia miltiorrhiza(AGW27195)、桑Morus alba(ALD83616)的4CL蛋白序列相似度較高為71%-72%。確定克隆得到的為珊瑚菜4CL基因的全長cDNA序列,將其命名為Gl4CL(GenBank序列號:KX924462)。
3.2 Gl4CL基因生物信息因分析
利用NCBI的ORF Finder(Open Reading Frame Finder)對Gl4CL基因進行分析,結果顯示1 951 bp的全長cDNA中包含一個1 635 bp的最大開放閱讀框(ORF),153 bp的5′非編碼區(5′UTR),163 bp的3′非編碼區(3′UTR),其中29 bp poly A尾(圖2)。利用DNAMAN軟件對此基因編碼的氨基酸序列進行分析,結果表明:Gl4CL蛋白分子量為59.481 kDa,等電點為8.20。

圖1 Gl4CL 3′RACE菌液PCR擴增結果
利用NCBI的Conserved Domains數據庫對Gl4CL蛋白保守結構域進行分析,結果表明Gl4CL基因序列屬于AFD class I基因家族,其中包含4CL保守結構域(214-1761,cd05904)、AMP結合位點(256-1494,pfam-00501)、AMP-binding C(1516-1743,pfa-m13193)和CoA結合位點(175-1761,PLN02574)4個重要結構域。Gl4CL蛋白包含SSGTTGVSKGV和GEICURG氨基酸序列,同已知物種的4CL蛋白序列中的保守結構域SSGTTGMPKGU和催化活化中心GEICIRG具有較高的相似性。但Gl4CL蛋白氨基酸序列N端保守域的第7、8個氨基酸位點,與常見的L和P不同,變為V和S,C端保守域的第5個氨基酸位點,與常見的I不同,變為U。
3.3 Gl4CL蛋白親疏水性
使用ExPASy的Protscale在線工具預測Gl4CL蛋白親疏水性(圖3),在第6個氨基酸位點,最低峰值是-1.967,在第239個氨基酸位點,最高峰值是2.744,總平均親水性0.075,說明該蛋白為疏水性蛋白。
3.4 Gl4CL蛋白系統進化分析
將珊瑚菜與NCBI上15種代表性植物的4CL蛋白進行氨基酸序列同源比對,并構建系統進化樹(圖4)。使用的物種序列有:紫穗槐Amorpha fruticosa(AAL35216)、大豆Glycine max(AAC97600)、東方山羊豆Galega orientalis(ACZ64784)、白樺Betula platyphylla(AAV65114)、覆盆子Rubus idaeus(AAF91310)、毛白楊Populus tomentosa(AAY84731)、煙草Nicotiana tabacum(NP001312554)、辣椒Capsicum annuum(AAG43823)、馬鈴薯Solanum tuberosum(AAD40664)、玉蜀黍Zea mays(NP001105258)、水稻Oryza sativa(AAA69580)、桑Morus alba(ALD83616)、大麻Cannabis sativa(AHA4-2444)、丹參Salvia miltiorrhiza(AGW27195)、水曲柳Fraxinus mandshurica(AHL44983)。從圖4可以看出:與同源性比對結果一致,珊瑚菜的Gl4CL蛋白與水曲柳、丹參、桑等植物的4CL蛋白親緣關系較近,而與玉蜀黍、水稻等植物的4CL蛋白親緣關系較遠。
3.5 Gl4CL蛋白的二維、三維結構預測
蛋白質的結構模型對于理解蛋白質的折疊機理和生物功能非常重要,利用在線軟件Sompa預測蛋白的二級結構參數。圖5表明,Gl4CL蛋白544個氨基酸中有170個氨基酸參與形成的α-螺旋(α-helix),占31. 25%;139個氨基酸參與形成β-折疊(β-sheet),占25.55%;64個氨基酸參與形成β-轉角(β-turn),占11.76%;171個氨基酸參與形成無規則卷曲(Random Coil),占31.43%。α-螺旋>30%、β-折疊>20%,該蛋白二級結構為α-β型。在第73、123、239、278、330、445、508、544個氨基酸處可能存在α-螺旋結構;28、44、433、529等處可能存在無規卷曲。對Gl4CL蛋白二級結構的四個項目與常見3個物種進行比較(表2),發現Gl4CL蛋白二級結構中α-螺旋、β-折疊、β-轉角、無規則卷曲與其他物種均有不同,表明其在空間結構上發生了一定的變異。

圖2 Gl4CL基因cDNA序列及氨基酸序列
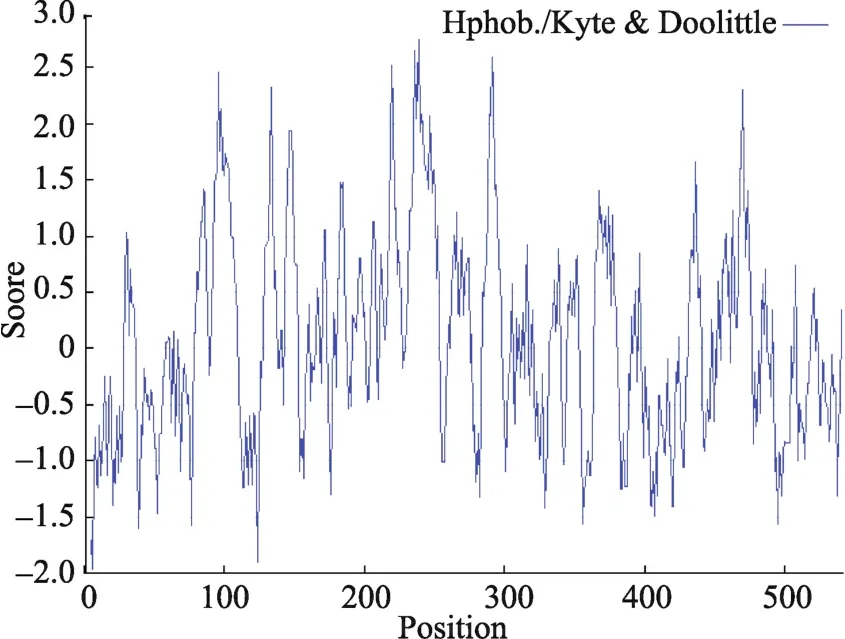
圖3 Gl4CL蛋白的親疏水性預測結果

圖4 4CL蛋白的系統進化樹

圖5 Gl4CL蛋白的二級結構

表2 珊瑚菜與其他3個物種的4CL蛋白二級結構比較
利用SWISS-MODEL在線軟件模建蛋白三級結構(圖6)。以5bsr.1.A蛋白作為模體分析,蛋白相似度為44.26%,預測模建可信度較高。預測Gl4CL蛋白三級結構包含了536個氨基酸,占編碼氨基酸總數的98.53%;結構中含有較多的α-螺旋和無規則卷曲,推測蛋白在生物體內合成后,其中某些氨基酸可能被酶進一步催化,改變其側鏈的化學結構,產生新的氨基酸,需進一步通過實驗來驗證預測三級結構的準確性。
3.6 Gl4CL基因的組織特異性表達
利用熒光定量PCR,獲得Ct值,利用2-ΔΔct計算方法[26]分析基因相對表達量。結果表明(圖7)Gl4CL基因的轉錄本在根和葉均能檢測到,但在根和葉兩組織中的表達量存在極顯著差異,在根中表達量高,在葉片中表達量少。
4 討論
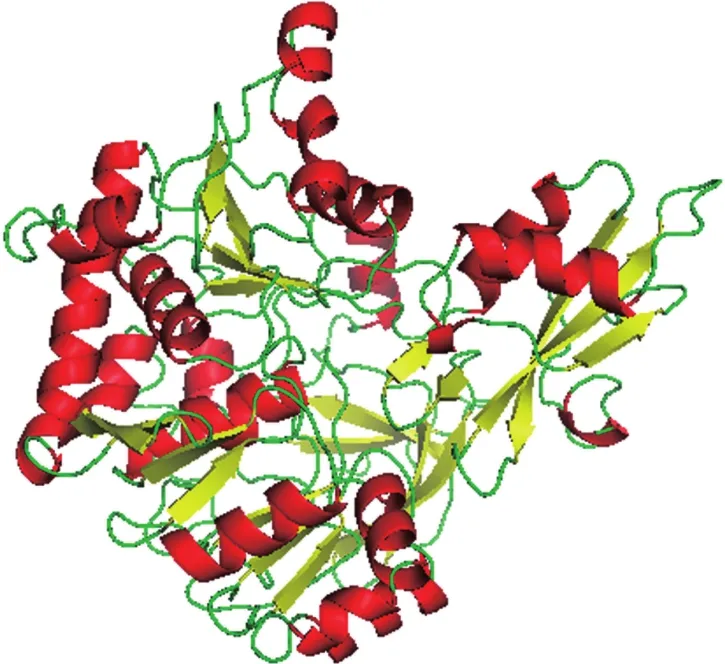
圖6 Gl4CL蛋白的三級結構
本研究通過RACE技術獲取Gl4CL基因的全長cDNA序列,將其與NCBI上植物的同源序列進行4CL蛋白比對,相似度達71%-72%;Gl4CL蛋白中存在4CL蛋白的氨基酸保守結構域。以上結果證明本研究獲得的基因序列即為珊瑚菜Gl4CL基因的cDNA序列。
研究發現,Gl4CL蛋白的氨基酸序列發生了3個氨基酸位點的變異,這種變異在桂花、覆盆子、黑麥草的4CL中也有報道。桂花N端保守域中的N和V替代了保守基序中P;C端的保守域由C、I,分別突變為保守域中的W和L[19];在覆盆子的C端保守域中的L被V替代[27];Stuible等[28]將擬南芥At4CL2的GEICIRG保守區半胱氨酸突變成丙氨酸,結果顯示酶活依然存在但有所下降,證明了半胱氨酸Cys未直接參與酶反應。以上研究結果表明4CL蛋白的保守區域是相對的,在外部環境、內部基因等條件影響下,不同物種的4CL蛋白保守區域可能發生輕微的變異。
4CL基因在植物中多是以基因家族的形式存在的,目前發現的4CL基因分為2類:Ⅰ類主要與木質素的生物合成有關,Ⅱ類主要調控類黃酮的生物合成。序列比對結果顯示Gl4CL基因序列與擬南芥(At4CL1、At4CL2)、楊樹(Ptd4CL1、Ptd4CL2、Pt4CL1)等同屬于ADF classⅠ基因家族。已有報道顯示,不同植物的4CL基因數量存在差異,小立碗蘚含有4個,歐芹含有2個,大豆含有4個,煙草含有3個,擬南芥含有4個,還有12個4CL-like基因[29]。本研究通過珊瑚菜的轉錄組測序結果篩選得到多條4CL基因片段,同時利用熒光定量PCR檢測發現Gl4CL基因在珊瑚菜根中表達量顯著高于葉中表達量,這與前人研究的東方山羊豆Go4CL基因表達模式相同[30]。由此推測,在珊瑚菜中4CL也是由一個基因家族控制的,該基因家族存在多名成員,不同成員表達特性不同,定位組織不同,參與的代謝途徑不同,有待后續實驗驗證。
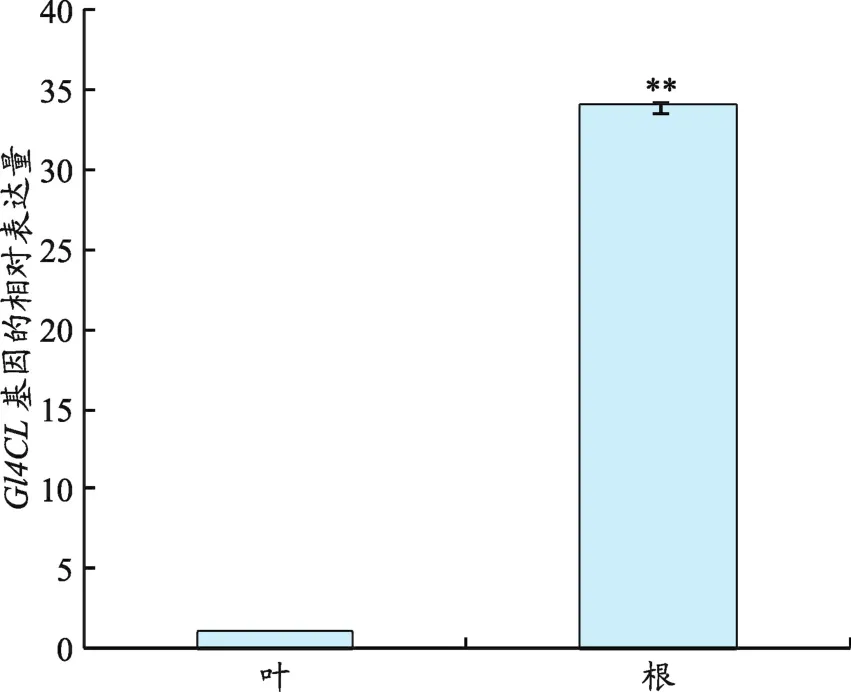
圖7 Gl4CL基因的組織特異性表達
北沙參是一味重要的山東道地藥材,其基源植物珊瑚菜在生產中病蟲害侵害嚴重。因此,珊瑚菜的抗逆育種十分重要。通過基因工程對珊瑚菜木質素合成途徑關鍵酶基因Gl4CL的過表達有望增加木質素含量、改良木質素組成,從而提高珊瑚菜植株的抗逆特性,可作為珊瑚菜抗逆育種的一種策略。本研究中對于Gl4CL基因的成功克隆及表達模式初步分析為進一步開展Gl4CL基因功能研究、遺傳調控和轉基因研究奠定基礎,通過深入探討Gl4CL基因的表達調控與木質素、植株生長表型等關系,有望獲得抗病蟲害、抗逆性強的北沙參高產優質品系。
1崔海燕,許一平.北沙參的化學成分及藥理作用研究綜述.中國科技信息,2009,(19):203-204.
2苗曉燕,朱維紅,張筱梅,等.水楊酸、茉莉酸甲酯及超聲波對北沙參愈傷組織生長及產香豆素的影響.食品科學,2016,37(9):181-185.
3王紅娟.萊陽北沙參藥材質量標準研究.濟南:山東中醫藥大學碩士學位論文,2010.
4董芳.三年生北沙參中化學成分的初步研究.青島:青島農業大學碩士學位論文,2010.
5張永清.山東省北沙參生產情況調查.山東中醫雜志,2001,20(3): 169-171.
6范丙友,陸海,蔣湘寧.維管植物4-香豆酸:輔酶A連接酶(4CL)研究進展.林業科學,2007,43(2):96-103.
7周龔,王玉鋒,婁來清,等.甜高粱4-香豆酸輔酶A連接酶基因(4CL)的克隆與鑒定及時空表達分析.南京農業大學學報,2014,37(3):9-19.
8朱海浩.木質素與抗病性的關系以及小麥Rop基因的調控作用分析.北京:中國科學院大學碩士學位論文,2013.
9程智慧,李玉紅,孟煥文,等.BTH誘導黃瓜幼苗對霜霉病的抗性與細胞壁HRGP和木質素含量的關系.中國農業科學,2006,39(5): 935-940.
10劉曉燕,金繼運,何萍,等.氯化鉀對玉米木質素代謝的影響及其與莖腐病抗性的關系.中國農業科學,2007,40(12):2780-2787.
11王媛,楊紅玉,程在全.SA誘導擬南芥對灰霉病的抗性與木質素含量的關系.植物保護,2007,33(4):50-54.
12李惠霞,王蒂.馬鈴薯晚疫病抗性反應中木質素及防御酶活性的變化.甘肅農業大學學報,2006,41(3):52-56.
13鄭喜清,王國,宋青山.不同哈密瓜品種對細菌性果斑病的抗性與木質素之間的關系.河套大學學報,2010,7(2):42-45.
14雒珺瑜,崔金杰,辛惠江.棉花葉片纖維素和木質素含量與綠盲蝽抗性的關系.西北農林科技大學學報:自然科學版,2012,40(4):81-85.
15 Saballos A,Sattler S E,Sanchez E,et al.Brown midrib2(Bmr2)encodes the major 4-coumarate:coenzyme A ligase involved in lignin biosynthe?sis in sorghum(Sorghum bicolor(L.)Moench).Plant J,2012,70(5): 818-830.
16 Humphreys J M,Chapple C.Rewriting the lignin roadmap.Curr Opin Plant Biol,2002,5(3):224-229.
17 Boerjan W,Ralph J,Baucher M.Lignin biosynthesis.Annu Rev Plant Biol,2003,54:519-546.
18 Lee D,Meyer K,Chapple C,et al.Antisense Suppression of 4-Couma?rate:Coenzyme A Ligase Activity in Arabidopsis Leads to Altered Lignin Subunit Composition.Plant Cell,1997,9(11):1985-1998.
19母洪娜,孫陶澤,徐晨,等.桂花(Osmanthus fragrans Lour.)4-香豆酸輔酶A連接酶(4CL)基因克隆與表達分析.分子植物育種,2016,14 (3):536-541.
20霍松,陳慧,朱瓊華,等.象草4CL基因片段的克隆及RNAi表達載體構建.草業學報,2012,21(1):296-301.
21饒國棟,張永卓,魏弘宜,等.毛白楊4-香豆酸:輔酶A連接酶(4CL3)的酶學特征研究.林業科學研究,2013,26(5):542-547.
22曹運鵬,方志,李姝妹,等.碭山酥梨4CL基因家族的全基因組鑒定與分析.遺傳,2015,37(7):711-719.
23 Li Y,Kim J I,Pysh L,et al.Four isoforms of Arabidopsis thaliana 4-cou?marate:CoA ligase(4CL)have overlapping yet distinct roles in phenyl?propanoid metabolism.Plant Physiol,2015,169(4):2409-2421.
24劉文彬,郭輝力,劉歡,等.煙草Nt4CL與虎杖PcSTS基因的融合表達與功能分析.生物技術進展,2016,6(2):91-97.
25雒軍,王引權,溫隨超,等.當歸苯丙氨酸解氨酶基因片段克隆和組織特異性表達分析.草業學報,2014,23(4):130-137.
26 Kenneth J K,Thomas D S.Analysis of relative gene expression data us?ing real-time quantitutive PCR and the 2-ΔΔctmethod.Methods,2001,25 (4):402-408.
27 Kumar A,Ellis B E.4-coumarate:CoA ligase gene family in Rubus idae?us:cDNA structures,evolution,and expression.Plant Mol Biol,2003,51 (3):327-340.
28 Schneider K,H?vel K,Witzel K,et al.The Substrate Specificity-Deter?mining Amino Acid Code of 4-Coumarate:CoA Ligase.Proc Natl Acad Sci U S A,2003,100(14):8601-8606.
29李珊珊.香鱗毛蕨4CL基因家族的克隆及功能驗證.哈爾濱:東北農業大學博士學位論文,2015.
30楊冬梅,王學敏,高洪文,等.東方山羊豆4香豆酸:輔酶A連接酶(4CL)基因的克隆和熒光定量表達分析.草地學報,2010,18(4):533-538.
Cloning and BioinformaticsAnalysis of Gl4CL Gene in Glehnia littoralis
Song Jiejie1,Luo Hongmei2,Zhu Xunzhi3,Zhang Yu1,Gao Ting1
(1.Key Laboratory of Plant Biotechnology in Universities of Shandong Province,College of Life Sciences, Qingdao Agricultural University,Qingdao 266109,China;2.Key Laboratory of Bioactive Substances and Resources Utilization of Chinese Herbal Medicine,Ministry of Education,Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences&Peking Union Medical College,Beijing 100193,China; 3.School of Biology and Chemical Engineering,Jiangsu University of Science and Technology,Zhenjiang 212018,China)
This study was aimed to clone and analyze the open reading frame(ORF)of 4-coumarate:coenzyme A ligase (Gl4CL)gene in Glehnia littoralis.Based on the high-throughput sequencing of G.littoralis,the full-length cDNA of Gl4CL gene was cloned by the rapid amplification of cDNA ends(RACE)method.Physical and chemical properties, secondary structure and three-dimensional structure of Gl4CL protein were predicted.Real-time PCR was used to detect the expression of Gl4CL gene in roots and leaves of G.littoralis.A total of 1951 bp full-length cDNA of Gl4CL gene was obtained,which encoded a protein of 544 amino acids with a predicted molecular weight of 59.481 kDa and the isoelectric point of 8.20.The cDNA of Gl4CL gene included 1 635 bp of ORF,153 bp of 5′untranslated regions(5′UTR) and 163 bp of 3′UTR.The result of real-time PCR showed that Gl4CL gene was both expressed in roots and leaves of G. littoralis,while the expression of gene in roots was significantly higher than that in leaves.It was concluded that the study will lay the foundation for further study of Gl4CL gene in function and gene regulation.Through in-depth study of therelationship between the expression of Gl4CL gene and lignin,as well as the plant growth phenotypes,it is expected to obtain high yield and quality lines of Glehniae Radix with strong resistance to diseases and insect pests.
Glehnia littoralis,4-coumarate,coenzyme A ligase,gene clone,bioinformatics analysis
10.11842/wst.2017.04.011
R282.6
A
(責任編輯:馬雅靜,責任譯審:王晶)
2017-03-14
修回日期:2017-04-20
*青島市科技局青島市應用基礎研究計劃項目(14-2-4-89-jch):北沙參轉錄組的高通量測序及香豆素合成關鍵酶的克隆研究,負責人:高婷;山東省青島市青年教師成長計劃經費資助項目。
**通訊作者:高婷,副教授,博士,主要研究方向:植物資源學和分子生物學研究。

