象草基因的克隆及生物信息學(xué)分析
吳娟子 錢晨 張建麗


摘要:從象草中克隆了基因,并對(duì)其進(jìn)行了初步的生物信息學(xué)分析。利用轉(zhuǎn)錄組測(cè)序獲得的表達(dá)序列信息,設(shè)計(jì)1對(duì)特異性的PCR引物,應(yīng)用RT-PCR技術(shù)從象草中擴(kuò)增出CpCCoAOMT2基因。對(duì)獲得的基因進(jìn)行序列同源性搜索、結(jié)構(gòu)域搜索及3D結(jié)構(gòu)預(yù)測(cè)、分子系統(tǒng)進(jìn)化樹構(gòu)建等生物信息學(xué)分析。測(cè)序結(jié)果表明,CCoAOMT2基因全長(zhǎng)926 bp,含110 bp的5′-UTR、741 bp的CDS和75 bp的3′-UTR,共編碼246個(gè)氨基酸,GenBank登錄號(hào)為BankIt1975804;Blastn和BlastT分析結(jié)果顯示,[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因及其編碼的蛋白在核苷酸水平和氨基酸水平上與多種植物的CCoAOMT基因和CCoAOMT蛋白具同源性;預(yù)測(cè)CpCCoAOMT2蛋白相對(duì)分子質(zhì)量為27 253,等電點(diǎn)為508,是一個(gè)不含跨膜結(jié)構(gòu)域、不含信號(hào)肽分子的親水性、穩(wěn)定蛋白。分子進(jìn)化樹分析結(jié)果表明,CpCCoAOMT2蛋白與禾本科植物的CCoAOMT蛋白親緣關(guān)系最近,與裸子植物和蕨類植物關(guān)系較近,而與雙子葉植物的關(guān)系較遠(yuǎn)。
關(guān)鍵詞:象草;咖啡酰酸輔酶A-O-甲基轉(zhuǎn)移酶;基因克隆;生物信息學(xué)分析
中圖分類號(hào): Q785文獻(xiàn)標(biāo)志碼:
文章編號(hào):1002-1302(2017)16-0033-04
收稿日期:2016-12-14
基金項(xiàng)目:國家自然科學(xué)基金(編號(hào):31302025);江蘇省農(nóng)業(yè)科技自主創(chuàng)新資金[編號(hào):CX(14)5035];國家牧草產(chǎn)業(yè)技術(shù)體系鹽城綜合試驗(yàn)站項(xiàng)目(編號(hào):CARS-35);浙江省寧波市科技計(jì)劃(編號(hào):2016C10018)。
作者簡(jiǎn)介:吳娟子(1977—),女,湖北京山人,博士,副研究員,主要從事牧草育種和產(chǎn)業(yè)化關(guān)鍵技術(shù)研究。E-mail:jzwu2014@jaasaccn。
象草(Cenchrus purpureus Schum)是一種多年生禾本科C4植物,具有高生物量,高光合效率、多用途、不占用耕地的優(yōu)點(diǎn)。它是世界上生物量最高的草本,年生物量可達(dá)45 t/hm2。它可作為草食畜禽的優(yōu)質(zhì)青飼料,理想的水土保持作物,高質(zhì)量人造板、紙漿和木質(zhì)纖維素類能源的優(yōu)質(zhì)原料[1-7]。大量研究表明,植物細(xì)胞壁組成和特征對(duì)木質(zhì)纖維素類的高效利用具有重要意義,例如,提高其細(xì)胞壁的纖維素含量,降低其木質(zhì)素含量,可以改善其飼料品質(zhì)和木質(zhì)纖維素轉(zhuǎn)化利用效率[8-10];提高其木質(zhì)素含量,有利于其作為板材的性能。因此,木質(zhì)素是限制細(xì)胞壁降解利用的關(guān)鍵因素之一,如何合理改變象草木質(zhì)素組成和含量對(duì)于有效利用象草具有重要意義。
木質(zhì)素是一種復(fù)雜的酚類聚合物,木質(zhì)素單體合成是木質(zhì)素合成的關(guān)鍵環(huán)節(jié),研究表明,主要有10種酶參與了木質(zhì)素單體的合成。目前,國內(nèi)外通過改變這10種酶基因的表達(dá),調(diào)控植物木質(zhì)素含量與組分的研究進(jìn)展迅速,已相繼從多種不同的植物中分離克隆了多個(gè)木質(zhì)素生物合成代謝中的關(guān)鍵酶基因[10-11]。咖啡酰輔酶A甲基轉(zhuǎn)移酶(caffeoyl CoA O-methyltransferase,CCoAOMT)是植物木質(zhì)素生物合成過程中一類重要的甲基轉(zhuǎn)移酶,催化咖啡酰CoA甲基化為阿魏酰CoA[12-14]。國內(nèi)外的研究表明,抑制楊樹和輻射松[14]等植物的CCoAOMT基因表達(dá),植物木質(zhì)素含量下降,同時(shí)伴隨S/G比值增大,木質(zhì)素結(jié)構(gòu)變得疏松,易于去除,且不影響植物的正常生長(zhǎng)。迄今為止,已經(jīng)從玉米、小麥、大麥、水稻、綠竹、南荻和柳枝稷等禾本科植物中克隆了CCoAOMT基因。NCBI中已有一個(gè)象草CCoAOMT基因(核酸登錄號(hào):KJ9957361)的記錄,但未見其他研究和相關(guān)報(bào)道。本研究從象草中分離克隆了另1個(gè)CCoAOMT基因,命名為[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ],并對(duì)其進(jìn)行了生物信息學(xué)分析,以期為研究象草CCoAOMT基因及其編碼蛋白在象草木質(zhì)素代謝中的功能提供理論依據(jù),為象草的木質(zhì)素改良提供基礎(chǔ)。
1材料和方法
11試驗(yàn)材料
象草品系為eg7,種植于江蘇省農(nóng)業(yè)科學(xué)院試驗(yàn)田,在自然條件下生長(zhǎng),待植株生長(zhǎng)5個(gè)月后取植株中部的莖段,迅速置于液氮中,帶回實(shí)驗(yàn)室-80 ℃保存?zhèn)溆谩?/p>
12試驗(yàn)方法
121引物設(shè)計(jì)
基于筆者所在課題組完成的象草轉(zhuǎn)錄組測(cè)序數(shù)據(jù),利用Primer50軟件設(shè)計(jì)1對(duì)特異性PCR擴(kuò)增引物F:5′-GAGTAGTGGGCGACCTGTGA-3′和R:5′-GAAGGGGAAGGAAAACTTATTG-3′,引物序列由通用生物公司合成。
122象草總RNA的提取、純化及cDNA的合成
采用TRIzol(Invitrogen)試劑提取象草莖總RNA,用RNase-free DNase Ⅰ(Fermentas)除去象草總RNA樣品中的DNA污染,12% 瓊脂糖凝膠電泳檢測(cè)總RNA純化效果,NanoDrop2000檢測(cè)濃度。參照RevertAid First Strand cDNA Synthesis Kit(Fermentas)說明書進(jìn)行cDNA的合成。
123象草基因的RT-PCR擴(kuò)增
25 μL PCR反應(yīng)體系為:10×LA PCR Buffer 25 μL,MgCl2 25 μL,dNTP Mixture 4 μL,正、反向引物各05 μL,LA Taq DNA酶025 μL,ddH2O 1325 μL,模板cDNA 15 μL。PCR反應(yīng)使用降落PCR,PCR反應(yīng)程序?yàn)椋?4 ℃ 5 min;94 ℃ 30 s,退火(1~2循環(huán)70 ℃,3~4循環(huán)69 ℃,5~6循環(huán)68 ℃,7~8循環(huán)67 ℃,9~10循環(huán)66 ℃,11~12循環(huán)65 ℃,13~14循環(huán)64 ℃,15~16循環(huán)63 ℃,17~18循環(huán)62 ℃,19~20循環(huán) 61 ℃,21~22循環(huán)60 ℃,23~24循環(huán)59 ℃,25~26循環(huán) 58 ℃,27~28循環(huán)57 ℃,29~30循環(huán)56 ℃)每個(gè)循環(huán)40 s,72 ℃ 90 s;72 ℃ 10 min,4 ℃ 10 min。1%瓊脂糖凝膠電泳檢測(cè)PCR擴(kuò)增產(chǎn)物。采用凝膠回收試劑盒回收純化目的條帶,擴(kuò)增產(chǎn)物經(jīng)PCR驗(yàn)證與目標(biāo)條帶一致后送通用生物公司測(cè)序。[JP]endprint
124合成序列拼接、比對(duì)和生物信息學(xué)分析
所得的序列用Sequencher 48進(jìn)行拼接并根據(jù)峰圖進(jìn)行手工校正。測(cè)序完成后,3次測(cè)序結(jié)果中的2次共同核苷酸合并為基因片段最終序列。利用DNAMAN V6和BioXM26軟件進(jìn)行序列比對(duì)和氨基酸序列的推導(dǎo),并通過NCBI的BLAST工具與GenBank中的已知序列比較,進(jìn)行蛋白序列的相似性分析。利用ProtParam軟件在線分析蛋白質(zhì)的分子結(jié)構(gòu)和理化性質(zhì);采用TMHMM在線分析軟件預(yù)測(cè)蛋白質(zhì)的跨膜結(jié)構(gòu)域;通過SignalIP分析軟件對(duì)信號(hào)肽進(jìn)行預(yù)測(cè);蛋白質(zhì)的二級(jí)結(jié)構(gòu)和三級(jí)結(jié)構(gòu)預(yù)測(cè)分別使用在線分析軟件NPSA和SWISS-MODEL完成。利用MEGA606軟件對(duì)氨基酸序列進(jìn)行系統(tǒng)進(jìn)化樹分析。
2結(jié)果與分析
21象草莖基因全長(zhǎng)cDNA克隆
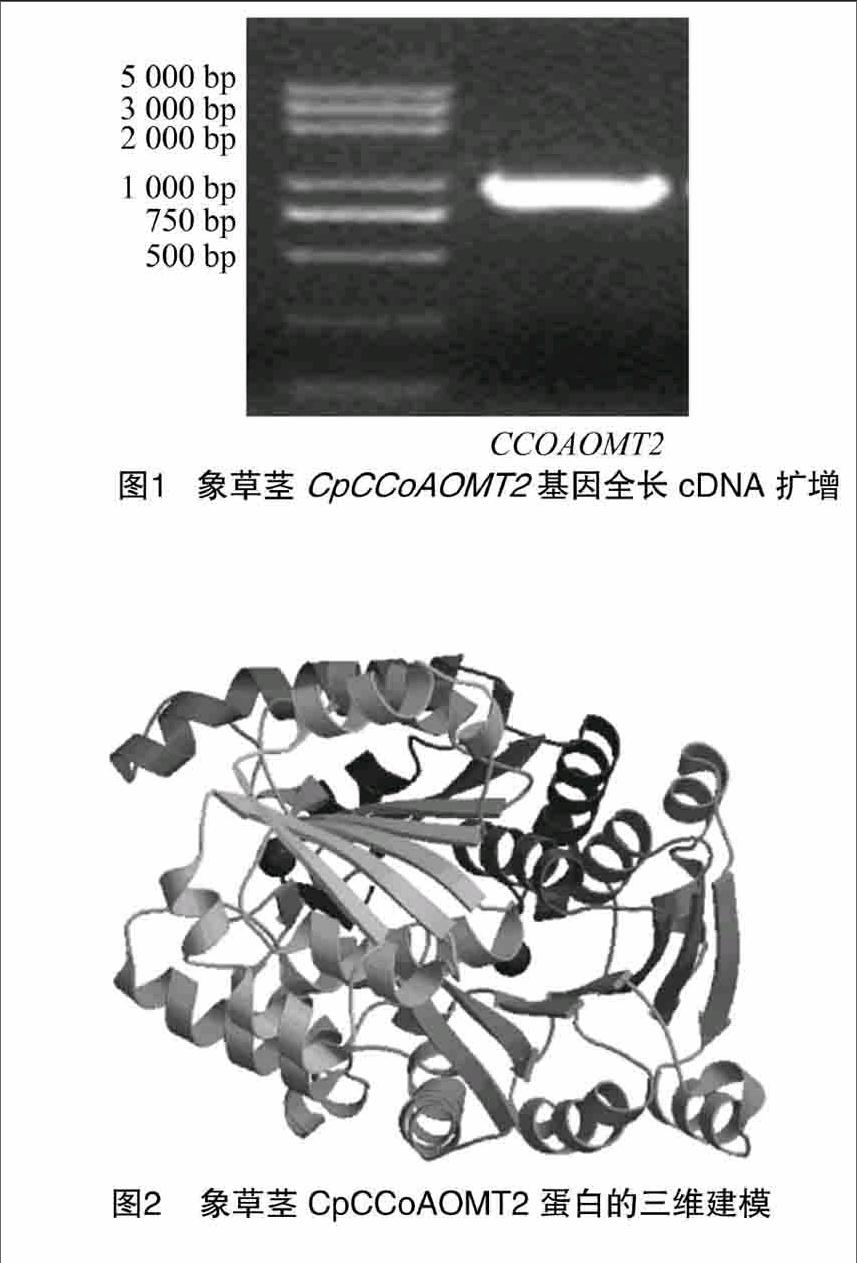
以象草莖中RNA為模板,利用F/R引物進(jìn)行RT-PCR擴(kuò)增,獲得了預(yù)期大小的cDNA片段(圖1)。將目的片段進(jìn)行回收、純化、驗(yàn)證并測(cè)序,獲得的CpCCoAOMT2基因全長(zhǎng)為926 bp,含110 bp的5′-UTR、75 bp的3′-UTR和1個(gè) 741 bp(111~851 bp)的開放閱讀框,起始密碼子為ATG,終止密碼子為TGA,G+C含量為649%,共編碼246個(gè)氨基酸,GenBank登錄號(hào)為BankIt1975804。
22象草CpCCoAOMT2蛋白的生物信息學(xué)分析
用在線網(wǎng)站(http://webExpasyorg/protparam)預(yù)測(cè)編碼蛋白質(zhì)的分子式為C1 212H1 926N328O365S10,總原子數(shù)為3 841個(gè),相對(duì)分子質(zhì)量為27 253。Leu、Ala和Asp在氨基酸組成中出現(xiàn)頻率較高,分別占總氨基酸的122%、102%和93%,而Trp、Cys和His出現(xiàn)的頻率較低,僅占 08%、12%和24%;理論pI為508,其中帶正電荷(Arg+Lys)和負(fù)電荷(Asp+Glu)殘基總數(shù)分別為27個(gè)和37個(gè);不穩(wěn)定系數(shù)為2605,是不穩(wěn)定蛋白;脂肪族系數(shù)為9793;親水性平均系數(shù)為-0100,屬親水性蛋白;半衰期為30 h。
TMHMM軟件(http://wwwcbsdtudk/services/TMHMM/)預(yù)測(cè)CpCCoAOMT2蛋白不存在跨膜結(jié)構(gòu)域。采用分析軟件SignalIP(http://wwwcbsdtudk/services/SignalP/)對(duì)獲得的CpCCoAOMT2氨基酸序列的信號(hào)肽序列進(jìn)行預(yù)測(cè),預(yù)測(cè)其沒有信號(hào)肽序列。通過NPS(https://npsa-prabiibcpfr/cgi-bin/secpred_hnnpl)預(yù)測(cè)CpCCoAOMT2氨基酸序列的二級(jí)結(jié)構(gòu),發(fā)現(xiàn)其多肽鏈中主要結(jié)構(gòu)元件是α螺旋和無規(guī)則卷曲,分別占4797%和3862%,延伸鏈占1341%;運(yùn)用SWISSMODEL工具(https://swissmodelexpasyorg)對(duì)CpCCoAOMT2的氨基酸序列進(jìn)行同源三維建模(圖2),模型以Automated mode方式構(gòu)建,建模范圍是從第19個(gè)到第245個(gè)氨基酸,其建模參考模板為高粱(Sorghum bicolor)咖啡酰輔酶A甲基轉(zhuǎn)移酶(登陸號(hào):5kva1A),目標(biāo)蛋白與參考蛋白的序列比對(duì)一致性為6344%。
23象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因推導(dǎo)酶蛋白氨基酸序列比較與分子進(jìn)化分析
在Pfam(http://pfamxfamorg)和NCBI上對(duì)獲得的[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]編碼的氨基酸序列進(jìn)行保守結(jié)構(gòu)域分析,發(fā)現(xiàn)CpCCoAOMT2具有Methyltransf_3(O-methyltransferase,PfamPF01596)和AdoMet_MTases(S-adenosylmethionine-dependent methyltransferases,cd02440)保守區(qū),是一種甲基轉(zhuǎn)移酶(圖3)。將[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]編碼的氨基酸序列與柳枝稷(Panicum virgatum,BAO208821)、玉米(Zea mays,NP_0011525111)和水稻(Oryza sativa,BAG925181)編碼的CCoAOMT氨基酸序列進(jìn)行多重比對(duì)分析,其與柳枝稷和玉米的CCoAOMT酶蛋白序列同源性高達(dá)93%、90%,具有高度的保守性,且具有植物甲基酶所共有的A、B、C保守序列元件和CCoAOMT基因家族中所特有的標(biāo)簽序列D、E、F、G、H等5個(gè)保守序列元件[15](圖4)。
象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因推導(dǎo)的酶蛋白氨基酸序列與其他植物已報(bào)道的相應(yīng)序列進(jìn)行多序列比較,并構(gòu)建系統(tǒng)樹(圖5)。結(jié)果顯示,象草CpCCoAOMT2酶蛋白氨基酸序列與禾本科的柳枝稷(Panicum virgatum:BAO208821)、玉米(Zea mays:NP_0011525111)的同源性相對(duì)較高;與禾本科的水稻(Oryza sativa:BAG925181、BAG984141)、小麥(Triticum aestivum:BAD063211)的同源性次之;象草CpCCoAOMT2酶蛋白氨基酸序列與NCBI中已有記錄的象草CpCCoAOMT(Cenchrus purpureus:AIN395351)的同源性相對(duì)較低;而CpCCoAOMT與禾本科中的美洲狼尾草(Cenchrus americanus:ALL436671)、 柳枝稷(Panicum virgatum:BAO208811) 和芒草(Miscanthus lutarioriparius:AEJ830601)的同源性相對(duì)較高。
在系統(tǒng)進(jìn)化樹分析中可見,本研究所克隆的象草[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]基因與禾本科植物的同類基因親緣關(guān)系最近,與裸子植物(臺(tái)灣杉,Taiwania cryptomerioides;臺(tái)灣扁柏,Chamaecyparis formosensis)和蕨類植物(江南卷柏,Selaginella moellendorffii)關(guān)系較近,而與雙子葉植物關(guān)系的較遠(yuǎn)(擬南芥,Arabidopsis thaliana;油菜Brassica napus;亞麻Camelina sativa)(圖5、表1),本結(jié)論與李雪平等在禾本科植物綠竹和南荻中的研究結(jié)果[16-17]類似,也符合象草分類學(xué)中的地位。endprint
3討論
本研究從象草品系eg7中克隆了1個(gè)[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]全長(zhǎng)基因,[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]編碼的氨基酸序列保守結(jié)構(gòu)域分析發(fā)現(xiàn),其具有Methyltransf_3和AdoMet_MTases保守結(jié)構(gòu)域,是一種甲基轉(zhuǎn)移酶。對(duì)[WTBX][STBX]CpCCoAOMT2[WTBZ][STBZ]編碼的氨基酸序列進(jìn)行多重比對(duì)發(fā)現(xiàn),CpCCoAOMT2不僅包含了植物甲基轉(zhuǎn)移酶中普遍存在的A、B和C 3個(gè)保守序列元件,還包含CCoAOMT基因家族中所特有的標(biāo)簽序列,即D、E、F、G和H 5個(gè)保守序列元件[CM(25],說明該基因具有CCoAOMT類基因所有的標(biāo)志性特征元
件[15],因此推測(cè)CpCCoAOMT2是CCoAOMT基因家族的一個(gè)成員,但其具體功能還有待于進(jìn)一步研究。在系統(tǒng)發(fā)育樹分析中,CpCCoAOMT2基因與禾本科植物的同類基因親緣關(guān)系最近,與裸子植物和蕨類植物關(guān)系較近,而與雙子葉植物較遠(yuǎn),本結(jié)論與李雪平等在禾本科植物綠竹和南荻的研究結(jié)果[16-17]類似,也與象草分類學(xué)中的位置一致。
參考文獻(xiàn):
[ZK(#]del Río J C,Prinsen P,Rencoret J,et al Structural characterization of the lignin in the cortex and pith of elephant grass (Pennisetum purpureum) stems[J] Journal of Agricultural and Food Chemistry,2012,60(14):3619-3634
Bhandari A P,Sukanya D H,Ramesh C R Application of isozyme data in fingerprinting Napier grass (Pennisetum purpureum Schum) for germplasm management[J] Genetic Resources & Crop Evolution,2006,53(2):253-264
[3]Strezov V,Evans T J,Hayman C Thermal conversion of elephant grass (Pennisetum purpureum Schum) to bio-gas,bio-oil and charcoal[J] Bioresource Technology,2008,99(17):8394-8399
[4]Harris K Genetic relationships among napiergrass (Pennisetum purpureum Schum) nursery accessions using AFLP markers[J] Plant Genetic Resources,2010,8(1):63-70
[5]Jakob K,Zhou F S,Paterson A H,et al Genetic improvement of C4 grasses as cellulosic biofuel feedstocks[J] Vitro Cellular & Developmental Biology Plant,2009,45(3):291-305
[6]Liu X H,Shen Y X,Lou L Q,et al Copper tolerance of the biomass crops elephant grass (Pennisetum purpureum Schumach),vetiver grass (Vetiveria zizanioides) and the upland reed (Phragmites australis) in soil culture[J] Biotechnology Advances,2009,27(5):633-640[HT][HJ][HT][FL)][LM]
[KH4D]
[HT8]
[7][ZK(#]Somerville C,Youngs H,Taylor C,et al Feedstocks for lignocellulosic biofuels[J] Science,2010,329(5993):790-792
[8]Himmel M E,Ding S Y,Johnson D K,et al Biomass recalcitrance:engineering plants and enzymes for biofuels production[J] Science,2007,315(5813):804-807
[9]Li X,Ximenes E,Kim Y,et al Lignin monomer composition affects Arabidopsis cell-wall degradability after liquid hot water pretreatment[J] Biotechnology for Biofuels,2010(3):27
[10][ZK(#]Simmons B A,Loqué D,Ralph J Advances in modifying lignin for enhanced biofuel production[J] Current Opinion in Plant Biology,2010,13(3):313-320
[11]Bonawitz N D,Chapple C The genetics of lignin biosynthesis:connecting genotype to phenotype[J] Genetics,2010,44(44):337-363
[12]Zhong R,Iii W H,Negrel J,et al Dual methylation pathways in lignin biosynthesis[J] Plant Cell,1998,10(12):2033-2046
[13]Zhong R,Morrison W H,Himmelsbach D S,et al Essential role of caffeoyl coenzyme A O-methyltransferase in lignin biosynthesis in woody poplar plants[J] Plant Physiology,2000,124(2):563-578
[14]Wagner A,Tobimatsu Y,Phillips L,et al CCoAOMT suppression modifies lignin composition in Pinus radiata[J] The Plant Journal,2011,67(1):119-129
[15]王華美,于延沖,付春祥,等 木質(zhì)素合成關(guān)鍵酶咖啡酰輔酶A氧甲基轉(zhuǎn)移酶的研究進(jìn)展[J] 基因組學(xué)與應(yīng)用生物學(xué),2014,32(2):458-466
[16][JP2]李雪平,高志民,彭鎮(zhèn)華,等 綠竹咖啡酰輔酶A-O-甲基轉(zhuǎn)移酶基因的克隆與分析[J] 分子植物育種,2008,6(3):587-592[JP]
[17]洪艷云,黃志剛,易圖永 南荻CCoAOMT基因的克隆與生物信息學(xué)分析[J] 中南林業(yè)科技大學(xué)學(xué)報(bào),2011,31(10):94-99endprint

