蛋白質復合物識別算法綜述
湯希瑋
(湖南第一師范學院信息科學與工程學院,湖南 長沙 410205)
蛋白質復合物識別算法綜述
湯希瑋
(湖南第一師范學院信息科學與工程學院,湖南 長沙 410205)
蛋白質復合物對理解細胞功能和細胞組織原則具有重要作用.高通量技術識別蛋白質復合物仍然相當不成熟.計算方法識別蛋白質復合物能在很大程度上彌補實驗方法的不足.為能給生物學家提供有價值的參考意見,匹配統計量評估方法被用于比較八種典型的計算方法.評估結果顯示,各種計算方法識別出的復合物均能較好地匹配真實的復合物.同時,不同的算法又具有各自不同的優勢和不足,這給蛋白質復合物識別算法研究提供了上升空間.
蛋白質相互作用;蛋白質復合物;識別算法
蛋白質復合物由蛋白質-蛋白質相互作用網絡中兩兩相互作用并緊密結合在一起的蛋白質組成.它能實現各種分子機制,進而執行大量生物功能,因此研究蛋白質復合物對理解細胞功能和細胞組織原則有著重要作用.如何從蛋白質-蛋白質相互作用網絡中識別蛋白質復合物是生物信息學研究中的關鍵問題.早期的研究注重通過生物實驗確定蛋白質復合物,但生物實驗不僅耗費巨大,效率不高,而且測不準.與此同時,隨著高通量技術的發展,大量蛋白質相互作用數據不斷涌現[1, 2],這為通過計算方法識別蛋白質復合物打下了堅實的基礎.因為蛋白質-蛋白質相互作用網絡可以轉化為一張圖,圖中的結點代表蛋白質,結點之間的邊表示蛋白質之間存在的相互作用關系,那么識別蛋白質復合物的科學問題就可以轉化為從圖中識別密度子圖的計算問題[3, 4].過去十年,出現了大量識別蛋白質復合物的計算方法.本文選擇八種典型的計算方法分別運行于蛋白質相互作用網絡,識別蛋白質復合物,然后采用被廣泛接受的評價方法來確定這些復合物的生物學意義.一方面是為生物學家在選擇計算工具時提供有價值的參考,另一方面也是為了蛋白質復合物識別算法研究提供新思路.
1 蛋白質復合物識別算法
在這一部分,我們按照算法發表的年代順序逐一描述八種典型的計算方法.
算法一:分子復合物偵測算法(Molecular Complex Detection,MCODE).MCODE算法依據蛋白質-蛋白質相互作用網絡中蛋白質之間的連通性確定蛋白質復合物,它能過濾非密度子圖并識別出有重疊的密度子圖(即蛋白質復合物)[5].該算法首先計算每個結點的局部鄰居密度,并用密度值給該結點打分,然后,選擇分值高的結點構建種子結點集合,最后從集合中的種子出發擴展成聚類.
算法二:馬爾科夫聚類算法(Markov Clustering,MCL).
馬爾科夫聚類算法在識別蛋白質復合物的過程中表現出了很強的健壯性[6].MCL算法在蛋白質-蛋白質相互作用網絡中模擬隨機游走,設計了膨脹和擴展兩個操作,控制加權或非加權鄰接矩陣.擴展操作將新概率值賦給所有的成對結點,膨脹操作通增加密度子圖內部的游走概率并降低密度子圖之間的游走概率來改變圖中的游走方向.算法迭代執行這兩個操作將網絡分離為許多的密度子圖.
算法三:邊緣密度圖聚類算法(Density-Periphery based graph Clustering ,DPClus).
Amin等人[7]提出的DPClus算法通過跟蹤密度子圖的邊緣識別蛋白質復合物.DPClus首先基于邊的兩個頂點之間的共同鄰居給每條邊加權,進一步通過已加權的度,再給頂點加權.為了形成蛋白質復合物,DPClus先將具有最高權值的節點(種子節點)選為初始聚類,然后按照優先級,從鄰居中選擇頂點,迭代擴大聚類.鄰居頂點的優先級由兩個條件確定:(1)鄰居頂點和聚類中每個頂點相連的邊的權值的總和;(2)鄰居頂點和聚類中每個頂點相連的邊的總數.該算法不能用于節點數超過5000個的大規模蛋白質-蛋白質相互作用網絡.
算法四:CFinder算法.
CFinder算法能識別蛋白質-蛋白質相互作用網絡中的功能模塊(或者蛋白質復合物)[8].CFinder算法將團的概念進一步擴展為K-團,K-團包括k個結點.兩個相鄰的K-團共享(k-1)個結點.該算法將所有相鄰的K-團連接在一起構成更大的密度子圖即K-團擴散聚類.這樣,CFinder算法借助團擴散方法能夠識別出重疊的密度子圖(蛋白質復合物).
算法五:基于極大團的聚類算法(Clustering method based on Maximal Clique,CMC).
CMC是劉桂梅等人[9]提出的基于極大團識別蛋白質復合物的聚類算法.CMC首先用極大團挖掘算法獲得所有極大團,然后基于文獻[10]中可靠性度量給每個相互作用指派一個分值.那么每個團都能通過其加權密度得到一個分值.最后,CMC移除或者合并高度重疊的團,生產蛋白質復合物.如果兩個團高度重疊,CMC或者合并兩個團為一個更大的團,或者簡單地刪除分值更低的團.
算法六:核心擴展算法(COre-AttaCHment,COACH).
為了深刻理解蛋白質復合物的組織結構,吳敏等人[11]提出了一個稱之為COACH的蛋白質復合物識別算法.該算法分兩個階段,在第一階段,COACH從鄰接圖中定義核心頂點,然后識別蛋白質復合物的核心;在第二階段,COACH將附加節點加入到核心中,形成有生物意義的結構.
算法七:速度與性能聚類算法(Speed and Performance In Clustering,SPICi).
SPICi是一種快速聚類算法,能夠從大規模生物網絡中識別蛋白質復合物[12].通過定義結點的密度和支持度兩個概念,SPICi算法給網絡中的結點加權并選擇加權度最高的結點作為種子,以種子為初始聚類,從其鄰居集合中選擇密度和支持度足夠大鄰居結點加入聚類,迭代執行以上規則直到新加入結點的支持度和密度低于給定閾值為止,最后輸出聚類(即蛋白質復合物)并從網絡中刪除聚類中的結點.
算法八:層次聚類算法(Hierarchical Clustering Protein Interaction Networks,HC-PINs).
HC-PINs是一種快速層次聚類算法[13].該算法提出了邊聚類值的概念,用以描述邊的鄰居構成團的過程.邊聚類值概念的提出,有效地避免了蛋白質-蛋白質網絡中邊聚類系數偏小的局限.又基于強模塊和弱模塊定義了更靈敏的λ模塊.算法執行時,將網絡中每個節點(蛋白質)初始化為聚類并計算網絡中每條邊的邊聚類值,然后依據邊聚類值降序排列所有邊并不斷將具有最大邊聚類值的邊添加到聚類中,直到λ模塊出現.最后所有邊都被分配到不同的λ模塊中形成蛋白質復合物.
2 復合物評估方法
匹配統計量分析是一種常見的評價方法.它將算法識別出的復合物與已知的標準復合物進行比較和匹配.已知復合物是指由生物學家經過生物實驗技術如串聯親和純化等獲得的復合物.一直以來, MIPS(Munich Information Center of Protein Sequences)數據庫對酵母蛋白質復合物的分類被廣泛用于產生蛋白質復合物參考集(即已知蛋白質復合物的標準集合).盡管這種分類是有價值的,但是,它已經不能適應該領域的最新發展現狀了.所以,我們從CYC2008[14]下載了408個典型的復合物作為已知復合物,采用與文獻[5]相同的得分方案以確定識別出的復合物怎樣有效地匹配已知復合物.該方案采用重疊得分(OverlapScore,OS)具體描述復合物的匹配程度.OS的計算公式如下.

(1)
公式(1)中,i表示算法識別出的復合物與已知復合物之間共有的蛋白質數,a表示算法識別出的復合物中蛋白質的數量,b是指已知復合物中蛋白質的數量.OS的值為0意味著算法識別出的復合物與已知復合物沒有共同的蛋白質,二者完全不匹配;OS為1表示算法識別出的復合物與已知復合物完全重疊,是最佳匹配.
真陽性數(TP)是在OS大于某一閾值的條件下,算法識別出的復合物集合中至少能夠匹配一個已知復合物的復合物數目,假陽性數(FP)指算法識別出的復合物總數減去真陽性數(TP).假陰性數(FN)表示不能被算法識別出的復合物所匹配的已知復合物數目.從而可以定義靈敏度(Sn)[5]為:

(2)
特異性(Sp)[5]為

(3)
由此可以定義二者的調和平均值(F-score).

(4)
基于以上公式,我們設計并實現了一個Perl程序,它能自動完成算法輸出的所有蛋白質復合物的匹配分析并計算三個評價指標.
3 使用的生物數據集
我們使用來自DIP(Database of Interacting Proteins,http://dip.doe-mbi.ucla.edu/dip/Download.cgi?SM=7/)數據庫中釀酒酵母的蛋白質相互作用網絡作為算法評價分析的原始數據.該網絡包含4950個節點(蛋白質)和21788條邊(相互作用).之所以沒有選擇其他數據庫或其他物種,是因為DIP數據庫中的基因產物的相互作用都是通過實際的生物實驗檢測到的,相對而言,其假陽性比較小,數據可靠性較高.與其他物種相比,釀酒酵母的蛋白質-蛋白質相互作用網絡數據是最全面的.
4 實驗結果比較和分析
我們分別用MCODE、MCL、CFinder、DPClus、CMC、COACH、SPICi和HC-PINs在釀酒酵母的蛋白質-蛋白質相互作用網絡上識別復合物.然后將各自得到的復合物與已知復合物進行匹配,從整體上評價算法識別蛋白質復合物的質量.由于生物學家主要關注計算方法所產生的處理結果的生物學意義,所以我們不比較這八種算法的時間復雜度和空間復雜度.各算法的控制參數值來自算法設計的推薦.
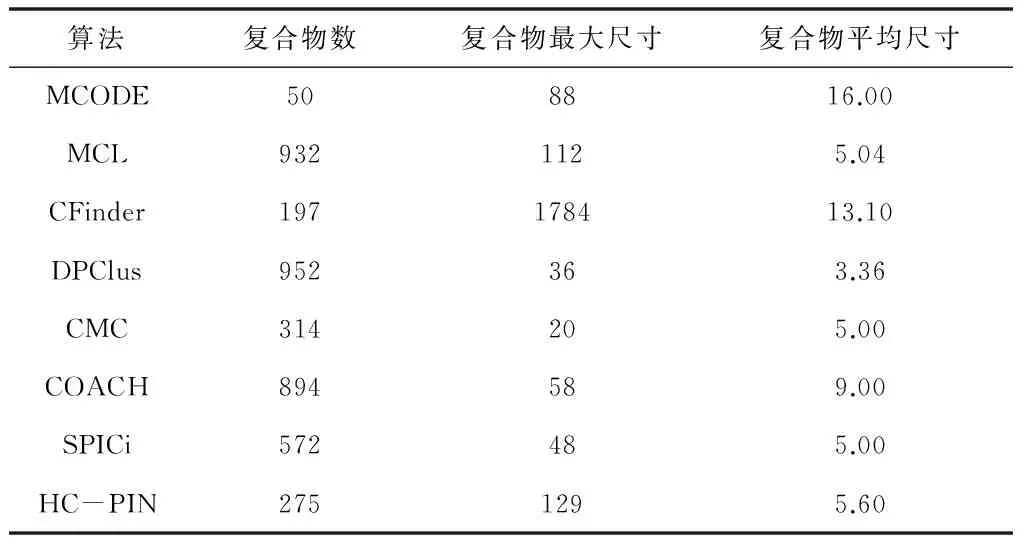
表1 八種算法識別出來的復合物的情況
表1顯示了八種算法從蛋白質-蛋白質相互作用網絡中識別出來的復合物的基本屬性.其中,復合物最大尺寸是指包含最多蛋白質的復合物中蛋白質的數量,復合物平均尺寸是某個算法識別出的所有復合物包含的蛋白質的平均數.
從表1中可以看出,MCODE算法識別了50個蛋白質復合物,其中最大的復合物有88個蛋白質,所有復合物平均擁有16個蛋白質.MCODE算法依據圖的局部密度擴充聚類,因此并不是蛋白質-蛋白質網絡中的所有蛋白質都被指派到復合物中.與MCODE算法不同,MCL算法將蛋白質-蛋白質相互作用網絡中的每個蛋白質都分配到相應的復合物中,它識別了932個復合物,僅次于DPClus算法的952個.CFinder算法識別了197個復合物,最大復合物覆蓋了1784個蛋白質.MCL算法和SPICi算法都是將蛋白質-蛋白質相互作用網絡分離為沒有重疊的復合物,而其他算法采用一步一步的方式產生的復合物集合中包含了重疊的復合物.

圖1 識別復合物與已知復合物在不同OS值上的匹配情況.
圖1顯示了匹配分析的綜合比較結果.圖1顯示在OS的主要區間上,MCODE算法產生的復合物匹配上的已知復合物的數量最少,算法性能最差,相反COACH算法匹配的已知復合物的數量最多,表現最佳.文獻[5]指出,當0.3≥OS≥0.2時,不具有生物學意義的蛋白質復合物已經被丟棄了.在這個范圍內COACH算法匹配的蛋白質復合物數量最多,表現出了最佳的性能.
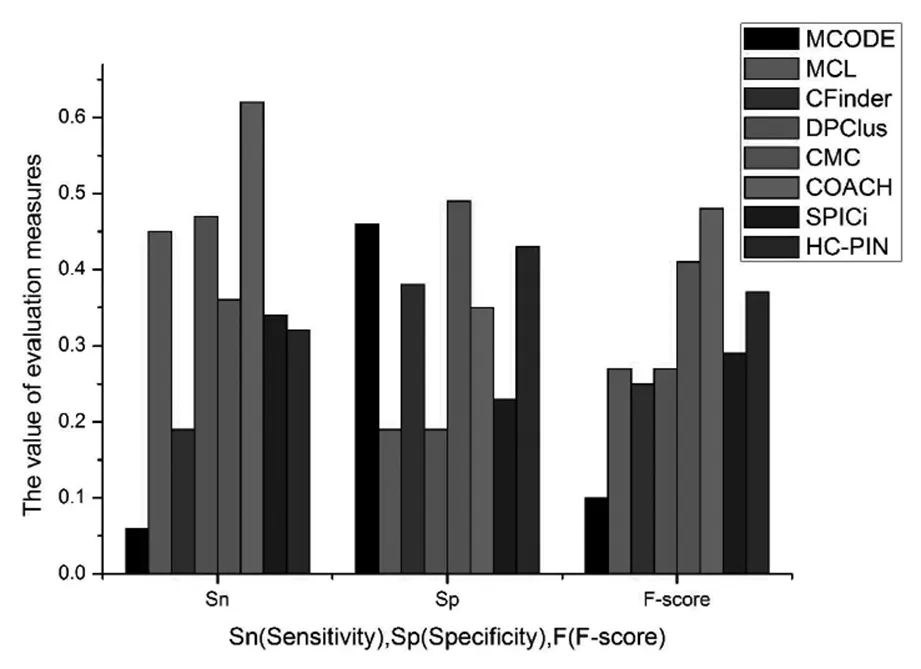
圖2 基于三種評價指標的算法性能比較.
另外,我們的評估結果還顯示 COACH算法產生了15個與已知復合物完全重疊的復合物,而HC-PINs產生了18個這樣的復合物.
既然當0.3≥OS≥0.2時,大部分復合物都有生物意義,所以,我們取SO=0.2為閾值,計算了八種算法的靈敏度Sn、特異性Sp與調和平均值F-score.從圖2可以看出,與其他算法相比,COACH、DPClus和MCL的靈敏度較高,這是由于它們識別出的復合物數量較多,則能夠與已知復合物進行匹配的復合物數量也越多,所以靈敏度較高,SPICi的靈敏度較高也是這個原因.CFinder識別出了一個巨大的復合物,該復合物能夠匹配的已知復合物數量應該是比較多的,導致其特異性較高,但其靈敏度并不高,可能的原因仍然是CFinder識別出的復合物數量偏少.CMC算法識別出的復合物數量遠少于SPICi,而且最大復合物包含的蛋白質數也比SPICi對應的復合物包含的蛋白質數要少,但其靈敏度反而比SPICi高.這說明,CMC算法識別出的復合物能與更多的已知復合物匹配.與其他算法相比,CMC算法具有最高的特異性,這正說明其識別的蛋白質復合物質量高.通過以上分析,可以看出,單純憑借靈敏度或特異性,都不足以說明算法的好壞.調和平均值F-score可以從一定程度上彌補了二者的不足,從數量和質量上,綜合評價算法的性能.從F-score來看,COACH算法的性能最好,CMC算法次之.
匹配分析結果顯示,盡管有些計算方法(如COACH、MCODE和HC-PIN)的性能在某些方面比其他計算方法要優異,但是,沒有一種方法能占住絕對主導地位,全面超越其他算法.相反,實際上每種方法都各有優勢以及不足,這也表明計算方法識別蛋白質復合物的研究仍有較大發展空間.
5 結束語
蛋白質復合物是執行細胞功能的關鍵分子實體.日益增加的大量蛋白質相互作用數據使得從蛋白質-蛋白質相互作用網絡中識別蛋白質復合物成為可能.但也僅僅只是可能,因為高通量技術雖然能通過生物實驗確定蛋白質-蛋白質之間的相互作用關系,但是并不能識別蛋白質復合物.幸運的是,計算方法正好能充當跨越鴻溝的橋梁.許多研究者對此展開了卓有成效的研究.本文選擇的八種蛋白質復合物識別算法正是這一研究領域的典型代表.我們的實驗結果表明各種算法雖然性能有高下之分,但是,識別出來的復合物都有一定的生物意義.這也暗示計算方法有助于生物學家繼續尋找新的蛋白質復合物.
[1]Hamp T, Rost B. Evolutionary profiles improve protein-protein interaction prediction from sequence[J]. Bioinformatics, 2015, (12): 1945-1950.
[2]Li T, Wernersson R, Hansen R B, et al. A scored human protein-protein interaction network to catalyze genomic interpretation[J]. Nature Methods, 2017, (1): 61-64.
[3]You Z H, Li X, Chan K C. An improved sequence-based prediction protocol for protein-protein interactions using amino acids substitution matrix and rotation forest ensemble classifiers[J]. Neurocomputing, 2017, 228: 277-282.
[4]Wang L, You Z H, Chen X, et al. An ensemble approach for large-scale identification of protein-protein interactions using the alignments of multiple sequences[J]. Oncotarget, 2017,(3): 5149-5159.
[5]Bader G, Hogue C. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, https://doi.org/10.1186/1471-2105-4-2.
[6]Van Dongen S. Graph Clustering by Flow Simulation[D]. Utrecht: PhD Thesis of Utrecht University, 2000.
[7]Altaf-Ul-Amin M, Shinbo Y, Mihara K, et al. Development and implementation of an algorithm for detection of protein complexes in large interaction networks[J]. BMC Bioinformatics, 2006,(1):1-13.
[8]Adamcsek B, Palla G, Farkas I J, et al.: locating cliques and overlapping modules in biological networks[J]. Bioinformatics, 2006, (8): 1021-1023
[9]Liu G M, Chua H N, Wong L. Complex discovery from weighted PPI networks[J]. Bioinformatics, 2009, (15):1891-1897.
[10]Liu G, Li J, Wong L. Assessing and Predicting Protein Interactions Using Both Local and Global Network Topological Metrics[J]. Genome Informatics, 2008, 21:138-149.
[11]Wu M, Li X L, Kwoh C K, et al. A core-attachment based method to detect protein complexes in PPI networks[J]. BMC Bioinformatics, 2009, (1):169.
[12]Peng J, Mona S. SPICi: A fast clustering algorithm for large biological networks[J]. Bioinformatics, 2010, (8): 1105-1111.
[13]Wang J, Li M, Chen J, et al. A fast hierarchical clustering algorithm for functional modules discovery in protein interaction networks[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2011,(3): 607-620.
[14]Pu S, Wong J, Turner B, et al. Up-to-date catalogues of yeast protein complexes[J]. Nucleic Acids Res, 2009,(3): 825-831.
ASurveyonComputationalApproachesforIdentifyingProteinComplex
TANG Xiwei
(School of Information Science and Engineering, Hunan First Normal University, Changsha Hunan 410205, China)
Protein complexes are important for understanding principles of cellular organization and function. The high-throughput technologies for detecting protein complexes remain relatively immature. Computational approaches for identifying protein complexes are useful complements to the limited experimental methods. Eight state-of-the-art techniques for computational prediction of protein complexes are evaluated by matching method. Results show that the complexes identified by the eight computational methods match well with actual protein complexes. Meanwhile, different algorithms have their own comparative advantages and disadvantages, indicating that more research should be done in identifying protein complexes by computing methods.
protein-protein interaction; protein complex; identify algorithm
TP301.6
A
1008-4681(2017)05-0019-05
2017-09-07
國家自然科學基金面上項目“基于生物網絡的人類疾病基因識別算法研究”(批準號:61472133).
湯希瑋(1973— ),男,湖南常德人,湖南第一師范學院信息科學與工程學院副教授,博士.研究方向:生物信息學.
(責任編校:晴川)

