普通小麥籽粒過氧化物酶活性全基因組關聯分析
時佳,翟勝男,劉金棟,魏景欣,白璐,高文偉,聞偉鍔,,何中虎,夏先春,耿洪偉
?
普通小麥籽粒過氧化物酶活性全基因組關聯分析
時佳1,翟勝男2,劉金棟2,魏景欣2,白璐3,高文偉1,聞偉鍔1,2,何中虎2,夏先春2,耿洪偉1
(1新疆農業大學農學院/新疆農業大學生物技術重點實驗室,烏魯木齊830052;2中國農業科學院作物科學研究所/國家小麥改良中心,北京100081;3新疆農業大學科學技術學院,烏魯木齊830091)
小麥籽粒過氧化物酶(peroxidase,POD)活性對面制品加工品質有重要影響,發掘控制籽粒POD活性重要位點,并篩選其候選基因,為小麥品質的改良奠定基礎。以151份黃淮冬麥區和82份北部冬麥區品種(系)為材料,分別利用來自于小麥90 K SNP芯片的18 189和18 417個高質量SNP標記,對POD活性進行全基因組關聯分析(genome-wide association study,GWAS)。供試材料中POD活性表現出廣泛的表型變異和多樣性,黃淮麥區材料的POD活性變異系數為15.4%—21.8%,遺傳力為0.79,北部麥區材料的POD活性變異系數為15.0%—19.9%,遺傳力為0.82。相關性分析表明,不同環境之間材料的POD活性表現出顯著的相關性,黃淮麥區相關系數為0.46—0.89(<0.0001),北部麥區相關系數為0.50—0.87(<0.0001)。多態性信息含量值為0.09—0.38,最小等位基因頻率MAF值為0.05—0.5。群體結構分析表明,黃淮麥區與北部麥區2個自然群體結構簡單,均可分為3個亞群。GWAS分析結果表明,在黃淮冬麥區材料中共檢測到20個與POD活性顯著關聯的位點(<0.001),分布在1A、2A、2B、2D、3A、3B、3D、4A、4B、5A、5B、6A、6D和7A染色體上,單個位點可解釋7.8%—13.3%的表型變異。在北部冬麥區材料中共檢測到20個與POD活性顯著關聯(<0.001)的位點,分布在1A、1B、1D、2A、2B、2D、3A、3B、4B、6A、6B、7A、7B和7D染色體上,單個位點可解釋14.4%—23.2%的表型變異。加性回歸分析表明,隨著優異等位基因數量的增多,小麥籽粒POD活性越高。在發現的所有POD活性相關位點中,2個位點在黃淮麥區和北部麥區材料中均能檢測到且穩定遺傳,可將其轉換為STARP(semi-thermal asymmetric reverse PCR)或CAPS標記,以應用于分子標記輔助育種。獲得3個與POD活性有關的候選基因,分別編碼磷酸甘露糖變位酶()、辣根過氧化物酶()和烷基氫過氧化物還原酶()。黃淮麥區與北部冬麥區2個自然群體遺傳多樣性豐富,群體結構簡單,適用于全基因組關聯分析。在2個自然群體中分別發現20個POD活性位點,并在顯著相關的位點區域內篩選到3個候選基因。含有越多優異等位變異的材料其POD活性越高。
普通小麥;POD活性;90 K SNP芯片;群體結構;候選基因
0 引言
【研究意義】面粉顏色是評價小麥品質的重要指標[1]。小麥籽粒中多酚氧化酶(polyphenol Oxidase,PPO)、過氧化物酶(peroxidase,POD)和脂肪氧化酶(lipoxidase,LOX)等是面粉和面制品在加工、儲藏過程中被漂白和發生褐變的主要原因[2-3]。POD具有與LOX類似的對胡蘿卜素等色素類物質的漂白作用,并可以作為主要的天然漂白劑與LOX同時加入到面粉中,使面團得以漂白,因此,高POD活性的小麥面粉白度更高[4]。另外,籽粒中的POD能催化阿魏酸等主要酚酸的氧化,并產生發色基團(如醌式結構),從而使面制品在制作和貯存過程中發生褐變[5-7]。目前,小麥籽粒LOX和PPO活性與顏色相關的指標在育種實踐中已受到重視,但對POD與顏色相關性的研究較少[8]。鑒于POD對面制品色澤存在雙重作用,深入開展普通小麥POD研究,對明確面制品顏色形成機制、影響因素及其改良策略具有重要意義。【前人研究進展】?ili?等[9]研究表明,普通小麥POD活性顯著高于硬粒小麥(<0.05)。在普通小麥的不同品種間POD活性可相差3—10倍[10]。因此,通過遺傳途徑改良POD活性是可行的。Wei等[11]利用豆麥/石4185重組自交系(recombinant inbred lines,RIL)群體的214個株系和7 391個SNP標記及一個新開發的STS標記對普通小麥POD活性進行了QTL分析,共檢測到3個QTL、和,它們在不同環境下分別解釋5.3%—9.2%、9.3%—21.2%和5.8%—11.7%的表型變異。連鎖分析理論上可以應用于所有性狀的遺傳解析,但受遺傳群體親本的差異度和群體大小等限制,定位QTL數目有限[12]。而關聯作圖則是一種能有效克服連鎖作圖局限的方法,連鎖作圖和關聯作圖相結合可以互為補充互為驗證[13]。近年來,關聯分析已被廣泛應用于各種作物復雜農藝性狀QTL的發掘。楊勝先等[14]、賴勇等[15]和張煥欣等[16]分別對大豆、大麥和玉米等作物進行了關聯分析研究。而在小麥育種工作中,陳廣鳳等[17]利用24 355個SNP標記對205份中國冬小麥品種進行關聯分析,共檢測到38個與株高相關的SNP,其中11個位點在2個以上環境穩定表達。Sun等[18]利用90 K芯片對163份黃淮麥區冬小麥品種進行了產量相關性狀GWAS,并發現了41個產量相關性狀的QTL位點。Dong等[19]利用90K芯片對166份黃淮麥區冬小麥品種進行了碳水化合物進行GWAS,并發現了23個產量相關性狀的QTL位點。【本研究切入點】雖然Wei等[11]用RIL群體對POD活性進行了全基因組連鎖分析,并發現了3個POD活性位點,但迄今為止尚未有POD活性GWAS的報道。【擬解決的關鍵問題】本研究以151份黃淮麥區和82份北部冬麥區小麥品種(系)2個自然群體為材料,利用Wheat 90K iSelect SNP芯片,對小麥籽粒POD活性進行GWAS分析,以發掘新的POD活性位點,解析小麥POD遺傳機制,為小麥POD活性的遺傳改良提供可用分子標記。
1 材料與方法
1.1 供試材料
供試材料包括233份冬小麥品種(系),其中151份來自黃淮冬麥區(Yellow and Huai River Valley Facultative Wheat Region, YHRVWWR),于2012— 2013和2013—2014年度種植于河南安陽和安徽濉溪;82份材料來自于北部冬麥區(Northern Winter Wheat Region,NWWR),于2012—2013和2013—2014年度種植于北京順義和河北石家莊。試驗采用隨機區組設計,3次重復,行長2 m,行距25 cm。上述試驗材料均由中國農業科學院作物科學研究所小麥品質課題組提供。
1.2 POD活性檢測
以25 μL的H2O2、5 μL 2%的愈創木酚和145 μL 0.05 mol·L-1的磷酸-檸檬酸緩沖液(pH 5.0)的混合液為底物,采用Wei等[13]的愈創木酚紫外分光光度法進行POD活性的檢測,每個小麥品種的POD活性重復檢測2次,2次檢測結果相差超過10%的進行重復檢測。
1.3 表型數據統計
采用SAS v9.2的PROC UNIVARIATE、RROC CORR程序分別進行描述性統計變量分析、相關分析和方差分析。利用R 3.31程序進行遺傳力計算。廣義遺傳力計算公式:h= σ/ (σ+ σ/r +σ/ re),其中σ、σ和σ分別表示基因型方差、基因型與環境互作方差和誤差方差,和分別表示環境個數和每個環境內的重復次數。
1.4 90 K SNP芯片基因型分型
基于Illumina測序平臺,應用小麥90 K iSelectSNP芯片(81 587個SNP)對233份小麥品種(系)進行SNP分型,由博奧生物技術有限公司(http://www. capitalbio.com)完成。利用Genome Studio軟件(http://www.illumina.com)進行SNP分型,具體操作參照Cavanagh等[20]方法。人工對分型結果進行質量控制,剔除數據缺失率>50%、雜合率>50%和最小等位基因頻率(minor allele frequency,MAF)<0.05的SNP標記,保留高質量的SNP標記進行關聯分析。使用Power Marker v3.25進行MAF和多態性信息含量(polymorphism information content,PIC)的運算[21]。
1.5 群體結構分析
關聯分析前進行群體結構分析可有效降低結果中假陽性概率。采用Structure v2.3.4的Admixture Ancestry模型分析2個自然群體的群體結構。
1.6 全基因組關聯分析
采用Tassel v5.0對2個自然群體進行kinship matrix(K matrix)運算。運用Tassel v5.0中的混合線性模型(mixed linear model,MLM),在考慮群體結構和親緣關系的情況下,進行SNP標記與POD活性的關聯分析。關聯分析結果中多個SNP標記是否位于同一位點由LD衰減距離決定。在<0.001水平進行SNP標記和籽粒POD活性顯著性檢測。利用R語言對關聯分析結果繪制Manhattan圖和quantile-quantile(Q-Q)圖。
1.7 候選基因篩選
以與POD活性顯著關聯的SNP標記序列為探針,在NCBI(http://www.ncbi.nlm.nih.gov/;National center for biotechnology information)和ENA(European Nucleotide Archive;http://www.ebi.ac.uk/ena)數據庫中進行BLASTx,篩選與POD活性相關的候選基因。-值<10-5,序列一致性大于75%。
2 結果
2.1 POD活性分析
151份黃淮麥區冬小麥材料在安陽2013、濉溪2013、安陽2014、濉溪2014及均值環境下的POD活性分別為507.0、513.8、781.4、和727.5 U·min-1g-1,變異系數為15.4%—21.8%。各環境之間的POD活性呈極顯著正相關,相關系數為0.46—0.89(<0.0001),遺傳力為0.79。83份北部冬麥區材料在北京2013、石家莊2013、北京2014、石家莊2014及均值環境下POD活性分別為591.6、567.5、899.5、858.9和729.4 U·min-1g-1,變異系數為15.0%—19.9%。各環境之間POD活性呈極顯著正相關,相關系數為0.50—0.87(<0.0001),遺傳力為0.82(表1)。以上結果表明中國冬小麥品種(系)的POD活性主要受遺傳因素控制,在早期世代對其進行選擇是有效的;且籽粒POD活性變異范圍廣,具有較大的選擇潛力。通過育種途徑選育高POD活性的品種,進而改良小麥面制品顏色是可行的。

表1 233份品種中POD含量統計分析
***表示在<0.0001水平差異顯著;SD:標準差;CV:變異系數
***Significant at<0.0001; SD: Standard deviation; CV: Variable coefficient
2.2 SNP標記的多態性及群體結構分析
利用90 K SNP芯片對151份黃淮麥區及82份北部冬麥區品種(系)進行檢測。其中黃淮麥區最終采用18 189個SNP標記進行GWAS分析,平均單條染色體包含866個標記,A、B和D組染色體分別包含1 007、1 338和254個標記;北部冬麥區品種GWAS分析選用18 417個標記,平均每條染色體含有877個標記,A、B和D組染色體平均包含1 005、1 357和269個SNP標記。在2個自然群體中,SNP標記圖譜長為3 700 cM,每個標記之間的平均遺傳距離為0.2 cM。在2個群體所選標記的A、B和D組染色體中,均表現為A與B組染色體標記密度顯著高于D組染色體(表2)。2個自然群體SNP標記值為0.09—0.38,MAF為0.05—0.50。
利用Structure v 2.3.4分別對2個自然群體進行遺傳結構分析。當ΔK=3時,2個自然群體的K值均達到最大值(圖1)。因此,2個群體均可分為3個亞群(圖2)。在黃淮麥區中,第一亞群(記為Pop1)含有57份品種,以山東品種(43.0%)為主,還包括陜西、河南及18份國外品種;第二亞群(記為Pop2)共含有48份品種,以河南品種(37.5%)為主,還包括部分陜西和安徽品種;第三亞群(記為Pop3)中含有46份品種,以河南品種(56.5%)為主。北部冬麥區自然群體的第一亞群(記為Grp1)含有39份品種,主要由國外品種(66.6%)組成,還包括8份北京和5份山西品種;第二亞群(記為Grp2)共有13份國內品種,主要由北京品種(84.6%)組成;第三亞群(記為Grp3)共有30份品種,以國外品種(46.6%)為主,還包含12份北京品種、1份河北品種、1份山西品種和1份寧夏品種。

表2 黃淮麥區與北部冬麥區群體關聯分析所用標記統計
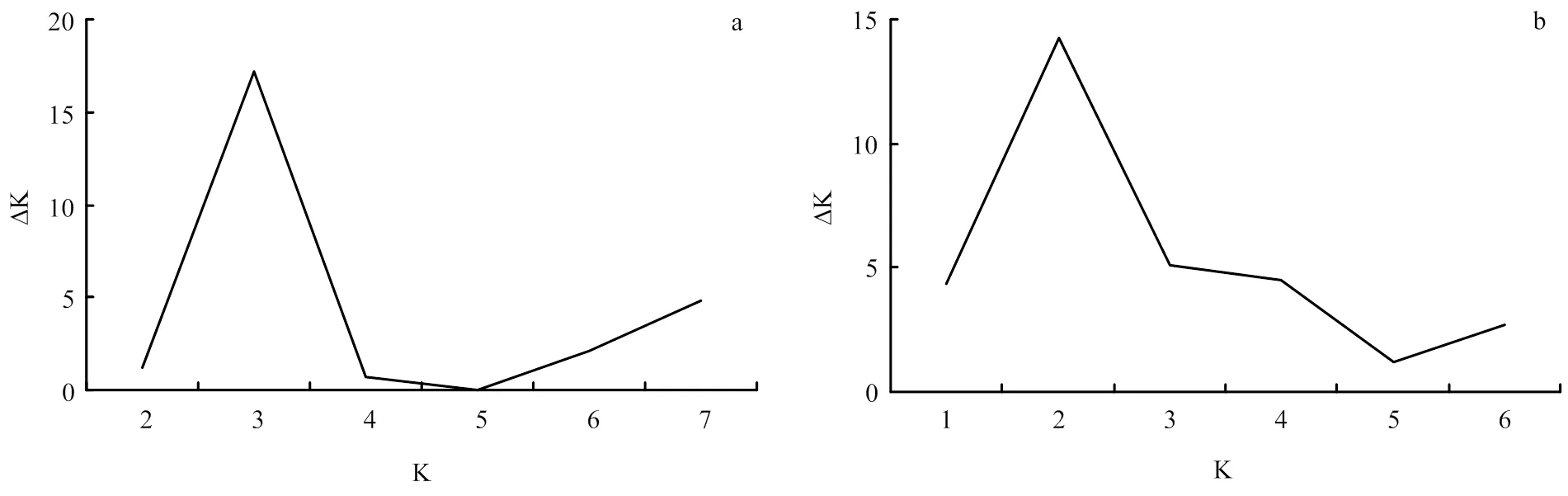
圖1 使用deltak值估計黃淮冬麥區(a)和北部冬麥區(b)品種亞群數

圖2 黃淮冬麥區(a)和北部冬麥區(b)品種的群體結構分析
2.3 POD活性全基因組關聯分析
對黃淮麥區151份品種(系)及北部冬麥區82份品種(系)的籽粒POD活性進行全基因組關聯分析。當≤0.001時,認為該標記與POD活性顯著關聯,多個環境檢測到的標記視為穩定遺傳標記(圖3)。由Q-Q圖可以看出,2個自然群體的群體結構控制較好,可以避免假陽性的出現(圖4)。黃淮麥區151份品種在A、B、D和全基因組水平下的LD衰減距離分別為5、7、11和7 cM,北部麥區82份品種在A、B、D和全基因組水平下的LD衰減距離分別為6、7、10和8 cM。GWAS結果中多個標記是否位于同一位點(Locus)由對應基因組的LD衰減距離決定。
在黃淮麥區共檢測到20個位點,包含86個與POD活性顯著關聯的SNP標記(<0.001),分別位于1A、2A、2B、2D、3A、3B、3D、4A、4B、5A、5B、6A、6D和7A染色體上,單個位點可解釋7.8%—13.7%的表型變異,其中4個位點至少在2個環境中穩定遺傳,分布在2A(177 cM)、2D(97—103 cM)、5A(80 cM)和6A(7 cM)染色體上。在北部冬麥區群體中篩選到20個位點,包含51個與POD活性顯著關聯的SNP標記(<0.001),分布在1A、1B、1D、2A、2B、2D、3A、3B、4B、6A、6B、7A、7B和7D染色體上,單個位點可解釋14.4%—23.2%的表型變異,其中4個位點至少在2個環境中穩定遺傳,分別位于2A(177 cM)、2D(97—99 cM)、7B(98—99 cM)和7D(182—184 cM)染色體上。在發現的所有POD活性相關位點中,2A(177cM,)和2D(99 cM,)2個位點在2個自然群體中均能檢測到,且在多個環境下穩定存在(表3)。
2.4 優異等位變異與POD活性的回歸分析
每個SNP標記含有2個等位變異,其中與高POD活性對應的等位變異則為優異等位變異。2個自然群體的優異等位變異的加性回歸分析表明,黃淮麥區151份冬小麥品種(系)中含有1—16個優異等位基因,POD活性與優異等位基因數量呈正相關(=25.493+499.52,2=0.9517);而北部冬麥區82份冬小麥品種(系)中含有3—17個優異等位變異,POD活性與優異等位基因數量呈正相關(= 34.022+536.06,2=0.7554)(圖5)。總之,優異等位基因數量越多,小麥籽粒POD活性越高。供試的233份材料中Soissons、SELYANKA、MV05-08、DONSKI-93、內鄉188、魯麥5號、魯麥11號和泰山5號等品種含有較多的優異等位基因和較高的POD活性。

X軸表示小麥21條染色體上的SNP標記,Y軸表示-log10(P-value)值,2013SHV:2013濉溪,2013AHV:2013安陽,2014SHV:2014濉溪,2014AHV:2014安陽和H-Average:黃淮麥區4個環境的平均值;2013BBY:2013北京,2013SBY:2013石家莊,2014BBY:2014北京,2014SBY:2014石家莊和B-Average:北部麥區4個環境的平均值

X軸表示經過負常數對數轉換的期望P值;Y軸表示經過負常數對數轉換觀察到的P值。2013SHV:2013濉溪,2013AHV:2013安陽,2014SHV:2014濉溪,2014AHV:2014安陽和Average:4個環境的平均值;2013BBY:2013北京,2013SBY:2013石家莊,2014BBY:2014北京,2014SBY:2014石家莊和Average:4個環境的平均值
2.5 候選基因分析
將51個穩定遺傳的與POD活性顯著關聯的SNP標記序列在NCBI數據庫中進行BLASTx,獲得3個候選基因,即編碼產物為磷酸甘露糖變位酶phosphomannomutase的,編碼辣根過氧化物酶和相關的分泌性的植物過氧化物酶的和編碼烷基氫過氧化物還原酶(AhpC)的(表4),為后續功能驗證及深入揭示POD活性表達機理奠定基礎。
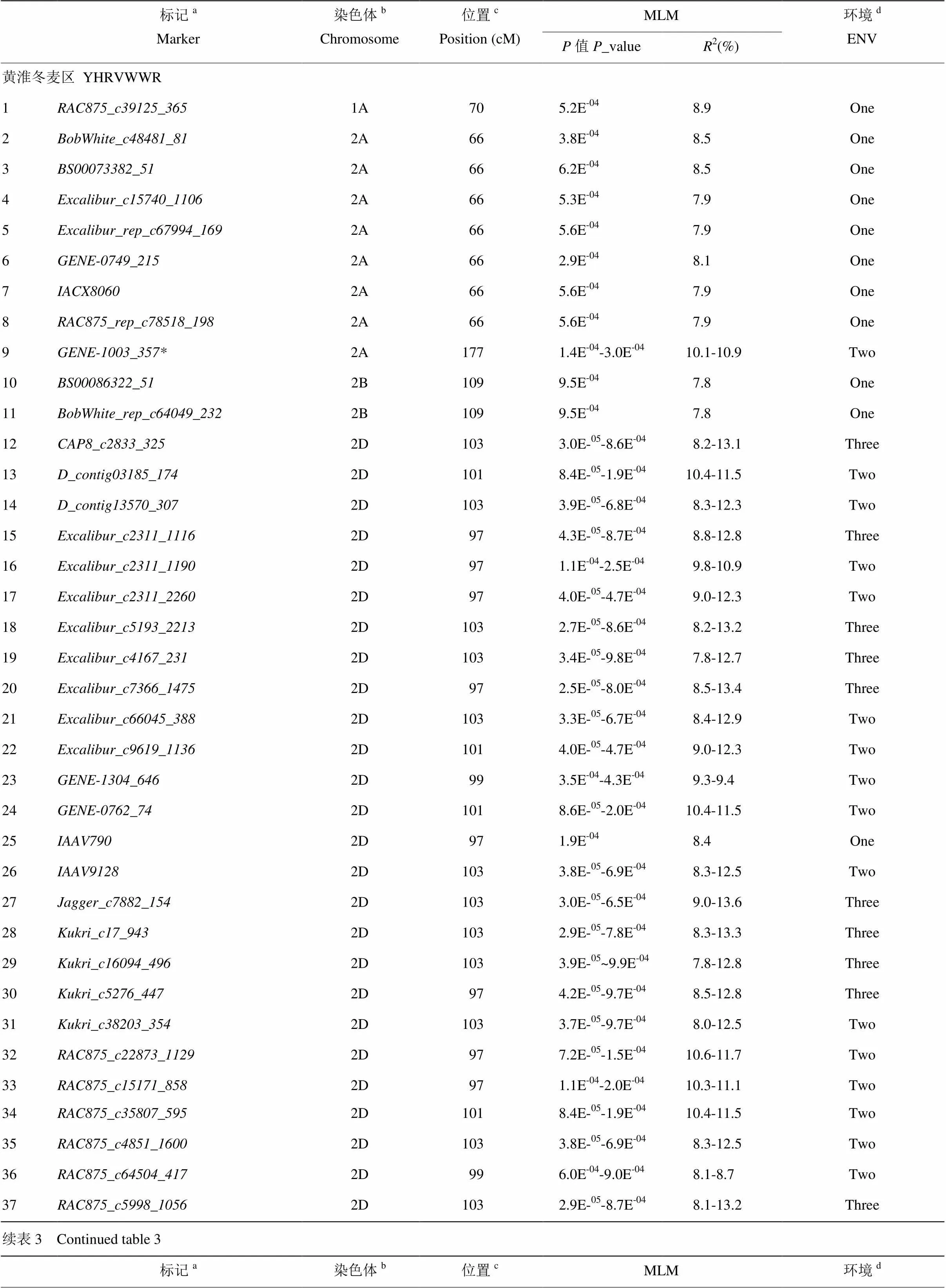
表3 黃淮冬麥區與北部冬麥區品種中與POD活性顯著關聯的SNP標記
aMarker混合線性模型(閾值為1E-03)檢測到的SNP標記;bChromosome染色體信息;cPosition連鎖圖譜中SNP標記的位置;dENV在4個環境中被檢測到的次數,One代表在1個環境中均檢測到,Two代表在2個環境中均檢測到,Three代表在3個環境中均檢測到
aMarker, shared markers were detected in MLM models at the threshold -log (P)=3.0;bChromosome;cPosition, the marker position on the linkage map;dENV, times of MTAs identified in four environments. One means MTA identified in one environments; Two means MTA identified in two environments; Three means MTA identified in three environments

圖5 黃淮冬麥區(a)和北部冬麥區(b)品種POD活性與優異等位基因數量的加性回歸分析
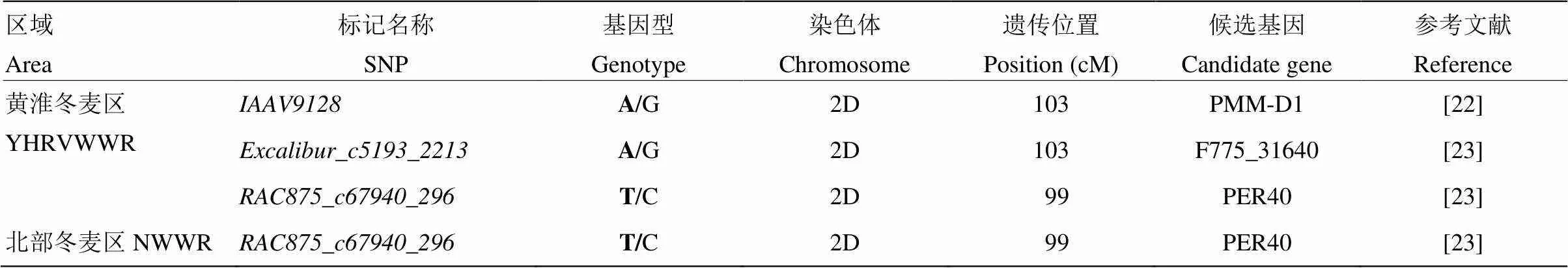
表4 黃淮冬麥區和北部冬麥區品種篩選獲得的候選基因
YHRVWWR和NWWR分別代表黃淮冬麥區和北部冬麥區。基因型粗體為優異等位變異
NWWR and YHRFWWR represent Northern China Plain Winter Wheat Region and Yellow & Huai Facultative Winter Wheat Region, respectively. Bold of genotype indicates favorable POD alleles
3 討論
3.1 POD活性定位及應用
小麥面粉顏色是衡量面制品品質的重要指標。影響面粉顏色的原因有很多,如小麥類胡蘿卜素合成限速酶八氫番茄紅素合酶(phytoene synthase,PSY)、多酚氧化酶PPO(polyphenol oxidase)與脂肪氧化酶LOX(lipoxygenase)。對PSY、PPO和LOX相關基因進行的研究已經較成熟,但對過氧化物酶POD的研究較少。POD也是影響面粉顏色的重要指標,因此,研究POD活性基因對小麥品質改良具有重要意義。
本研究利用90K SNP基因芯片對2個區域自然群體多個環境下的POD活性進行GWAS,檢測到2A、2D、5A、6A、7B和7D等多個穩定遺傳的位點。其中2D位點(97—100 cM)在3個環境中均能檢測到,是比較穩定的遺傳位點,可根據其連鎖標記開發新的標記,用于分子標記輔助育種。將與其緊密連鎖的12個SNP標記(、、、、、、、、、、和)轉化為STARP或CAPS標記[24],將為分子標記輔助育種提供有效的工具。在檢測到的多環境穩定遺傳的標記位點中,7D位點的貢獻率最大,為18.0%—21.4%。因此,在進一步研究中,可以考慮優先選擇2D與7D染色體的關聯位點進一步進行精細定位、圖位克隆及功能標記開發,為分子標記輔助育種提供有益信息。此外,由于單個位點標記的選擇效率有限,通過發現新的主效位點,將本研究發現的POD活性位點改進和開發的新的功能標記與小麥3A染色體的POD活性功能標記和[11]相結合,將多個主效位點結合起來進行分子標記輔助選擇,能顯著提高選擇效率,最終選出高POD活性的材料。
另外POD活性與優異等位變異含量顯著正相關,因此,含有較多的優異等位基因和較高的POD活性的品種,如Soissons、SELYANKA、MV05-08、DONSKI- 93、內鄉188、魯麥5號、魯麥11號和泰山5等品種(系),可作為優良親本用于育種工作,為POD活性改良工作提供基礎。
3.2 基于90 K SNP基因芯片的關聯分析與連鎖分析比較
隨著生物芯片技術的不斷發展,利用高密度芯片對基因定位的研究越來越廣泛。目前小麥中報道的芯片主要有小麥9K SNP芯片、小麥90K SNP芯片、小麥660K SNP芯片和小麥820K SNP芯片等。本研究所利用的小麥90K SNP 芯片:該芯片由美國Illumina公司與Akhunov E、Hayden M.及Cavanagh合作完成,包含81 587個SNP標記。其中有染色體信息的標記共42 104個,占51.61%[25]。90K芯片的分辨率已經很好,并且應用90K芯片來進行小麥基因定位的研究已經很成熟,雖然660K芯片與820K芯片的標記密度較90K芯片更高,但90K芯片較660K與820K芯片的成本較低,性價比更高。
Wei等[11]利用豆麥/石4185 RIL群體結合小麥90K SNP芯片,首次對普通小麥POD活性進行了連鎖分析,發現了3個主效QTL。其中5A位點與本研究一致;進一步證明關聯分析與連鎖分析均能得到與目標性狀顯著相關的QTL位點,通過自然群體進行關聯分析與連鎖分析技術互為補充,互為驗證。Zhai等[26]同樣用90K SNP芯片對面粉顏色性狀進行QTL定位,其中2D(84.3—94.0 cM)、5A(71.7—76.3 cM)和7B(109.2—114 cM)QTL與本研究檢測到的2D(97—103 cM)、5A(80 cM)和7B(98和99 cM)位點相近。2種方法同時能檢測到的重合位點是穩定遺傳的關鍵位點。但是本研究發現大部分POD活性位點與QTL定位的位點并不相同,如GWAS結果中的7D等位點在2個麥區均為穩定遺傳位點,但QTL定位并沒有檢測到,在排除假陽性和偽關聯的前提下,可見以自然群體為材料的關聯分析可以補充檢測到更多與POD活性相關的新位點。近年來利用連鎖分析進行QTL定位已經成為現代分子育種領域的研究重點,但受限于遺傳群體親本的差異度,單一以連鎖分析的方法進行QTL定位往往會遺漏大量微效甚至主效基因[27]。而關聯分析是一種利用自然群體中積累的歷史重組結果,發掘性狀關聯位點的方法,此方法能達到比雙親分離群體更高的分辨率,是連鎖分析的有益補充,能對連鎖分析結果進行驗證的同時,發現新的關鍵位點[28]。因此,基于高密度基因芯片,采用連鎖分析結合關聯分析技術發掘作物產量、品質、抗逆等數量性狀基因已經成為分子標記研究領域的發展趨勢[29-30]。
3.3 候選基因分析
編碼產物為磷酸甘露糖變位酶(phosphomannomutase,PMM),在高等植物中,PMM是抗氧化抗壞血酸合成的重要前體物質[31],在高等植物POD調節途徑中,PMM可以通過控制抗氧化抗壞血酸的合成來抑制POD活性[32]。編碼辣根過氧化物酶和相關的分泌性的植物過氧化物酶,分泌過氧化物酶屬于植物血紅素依賴性POD超家族Ⅲ類。編碼烷基氫過氧化物還原酶(AhpC),在植物體內,AhpC可以通過與POD競爭相同底物H2O2,來抑制POD活性[33]。以上候選基因編碼不同蛋白,在植物代謝途徑中直接或間接參與POD活性表達,故推測對小麥籽粒POD活性有調節作用,因此,在后續工作中對候選基因進行深入研究有助于為提高小麥籽粒POD活性提供依據。
4 結論
黃淮麥區與北部冬麥區2個自然群體遺傳多樣性豐富,群體結構簡單,適用于全基因組關聯分析。在2個自然群體中分別發現20個POD活性位點,其中有2個位點在2個自然群體中均能穩定遺傳,發現3個候選基因、和。含有越多優異等位變異的材料,其POD活性越高,可并發掘高POD活性品種為育種工作提供優質材料。
[1] 席德清. 對當前面粉生產中幾個問題的探討. 中國商辦工業, 1999, 11(7): 30-32.
XI D Q. Discussion on several problems in the current production of flour. Modern Business Trade Industry, 1999, 11(7): 30-32. (in Chinese)
[2] SIEDOW J N. Plant lipoxygenase: Structure and function. Annual Review of Plant Biology, 1991, 42(1): 145-188.
[3] BORRELLI G M, TROCCOLI A, DI FONZO N, FARES C. Durum wheat lipoxygenase activity and other quality parameters that affect pasta color. Cereal Chemistry, 1999, 76(3): 335-340.
[4] GELINAS P, POITRAS E, MCKINNON C M, MORIN A. Oxido-reductases and lipases as dough-bleaching agents. Cereal Chemistry, 1998, 75(6): 810-814.
[5] FRY S C. Cross-linking of matrix polymers in the growing cell walls of angiosperms. Annual Review of Plant Biology, 2003, 37(1): 165-186.
[6] TAHA S A, SAGI F. Relationships between chemical composition of durum wheat semolina and macaroni quality: Ⅱ. Ash, carotenoid pigments and oxidative enzymes. Cereal Research Communication, 1987, 15(2): 123-129.
[7] FRAIGNIER M P, MICHAUX-FERRIERE N, KOBREHEL K. Distribution of peroxidases in durum wheat (). Cereal Chemistry, 2000, 77(1): 11-17.
[8] HIDALGO A, BRANDOLINI A, POMPEI C. Carotenoids evolution during pasta, bread and water biscuit preparation from wheat flours., 2010, 121(3): 746-751.
[9] ?ili? S, DODIG D, ?ukalovi? V, MAKSIMOVIC M, SARATLIC G, SKRBIC B. Bread and durum wheat compared for antioxidants contents, and lipoxygenase and peroxidases activities. International Journal of Food Science &Technology, 2010, 45(7): 1360-1367.
[10] MCDONALD C E. Lipoxygenase and lutein bleaching activity of durum wheat semolina., 1979, 56(2): 84-89.
[11] WEI J X, GENG H W, ZHANG Y, LIU J D, WEN W E, ZHANG Y, XIA X C, HE Z H. Mapping quantitative trait loci for peroxidase activity and developing gene-specific markers foron wheat chromosome 3AL. Theoretical and Applied Genetics, 2015, 128(10): 2067.
[12] 張國華, 高明剛, 張桂芝, 孫金杰, 靳雪梅, 王春陽, 趙巖, 李斯深. 黃淮麥區小麥品種(系)產量性狀與分子標記的關聯分析. 作物學報, 2013, 39(7): 1187-1199.
ZHANG G H, GAO M G, ZHANG G Z, SUN J J, JIN X M, WANG C Y, ZHAO Y, LI S S. Association analysis of yield traits with molecular markers in Huang-Huai River Valley winter wheat region, China. Acta Agronomica Sinica, 2013, 39(7): 1187-1199. (in Chinese)
[13] WEI T M, CHANG X P, MIN D H, JING R L. Analysis of genetic diversity and tapping elite alleles for plant height in drought-tolerant wheat varieties. Acta Agronomica Sinica, 2010, 36(6): 895-904.
[14] 楊勝先, 牛遠, 李夢, 魏世平, 劉曉芬, 呂海燕, 章元明. 栽培大豆農藝性狀的關聯分析及優異等位變異挖掘. 中國農業科學, 2014, 47(20): 3941-3952.
YANG S X, NIU Y, Li M, WEI S P, LIU X F, Lü H Y, ZHANG Y M. Association mapping of agronomic traits in soybean (L. Merr.) and mining of novel alleles. Scientia Agricultura Sinica, 2014, 47(20): 3941-3952. (in Chinese)
[15] 賴勇, 王鵬喜, 范貴強, 司二靜, 王晉, 楊軻, 孟亞雄, 李葆春, 馬小樂, 尚勛武, 王化俊. 大麥SSR標記遺傳多樣性及其與農藝性狀關聯分析. 中國農業科學, 2012, 46(2): 233-242.
LAI Y, WANG P X, FAN G Q, SI E J, WANG J, YANG K, MENG Y X, LI B C, MA X L, SHANG X W, WANG H J. Genetic diversity and association analysis using SSR markers in barley. Scientia Agricultura Sinica, 2012, 46(2): 233-242. (in Chinese)
[16] 張煥欣, 翁建峰, 張曉聰, 劉昌林, 雍洪軍, 郝轉芳, 李新海. 玉米穗行數全基因組關聯分析. 作物學報, 2014, 40(1): 1-6.
ZHANG H X, WENG J F, ZHANG X C, LIU C L, YONG H J, HAO Z F, LI X H. Genome-wide association analysis of kernel row number in maize. Acta Agronomica Sinica, 2014, 40(1): 1-6. (in Chinese)
[17] 陳廣鳳, 陳建省, 田紀春. 小麥株高相關性狀與SNP標記全基因組關聯分析. 作物學報, 2015, 41(10): 1500-1509.
CHEN G F, CHEN J X, TIAN J C. Genome-wide association analysis between SNP markers and plant height related traits in wheat. Acta Agronomica Sinica, 2015, 41(10): 1500-1509. (in Chinese)
[18] SUN C, ZHANG F, YAN X, ZHANG X, DONG Z, CUI D, CHEN F. Genome-wide association study for 13 agronomic traits reveals distribution of superior alleles in bread wheat from the Yellow and Huai Valley of China.Plant Biotechnology Journal, 2017, doi: 10.1111/pbi.12690.
[19] DONG Y, LIU J, ZHANG Y, GENG H, RASHEED A, XIAO Y, CAO S, FU L, YAN J, WEN W. Genome-wide association of stem water soluble carbohydrates in bread wheat. Plos One, 2016, 11(11): e0164293.
[20] CAVANAGH C R, CHAO S, WANG S, HUANG B E, STEPHEN S, KIANI S, FORREST K, SAINTENAC C, BROWN-GUEDIRA G L, AKHUNOVA A, SEE D, BAI G, PUMPHREY M, TOMAR L, WONG D, KONG S, REYNOLDS M, DA SILVA M L, BOCKELMAN H, TALBERT L, ANDERSON J A, DREISIGACKER S, BAENZIGER S, CARTER A, KORZUN V, MORRELL P L, DUBCOVSKY J, MORELL M K, SORRELLS M E, HAYDEN M J, AKHUNOV E. Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars. Proceedings of the National Academy of Sciences of the USA, 2013, 110(20): 8057-8062.
[21] LUI K, MUSE S V. Power Marker: integrated analysis environment for genetic marker data. Bioinformatics, 2005, 21(9): 2128-2129.
[22] KAWAURA K, MOCHIDA K, ENJU A, TOTOKI Y, TOYODA A, SAKAKI Y, SHINOZAKI K. Assessment of adaptive evolution between wheat and rice as deduced from full-length common wheat cDNA sequence data and expression patterns. BMC Genomics, 2009, 10(1): 271.
[23] JIA J, ZHAO S, KONG X, LI Y, ZHAO G, HE W, JING R.draft genome sequence reveals a gene repertoire for wheat adaptation. Nature, 2013, 496(7443): 91-95.
[24] LONG Y M, CHAO W S, MA G J, XU S S, QI L L. An innovative SNP genotyping method adapting to multiple platforms and throughputs. Theoretical and Applied Genetics, 2017, 130(3): 597-607.
[25] WANG S, WONG D, FORREST K, ALLEN A, CHAO S, HUANG B E, MACCAFERRI M, SALVI S, MILNER S G, CATTIVELLI L. Characterization of polyploid wheat genomic diversity using a high-density 90,000 single nucleotide polymorphism array. Plant Biotechnology Journal, 2014, 12(6): 787.
[26] ZHAI S N, HE Z H, WEN W E, JIN H, LIU J D, ZHANG Y, LIU Z Y, XIA X C. Genome-wide linkage mapping of flour color-related traits and polyphenol oxidase activity in common wheat. Theoretical and Applied Genetics, 2016, 129(2): 377-394.
[27] XIE J Z, HUO N X, ZHOU S G, WANG Y, GUO G H, DEAL K R, QUYANG S H, LIANG Y, WANG Z Z, XIAO L C, ZHOU T T, HU T Z, TIWARIV, ZHANG J W, LI H X, NI Z F, YAO Y Y, RENG H R, ZHANG S L, ANDERSON O D, MCGUIRE P E, DVORAK J, LUO M C, LIU Z Y, GU Y Q, SUN Q X. Sequencing and comparative analyses ofchromosome arm 3DS reveal rapid evolution of Triticeae genomes. Journal of Genetics and Genomics, 2017, 44(1): 51-61.
[28] 周生輝, 吳秋紅, 謝菁忠, 陳嬌嬌, 陳永興, 傅琳, 王國鑫, 于美華, 王振忠, 張德云, 王令, 王麗麗, 張艷, 梁榮奇, 韓俊, 劉志勇. 小麥燕大1817×北農6號重組自交系群體在正常和鹽脅迫水培條件下苗期性狀的QTL定位. 作物學報, 2016, 42(12): 1764-1778.
ZHOU S H, WU Q H, XIE J Z, CHEN J J, CHEN Y X, FU L, WANG G X, YU M H, WANG Z Z, ZHANG D Y, WANG L, WANG L L, ZHANG Y, LIANG R Q, HAN J, LIU Z Y. Mapping QTLs for wheat seedling traits in RILs population of Yanda 1817×Beinong 6 under normal and salt-stress conditions. Acta Agronomica Sinica, 2016, 42(12): 1764-1778. (in Chinese)
[29] HUANG X, WEI X, SANG T, ZHAO Q, FENG Q, ZHAO Y, LI C, ZHU C, LU T, ZHANG Z, LI M, FAN D, GUO Y, WANG A, WANG L, DENG L, LI W, LU Y, WENG Q, LIU K, HUANG T, ZHOU T, JING Y, LI W, LIN Z, BUCKLER E S, QIAN Q, ZHANG Q F, LI J, HAN B. Genome-wide association studies of 14 agronomic traits in rice landraces. Nature Genetics, 2010, 42(11): 961-967.
[30] WRIGHT S I, BI I V, SCHROEDER S G,YAMASAKI M, DOEBLEY J F, MCMULLEN M D, GAUT B S. The effects of artificial selection on the maize genome. Science, 2005, 308(5726): 1310-1314.
[31] YU C, LIU X, ZHANG Q, HE X, HUAI W, WANG B, ZHOU R. Molecular genetic analysis of phosphomannomutase genes in Triticummonococcum. The Crop Journal, 2015, 3(1): 29-36.
[32] 劉敏, 曹志軍, 李正英, 敖萬鳳, 李艷梅. 木耳菜過氧化物酶活性及其影響因素的研究. 中國釀造, 2009, 28(11): 50-52.
LIU M, CAO Z J, LI Z Y, AO W F, LI Y M. Activity and its influence factor of peroxidase from. China Brewing, 2009, 28(11): 50-52. (in Chinese)
[33] HUSSAIN R M, ABDULLAH N F, ABDULLAH N F, AMOM Z, Killing ofby allylpyrocatechol is potentiated by induction of intracellular oxidative stress and inhibition of catalase activity. Journal of Integrative Medicine, 2016, 14(6): 456-464.
(責任編輯 李莉)
附表1 黃淮麥區POD活性數據
Table S1 The POD activity in Yellow & Huai Facultative Winter Wheat Region (U·min-1g-1)

品種名稱Name來源Origin2013濉溪2013SHV2013安陽2013AHV2014安陽2014AHV2014濉溪2014SHV平均值mean 185中3385 Zhong 33中國河南Henan, China547.5559.8780.3984.9718.1 2Aca 601阿根廷Argentina652.4550.8857.7930.0747.7 3Aca 801阿根廷Argentina417.1387.8797.4610.7553.2 4Barra意大利Italy627.0559.51015.11136.4834.5 5Dorico意大利Italy585.8477.9910.71010.1746.1 6Genio意大利Italy729.6509.9697.7670.3651.9 7Hk1/6/Nvsr3/5/Bez/Tvr/5/Cfn/Bez//Su92/Ci13645/3Nai60土耳其Turkey401.1530.6761.4883.4644.1 8Kanto 107日本Japan498.4434.8764.0781.4619.6 9Kitanokaori日本Japan459.4505.0868.7863.1674.0 10Klein Flecha阿根廷Argentina302.9447.5721.1668.0534.8 11Klein Jabal 1阿根廷Argentina439.0379.4584.7694.4524.4 12Libero意大利Italy416.7535.5807.0876.0658.8 13Mantol意大利Italy496.2476.9783.21182.8734.8 14Nidera Baguette 20阿根廷Argentina322.8846.9899.3946.4753.8 15Norin 61日本Japan632.7444.8785.1952.4703.7 16Norin 67日本Japan491.0705.6956.11046.6799.8 17ProintaColibr 1阿根廷Argentina430.3494.0685.8782.4598.1 18Sagittario意大利Italy392.2398.5761.1707.0564.7 19Abbondanza意大利Italy527.8546.8699.4786.1640.0 20Funo意大利Italy646.3635.9825.5989.7774.3 21矮豐3 Aifeng 3中國陜西Shaanxi, China479.2479.5713.71032.8676.3 22矮抗58 Aikang 58中國河南Henan, China383.4425.8754.5831.3598.8 23安1331 An 1331中國安徽Anhui, China352.2416.1690.6827.0571.5 24百農3217 Bainong 3217中國河南Henan, China416.8406.8537.1780.9535.4 25百農64 Bainong 64中國河南Henan, China351.0404.1625.2713.4523.4 26碧螞1號 Bima 1中國陜西Shaanxi, China466.3438.1768.3838.7627.8 27碧螞4號 Bima 4中國陜西Shaanxi, China561.6475.8879.0855.2692.9 28豐產3 Fengchan 3中國陜西Shaanxi, China370.9526.9679.5857.9608.8 29阜936 Fu 936中國安徽Anhui, China463.9655.7644.6886.8662.7 30高優503 Gaoyou 503中國河北Hebei, China503.2546.6669.6822.8635.5 31藁城8901 Gaocheng 8901中國河北Hebei, China365.5188.6524.7646.5431.3 32觀35 Guan 35中國河北Hebei, China465.1422.4623.2625.1533.9 33邯6172 Han 6172中國河北Hebei, China483.5594.4795.6898.4693.0 34衡7228 Heng 7228中國河北Hebei, China479.9444.6710.3756.5597.8 35衡觀33 Hengguan 33中國河北Hebei, China455.7396.2650.8851.9588.7 36花培5號 Huapei 5中國河南Henan, China393.5336.2660.8693.0520.9 37淮麥18 Huaimai 18中國安徽Anhui, China448.5458.5613.0712.8558.2 38淮麥20 Huaimai 20中國安徽Anhui, China521.3437.0782.4999.5685.0 39淮麥21 Huaimai 21中國安徽Anhui, China361.7452.7752.3802.9592.4 40濟麥19 Jimai 19中國山東Shandong, China359.0450.8571.0687.7517.1 41濟麥20 Jimai 20中國山東Shandong, China528.2512.6757.4825.0655.8 42濟麥21 Jimai 21中國山東Shandong, China644.8710.9772.51203.2832.8 43濟麥22 Jimai 22中國山東Shandong, China758.9670.11041.01176.6911.6 44濟南17 Jinan 17中國山東Shandong, China468.5416.6661.8824.4592.8 45濟寧16 Jining 16中國山東Shandong, China430.7405.6699.6747.3570.8 46冀師02-1 Jishi 02-1中國河北Hebei, China631.9512.8747.6927.5704.9 47金禾9123 Jinhe 9123中國河北Hebei, China573.0390.7671.9757.4598.2 48濟麥61 Jinmai 61中國山東Shandong, China460.9341.6642.6879.3581.1 49蘭考24 Lankao 24中國河南Henan, China852.2758.71016.91136.4941.1 50蘭考2 Lankao 2中國河南Henan, China658.1659.9775.21089.9795.8 51蘭考906 Lankao 906中國河南Henan, China784.2489.31081.41151.3876.5 52良星66 Liangxing 66中國山東Shandong, China839.3705.11048.81226.0954.8 53良星99 Liangxing 99中國山東Shandong, China528.1644.41131.51090.4848.6 54臨旱2號Linhan 2中國山西Shanxi, China460.3438.2705.9861.8616.6 55臨抗12 Linkang 12中國山西Shanxi, China421.6465.0664.4891.0610.5 56臨麥2號 Linmai 2中國山東Shandong, China487.2654.51054.41252.4862.1 57臨麥4號 Linmai 4中國山東Shandong, China592.3574.5776.41150.5773.4 58魯麥11 Lumai 11中國山東Shandong, China385.3534.2559.0659.8534.6 59魯麥14 Lumai 14中國山東Shandong, China473.6633.31144.51228.7870.0 60魯麥15 Lumai 15中國山東Shandong, China506.3447.2712.4853.7629.9 61魯麥21 Lumai 21中國山東Shandong, China612.0673.8774.5846.0726.6 62魯麥23 Lumai 23中國山東Shandong, China527.6558.3898.71014.5749.8 63魯麥5 Lumai 5中國山東Shandong, China510.3509.9832.8941.3698.6 64魯麥6 Lumai 6中國山東Shandong, China535.9505.1770.31039.8712.8 65魯麥7 Lumai 7中國山東Shandong, China571.1647.1808.41043.1767.4 66魯麥8 Lumai 8中國山東Shandong, China436.1601.0593.9830.9615.5 67魯麥9 Lumai 9中國山東Shandong, China646.5705.2930.81085.0841.8 68魯原502 Lunyuan 502中國山東Shandong, China558.6671.11003.2914.7786.9 69洛旱2號 Luohan 2中國河南Henan, China349.6415.2646.1871.8570.7 70洛麥21 Luomai 21中國河南Henan, China451.2565.6776.3921.9678.7 71內鄉188 Neixiang 188中國河南Henan, China561.8605.8827.0924.3729.7 72山農20 Shannong 20中國山東Shandong, China498.7650.9931.4799.7720.2 73陜229 Shaan 229中國陜西Shaanxi, China452.4456.4677.9917.1626.0 74陜223 Shaan 253中國陜西Shaanxi, China397.6469.6719.3862.4612.2 75陜354 Shaan 354中國陜西Shaanxi, China519.5471.0760.1726.7619.3 76陜512 Shaan 512中國陜西Shaanxi, China368.4350.4714.5706.7535.0 77陜715 Shaan 715中國陜西Shaanxi, China299.9318.2722.3629.2492.4 78陜麥509 Shaanmai 509中國陜西Shaanxi, China634.9423.4844.8805.1677.0 79陜麥94 Shaanmai 94中國陜西Shaanxi, China570.1564.5873.2998.6751.6 80陜農78-59 Shaannong 78-59中國陜西Shaanxi, China507.7434.6777.0671.4597.7 81陜農981 Shaannong 981中國陜西Shaanxi, China434.6327.0591.8581.1483.6 82陜優225 Shaanyou 225中國陜西Shaanxi, China378.1385.0715.1746.2556.1 83石4185 Shi 4185中國河北Hebei, China443.1392.9796.2665.5574.4 84石家莊15 Shijiazhuang 15中國河北Hebei, China383.1564.2814.2814.9644.1 85石家莊8 Shijiazhuang 8中國河北Hebei, China494.6394.5876.3878.9661.1 86石新733 Shixin 733中國河北Hebei, China525.0396.5622.0819.0590.6 87石新828 Shixin 828中國河北Hebei, China576.8436.9711.6803.0632.1 88石優17 Shiyou 17中國河北Hebei, China520.0366.7955.21079.4730.3 89宿0663 Su 0663中國安徽Anhui, China535.2630.8657.8805.5657.3 90宿農6號 Sunong 6中國安徽Anhui, China518.3739.5982.11049.4822.3 91泰山1號 Taishan 1中國山東Shandong, China692.6607.61013.11183.5874.2 92泰山5號 Taishan 5中國山東Shandong, China512.2579.2780.91078.5737.7 93皖23094 Wan 23094中國安徽Anhui, China411.6667.6753.0870.9675.8 94皖麥29 Wanmai 29中國安徽Anhui, China333.8400.3545.0734.4503.4 95皖麥33 Wanmai 33中國安徽Anhui, China456.5471.5658.6929.0628.9 96皖麥38 Wanmai 38中國安徽Anhui, China517.4407.1695.1862.5620.5 97皖麥50 Wanmai 50中國安徽Anhui, China539.7521.3825.81108.6748.8 98皖麥52 Wanmai 52中國安徽Anhui, China499.1503.3759.9886.5662.2 99皖麥53 Wanmai 53中國安徽Anhui, China470.9544.2746.6805.4641.7 100汶農14 Wennong 14中國山東Shandong, China477.4561.21085.11115.1809.7 101汶農5 Wennong 5中國山東Shandong, China551.4528.1809.4788.7669.4 102汶農148 Wunong 148中國山東Shandong, China410.6417.5664.1836.3582.1 103西農1376 Xinong 1376中國陜西Shaanxi, China478.6478.6699.3804.0615.1 104西農2000-7 Xinong 2000-7中國陜西Shaanxi, China409.4379.8654.8633.3519.3 105西農291 Xinong 291中國陜西Shaanxi, China509.0508.21016.0855.4722.2 106西農88 Xinong 88中國陜西Shaanxi, China458.5522.3715.1775.9617.9 107西農975-005 Xinong 979-005中國陜西Shaanxi, China484.4438.7660.3770.2588.4 108小偃22 Xiaoyan 22中國陜西Shaanxi, China630.7519.9762.3983.4724.1 109小偃54 Xiaoyan 54中國陜西Shaanxi, China499.6424.5737.4790.1612.9 110小偃81 Xiaoyan 81中國陜西Shaanxi, China568.7584.2848.9905.2726.7 111新麥19 Xinmai 19中國新疆Xinjiang, China750.0482.2733.4980.1736.4 112新麥9408 Xinmai 9408中國新疆Xinjiang, China594.8562.4741.51068.3741.7 113新麥9 Xinmai 9中國新疆Xinjiang, China497.6564.0813.5948.0705.8 114煙農15 Yannong 15中國山東Shandong, China751.9520.2880.2968.1780.1 115煙農19 Yannong 19中國山東Shandong, China432.2644.3835.41118.1757.5 116偃展4110 Yanzhan 4110中國河南Henan, China650.5627.7788.4952.1754.7 117豫麥13 Yumai 13中國河南Henan, China367.6361.6677.6737.3536.0 118豫麥18 Yumai 18中國河南Henan, China420.3484.6806.9820.8633.2 119豫麥21 Yumai 21中國河南Henan, China356.9367.9477.5643.3461.4 120豫麥2 Yumai 2中國河南Henan, China435.0536.8787.2780.3634.8 121豫麥34 Yumai 34中國河南Henan, China575.1555.1854.31184.6792.3 122豫麥35 Yumai 35中國河南Henan, China564.3487.8879.0992.3730.8 123豫麥47 Yumai 47中國河南Henan, China639.9463.9727.5942.8693.5 124豫麥49 Yumai 49中國河南Henan, China536.0660.3944.41040.0795.2 125豫麥50 Yumai 50中國河南Henan, China495.6589.6895.7859.7710.1 126豫麥63 Yumai 63中國河南Henan, China336.3613.8823.7864.9659.7 127豫麥7 Yumai 7中國河南Henan, China445.8419.2599.6905.9592.6 128鄭9023 Zheng 9023中國河南Henan, China468.4544.5783.3842.1659.6 129鄭引1號 Zhengyin 1中國河南Henan, China537.2685.0972.6878.9768.4 130鄭州3號 Zhengzhou 3中國河南Henan, China791.9695.2932.41042.1865.4 131中892 Zhong 892中國河南Henan, China797.8561.5978.01066.4850.9 132中麥871 Zhongmai 871中國河南Henan, China531.6474.2781.7860.4662.0 133中麥875 Zhongmai 875中國河南Henan, China491.6495.8808.2960.4689.0 134中麥895 Zhongmai 895中國河南Henan, China460.7478.6745.7922.6651.9 135中育5號 Zhongyu 5中國河南Henan, China306.7398.9821.4530.2514.3 136中育9號 Zhongyu 9中國河南Henan, China401.9517.9757.1761.3609.6 137周8425B Zhou 8425B中國河南Henan, China554.8820.51061.11199.6909.0 138周麥11 Zhoumai 11中國河南Henan, China478.3449.2895.2960.3695.7 139周麥12 Zhoumai 12中國河南Henan, China608.3712.6926.91015.7815.9 140周麥13 Zhoumai 13中國河南Henan, China713.4858.6725.01126.4855.8 141周麥16 Zhoumai 16中國河南Henan, China542.3413.7713.11037.9676.7 142周麥18 Zhoumai 18中國河南Henan, China535.7513.0804.01023.2719.0 143周麥19 Zhoumai 19中國河南Henan, China449.8349.0675.0961.5608.8 144周麥22 Zhoumai 22中國河南Henan, China515.4492.0747.5962.7679.4 145周麥23 Zhoumai 23中國河南Henan, China491.9580.2701.1830.7651.0 146周麥25 Zhoumai 25中國河南Henan, China529.3433.6611.8757.5583.0 147周麥26 Zhoumai 26中國河南Henan, China512.4533.4902.6803.6688.0 148周麥31 Zhoumai 31中國河南Henan, China391.7464.6707.1765.9582.3 149周麥32 Zhoumai 32中國河南Henan, China514.7476.5670.2888.9637.6 150淄麥12 Zimai 12中國山東Shandong, China583.3509.7854.11077.9756.2 151淄選2號 Zixuan 2中國山東Shandong, China588.5541.4834.5870.3708.7
附表2 北部冬麥區POD活性數據
Table S2 The POD activity in Northern China Plain Winter Wheat Region (U·min-1g-1)

品種名稱Name來源Origin2013北京2013 BBV2013石家莊2013SBV2014北京2014 BBV2014石家莊2014 SBV平均mean 1Batjko俄羅斯Russia781.2649.81037.41130.9899.8 2Bruta法國France238.1577.6820.1908.9636.2 3C英國Britain482.3368.8849.5698.3599.7 4C 39英國Britain505.9572.9907.5799.7696.5 5CA1119中國北京Beijing, China596.0602.4788.9951.8734.8 6Carimulti法國France717.2636.11155.61128.3909.3 7D法國France669.1523.8900.1975.3767.1 8Darius法國France584.6598.6845.1820.5712.2 9E意大利Italy758.4811.91202.11154.9981.8 10F498U1-1021/Boema羅馬尼亞Romania619.6624.81065.8777.3771.9 11F98047G14-2INC羅馬尼亞Romania694.5549.1962.4869.7768.9 12Festin法國France521.6521.61033.8984.8765.4 13Fr03717 法國France786.6424.6863.9825.3725.1 14Fr03724法國France627.7428.2819.0918.6698.4 15Fr03725法國France630.0516.9848.1567.3640.6 16Fr3713法國France508.6502.81039.3743.4698.5 17Insignia法國France604.8689.4787.7787.7717.4 18Jagger/W94-244-132美國USA556.4595.0838.7801.9698.0 19Kniish 46俄羅斯Russia767.8639.11014.3736.1789.3 20Lovrin13 羅馬尼亞Romania780.9595.7842.7964.8796.0 21Magnus德國Germany525.9525.9825.1825.1675.5 22Manital法國France425.9425.9778.2778.2602.0 23Mason/Jagger意大利Italy440.0436.9831.2824.6633.2 24Mesofold法國France415.3394.4621.6635.1516.6 25NSA09-3645法國France619.7619.71052.7912.5801.1 26RE714挪威Norway629.8527.9990.8891.5760.0 27Salmone法國France478.2486.2857.3801.6655.8 28Soissons法國France883.9772.71590.51200.01111.8 29Thesee法國France675.7537.51175.9851.4810.1 30Wgrc10/3/KS93U69sib/TA2455//KS93U69/4/Jagger美國USA608.0502.7952.1874.5734.3 31北京0045Beijing 0045中國北京Beijing, China595.6540.0826.9789.3688.0 32長武134Changwu 134中國陜西Shaanxi, China571.1766.5914.1743.4748.8 33晉麥45Jinmai 45中國山西Shanxi, China675.6731.1919.9884.1802.7 34京9428Jing 9428中國北京Beijing, China578.1626.0872.1901.2744.4 35農大139Nongda 139中國北京Beijing, China685.0511.3843.5961.4750.3 36秦農151Qinnong 151中國陜西Shaanxi, China513.5574.3956.9708.3688.3 37秦農731Qinnong 731中國陜西Shaanxi, China602.1626.4999.8795.2755.9 38洋小麥Yangxiaomai中國地方種Local species, China801.5561.8881.6989.3808.5 39中優206Zhongyou 206中國北京Beijing, China496.9493.9773.0772.8634.1 40中優9507Zhongyou 9507中國北京Beijing, China738.4729.51031.6901.7850.3 41CA0548中國北京Beijing, China576.0741.4805.4903.9756.7 42CA0816 (white)中國北京Beijing, China488.7649.6853.4880.4718.0 43CA0816 (red)中國北京Beijing, China529.2567.7855.4677.1657.3 44CA0958中國北京Beijing, China546.8654.0775.5990.0741.6 45CA0998中國北京Beijing, China432.5417.8704.3613.2542.0 46CA1055中國北京Beijing, China611.2447.7514.7773.3586.7 47CA1090中國北京Beijing, China605.3646.7905.1933.6772.7 48CA1133中國北京Beijing, China643.4833.61161.91104.0935.7 49CA1135中國北京Beijing, China659.9773.91033.61167.2908.7 50京411Jing 411中國北京Beijing, China690.5715.5924.6841.8793.1 51寧冬11Ningdong 11中國寧夏Ningxia, China619.4592.7958.4912.0770.6 52新麥37Xinmai 37中國新疆Xinjiang, China446.5491.2555.0612.9526.4 53中麥175Zhongmai 175中國北京Beijing, China760.2887.0921.01190.4939.6 54中麥415Zhongmai 415中國北京Beijing, China437.9445.9657.6647.2547.1 5598039G5-103羅馬尼亞Romania694.3461.9803.4891.2712.7 56Aztec法國France597.3595.8995.8909.6774.6 57Azulon法國France508.7467.3927.3938.1710.4 58CA1062中國北京Beijing, China502.2354.1695.8540.9523.2 59CA9722中國北京Beijing, China528.7515.3728.3800.9643.3 60Donski-93俄羅斯Russia676.3676.31214.4987.6888.6 61F92080G1-1/F93042G2-1羅馬尼亞Romania587.5448.6996.3996.3757.2 62LASEN法國France561.2405.2700.4786.8613.4 63Lovrin10 羅馬尼亞Romania625.1649.21101.2915.5822.8 64MV05-08匈牙利Hungary580.5555.71189.0926.6812.9 65Palpich俄羅斯Russia562.9467.2924.3936.6722.7 66Selyanka俄羅斯Russia711.0587.81124.5939.0840.6 67Starshina俄羅斯Russia609.8546.1903.0772.1707.7 68TX03A0148 美國USA610.4610.4834.2881.9734.2 69YANA法國France556.0488.2922.3676.8660.8 70北京841Beijing 841中國北京Beijing, China484.0471.5827.8755.7634.8 71豆麥Doumai中國地方種Local species, China490.9419.4653.8706.2567.6 72豐抗2Fengkang 2中國北京Beijing, China477.4541.5760.1808.2646.8 73晉麥67Jinmai 67中國山西Shanxi, China631.0616.3958.3908.7778.6 74京冬17Jingdong 17中國北京Beijing, China398.4600.0778.5845.1655.5 75京冬22Jingdong 22中國北京Beijing, China744.4613.71086.2920.4841.2 76京冬8Jingdong 8中國北京Beijing, China682.3588.7914.0859.2761.1 77京雙16Jingshuang 16中國北京Beijing, China567.5628.8937.5934.5767.1 78科衡6654Keheng 6654中國河北Hebei, China560.1472.6646.8682.2590.4 79輪選987Lunxuan 987中國北京Beijing, China406.7323.4665.2745.1535.1 80寧冬10Ningdong 10中國寧夏Ningxia, China626.3549.1866.1823.2716.2 81農大211Nongda 211中國北京Beijing, China544.7557.2853.4834.5697.4 82農大212Nongda 212中國北京Beijing, China548.8635.2807.6871.8715.9
Genome-Wide Association Study of Grain Peroxidase Activity in Common Wheat
SHI Jia1, ZHAI ShengNan2, LIU JinDong2, WEI JingXin2, BAI Lu3, GAO WenWei1, WEN WeiE1,2, HE ZhongHu2, XIA XianChun2, GENG HongWei1
(1College of Agronomy, Xinjiang Agricultural University/Key Laboratory of Agricultural Biological Technology, Xinjiang Agricultural University, Urumqi 830052;2Institute of Crop Science, Chinese Academy of Agricultural Sciences (CAAS)/National Wheat Improvement Center, Beijing 100081;3College of Science and Technology, Xinjiang Agricultural University, Urumqi 830091)
Peroxidase (POD) activity has browning and bleaching effects on the color of flour and flour-based products during processing and storage. Identification of associated loci and candidate genes for grain POD activity is important for molecular marker-assisted selection (MAS) in wheat quality breeding.In the present study, the POD activities were surveyed with 151 and 82 Chinese bread wheat cultivars from Yellow & Huai Winter Wheat Region (YHRVWWR) and Northern China Plain Winter Wheat Region (NWWR), respectively, and each set of cultivars was planted in four environments. A genome-wide association study (GWAS) was performed using the mixed linear model (MLM) based on 18 189 and 18 417 high-quality SNP markers from 90K SNP array for two sets of cultivars, respectively.The POD activity of the tested materials showed extensive phenotypic variation and diversity. The variation coefficient of YHRVWWR was 15.4%-21.8%, the heritability was 0.79, and the variation coefficient of NWWR was 15.0%-19.9%, the heritability was 0.82. The POD activity of the materials in different environments showed a significant correlation, and the correlation coefficients were 0.46-0.89 (<0.0001) and 0.50-0.87 (<0.0001) in YHRVWWR and NWWR, respectively. The polymorphic information content of value was between 0.09-0.38, and the minimum allele frequency was between 0.05-0.5. The population structure analysis showed that the two natural populations in YHRVWWR and NWWR were simple and could be divided into three subgroups. In the YHRVWWR cultivars, 20 loci were found to be associated with POD activity (<0.001), which were located on chromosomes 1A, 2A, 2B, 2D, 3A, 3B, 3D, 4A, 4B, 5A, 5B, 6A, 6D and 7A, and each explained 7.8%-13.3% of phenotypic variation. In the NWWR cultivars, 20 loci showed significant association with POD activity (<0.001), which were located on chromosomes 1A, 1B, 1D, 2A, 2B, 2D, 3A, 3B, 4B, 6A, 6B, 7A, 7B and 7D, explaining 14.4%-23.2% of phenotypic variation. Two loci were detected in both the YHRVWWR and NWWR cultivars, and the associated SNPs could be used to develop STARP (Semi-thermal asymmetric reverse PCR) or CAPS markers. The regression analysis showed that the POD activity of wheat grain was higher with the increasing number of favorable alleles. Meanwhile, three candidate genes,,andwere scanned, encoding phosphomannomutase, horseradish peroxidasesand alkyl hydro peroxide reductase, respectively.The genetic diversity of the two natural populations in YHRVWWR and NWWR are rich in genetic structure and were suitable for genome-wide association analysis. Twenty POD activity loci were found in 2 natural populations, respectively, and three candidate genes were detected. Regression analysis showed that the more favorable alleles variation, the higher the POD activity.
common wheat; POD activity; SNP; population structure; candidate gene
2017-04-24;接受日期:2017-06-02
國家自然科學基金(31771786)、2016年南京農業大學-新疆農業大學聯合基金(KYYJ201602)
時佳,E-mail:shijia0401@126.com。通信作者耿洪偉,E-mail:hw-geng@163.com

