番茄附生和內生細菌分離與群落相似性分析
王玲玲 尚慶茂 董春娟
(中國農業科學院蔬菜花卉研究所 農業部園藝作物生物學與種質創制重點實驗室,北京 100081)
植物自身是一個復雜的微生態系統,棲息著大量微生物,其中尤以細菌數量最多,包括定殖于植物根際的根際細菌、附生于植物組織(莖、葉、花及果實等)表面的附生細菌、以及分布于各器官或組織內部的內生細菌[1-2],它們與宿主協同進化,在促進植物生長、抵御病蟲害、增強寄主對環境脅迫的抗性方面具有重要作用。植物附生和內生細菌現已作為新型的微生物資源,研究者們在水稻、玉米、番茄、辣椒、白菜等多種作物中分離到具有固氮、促生和生防作用的菌株[3-5]。研究土壤以及植物附生和內生細菌的種類、分布、定殖規律,并比較它們相互間的異同,對篩選植物促生菌具有重要意義。
研究植物附生和內生細菌群落主要采用傳統的分離培養方法,依靠形態特征、生理生化特性等進行分類鑒定和計數。但是,可培養細菌僅占自然界細菌總數的1%-10%,傳統方法對細菌群落結構及多樣性的認識是不全面和有選擇性的[6]。近年來,以16S rRNA分析為基礎的分子生物學手段的應用為細菌群落結構的解析提供了更高效的研究技術,如末端限制性片段長度多樣性(Terminal restriction fragment length polymorphism,T-RFLP)、變性梯度凝 膠 電 泳(Denaturing gel gradient electrophoresis,DGGE)等。這些不依賴于培養的分子生態學方法,通過對條帶圖譜和序列的分析,更加快速、完整地反映植物附生和內生細菌的群落結構和多樣性[7-8]。
本實驗以番茄(Solanum lycopersicum cv.)為研究對象,采用傳統平板培養法結合DGGE技術對番茄根區和根際土壤以及各組織附生和內生細菌的數量、豐富度、多樣性等進行定量和比較分析,明確土壤細菌以及組織附生、內生細菌間的分布規律及相關性,為篩選植物促生菌,提高植物的健康水平提理論依據。
1 材料與方法
1.1 材料
供試番茄選用中雜302(S. lycopersicum cv. Zhongza No.302)由中國農業科學院蔬菜花卉研究所選育,番茄于2016年2月播種,4月定植于該所日光溫室中,常規栽培管理,植株長勢良好,至7月當番茄第四穗位果成熟時進行取樣。
1.2 方法
1.2.1 番茄組織樣品采集 隨機選取20株健康番茄植株,依次收集根系、成熟葉片、位于第2至第4葉片間的莖段、以及第2和第3穗成熟果實,各組織樣品均無明顯的損傷。根區土壤的收集采用抖落法[9],將松散與根系結合的土壤抖落后,過2 mm篩,去除根組織及大塊土壤、石頭塊等。將各土壤樣品和植物組織樣品分別裝入無菌自封袋中,4℃冰箱保存待用。3次生物學重復。
1.2.2 根區土壤中細菌的分離培養 取番茄根區土壤(0.3 g),置于無菌三角瓶中,加入無菌PBS溶液(NaCl 8 g/L、KCl 0.2 g/L、Na2HPO41.44 g/L、KH2PO40.24 g/L,pH 7.4),于25℃、150 r/min振蕩1 h后,靜置10 min,取上清液用無菌水按10倍梯度稀釋(10-1-10-6),稀釋后的懸液分別取100 μL涂布于TSA培養基(胰蛋白胨15 g/L、大豆胨5 g/L、NaCl 5 g/L、瓊脂15 g/L,pH 7.0),每個梯度懸液涂布3個平行,將培養皿置于37℃培養箱中倒置培養,12 h后觀察細菌生長情況并計數。
1.2.3 根際和番茄組織附生細菌的分離培養 取抖落根區土壤的番茄根以及莖、葉片等組織,置于無菌三角瓶中,加入含有0.01% Triton X-100的PBS溶液中,25℃、150 r/min振蕩3 h,將振蕩后的液體經5000 r/min離心20 min,用無菌PBS溶液重懸沉淀,分別得到根際、莖和葉片的附生細菌懸液。將菌懸液用無菌水按10倍梯度稀釋后,分別涂布于TSA培養基上,37℃倒置培養,并觀察、計數。
1.2.4 番茄組織內生細菌的分離培養 對上述經PBS溶液清洗的番茄根、莖和葉片,參照文獻[10]方法進行表面滅菌(80%乙醇,2 min;5% NaClO溶液,10 min),用無菌水清洗3遍,并將清洗液涂布于TSA培養基,以確認表面滅菌是否徹底。參照文獻[11-13]分離番茄組織內生細菌,用無菌刀片將各滅菌后的組織切成約0.5 cm × 0.5 cm的小塊,置于無菌研缽中研磨,研磨物中加入PBS溶液于150 r/min振蕩1 h后,經4層無菌紗布過濾,濾液5000 r/min離心20 min,用無菌PBS溶液重懸沉淀,得各組織的內生細菌懸液。將細菌懸液用無菌水梯度稀釋后涂布于TSA培養基上,37℃倒置培養后觀察、計數。
對于果實中內生細菌的分離,將番茄果實經表面滅菌后(80%乙醇,2 min;5% NaClO溶液,10 min),在中部橫切,分離其中果皮、胎座、種子及其表面膠狀物等4個部分。將各部分研磨后,過濾、離心,分離各部分的內生細菌,并涂布TSA平板,觀察、計數,方法同上。
1.2.5 番茄組織附生和內生細菌的16S rRNA拷貝數分析 利用實時熒光定量PCR法計算各樣品中細菌的 16S rRNA 拷貝數[14]。
16S rRNA拷貝數標準曲線的繪制:采用Easy-Pure Bacteria Genome DNA Kit(全式金,北京)提取大腸桿菌DH5α(Escherichia coli DH5α)的基因組DNA。采用細菌通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和 1492R(5'-GGTTACCTTGTTACGACTT-3')擴增16S rRNA序列,采用Zymoclean Gel DNA Recovery Kit(ZYMO,USA)對PCR產物進行切膠回收,采用NanoDrop 2000測定其濃度。使用 ddH2O 稀 釋 DNA 至 濃 度 為 1、10-1、10-2、10-3、10-4、10-5和 10-6ng/μL(1 ng 模板對應的 16S rRNA拷貝數為6.14 × 108),并以此為模板,采用細菌通用 引 物 SRV3-1(5'-CGGTCCAGACTCCTACGGG-3')和 SRV3-2(5'-TTACCGCGGCTGCTGGCAC-3') 對16S rRNA的V3區進行PCR擴增。Real-time PCR反應體系(20 μL):2 × Top Green qPCR SuperMix(全式金,北京)10 μL、引物(10 μmol/L)各 0.5 μL、模板(10 ng/μL)1.0 μL,用 ddH2O 補足 20 μL ;反應程序為 95℃ 5 min;95℃ 10 s,55℃ 10 s,72℃ 10 s,45個循環。以模板中16S rRNA拷貝數的lg值為橫坐標,以Real-time PCR所得的Ct值為縱坐標,繪制標準曲線。
附生和內生細菌基因組DNA提取:將分離得到的番茄根區土壤、根際、組織附生和內生細菌懸液,利用FastDNATMSpin Kit for Soil試劑盒(MPbio,USA)進行基因組DNA提取,并采用DNA產物純化試劑盒(天根,北京)對提取的DNA進行純化。采用NanoDrop 2000(Thermo Scientific,USA)測定DNA濃度,用0.8%的瓊脂糖凝膠電泳檢測DNA的完整性。
細菌16S rRNA拷貝數分析:以純化后的細菌基因組DNA為模板,利用細菌通用引物SRV3-1和SRV3-2進行PCR擴增,PCR反應體系和程序同1.2.5。根據Real-time PCR反應的Ct值,結合標準曲線計算出番茄各組織附生和內生細菌的16S rRNA拷貝數。
1.2.6 番茄組織附生和內生細菌的DGGE分析 以番茄根區土壤、根際、組織附生和內生細菌基因組DNA為模板,采用通用引物341F-GC(5'-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGC ACGGGGGGCCTACGGGAGGCAGCAG-3')和518R(5'-ATTACCGCGGCTTGCTGG-3') 擴 增 細 菌16S rRNA的V3區片段。PCR反應體系(50 μL):2×Fast Pfu SuperMix(全式金,北京)25 μL、引物(10 μmol/L)各 2 μL、模板(10 ng/μL)1 μL,用 ddH2O補足至 50 μL。PCR 反應條件:95℃ 5 min;93℃ 30 s,65℃ 40 s,72℃ 1 min,10 個循環 ;93℃ 30 s,60℃40 s,72℃ 1 min,10 個循環 ;93℃ 30 s,55℃ 40 s,72℃ 1 min,9 個循環 ;93℃ 30 s,55℃ 40 s,72℃5 min。PCR產物經0.8%瓊脂糖凝膠電泳檢測后,-20℃保存備用。
DGGE試驗參照房嫚嫚等[15]方法并進行適當修改。PCR產物采用DcodeTM基因突變檢測系統(Bio-Rad,USA)進行DGGE電泳,聚丙烯酰胺梯度為6%-12%,變性劑梯度為20%-55%(以尿素7 mol/L、甲酰胺40%時的變性劑濃度為100%)。電泳結束后,凝膠經SYBR Green I(Invitrogen,USA)染色后在成像系統中觀察并拍照。使用Quantity One(v4.6.2,Bio-Rad)對DGGE圖譜進行分析,2個樣本的相似性通過戴斯系數(Cs)條帶模式進行估測(公式1),用非加權平均法(UPGMA)檢驗各番茄樣品的相似性。

其中,j是兩條DGGE泳道中共有的條帶數,a和b分別為兩條DGGE泳道各自的條帶數。
采用多樣性指數(H)、豐富度指數(S)和均勻度指數(EH)等指標比較各樣品的細菌多樣性。H的計算公式如下:

其中,S為樣品中所有條帶的數目總和,Pi為樣品中某一條帶(i)的強度在該樣品所有條帶總強度中所占的比率(%)。
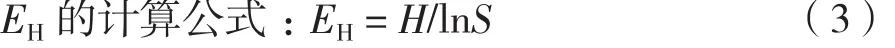
1.2.7 數據處理 試驗結果采用3次獨立生物學重復的平均值 ± 標準差(x-±s)表示,采用Microsoft Excel 2010進行數據處理和作圖,用DPS 2000軟件對數據進行方差分析,運用鄧肯氏新復極差法進行差異顯著性(P < 0.05)檢驗。
2 結果
2.1 番茄組織附生和內生可培養細菌的分離與計數
對番茄根區和根際土壤中的細菌進行分離培養并計數(圖1-A),根區和根際土壤中可培養細菌數量分別為8.19×107和1.81×109CFU/g土壤,根際土壤中的細菌數量顯著高于根區土壤,是根區土壤的約22倍。對番茄根、莖和葉片附生的可培養細菌進行計數(圖1-B),根附生的細菌數量最多(3.05×107CFU/g FW),莖和葉片附生的細菌數量次之,分別為8.53×106和1.12×107CFU/g FW。
圖1-C表示番茄各組織內生的可培養細菌數量,內生細菌的數量遠低于附生細菌。各組織內生細菌數量依次為 :根 > 果皮 > 葉片 > 胎座 ≈ 種子 > 莖 >種子表面膠狀物。根內生細菌數量最多,為1.16×105CFU/g FW,而種子表面膠狀物中細菌數量最少,只有6.62×102CFU/g FW。
2.2 番茄組織附生和內生細菌的16S rRNA拷貝數分析
采用Real-time PCR檢測番茄土壤及各組織附生和內生的細菌16S rRNA拷貝數(圖2)。根區和根際土壤中16S rRNA拷貝數分別為1.96×109/g和5.13×109/g(圖2-A);根、莖和葉片的附生細菌16S rRNA拷貝數分別為3.05×108/g、2.22×108/g和1.60×108/g(圖2-B),與傳統細菌培養法計數結果相比,變化趨勢一致,但數量上較傳統培養法多1-2個數量級。對于內生細菌16S rRNA拷貝數,較傳統培養法,在數量上多2-4個數量級。其中,根中拷貝數最多,葉片與莖內生數量相當。果實的內生細菌 16S rRNA 拷貝數依次為果皮 > 種子 > 胎座 > 種子表面膠狀物(圖2-C)。
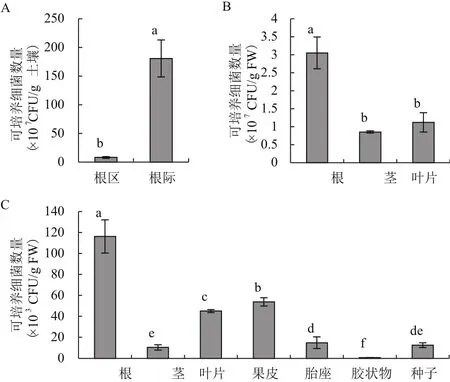
圖1 番茄根區和根際土壤(A)、組織附生(B)和內生(C)可培養細菌數量
圖2 番茄根區和根際土壤(A)、組織附生(B)和內生(C)細菌16S rRNA拷貝數
2.3 番茄組織附生和內生細菌群落相似度和多樣性分析
采用DGGE技術對番茄根區和根際土壤及各組織附生和內生細菌進行多樣性分析,各樣品的條帶幾乎布滿整個泳道(圖3),表明DGGE的變性劑梯度范圍合理,且各樣品均具有較高的細菌多樣性。各樣品所含條帶數目不等,其中番茄根際土壤條帶數最多,說明根際微生物最豐富。
在DGGE圖譜中,條帶a、e、f、n、o、p、u、v出現在所有樣品中,其中條帶a、e、o在各樣品中位置相同,亮度也相近,條帶f、n、p、u、v在內生菌樣品中條帶亮度較高,而在土壤和附生菌樣品中較弱,說明這些條帶代表的細菌種類在番茄組織內生菌中占有優勢地位。在7個番茄組織(根、莖、葉片、果實)內生細菌樣品中,檢測到共有條帶10個,即 a、e、f、l、n、o、p、u、v、w,其中條帶 l、w僅在內生菌樣品中出現,在土壤和附生菌中均未檢測到。在4種果實(果皮、胎座、種子及其表面膠狀物)的內生細菌樣品中共檢測到12個共有條帶,即條帶 a、e、f、k、l、n、o、p、t、u、v、w,其中 k、t為僅在果實樣品中出現的特征條帶。

圖3 番茄組織附生和內生細菌DGGE圖譜
根據DGGE圖譜分析各樣品細菌群落間的相似度(表1),并進行聚類分析,結果(圖4)表明,進化樹被分為3個分支,分別為根區土壤細菌分支、附生細菌分支和內生細菌分支。在附生菌分支中,葉片附生和莖附生細菌群落間相似度為0.56,高于二者與根際細菌間的相似度。而在內生菌分支中,莖內生、葉片內生和根內生樣品間的細菌群落相似度達到0.62以上,三者聚類在一起。此外,果實中胎座內生、種子內生和種子表面膠狀物內生的細菌群落間的相似度分別為0.61、0.67和0.65,三者聚為一類。
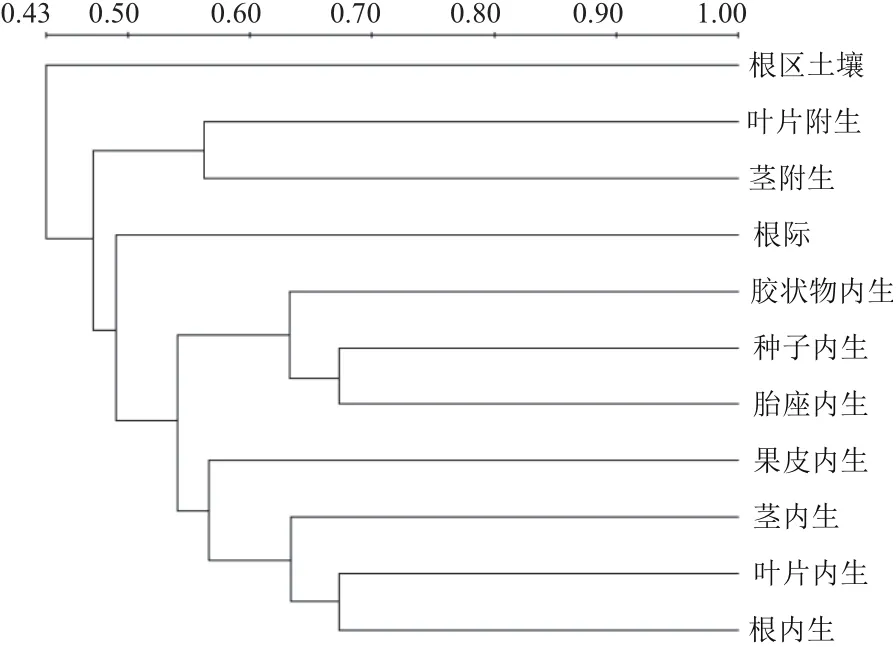
圖4 番茄組織附生和內生細菌DGGE圖譜的聚類分析
根據DGGE圖譜計算各樣品中細菌多樣性指數H、豐富度指數S和均勻度指數EH(表2)。在11個番茄樣品中,根際細菌的H指數和S指數最高;其次為根區土壤;莖和葉片附生細菌的H指數和S指數低于土壤樣品中;內生細菌樣品中,除根和果皮內生細菌的H指數和S指數與附生菌相當,其余樣品的H指數和S指數顯著低于附生細菌,其中尤以種子表面膠狀物內生細菌的H指數和S指數最低。番茄各樣品的均勻度指數均約0.95,差異不顯著。
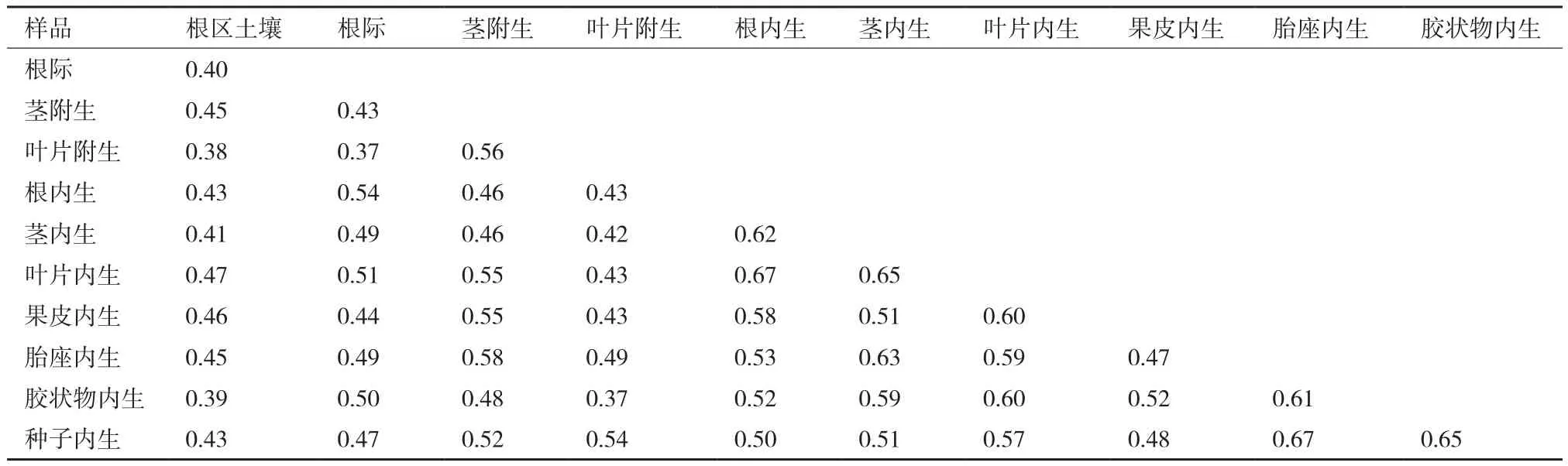
表1 番茄組織附生和內生細菌群落間的相似度
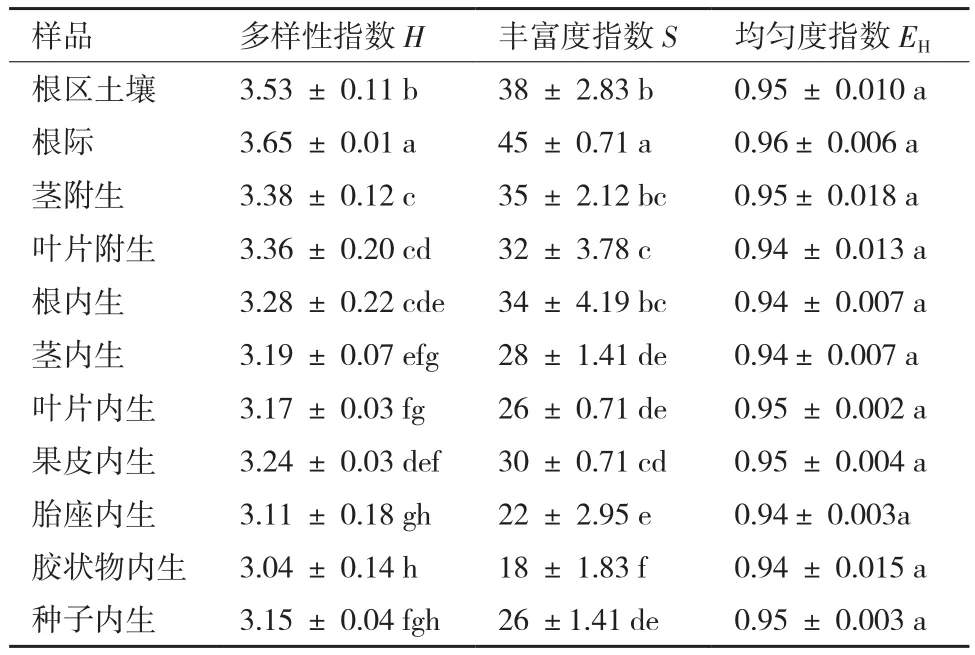
表2 番茄組織附生和內生細菌的多樣性指數
3 討論
植物組織器官表面和內部著生的各類細菌,是植物生態系統的重要組成,與植物密切互作,消長相關。植物的種類、生長發育情況等影響著這些細菌的群落結構,而這些細菌又通過自身的生命活動影響植物的生長發育[16]。本實驗中,采用傳統平板培養方法結合16S rRNA拷貝數分析發現,對于日光溫室中栽培的番茄,根際細菌數量最多,遠高于根區土壤,根系分泌物中豐富的糖、蛋白、脂肪等有機物質為微生物生存提供有機碳源,使得根際微生物數量比根際外的土壤微生物數量高幾倍到幾十倍[17]。對于番茄各組織的附生細菌數量,以根部最多,莖和葉片次之。這是由于在根周圍的土壤中,細菌可以存活幾年甚至幾十年,而莖和葉片等組織的生活周期相對較短[18],且相較根部,莖、葉表面細菌生存環境相對嚴苛,可被利用的營養成分較少,溫濕度波動較大,且紫外線輻射對細菌的生存也會有較大影響[19],從而導致根系附生的細菌數量顯著大于莖、葉片表面附生的細菌數量。
研究還發現,番茄內生細菌的數量顯著低于附生細菌,且在不同組織中有顯著不同,以根內生細菌數量最多,這與之前在辣椒、番茄、茄子等植物中的報道結果一致[20-22],主要是由于根所在的土壤環境含有比空氣中更為豐富的細菌資源,且根的表面積顯著大于其他組織或器官,使根成為細菌進入植物的主要入口,導致根內生細菌數量最多,種類最豐富。果皮內生細菌的數量和多樣性僅次于根,推測果皮中富含葡萄糖、果糖、蔗糖、半乳糖等糖類物質,以及蘋果酸、檸檬酸、琥珀酸、丁酸、脯氨酸等有機酸和氨基酸[23],可為細菌的生長繁殖提供良好的營養條件,引起細菌在果皮中富集。
本實驗進一步采用DGGE技術對土壤細菌、以及番茄各組織附生和內生細菌的多樣性進行了比較分析。DGGE圖譜顯示番茄各樣品均具有較高的細菌多樣性,但以根際細菌的多樣性指數和豐富度指數最高,根區土壤次之,附生和內生樣品最低,其中尤以種子表面膠狀物中內生細菌的多樣性指數和豐富度指數最低。有8條條帶存在于番茄所有樣品中,其中3條條帶在各樣品中位置相同,亮度相近,說明這些條帶代表的細菌在土壤和番茄各組織表面和內部均占有優勢地位;有5條條帶在番茄內生組織中亮度較高,而在其他樣品中較弱,說明這些條帶代表的細菌種類在番茄組織內生菌中占有優勢地位。對DGGE圖譜的聚類分析發現,根際細菌與組織附生和內生細菌的群落相似度顯著高于根區土壤,根際與莖、葉片附生細菌聚為一個分支,7個組織內生樣品聚為一個內生分支,暗示細菌在番茄植株表面和內部的定殖具有選擇性。番茄植株不同組織內生細菌間的相似度最高,根、莖和葉片內生細菌群落的相似度約為0.65,果實胎座、種子表面膠狀物以及種子內生細菌群落的相似度也達到0.6以上。這些結果表明,番茄植株各組織,尤其是相鄰組織的內生細菌間可能具有流動性[24]。相似度分析還發現,相較于附生細菌,根際細菌與組織內生細菌群落間的相似度更高,推測番茄內生細菌可能主要來源于根際,通過根部吸收,并在進入植物內部后在其體內遷移,最終在各個組織間選擇性地定殖[25-26]。
本實驗通過平板培養計數結合分子生物學手段,明確了番茄根區、根際土壤以及根、莖、葉片、果實、種子等組織的附生和內生細菌的數量、多樣性及各樣品間的相似度。但上述方法也存在一定的局限性,未能在種屬水平上鑒定各樣品的細菌群落組成。故采用高通量測序技術鑒定番茄各樣品的細菌群落結構,全面解析土壤細菌以及組織附生和內生細菌間的相互關系,將是下一步開展的工作重點。
4 結論
番茄根區、根際土壤以及組織附生和內生細菌數量依次為 :根際土壤 > 根區土壤 > 附生 > 內生,組織內生中以根和果皮內生細菌數量最多,葉片、莖和種子次之,種子表面膠狀物內生細菌數量最少。DGGE分析表明細菌在番茄植株表面和內部的定殖具有選擇性,且組織內生細菌與根際細菌群落密切相關,相鄰組織的內生細菌群落結構相似,表明內生細菌在組織間具有流動性。

