山西運城漢族群體23個STR基因座遺傳多態性研究
陳亞明,王雪琴,高紅艷,白慧茹,賈富全
(1.山西省運城市公安局,山西 運城 044000;2.重慶市公安局渝北區分局,重慶 401100;3.遵義醫學院附屬醫院司法鑒定中心,貴州 遵義 563000;4.內蒙古醫科大學,內蒙古自治區 呼和浩特010000)
短串聯重復序列(Short tandem repeats,STRs)是法醫個人識別和親子鑒定應用最為廣泛的常規遺傳標記之一[1]。隨著STR分型技術的發展,目前國內外已經開發了大量STR分型試劑盒,所采用的基因座數目不斷增多,鑒別能力也進一步提高[2]。新開發的華夏TM白金PCR擴增試劑盒(美國,賽默飛世爾科技)針對中國人群專門設計,采用六色熒光標記系統可同時檢測23個常染色體STR基因座以及確定性別的1個Y-InDel(rs2032678)和牙釉基因座。該試劑盒包含了國際上通用的美國聯合DNA檢索系統(CODIS)、歐洲標準基因座(ESS)以及中國國家DNA數據庫推薦使用的全部的基因座,在具有更高鑒別能力的同時可兼容絕大多數既往開發的試劑盒,有效實現DNA數據庫中歷史數據的共享[3]。
針對特定群體遺傳多態性數據的調查是法醫學應用的基礎,然而華夏TM白金試劑盒在中國不同群體中的數據較為缺乏。本研究采用華夏TM白金復合STR檢測系統,對山西運城地區漢族人群23個STR基 因 座 (D3S1358,vWA,D16S539,CSF1PO,TPOX,D8S1179,D21S11,D18S51,Penta E,D2S441,D19S433,TH01,FGA,D22S1045,D5S818,D13S317,D7S820,D6S1043,D10S1248,D1S1656,D12S391,D2S1338和Penta D)遺傳多態性進行了調查,為該地區人群的法醫學應用及其他群體遺傳研究提供了基礎數據。
1 對象與方法
1.1 研究對象
經知情同意,采集山西運城漢族397名健康、無血緣關系個體(男242名,女155名)外周血1~2 mL,EDTA抗凝,Chelex-100法提取基因組DNA,-20℃保存備用。
1.2 PCR擴增
按照產品指南,使用華夏TM白金PCR擴增試劑盒(美國賽默飛世爾科技)對23個STR基因座、以及Y-InDel(rs2032678)和牙釉基因進行復合擴增,PCR反應在ABI 9700型擴增儀上進行。
1.2 分型和命名
擴增產物采用ABI 3500型遺傳分析儀電泳分離,使用 GeneMapper ID-X1.4進行等位基因分型。在對每一批次樣本檢測中,以 DNA9948作為陽性對照,ddH2O作為陰性對照。等位基因命名通過與廠家提供的等位基因分型標準物比較,并遵照國際法醫遺傳學會(International Society for Forensic Genetics,ISFG)推薦的原則進行命名。
1.3 統計學分析
采用Powerstats V1.2軟件對各基因座進行Hardy-Weinberg平衡檢驗,采用SHEsis[4]軟件包進行23個STR基因座的配對連鎖不平衡檢驗。采用Powerstats V1.2(Promega美國)計算等位基因頻率、雜合度(heterozygosity,H)、多態性信息量(polymorphism information content,PIC)、匹配概率(probability of match,Pm)、典型父權指數(typical paternal index,TPI)、個體識別率(discrimination power, DP)和非父排除率(probability of exclusion,PE)。應用公式[5]計算累積個體識別率(cumulative power of discrimination,CDP)和累積非父排除率(cumulative probability ofexclusion,CPE)。
對已公開報道來自不同區域的18個漢族在群體,采用19個共有的基因座進行群體間遺傳關系分析。采用 Phylip 3.695軟件包計算配對的群體間Nei’s遺傳距離,采用Mega 7.0構建相鄰連接(Neighbour-Joining,NJ)系統發生樹。
2 結果
2.1 等位基因頻率分布
在受檢的397例山西運城漢族無關個體中,23個STR基因座上共檢出261個等位基因,等位基因頻率分布在 0.0010~0.5430 (表 1)。
2.2 法醫學參數
對 23個STR基因座Hardy-Weinberg平衡檢驗的P值為 0.061(D3S1358)~0.968(D5S818)(表2),配對連鎖不平衡檢驗D'值為 0.081~0.304(表 3),r2為 0.001~0.008(表 4),H 值為 0.5869(TPOX)~0.9194(PentaE),PIC 值為 0.5419(TPOX)~0.9194(PentaE),DP 值為 0.7810(TPOX)~0.9853(PentaE),PE 值為0.2755(TPOX)~0.8352(PentaE),累積個體識別率為1-3.6566×10-10,累積非父排除率為 1-2.7171×10-27。
2.3 群體關系分析
根據19個STR基因座計算得到19個不同地區漢族群體的Nei’s遺傳距離(表5),山西運城漢族與其他群體遺傳距離值范圍為0.0057(河南)~0.0224(廣西)。
根據Nei’s遺傳距離構建的系統發生樹(圖1)顯示,山西運城漢族與河南漢族首先聚類,隨后與甘肅、山東、河北的漢族聚集為一個分支。其余來自中國南方的漢族聚集為另一個大的分支,包括四川、重慶、湖北、湖南、福建、廣東、廣西、海南。來自廣東不同區域的漢族較其他省份漢族更加集中地聚集在一起。
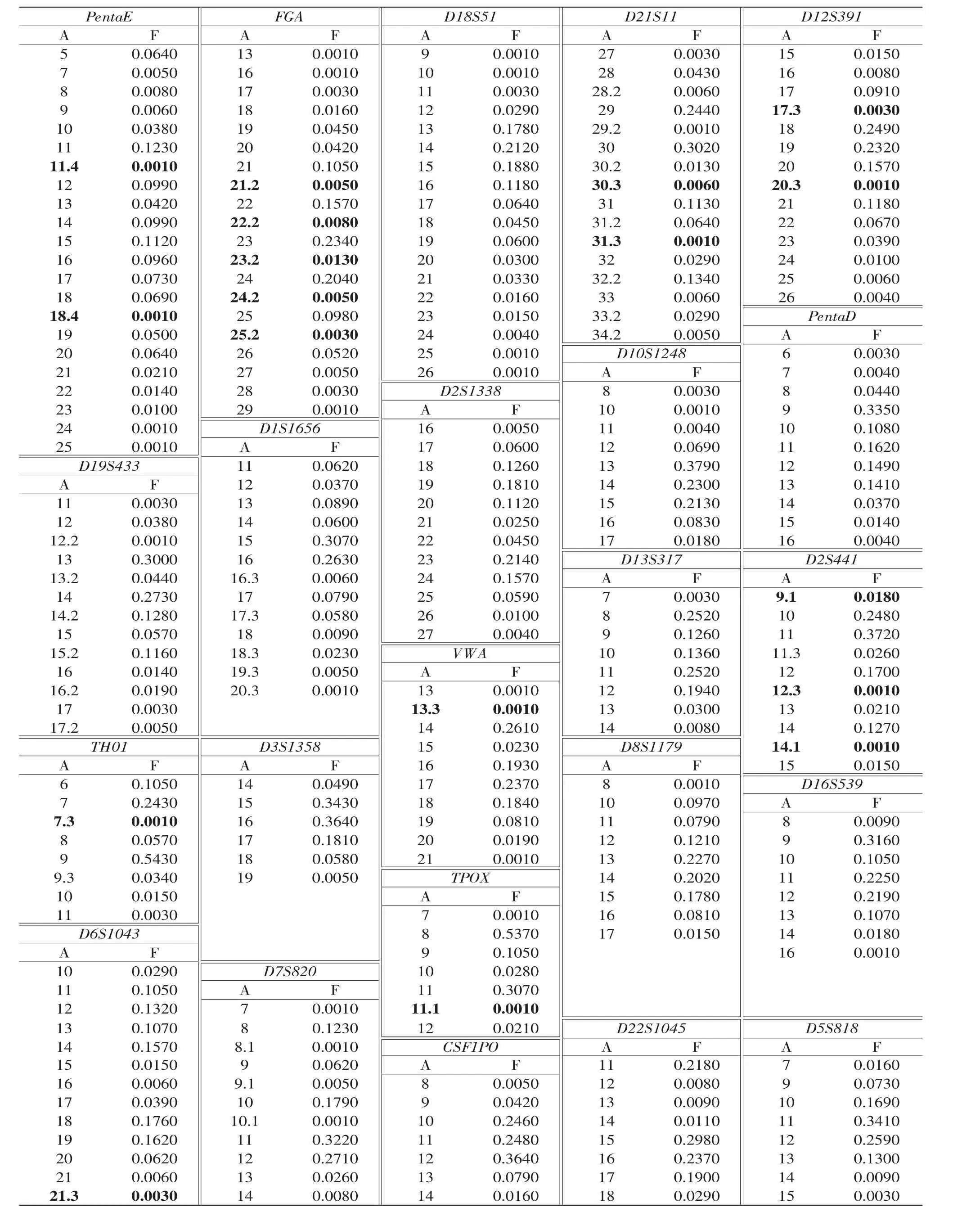
表1 山西運城地區漢族人群23個STR基因座等位基因頻率分布(n=397)
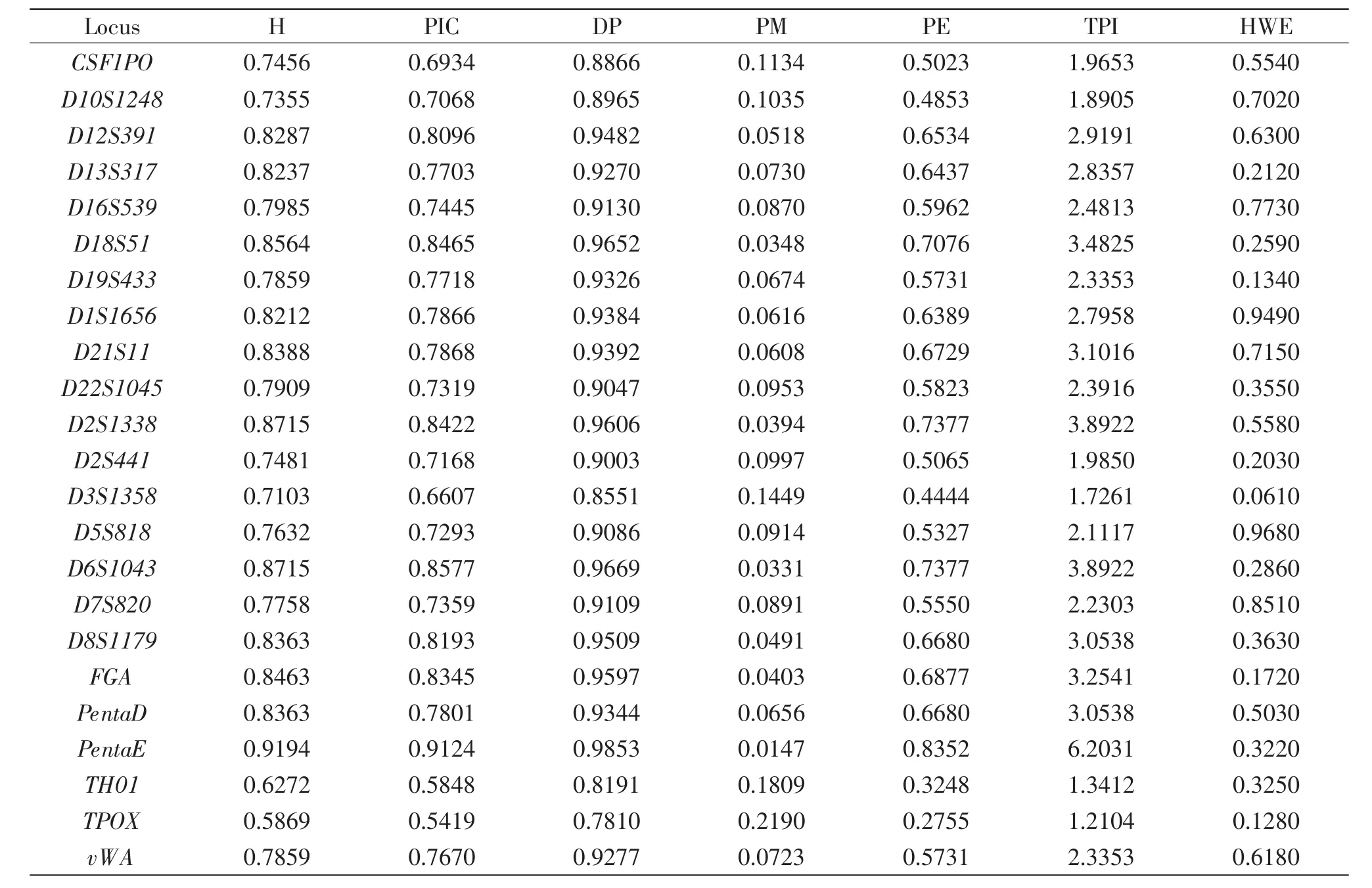
表2 山西運城漢族群體23個STR基因座法醫學參數
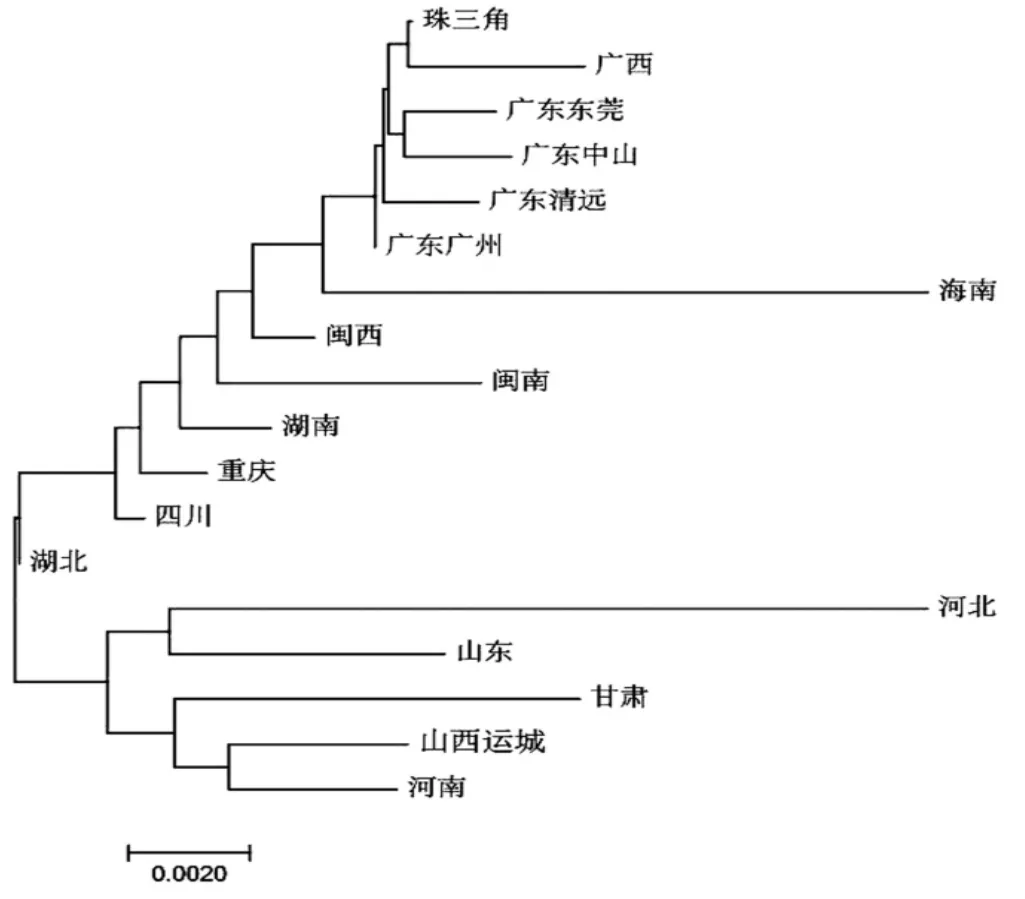
圖1 基于Nei’s遺傳距離構建的山西運城漢族和其他18個中國漢族人群的相鄰連接系統發生樹

值'D驗檢衡平不鎖連對配座因基R T S個3 2 3表A v W X T P O 0 1 T H E P e n t a D P e n t a A F G 8 S 1 1 7 9 D 7 S 8 2 0 D 6 S 1 0 4 3 D 5 S 8 1 8 D 3 S 1 3 5 8 D 2 S 4 4 1 D 2 S 1 3 3 8 D 2 2 S 1 0 4 5 D 2 1 S 1 1 D 1 S 1 6 5 6 D 1 9 S 4 3 3 D 1 8 S 5 1 D 1 6 S 5 3 9 D 1 3 S 3 1 7 D 1 2 S 3 9 1 D 1 0 S 1 2 4 8 D C S F 1 P O'D 0.1 4 8 0 0.1 4 4 0 0.0 9 5 0 0.1 8 4 0 0.1 2 6 0 0.1 5 7 0 0.1 5 8 0 0.1 3 6 0 0.1 7 5 0 0.1 7 1 0 0.0 8 3 0 0.1 4 2 0 0.1 8 9 0 0.1 5 1 0 0.1 8 3 0 0.1 6 5 0 0.1 2 4 0 0.1 6 8 0 0.1 5 0 0 0.1 3 9 0 0.1 3 1 0 0.1 5 0 0-C S F 1 P O 0.1 0 6 0 0.1 0 3 0 0.1 6 0 0 0.2 5 5 0 0.1 7 0 0 0.1 7 1 0 0.1 2 8 0 0.1 3 3 0 0.1 3 5 0 0.1 2 7 0 0.1 3 1 0 0.1 4 7 0 0.1 2 5 0 0.1 1 6 0 0.1 5 4 0 0.1 5 4 0 0.1 1 3 0 0.1 7 1 0 0.1 2 0 0 0.1 2 1 0 0.1 4 5 0--1 0 S 1 2 4 8 D 0.1 5 1 0 0.1 2 2 0 0.1 3 6 0 0.2 6 5 0 0.1 8 9 0 0.2 3 7 0 0.1 7 1 0 0.1 6 0 0 0.2 0 9 0 0.1 8 4 0 0.1 3 3 0 0.1 7 6 0 0.2 2 2 0 0.1 4 9 0 0.1 7 2 0 0.1 9 2 0 0.2 0 2 0 0.1 9 4 0 0.1 7 8 0 0.1 5 4 0---1 2 S 3 9 1 D 0.1 3 1 0 0.1 3 9 0 0.1 5 6 0 0.2 4 5 0 0.1 6 7 0 0.2 0 7 0 0.1 4 3 0 0.1 5 5 0 0.2 1 8 0 0.1 5 1 0 0.1 2 6 0 0.1 0 8 0 0.1 7 2 0 0.1 0 1 0 0.1 7 3 0 0.1 6 4 0 0.1 6 9 0 0.1 7 4 0 0.1 2 5 0----1 3 S 3 1 7 D 0.1 1 7 0 0.0 8 1 0 0.1 6 2 0 0.2 0 6 0 0.1 3 7 0 0.1 9 3 0 0.1 3 1 0 0.1 5 3 0 0.2 0 5 0 0.1 1 6 0 0.1 1 2 0 0.1 6 2 0 0.1 8 0 0 0.1 5 6 0 0.1 6 7 0 0.1 4 4 0 0.1 4 4 0 0.1 9 6 0-----1 6 S 5 3 9 D 0.2 0 9 0 0.1 6 8 0 0.1 7 9 0 0.2 8 8 0 0.1 8 6 0 0.2 1 1 0 0.2 4 1 0 0.1 9 5 0 0.2 3 8 0 0.1 6 7 0 0.1 5 2 0 0.1 8 4 0 0.2 3 9 0 0.2 0 3 0 0.2 5 0 0 0.2 2 6 0 0.2 1 2 0------1 8 S 5 1 D 0.1 9 0 0 0.1 7 4 0 0.1 4 5 0 0.2 6 1 0 0.2 0 7 0 0.2 2 1 0 0.1 7 0 0 0.1 9 0 0 0.2 1 7 0 0.1 4 8 0 0.1 3 8 0 0.1 6 7 0 0.1 7 5 0 0.1 6 2 0 0.2 1 0 0 0.1 8 5 0-------1 9 S 4 3 3 D 0.1 8 8 0 0.1 4 1 0 0.1 8 4 0 0.2 8 9 0 0.1 8 3 0 0.1 7 8 0 0.1 9 3 0 0.1 6 9 0 0.2 2 5 0 0.1 6 7 0 0.1 4 6 0 0.1 3 6 0 0.2 0 7 0 0.1 3 9 0 0.2 0 6 0--------1 S 1 6 5 6 D 0.1 9 2 0 0.1 5 6 0 0.1 8 6 0 0.2 4 9 0 0.2 3 4 0 0.2 3 3 0 0.1 7 4 0 0.1 5 2 0 0.2 3 2 0 0.1 6 5 0 0.1 7 7 0 0.1 4 9 0 0.2 2 0 0 0.1 4 1 0---------2 1 S 1 1 D 0.1 3 3 0 0.0 9 3 0 0.1 6 4 0 0.2 4 7 0 0.1 3 0 0 0.1 6 5 0 0.1 0 9 0 0.1 3 0 0 0.1 4 6 0 0.1 1 1 0 0.1 2 5 0 0.1 1 6 0 0.2 1 0 0----------2 2 S 1 0 4 5 D 0.2 0 2 0 0.1 0 9 0 0.1 8 6 0 0.3 0 4 0 0.1 9 4 0 0.2 2 4 0 0.1 9 7 0 0.1 8 0 0 0.1 7 4 0 0.1 6 0 0 0.1 7 8 0 0.1 6 6 0-----------2 S 1 3 3 8 D 0.1 0 6 0 0.1 6 5 0 0.1 1 4 0 0.1 8 8 0 0.1 5 1 0 0.1 8 2 0 0.1 6 0 0 0.1 3 2 0 0.1 7 7 0 0.1 6 7 0 0.1 3 2 0------------2 S 4 4 1 D 0.1 3 2 0 0.0 8 2 0 0.1 3 5 0 0.2 0 6 0 0.1 5 0 0 0.1 5 8 0 0.1 3 8 0 0.1 2 4 0 0.1 6 2 0 0.0 9 7 0-------------3 S 1 3 5 8 D 0.1 3 1 0 0.1 0 6 0 0.1 2 7 0 0.1 8 2 0 0.1 6 4 0 0.1 9 2 0 0.1 2 4 0 0.1 6 1 0 0.1 6 1 0--------------5 S 8 1 8 D 0.1 6 3 0 0.1 4 6 0 0.2 2 9 0 0.2 6 2 0 0.1 9 7 0 0.2 2 2 0 0.1 6 8 0 0.1 8 5 0---------------6 S 1 0 4 3 D 0.1 3 5 0 0.1 1 4 0 0.1 6 9 0 0.2 4 8 0 0.1 9 8 0 0.1 5 2 0 0.1 9 0 0----------------7 S 8 2 0 D 0.1 6 9 0 0.1 6 5 0 0.1 4 5 0 0.2 5 4 0 0.1 6 0 0 0.2 1 1 0-----------------8 S 1 1 7 9 D 0.1 8 5 0 0.1 5 6 0 0.2 3 1 0 0.2 5 9 0 0.2 0 1 0------------------A F G 0.1 4 6 0 0.1 8 5 0 0.1 4 3 0 0.2 3 6 0-------------------D P e n t a 0.2 1 5 0 0.2 3 0 0 0.1 9 8 0--------------------E P e n t a 0.1 5 0 0 0.1 2 8 0---------------------0 1 T H 0.1 2 0 0----------------------X T P O 0.1 2 0 0----------------------A v W

2值r驗檢衡平不鎖連對配座因基R T S個3 2 4表A v W X T P O 0 1 T H E P e n t a D P e n t a A F G 8 S 1 1 7 9 D 7 S 8 2 0 D 6 S 1 0 4 3 D 5 S 8 1 8 D 3 S 1 3 5 8 D 2 S 4 4 1 D 2 S 1 3 3 8 D 2 2 S 1 0 4 5 D 2 1 S 1 1 D 1 S 1 6 5 6 D 1 9 S 4 3 3 D 1 8 S 5 1 D 1 6 S 5 3 9 D 1 3 S 3 1 7 D 1 2 S 3 9 1 D 1 0 S 1 2 4 8 D C S F 1 P O 2 r 0.0 0 3 0 0.0 0 5 0 0.0 0 1 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0 0.0 0 5 0 0.0 0 3 0 0.0 0 3 0 0.0 0 5 0 0.0 0 1 0 0.0 0 4 0 0.0 0 3 0 0.0 0 6 0 0.0 0 6 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 5 0 0.0 0 3 0 0.0 0 1 0 0.0 0 4 0-C S F 1 P O 0.0 0 2 0 0.0 0 2 0 0.0 0 5 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 1 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 1 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0 0.0 0 1 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0--1 0 S 1 2 4 8 D 0.0 0 2 0 0.0 0 1 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0 0.0 0 4 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0---1 2 S 3 9 1 D 0.0 0 2 0 0.0 0 4 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 4 0 0.0 0 3 0 0.0 0 4 0 0.0 0 5 0 0.0 0 4 0 0.0 0 3 0 0.0 0 1 0 0.0 0 2 0 0.0 0 1 0 0.0 0 3 0 0.0 0 2 0 0.0 0 4 0 0.0 0 3 0 0.0 0 2 0----1 3 S 3 1 7 D 0.0 0 2 0 0.0 0 1 0 0.0 0 5 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0-----1 6 S 5 3 9 D 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0------1 8 S 5 1 D 0.0 0 4 0 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0-------1 9 S 4 3 3 D 0.0 0 5 0 0.0 0 3 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 1 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0--------1 S 1 6 5 6 D 0.0 0 3 0 0.0 0 4 0 0.0 0 3 0 0.0 0 2 0 0.0 0 8 0 0.0 0 3 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0 0.0 0 5 0 0.0 0 2 0---------2 1 S 1 1 D 0.0 0 3 0 0.0 0 2 0 0.0 0 5 0 0.0 0 4 0 0.0 0 2 0 0.0 0 2 0 0.0 0 1 0 0.0 0 2 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 2 0 0.0 0 5 0----------2 2 S 1 0 4 5 D 0.0 0 5 0 0.0 0 1 0 0.0 0 4 0 0.0 0 4 0 0.0 0 3 0 0.0 0 3 0 0.0 0 4 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0-----------2 S 1 3 3 8 D 0.0 0 2 0 0.0 0 7 0 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 5 0 0.0 0 3 0------------2 S 4 4 1 D 0.0 0 3 0 0.0 0 1 0 0.0 0 6 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 1 0-------------3 S 1 3 5 8 D 0.0 0 2 0 0.0 0 2 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0 0.0 0 4 0 0.0 0 2 0--------------5 S 8 1 8 D 0.0 0 2 0 0.0 0 2 0 0.0 0 5 0 0.0 0 3 0 0.0 0 3 0 0.0 0 3 0 0.0 0 2 0 0.0 0 4 0---------------6 S 1 0 4 3 D 0.0 0 2 0 0.0 0 2 0 0.0 0 5 0 0.0 0 4 0 0.0 0 6 0 0.0 0 1 0 0.0 0 5 0----------------7 S 8 2 0 D 0.0 0 4 0 0.0 0 4 0 0.0 0 2 0 0.0 0 3 0 0.0 0 2 0 0.0 0 3 0-----------------8 S 1 1 7 9 D 0.0 0 3 0 0.0 0 2 0 0.0 0 6 0 0.0 0 2 0 0.0 0 3 0------------------A F G 0.0 0 2 0 0.0 0 5 0 0.0 0 3 0 0.0 0 3 0-------------------D P e n t a 0.0 0 3 0 0.0 0 4 0 0.0 0 2 0--------------------E P e n t a 0.0 0 3 0 0.0 0 3 0---------------------0 1 T H 0.0 0 2 0----------------------X T P O 0.0 0 3 0----------------------A v W
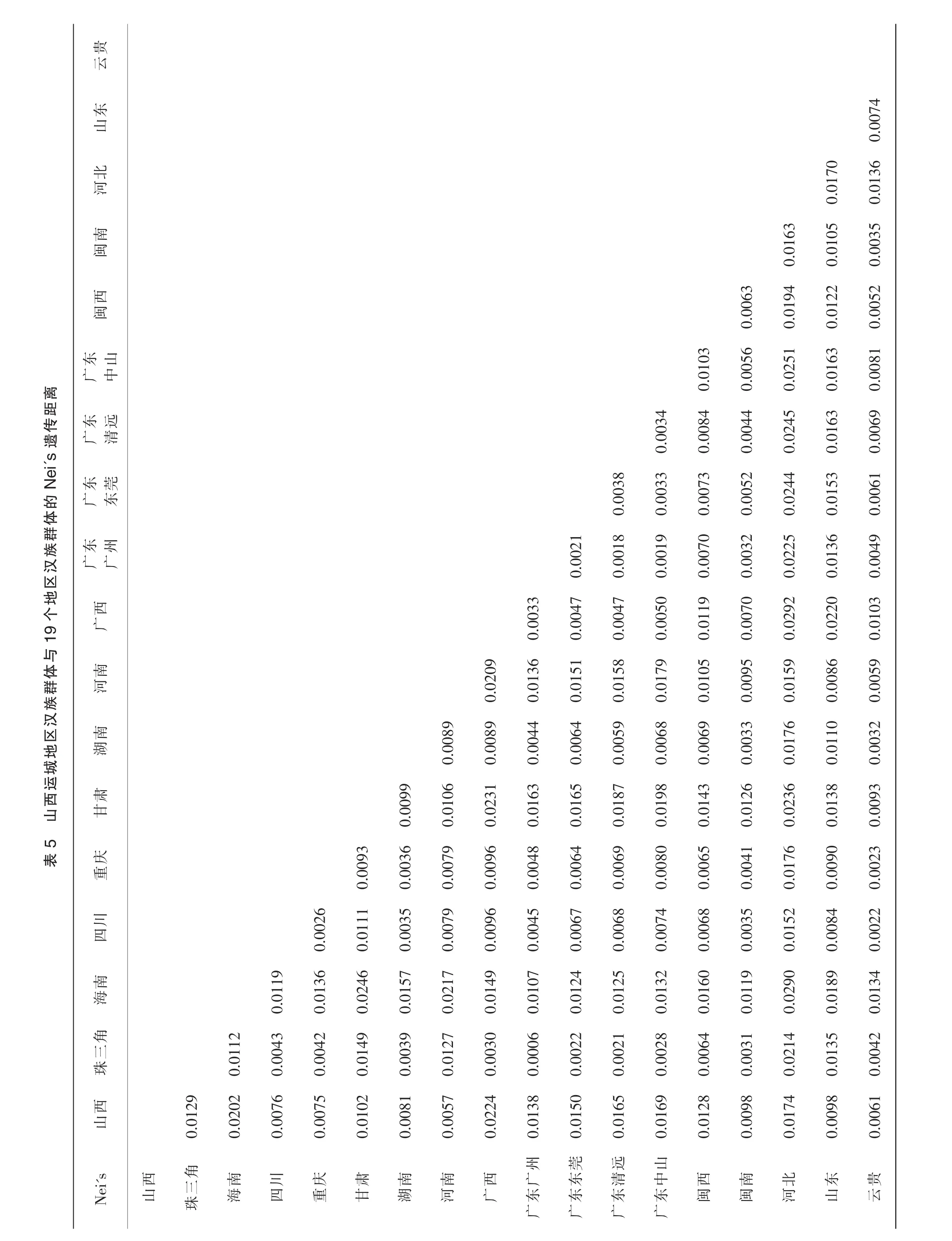
離距傳遺i's e N的體群族漢區地個9 1與體群族漢區地城運西山5表貴云東山北河南閩西閩東山廣中東遠廣清東莞廣東東州廣廣西廣南河南湖肅甘慶重川四南海角三珠西山e i's N西山2 9 0.0 1角三珠1 1 2 0.0 0 2 0.0 2南海9 1 1.0 0 0 4 3 0.0 7 6 0.0 0川四2 6 0.0 0 6 1 3.0 0 0 4 2 0.0 7 5 0.0 0慶重9 3 0.0 0 1 1 0.0 1 6 2 4.0 0 1 4 9 0.0 0 2 0.0 1肅甘9 0 9.0 0 3 6 0.0 0 3 5 0.0 0 7 1 5.0 0 0 3 9 0.0 8 1 0.0 0南湖8 9 0.0 0 6 1 0.0 0 7 9 0.0 0 7 9 0.0 0 7 2 1.0 0 1 2 7 0.0 5 7 0.0 0南河0 9 0.0 2 8 9 0.0 0 1 2 3.0 0 9 6 0.0 0 9 6 0.0 0 9 1 4.0 0 0 3 0 0.0 2 4 0.0 2西廣3 0 3.0 0 3 6 0.0 1 4 4 0.0 0 3 1 6.0 0 4 8 0.0 0 4 5 0.0 0 7 1 0.0 0 0 0 6 0.0 3 8 0.0 1州廣東廣1 0.0 0 2 7 0 4.0 0 5 1 0.0 1 6 4 0.0 0 5 1 6.0 0 6 4 0.0 0 6 7 0.0 0 4 1 2.0 0 0 2 2 0.0 5 0 0.0 1莞東東廣3 8 0.0 0 8 0.0 0 1 7 0 4.0 0 5 8 0.0 1 5 9 0.0 0 7 1 8.0 0 6 9 0.0 0 6 8 0.0 0 5 1 2.0 0 0 2 1 0.0 6 5 0.0 1遠清東廣4 0 3.0 0 3 3 0.0 0 9 0.0 0 1 0 0 5.0 0 7 9 0.0 1 6 8 0.0 0 8 1 9.0 0 8 0 0.0 0 7 4 0.0 0 2 1 3.0 0 0 2 8 0.0 6 9 0.0 1山中東廣3 0.0 1 0 4 0 8.0 0 7 3 0.0 0 0 0.0 0 7 9 1 1.0 0 0 5 0.0 1 6 9 0.0 0 3 1 4.0 0 6 5 0.0 0 6 8 0.0 0 0 1 6.0 0 0 6 4 0.0 2 8 0.0 1西閩6 3 0.0 0 6 0.0 0 5 4 0 4.0 0 5 2 0.0 0 2 0.0 0 3 0 0 7.0 0 9 5 0.0 0 3 3 0.0 0 6 1 2.0 0 4 1 0.0 0 3 5 0.0 0 9 1 1.0 0 0 3 1 0.0 9 8 0.0 0南閩1 6 3.0 0 9 4 0.0 1 1 0.0 2 5 5 2 4.0 0 4 4 0.0 2 5 0.0 2 2 2 2 9.0 0 5 9 0.0 1 7 6 0.0 1 6 2 3.0 0 7 6 0.0 1 5 2 0.0 1 0 2 9.0 0 2 1 4 0.0 7 4 0.0 1北河0.0 1 7 0 1 0 5.0 0 2 2 0.0 1 3 0.0 1 6 3 1 6.0 0 5 3 0.0 1 6 0.0 1 3 0 2 2.0 0 8 6 0.0 0 1 0 0.0 1 8 1 3.0 0 9 0 0.0 0 8 4 0.0 0 9 1 8.0 0 1 3 5 0.0 9 8 0.0 0東山7 4 0.0 0 6.0 1 3 0 0 3 5.0 0 5 2 0.0 0 1 0.0 0 8 9 0 6.0 0 6 1 0.0 0 9 0.0 0 4 3 1 0.0 0 5 9 0.0 0 3 2 0.0 0 3 0 9.0 0 2 3 0.0 0 2 2 0.0 0 4 1 3.0 0 0 4 2 0.0 6 1 0.0 0貴云
3 討論
短串聯重復序列(Short tandem repeats,STRs)是一類廣泛存在于人類基因組中,重復單位為2~6 bp的DNA水平遺傳標記,由于其具有高度的多態性、分型簡便快速、易于標準化和自動化,是法醫個體識別和親子鑒定應用最為廣泛的遺傳標記。為了提高鑒別能力并促進國際DNA數據庫的建立,法醫STR分型體系不斷更新,如美國聯邦調查局將1997年建立的13個STR的CODIS系統于2011年更新至19個,國際刑警組織于1998年建立的4個歐洲標準基因座(ESS)不斷更新至2009年的12個。近年國內外開發的高鑒別能力的試劑盒如Globalfiler、PowerPlex 21、Powerplex Fusion、Goldeneye 25A 等都包含了這些基因座。華夏TM白金試劑盒是最新開發的針對中國人群體遺傳特征的復合STR分型體系,包含了最新的CODIS、ESS和中國國家數據庫中推薦使用的全部基因座[6]。在提供高效的鑒別能力的同時能夠更好地與以往試劑盒兼容,實現數據庫中與其他試劑盒的歷史分型數據的比對。
目前華夏TM白金試劑盒群體遺傳多態性調查數據僅見于廣東、四川、遼寧部分地區的漢族,以及西藏藏族、新疆維吾爾族、四川彝族等少數幾個群體[7-8]。運城地區位于山西省西南部,古稱河東,因“鹽運之城”得名,是中華文明的重要發祥地之一,上古時期便是堯、舜、禹活動的中心區域,特別是垣曲縣“中華世紀曙猿”化石的發現,為人類起源學說提供了更加豐富的論據。運城也是人類最早食用鹽、開始冶煉和農耕文明的地方。其人口數居山西首位,達522.39萬,漢族占99.7%。運城南向與河南濟源毗鄰,西、南隔黃河與陜西渭南、河南三門峽及洛陽相望。因此,采用最新的華夏TM白金PCR擴增試劑盒調查山西運城漢族群體23個基因座進行遺傳多態性,可為其在該地區人群的法醫學應用以及中國人群的群體遺傳研究提供基礎數據。
統計結果顯示,23個基因座都達到了Hardy-Weinberg平衡,說明群體樣本采集符合統計學要求,群體具有代表性,分型數據可靠。23個 STR配對連鎖不平衡檢驗均符合連鎖平衡,說明基因座之間無連鎖關系,可以應用乘積定律計算23個STR組成的復合系統累積的法醫學參數。在本群體研究的23個基因座中,除TPOX、D10S1248、D3S1358、TH01,其余19個STR基因座均為高度多態性的遺傳標記(DP>0.80、PE>0.50),其中Pent E的多態性最高(DP=0.9853,PE=0.8352),其次為D6S1043(DP=0.9670,PE=0.7377),說明在法醫學個人識別、親子鑒定及群體遺傳研究中具有較高的應用價值。23個STR基因座累積個體識別率為1-2.7171×10-27,累積的非父排除率為 1-3.6566×10-10,說明由23個STR組成的華夏TM白金PCR復合檢測體系在山西運城漢族人群中具有高度的多態性和鑒別能力,適用于該地區人群法醫學個人識別和親子鑒定。
本研究在檢測過程發現了大量未包含于ladder中而不能被分型軟件自動命名的等位基因(off-ladder,OL),在9個基因座上共檢出了18個OL等位基因(表1),均為含有不完整重復單位的中間等位基因,其中PentaE(11.4,18.4)、D21S11(31.3)、D12S391(20.3)、vWA(13.3)、TH01(7.3)、D2S441(12.3,14.1)和TPOX(11.1)的頻率低于 0.1%,為稀有等位基因。大量OL等位基因的出現,可能是試劑盒設計時未將部分已知的等位基因納入ladder,也有可能是在新檢測群體中發現的新等位基因。新檢出的等位基因可以在一定程度上反映被檢測群體的多態性特征[9],而OL等位基因真實的序列結構的則有待于通過進一步測序闡明。
遺傳距離計算和聚類關系分析顯示山西運城漢族和地理上最為毗鄰的河南漢族的遺傳關系最近,與 廣東、廣西等南方漢族群體的遺傳關系較遠。所有19個不同區域的中國漢族群體中,北方與南方漢族分別聚集為兩個不同分支,呈現出一定的南北方地理分布的差異及連續過渡的特征。同時在地理位置上接近的群體,如廣東省東莞、中山、清遠、廣州幾個地區的漢族明顯聚集,廣東與珠三角、廣西漢族緊密聚集,山東-河北省、甘肅-山西-河南等相鄰省份聚集為不同的分支,說明地理上越接近的群體,遺傳關系越近。

