基于CRISPR-Cas9系統的Saccharomyces cerevisiae基 因刪除
牛瀟迪,李天明,劉金雷,楊天勇,李子怡,馮惠勇,*
(1.河北科技大學生物科學與工程學院,河北 石家莊 050018;2.馬里蘭大學帕克分校文理科學學院,美國 馬里蘭 20742)
釀酒酵母(Saccharomyces cerevisiae)是食品級GRAS(generally regarded as safe)微生物,是釀造業、焙烤業廣泛使用的微生物,也是重組蛋白高效表達的理想宿主菌之一。另外,由于其具有魯棒性好、細胞量大、在低pH值條件(甚至pH值小于3.0)下生長良好、能耐受高濃度的底物和發酵產品具有安全性等優點,作為“細胞工廠”的底盤細胞,被廣泛應用于食品添加劑、有機酸、飼料添加劑等生產[1-5]。基因組改造是提高生產性能的有效途徑。常規的S. cerevisiae的基因改造方法是采用Cre-loxP系統[6],該方法利用同源重組機制采用抗生素或者營養缺陷型進行篩選,一般一次只能進行單個基因的刪除;由于適用于S. cerevisiae的抗生素和營養缺陷型種類數量的限制,當進行多個基因刪除時,還要進行第二次轉化,借助Cre-loxP系統去除篩選標記后,才能進行;因此此方法在打靶效率、操作簡便性及工作效率等方面,都有待改進提高。近年來,迅速發展完善的CRISPRCas9基因編輯技術,可以依靠向導RNA(guide RNA,gRNA)識別目標序列,利用Cas9蛋白切割序列[7-11],實施靶位點特異性切割產生DNA雙鏈斷裂,激發細胞同源重組修復機制或者非同源末端連接修復機制,無需利用篩選標記,就可實現高效的基因組的定點敲除、定點敲入和基因修飾[12-13]。CRISPR-Cas9系統相較于重組鋅指核酸酶(zinc-finger nucleases,ZFNs)技術[14]和轉錄激活因子樣效應物核酸酶(transcription activator-like effector nucleases,TALENs)基因編輯技術,有操作簡單[15-17]、設計靈活、價格低廉、能實現多點編輯、可擴展性強等優點[18-20]。Church[20]和Bao Zehua[21]等先后報道了利用CRISPR-Cas9進行S. cerevisiae單倍體細胞基因組編輯的方法,但是利用該方法對S. cerevisiae雙倍體細胞進行高效基因組編輯的方法還鮮見報道。
本研究通過構建Cas9和gRNA的表達質粒,在S. cerevisiae雙倍體細胞中建立了CRISPR-Cas9系統,借助供體DNA(donor DNA)[22-24],實現了can1、pdc、adh3、adh2、adh1、pdc等多個單基因的高效敲除;建立了基因連續敲除和兩個以上基因同時敲除的方法和程序;為在S. cerevisiae中進行大規模的基因編輯提供了方法學基礎,同時也為CRISPR-Cas9技術在其他工業微生物中的應用提供了借鑒。
1 材料與方法
1.1 材料與試劑
1.1.1 菌株與質粒
本實驗所用的菌株、質粒分別如表1所示。

表1 本實驗所用的菌株、質粒Table 1 Strains and plasmids used in this study

續表1
1.1.2 引物與試劑
本實驗所用引物如表2所示。

表2 本實驗所用引物Table 2 PCR primers used in this study
構建及驗證所用聚合酶鏈式反應(polymerase chain reaction,PCR)引物由生工生物工程(上海)股份有限公司合成;FastPfuDNA聚合酶、High Fidelity DNA聚合酶 北京全式金生物技術有限公司;限制性內切酶KpnI、XhoI、SacI、NotI NEB(北京)公司;T4 DNA ligse 大連寶生物工程公司;瓊脂糖凝膠DNA回收試劑盒、質粒小提中量試劑盒、超薄DNA產物純化試劑盒和基因組提取試劑盒 天根生化科技(北京)有限公司。
轉化過程中用到10×TE、1×TE、10×LiAc、1×LiAc(0.1 mol/L),1×LiAc/0.5×TE為10 mL 10×LiAc和5 mL 10×TE用去離子水定容100 mL,1×LiAc/40%聚乙二醇(polyethylene glycol,PEG)3350/1×TE(現用現混)為100 μL 10×LiAc+100 μL 10×TE+800 μL 50% PEG3350混合。
1.1.3 培養基
LB(Luria-Bertani)培養基:胰蛋白胨10 g/L、酵母提取物5 g/L、NaCl 10 g/L,調pH值為7.4左右,需要時加入卡那霉素至終質量濃度0.05g/L。固體培養基則添加瓊脂粉至20 g/L。
SOC(super optimal broth with catabolite repression)培養基:2 g/100 mL胰蛋白胨、0.5 g/100 mL酵母粉、0.05 g/100 mL氯化鈉、2.5 mmol/L氯化鉀、10 mmol/L氯化鎂、20 mmol/L葡萄糖。
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培養基:1 g/100 mL酵母膏、2 g/100 mL 蛋白胨、2 g/100 mL葡萄糖,若制固體培養基,加入2 g/100 mL瓊脂粉。
選擇(synthetic dropout,SD)培養基:無氨基酵母氮源(yeast nitrogen base,YNB)培養基(成分見Solarbio YNB培養基產品介紹)6.7 g,另取葡萄糖5 g加熱溶解于100 mL蒸餾水中,制成10 倍溶液,過濾除菌。使用時,取0.5 mL 10 倍溶液加入4.5 mL無菌蒸餾水中,備用。根據不同的篩選標記添加不同的氨基酸。
1.2 儀器與設備
Finnpipette移液槍 美國Thermo公司;SW-CJ型超凈工作臺 蘇凈集團安泰公司;PCR儀 美國Bio-Rad公司;Jl003A型電子天平 常熟雙杰測試儀器廠;HWS型培養箱 寧波江南儀器廠;F1-F2凝膠成像儀上海天能科技有限公司;Neofuge23R冷凍離心機 力新儀器(上海)有限公司;pH211酸度計 意大利Hanna公司;HYG型搖床 上海欣蕊自動化設備有限公司;PoweWave HT酶標儀 美國伯騰儀器有限公司;DZKW-D水浴鍋 河北黃驊航天儀器廠;BCD-191W/HC冰箱 上海海爾股份有限公司;G80W23YCsL-03(RO)微波爐 上海申安醫療器械廠。
1.3 方法
1.3.1 菌株培養
本研究所用的S. cerevisiae菌株為4 種氨基酸缺陷的雙倍體菌株,缺陷型為Trp-、Leu-、Ura-、His-。親本S. cerevisiae菌株Δura3Δtrp1Δleu2Δhis3(下文均稱為WT)轉化前在YPD培養基,30 ℃、180 r/min條件下培養。轉化后的篩選培養基采用缺乏相應質粒的篩選標記的篩選培養基,30 ℃、200 r/min條件下培養。
1.3.2 CRISPR-Cas9系統的構建

圖1 pRS424-TRP-cas9構建示意圖Fig. 1 Schematic illustration of construction of pRS424-TRP-cas9
CRISPR-Cas9系統需要Cas9核酸酶和gRNA的共同作用才能發揮基因編輯作用,需要構建一個能夠表達cas9的質粒,轉化進酵母菌中使其表達Cas9核酸酶[25-30]。表達Cas9核酸酶的質粒構建方法如下:首先以實驗室保存質粒pMD18-cas9為模板,通過引物cas9-F、cas9-R利用PCR技術擴增出cas9基因片段;以S. cerevisiae288c野生型菌株基因組為模板,通過引物cas9-tef-F、cas9-tef-R利用PCR技術擴增出tef啟動子;以S. cerevisiae288c野生型菌株基因組為模板,通過引物cas9-cyc-F、cas9-cyc-R利用PCR技術擴增出cyc1終止子。為使在細胞質中表達的Cas9蛋白質能夠透過核膜到達細胞核內參與DNA剪切,在Cas9表達質粒中添加一段表達核定位信號(nuclear localization signal,NLS)的基因序列,通過在tef啟動子基礎上利用延伸PCR擴增得到,將所得片段利用Overlapping PCR技術融合得到目的片段,凝膠純化目的片段后與pRS424-TRP空質粒利用KpnI和XhoI進行雙酶切,目的片段與pRS424-TRP空質粒的酶切產物通過超薄DNA產物回收試劑盒回收,利用T4連接酶過夜連接。產物轉化E.coliTransT1感受態中,轉化子提取質粒,經酶切驗證后獲得Cas9表達質粒pRS424-TRP-cas9。構建示意圖如圖1所示。質粒pRS424-TRP帶有色氨酸(Trp)表達基因,WT為Δura3Δtrp1Δleu2Δhis3基因缺陷型,將pRS424-TRP質粒轉化進WT細胞后,可利用Trp營養缺陷的培養基篩選。
被敲除基因的靶位點gRNA表達質粒構建及其donor DNA的設計:以敲除can1基因的打靶質粒為例,參照NCBI中S. cerevisiae模式菌株S. cerevisiae288c的基因組信息,選取編碼精氨酸透酶的基因can1為靶點基因,利用軟件找到cas9可以識別的間隔序列前體旁基序(protospacer adjacent motif,PAM)位點(5-NGG或NAG結構),通過引物can1-gRNA-1F、can1-gRNA-1R利用PCR擴增出SNR52啟動子、HINDLE,通過引物can1-gRNA-2F、can1-gRNA-2R利用PCR擴增出SUP43FLANK,利用Overlapping PCR技術融合啟動子SNR52、20 nt can1靶點序列、HINDLE及SUP43FLANK,將所得片段純化后與pESCG-LEU質粒骨架利用SacI和NotI進行雙酶切,目的片段與pESCGLEU空質粒的酶切產物通過超薄DNA產物回收試劑盒回收,利用T4連接酶過夜連接,產物轉化E. coli TransT1感受態中,轉化子提取質粒,經酶切驗證后獲得打靶質粒pESCG-LEU-gRNA-can1。構建示意圖如圖2所示。

圖2 pESCG-LEU-gRNA-can1構建示意圖Fig. 2 Schematic illustration of construction of pESCG-LEU-gRNA-can1
設計引物融合靶位點上下游的一段同源序列,以S.cerevisiae 288c基因組為模板,通過PCR技術擴增出donor DNA。構建示意圖如圖3所示。
本實驗還成功敲除了adh1、adh2、adh3、pdc、pdh等多個基因,其打靶質粒詳見表1。本實驗所用的菌株、質粒。打靶質粒的構建方法和donor DNA的構建方法與前文所述類似,此外,一次轉化兩個打靶質粒pRS423-HIS-gRNA-qadh5及pESCG-LEU-gRNA-lip的構建方法和donor DNA的構建方法也與之類似。
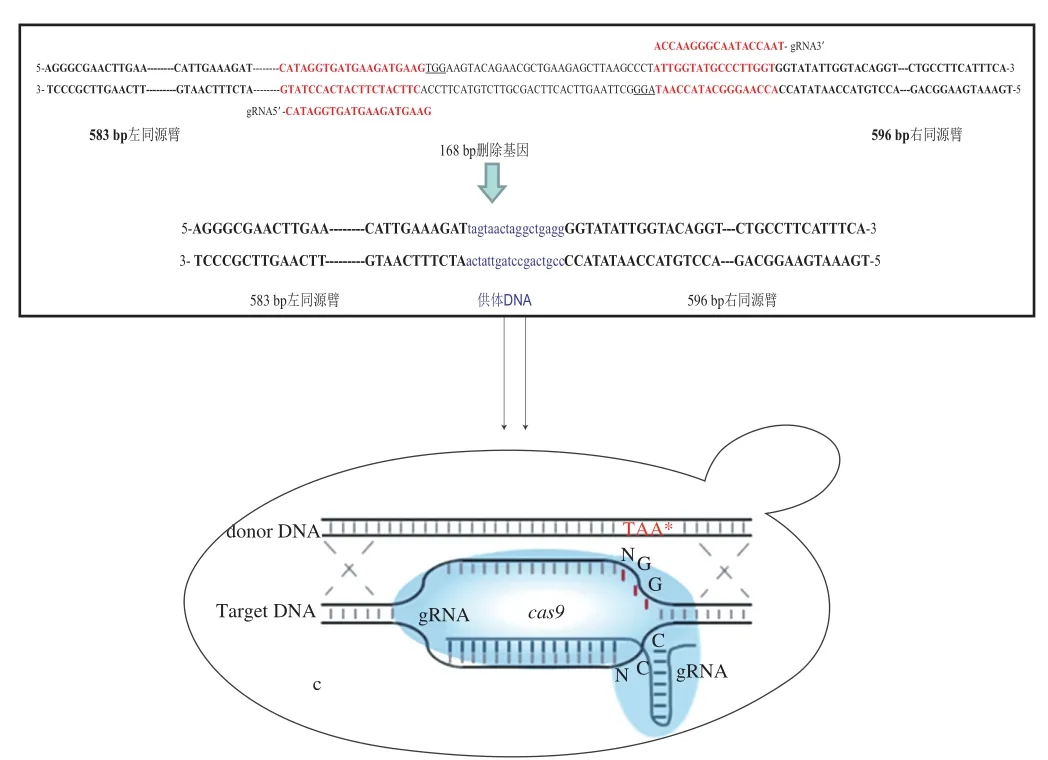
圖3 donor DNA構建示意圖Fig. 3 Schematic illustration of construction of donor DNA
1.3.3 S. cerevisiae感受態的制備及轉化
S. cerevisiae感受態的制備方法參考文獻[31]酵母高效轉化,稍作修改。將過夜培養的菌株WT以10%體積比接種于新鮮的YPD培養基,30 ℃、180 r/min培養3~4 h,當OD600nm達到0.5~1時,利用冷凍離心機在4 ℃條件下收集菌體,棄去上清液,加1 mL 1×TE重懸,棄去上清液,30 μL 1×LiAc/0.5×TE重懸,之后室溫靜置10 min,加入1 μg pRS424-TRP-cas9,1 μg打靶質粒,10 μL donor DNA和10 μL ss-DNA,加入250 μL 1×LiAc/40% PEG3350/1×TE,混合均勻,30 ℃孵育30 min,加入30 μL二甲基亞砜,混合均勻,42 ℃熱擊7 min,4 000 r/min離心2 min,棄去上清液,加入1 mL 1×TE重懸漂洗,最后離心收集菌體涂布篩選平板。30 ℃培養約72 h后,挑選固體培養基平板上長出的第3批較小的單菌落進行篩選驗證。
1.3.4 can1靶點的篩選驗證
由于can1基因編碼一種質膜精氨酸(Arg)通透酶,所以在敲除can1基因的實驗中,將轉化進打靶質粒的酵母感受態涂布在添加精氨酸的培養基上。待長出轉化子后挑取單菌落影印在含有刀豆素(一種有毒精氨酸類似物)和不含刀豆素的培養基上,若can1基因被敲除,則菌在刀豆板上生長,若沒有敲除can1基因,則菌在刀豆板上不長。由此可以挑選出正確的轉化子。
之后設計驗證引物進行分子驗證,敲除基因不能擴增出來,敲入基因能擴增出來的為陽性轉化子。
1.3.5 其他基因敲除的篩選驗證
挑選培養72 h后第3批長出的較小的菌落擴菌到相同篩選標記的固體培養基平板上,第2天振蕩裂解進行菌落PCR驗證,確認打靶細胞經過同源重組正確地整合到宿主細胞中。將驗證正確的菌株再次擴菌,提取基因組進一步驗證確認。
1.4 數據及圖像處理
基因敲除效率的計算方法:將PCR驗證正確的陽性轉化子的個數與所有被驗證轉化子的比例作為這一基因的敲除效率。凝膠成像圖利用visio軟件處理并標注。
2 結果與分析
2.1 Cas9系統的構建
2.1.1 pRS424-TRP-cas9質粒的構建
以pMD18-cas9的基因組為模板經PCR擴增出cas9基因片段;以S. cerevisiae 288c野生型菌株基因組為模板,利用PCR技術擴增出tef啟動子,cyc1終止子,NLS在tef啟動子基礎上通過延伸PCR擴增得到,利用Overlapping PCR技術融合得到目的片段,將目的片段與空質粒pRS424-TRP雙酶切后連接,轉化到E. coli,轉化子提取質粒進行酶切驗證,獲得5 616 bp載體片段和4 860 bp目的片段2 條條帶,結果如圖4所示,質粒pRS424-TRP-cas9構建成功。
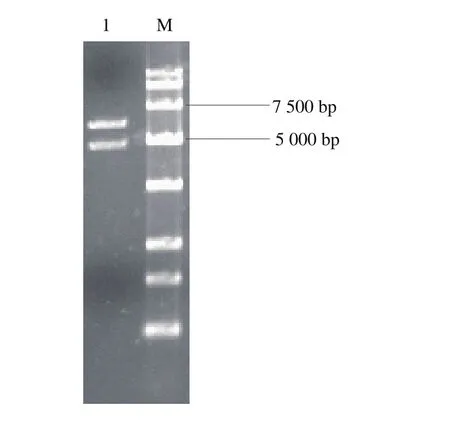
圖4 pRS424-TRP-cas9酶切驗證Fig. 4 Identification of pRS424-TRP-cas9 by double enzymatic digestion
2.1.2can1基因gRNA打靶質粒的構建
以S. cerevisiae288c基因組為模板,利用PCR擴增技術得到can1靶點基因、SNR52啟動子、HINDLE及SUP43FLANK,利用Overlapping PCR技術融合得到目的片段,將目的片段與空質粒pESCG-LEU雙酶切后連接,轉化到大腸桿菌,轉化子提取質粒進行酶切驗證,得到400 bp大小目的片段和7 200 bp大小載體片段,如圖5所示,質粒pESCG-LEU-gRNA-can1構建成功。
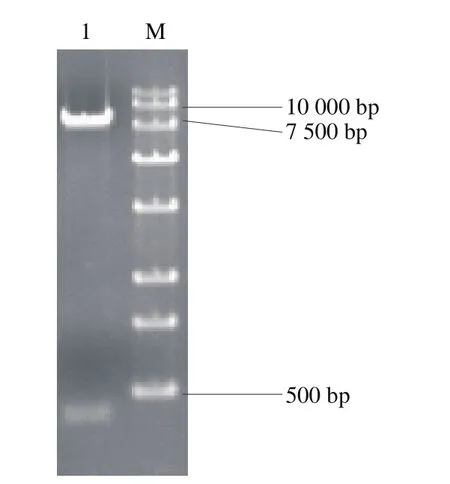
圖5 打靶質粒pESCG-LEU-gRNA-can1酶切驗證Fig. 5 Identification of target plasmid pESCG-LEU-gRNA-can1 by double enzymatic digestion
donor DNA的構建:以S. cerevisiae288c基因組為模板,通過引物融合靶位點上下游的一段同源序列,得到一段大小為1 450 bp的基因片段,凝膠回收備用。
2.2 can1靶點基因敲除的結果
將表達Cas9核酸酶的表達質粒pRS424-TRP-cas9、打靶質粒pESCG-LEU-gRNA-can1以及donor DNA共同轉化進S. cerevisiae感受態細胞內,Cas9核酸酶與gRNA共同作用實現細胞體內重組。將單菌落用0.9%生理鹽水稀釋,在含有刀豆素(CAN+)和不含刀豆素(CAN-)的培養基上影印,含刀豆的板上長的即為陽性轉化子。如圖6所示,統計得到陽性抗刀豆氨酸的突變子大約22%。將影印結果中陽性轉化子的對應菌液振蕩裂解做PCR菌落驗證,根據待敲除基因can1以及donor DNA設計敲除驗證引物,驗證被敲除基因的引物為YZQCF/YZQCR,其中,F為被敲除基因的23 bp大小的基因序列(AGGATGGCATAGGTGATGAAGAT,GenBank號為NC_001133.9),R為下同源臂下游染色體27 bp大小基因序列(GCAGTGATACCAACTAGTTCAGTACCT,G e n B a n k號為N C_0 0 1 1 3 3.9)。驗證敲入基因的引物為Y Z Q R F/Y Z Q R R,其中F為d o n o r D N A中含有終止密碼子的2 1 b p序列(AATAACGGCAAACAGCAAAGG,GenBank號為NC_001133.9),R為下同源臂下游染色體27 bp大小基因序列(GCAGTGATACCAACTAGTTCAGTACCT,GenBank號為NC_001133.9)分別以can1-/WT菌株與WT菌株的基因組為模板,進行PCR驗證,結果如圖7所示,敲除基因沒有擴增出來,敲入基因擴增出來,大小為750 bp為陽性轉化子,22 個中有4 個為基因編輯的轉化子。生理突變株和分子驗證的突變株數量不一致的原因,可能是S. cerevisiae為真核細胞,存在著非同源性末端接合,雖然沒有借助同源重組也得到了can1基因改變的突變株。

圖6 can1基因敲除影印結果Fig. 6 Photographic results of knocking-down can1 gene

圖7 can1缺失基因PCR驗證Fig. 7 Identification of can1 deletion by PCR
2.3 其他單基因敲除的結果
為驗證建立的CRISPR-Cas9系統在S. cerevisiae中進行基因編輯的有效性,本研究分別構建pdc、adh3、adh2、adh1、pdh基因gRNA的表達質粒及相應的打靶donor DNA(其他基因敲除表達質粒的構建結果略)分別轉化S. cerevisiae感受態細胞,Cas9核酸酶與gRNA共同作用實現細胞基因編輯。待培養72 h后,挑取最小一批的單菌落擴菌,PCR驗證,匯總實驗結果,總體基因編輯效率如表3所示。基因pdc敲除效率為4/48;adh3敲除效率為3/48;adh2敲除效率為1/48;adh1敲除效率為3/28;pdh敲除效率為1/16。

表3 各基因敲除效率Table 3 Gene knockdown efficiency
2.4 基因連續敲除的結果
為在敲除掉某一個基因后能夠快速順利地敲除下一個基因,采用篩選標記交替使用的方法:敲除第1個基因的gRNA構建在Leu作為篩選標記的質粒上,第2個敲除的基因構建在His作為篩選標記的質粒上,第1個基因敲除后,使用添加第1種篩選標記的Leu的培養基,繼續進行第2個基因的敲除操作,這樣可以在進行敲除基因的同時,丟掉第1個基因的pESCG-LEU-gRNA質粒,以便進行第3個基因敲除操作,這樣交替進行,提高了多個基因敲除的操作效率,簡化了操作步驟。連續基因敲除流程如圖8所示。采用上述方法,本研究在pdc基因敲除后,繼續敲除了adh3基因,又繼續敲除了adh2,敲除3 個基因整個操作過程為17 d。根據待敲除基因以及donor DNA設計敲除驗證引物,分別以待驗證的敲除基因菌株與WT菌株的基因組為模板,進行PCR驗證,第1個基因pdc敲除的驗證結果如圖9所示,敲入基因擴增出來,大小為560 bp為陽性轉化子;第2個基因adh3敲除的驗證結果如圖10所示,敲入基因擴增出來,大小為850 bp為陽性轉化子;第3個基因adh2敲除的驗證結果如圖11所示,敲除基因擴增不出來,敲入基因擴增出來,大小為570 bp為陽性轉化子。
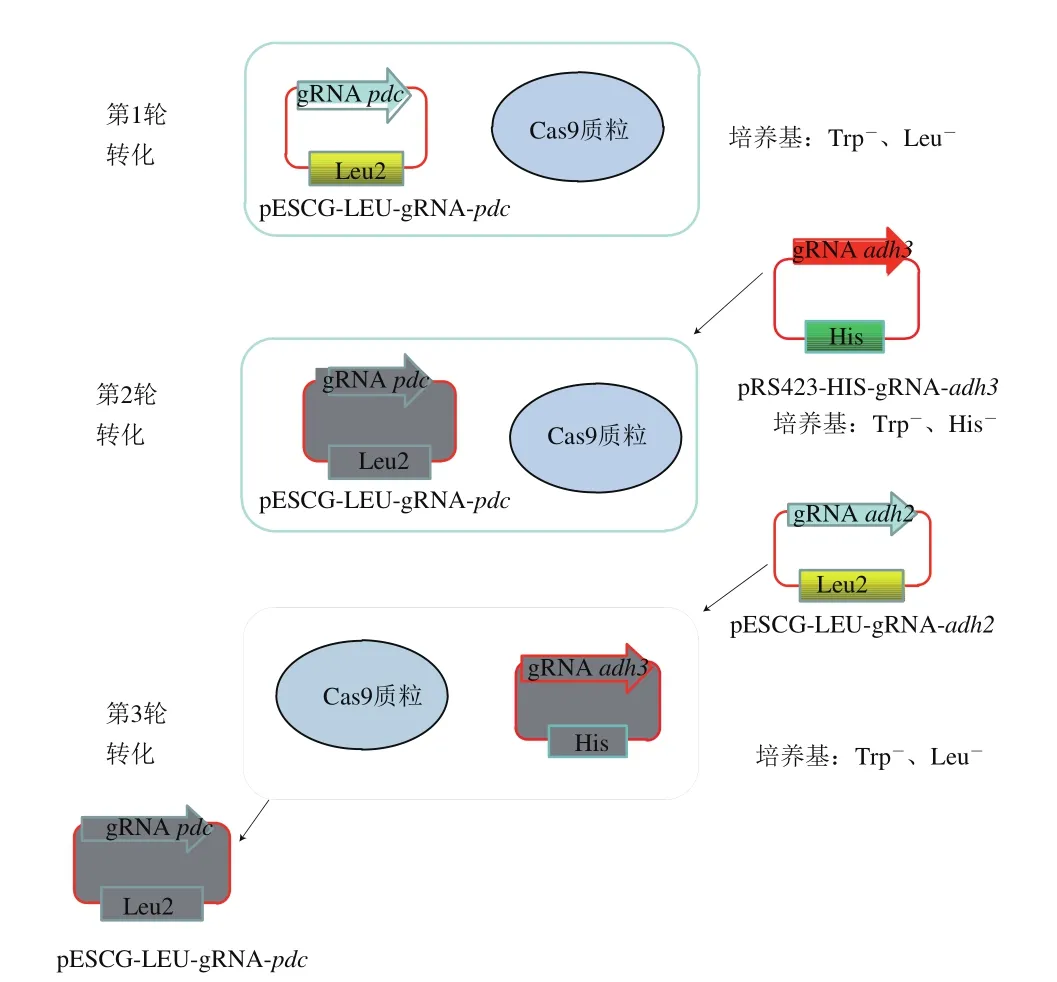
圖8 連續基因敲除流程圖Fig. 8 Flow chart of sequential knockout
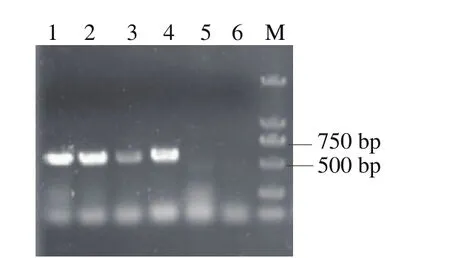
圖9 pdc缺失基因PCR驗證Fig. 9 Identification of pdc deletion by PCR

圖10 adh3缺失基因PCR驗證Fig. 10 Identi fi cation of adh3 deletion by PCR

圖11 adh2缺失基因PCR驗證Fig. 11 Identification of adh2 deletion by PCR
2.5 雙基因敲除的篩選驗證結果
為進一步提高CRISPR-Cas9系統的基因刪除效率,本研究將pRS424-TRP-cas9、gRNA表達質粒pRS423-HIS-gRNA-adh5、pESCG-LEU-gRNA-lip以及donor DNA共同轉化進S. cerevisiae感受態細胞內,Cas9核酸酶與gRNA共同作用實現細胞體內重組。待培養72 h后挑取最小的一批單菌落擴菌驗證,根據待敲除基因lip以及donor DNA設計敲除驗證引物,分別以lip-/WT菌株與WT菌株的基因組為模板,進行PCR驗證,結果如圖12所示,敲除基因沒有擴增出來,敲入基因擴增出來,大小為750 bp為陽性轉化子,統計得到轉化效率為9/32。根據待敲除基因adh5以及donor DNA設計敲除驗證引物,分別以adh5-/WT菌株與WT菌株的基因組為模板,進行PCR驗證,結果如圖13所示,敲除基因沒有擴增出來,敲入基因擴增出來,大小為1 400 bp為陽性轉化子,統計得到轉化效率為10/32。

圖12 lip缺失基因PCR驗證Fig. 12 Identification of lip deletion by PCR
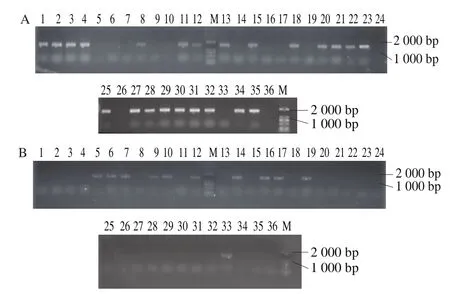
圖13 adh5缺失基因PCR驗證Fig. 13 Identification of adh5 deletion by PCR
3 討 論
酵母是一種單細胞真核生物,其清晰的遺傳背景、簡易的分子遺傳操作、可控的發酵過程及低廉的發酵生產成本,在外源蛋白表達生產及作為細胞工廠生產生物能源和化學品中發揮了極大的優勢。傳統的酵母基因編輯通常采用同源片段打靶抗性篩選的方法,往往因抗性標記在染色體上的整合而無法重復使用和無法去除,影響其在工業上的應用。借助酵母的營養缺陷底盤細胞和Cre重組酶的酵母基因修飾改造系統[6],雖然可以實現基因組的無痕改造,但需要兩次轉化,而且反向篩選假陽性多,造成操作繁瑣、周期長,也影響了酵母基因編輯的大規模實施。本研究采用CRISPR-Cas9基因編輯技術,只需構建gRNA和Cas9表達質粒,共轉化S. cerevisiae細胞,造成靶位點特異性切割,大大提高了供體DNA的重組效率,無需抗性篩選標記的篩選,就可高效率進行基因組基因的刪除、插入及修飾。與鋅指核酸酶ZFN[15-16]和轉錄激活因子效應物核酶TALEN[17]的基因編輯技術相比,CRISPR-Cas9系統可以有效地實現多個基因的編輯,在應用中更低成本、更簡便、更高效[18-19],是解決酵母基因編輯上述瓶頸問題的有效工具[32-34]。
酵母工業菌株都是雙倍體,對于雙倍體S. cerevisiae基因編輯的難度大于單倍體細胞。Church等[20]第一次利用CRISPR-Cas9基因編輯技術將單倍體S. cerevisiae中的can1基因敲除,效率為0.7%。Bao Zehua等[21]利用CRISPR-Cas9系統一次轉化單倍體S. cerevisiae后,通過液體長程培養6 d后再進行涂板篩選,可以同時敲除3 個基因。兩位研究者,基因編輯的均是單倍體S. cerevisiae細胞,采用上述方法必須將雙倍體酵母細胞分離為單倍體,基因修飾后再復性為雙倍體,操作繁瑣。本研究借鑒其研究,將CRISPR-Cas9基因編輯技術拓展應用到雙倍體S. cerevisiae細胞的基因定向改造中,成功地將can1、pdc、adh3、adh2、adh1、pdh基因敲除,這種針對工業雙倍體菌株的基因編輯方法還鮮見報道。本研究還探索高效率連續敲除基因的方法流程,完全敲除3 個基因整個操作過程為17 d。利用2 個篩選標記的gRNA一次轉化同時刪除兩個lip和adh5基因也成功實現。本研究也探索了3 個基因同時敲除的方法,但由于2 個gRNA表達質粒及Cas9表達質粒采用4 種篩選標記篩選,而且3 種供體DNA同時轉化,影響轉化DNA的通量,轉化子較少,造成陽性突變株很少或沒有(數據略)。在以后研究中可改進為:采用不同基因的靶點gRNA構建在同一個表達質粒上,就可解決轉化子少的問題,實現多個基因的同時刪除及修飾。

