利用SLAF-Seq結合BSA方法分子標記‘小白冬麥’抗白粉病基因mlxbd
金彥龍,李艷軍,張新宇,孫 杰,薛 飛
(石河子大學 農學院,石河子大學新疆生產建設兵團綠洲生態農業重點實驗室,新疆石河子 832003)
小麥(TriticumaestivumL.)是全世界最重要的糧食作物之一,在其生產過程中常面臨銹病、赤霉病和白粉病的危害。其中小麥白粉病是由子囊菌亞門的布氏白粉菌Blumeriagraminis(DC) Speer f.sp.tritici引發的世界性小麥病害。由于小麥白粉菌生理小種變異速度快,以及單一抗性品種的大面積推廣,導致品種抗性極易喪失。選擇和配置多基因聚合的抗病品種是最經濟、有效且環境友好的防治方法[1]。發掘有效且持久的抗性基因資源,并進行精細定位,將有助于現有抗源的合理利用,最終實現小麥的持久抗 病性。
對于大規模SNP和InDel標記開發,SLAF-Seq(Specific length amplified fragment sequencing)具有低成本和高通量的特性,彌補了SSR標記在基因組分布不均的不足。SLAF-Seq技術結合BSA(Bulked segregant analysis)方法已被廣泛應用于玉米花序[2]、擬南芥疫霉病[3]、黃瓜果肉[4]、大麥葉色[5]、油菜種子[6]、桃樹溫敏半矮化[7]、甜瓜風味[8]、番茄葉霉病[9]、水稻分蘗[10]、棉花雄性不育[11]等性狀的分子標記開發、QTLs鑒定和候選基因關聯分析等。
中國小麥農家品種非常豐富,其中‘小白冬麥’經歷漫長年代至今仍保持著一定抗白粉病能力。然而,‘小白冬麥’所持有的抗白粉病基因mlxbd并沒有得到充分的研究和利用。本研究將通過構建‘小白冬麥’與感病材料‘陜優225’的F2:3作圖群體,基于SLAF-Seq結合BSA方法進一步縮小mlxbd所在染色體候選區域,基于序列差異查找SNP和Indel。并通過生物信息學數據庫對定位染色體區間內的候選基因進行分析,為進一步圖位克隆抗白粉病基因mlxbd以及深入研究Pm5抗病基因位點提供理論依據。
1 材料與方法
1.1 材 料
農家品種‘小白冬麥’作為抗病親本與感病材料‘陜優225’雜交,獲得F1代,自交獲得F2:3分離群體。小麥白粉病生理小種E09由中國農業科學院植物保護研究所麥類病害組提供。
1.2 白粉病反應型鑒定
對‘小白冬麥’和‘陜優225’組合的F2:3分離群體接種BgtE09進行苗期(1到2葉期)抗病性鑒定,待對照組‘陜優225’充分發病時對F2:3分離群體進行鑒定,按照0~4級分級標準調查小麥白粉病反應型[12]。
1.3 基因組DNA提取
選取經鑒定的F2:3免疫及近免疫(等級為0;和0)和高感(等級為4)兩個極端個體各50株,抗病親本‘小白冬麥’與感病親本‘陜優225’各1株,分別取植株的第1片或第2片葉,液氮冷凍研磨,采用優化的CTAB法[13]提取基因組DNA。用Nanodrop 1000紫外分光光度計檢測樣品:質量濃度≥30 ng/μL,OD260/280為1.8~2.2, 10 g/L的瓊脂糖凝膠電泳檢測DNA質量。
1.4 SLAF文庫構建及測序
根據小麥的基因組大小以及GC含量等信息,以小麥CSS(Chromosome survey sequence)基因組(http://plants.ensembl.org/Triticum_aestivum/Info/Annotation/)作為參考基因組(IWGSC,2014),并組裝到染色體水平(ftp://ftp.ensemblgenomes.org/pub/plants/release30/ fasta/Triticumaestivum/dna/)。
為評估酶切試驗方案的準確性,選用水稻(Oryzasativa)作為對照進行測序。通過對照(水稻)測序數據的評估監控試驗過程是否正常,確定酶切方案實施的有效性(http://rapdb. dna.affrc.go.jp/)。文庫質檢合格后用Illumina HiSeqTM,PE125bp進行測序。對得到的序列進行GC含量、Q30以及原始reads進行分析,以保證測序質量。
1.5 SLAF-Seq數聚類和關聯分析
利用Dual-index[14]對測序原始數據進行識別,得到各個樣品的reads。過濾測序reads的接頭后,進行測序質量和數據量評估。通過將reads與參考基因組比對,在親本和混池中開發SLAF標簽,尋找在親本中存在多態性的SLAF標簽和在reads覆蓋區域的SNP。利用歐氏距離(Euclidean Distance,ED)關聯分析和SNP-index關聯分析兩種方法,獲得與性狀緊密關聯的位點,并根據關聯閾值確定候選區域。
1.6 候選區域基因分析
依據中國春IWGSC RefSeq v1.0基因組(IWGSC,2018)(https://wheat-urgi.versailles.inra.fr/ Seq-Repository/Assemblies)獲得候選區域位置。基因功能參照IWGSC RefSeq v1.0注釋(https://urgi.versailles.inra.fr/download/iwgsc/IWGSC_RefSeq_Annotations/v1.0/),在候選區域內篩選出含有核苷酸結合位點(Nucleotide binding site,NBS)或富亮氨酸(Leucine rich repeat,LRR)等抗病基因保守結構域的基因。利用小麥表達數據庫expVIP[15-16](http://www.wheat-expression.com/)進行候選基因表達分析并用HemI軟件制作熱圖。
2 結果與分析
2.1 表型和抗白粉病基因遺傳分析


圖1 親本‘小白冬麥’和‘陜優225’ 接種Bgt E09后第8天的表型Fig.1 Phenotype of resistant parent ‘Xiaobaidongmai’ and susceptible parent ‘Shaanyou 225’ on 8th day post-inoculation with Bgt isolate E09
2.2 SLAF標記開發和多態性分析
通過SLAF文庫構建和高通量測序,88.61 M的數據被開發,其中雙端作圖reads占 73.6%,單端作圖reads占9.07%,非作圖reads占17.33%。Q30的比例為87.87%~ 89.38%,平均比率為88.53%,GC含量為 45.72%。SLAF標記在每個染色體上分布均勻且完整性和準確性高。利用參考基因組開發31 090個SLAF標簽。根據基因組SLAF-Seq定位的數據,獲得每個染色體上SLAF標簽數量(表2)。依據等位基因分析和基因序列之間的差異,其中99 990個具有多態性,SLAF多態率為32.16%。SLAF標簽的雙親平均測序深度為30.07x,混合池為65.42x。

表1 抗白粉病基因mlxbd的遺傳分析Table 1 Genetic analysis of powdery mildew resistance gene mlxbd

表2 每個染色體上SLAF標簽和SNP標記的分布統計Table 2 Distribution statistics of SLAF labels and SNP markers on each chromosomes
2.3 SNP位點的開發
SNP標記在每個染色體上分布良好且完整性和準確性高。SNP的檢測主要使用GATK軟件工具包[17]實現,不同染色體上的SNP標記分布如表2。
在關聯分析之前,首先對2 069 543個低質量的SNP進行過濾,過濾多個基因型的SNP位點 3 938個,其次過濾reads支持度小于4的SNP位點1 496 393個,再次過濾混池之間基因型一致的SNP位點270 832個以及隱性混池基因不是來自于隱性親本的SNP位點198 390個,最終得到高質量可信SNP位點99 990個用于后續關聯分析。
2.4 關聯分析
通過歐氏距離算法[17],共有99 990個候選多態性SLAFs標簽用于關聯分析,關聯閾值為 0.08(圖2)。依據關聯結果,在染色體7B上共得到2個關聯區域,總長度為18.47 Mb。
通過SNP_index方法[18-19],對99 990個候選多態性SLAFs標簽進行關聯分析,關聯閾值為0.67(圖3)。依據關聯結果,在染色體2B、3B和7B上共得到6個區域,總長度為37.04 Mb。
取兩種關聯方法交集,將其縮小到染色體7B的3個候選區域內(表3)。對候選區域內開發的SLAF標簽進行分析,發現有788個SNP和47個InDel。
2.5 候選區域抗病基因分析
通過關聯分析,候選區域被定位在小麥染色體7BL上220 006 508~245 380 515[(參照小麥CSS(Chromosome survey sequence)基因組(IWGSC,2014)],將其對應到‘中國春’IWGSC RefSeq v1.0基因組(IWGSC,2018)上,位置是611 088 744~723 157 603,包含3 043個基因(高置信度基因1 134個(TraesCS7B01G353300.1~Traes CS7B01G466600.1)和低置信度基因 1 909個(TraesCS7B01G590100LC.1~TraesCS7B 01G780900LC.1),其中含有NBS或LRR抗病結構域的基因109個。利用轉錄組數據庫(expVIP)對候選基因的表達情況進行分析,發現56個基因受白粉病菌誘導,其中42個基因在接種后上調表達,14個基因在接種后下調表達(圖4)。
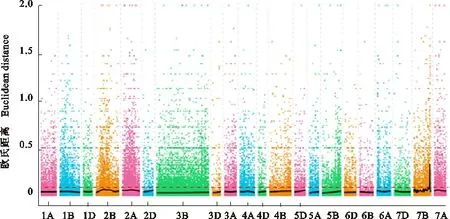
橫坐標代表染色體名稱,彩色的點代表每個SNP位點的ED值,黑色的線為擬合后的ED值,紅色的線代表顯著性關聯閾值。ED值越高,代表該點關聯效果越好 X-axis represents wheat chromosomes and colored dots represents the ED value of each SNP locu.Black lines show all fitting results of ED,red lines show the threshold of ED. The larger the ED value is,the stronger the association is.
圖2 ED關聯分析在染色體上的分布結果
Fig.2 Distribution results of ED association analysis on chromosomes
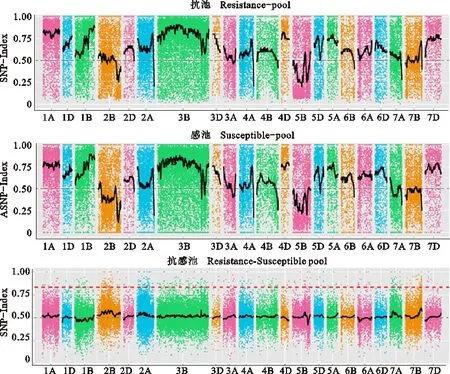
橫坐標代表染色體名稱,彩色的點代表計算出來的ΔSNP-index值。黑色線為擬合后的ΔSNP-index,紅色線為ΔSNP-index的關聯閾值,ΔSNP-index值越高,代表該點關聯效果越好 X-axis represents wheat chromosomes and the colored dots represent the SNP-index value. The black lines show all fitting results of ΔSNP-index,the red lines show the threshold of ΔSNP-index. The larger the ΔSNP-index value is,the stronger the association is.
圖3 SNP-index關聯分析在染色體上的分布結果
Fig.3 Distribution results of SNP-index association analysis on chromosomes
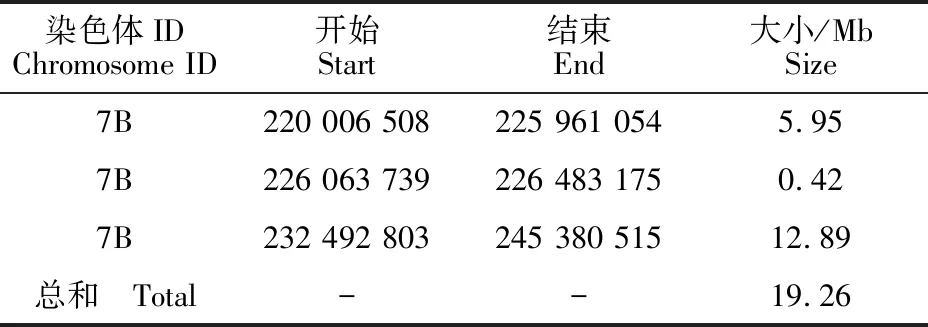
表3 兩種關聯分析結果交集的信息統計Table 3 Intersection Information statistics of two association analysis results
結合mlxbd和mlmz的遺傳連鎖圖譜[20-21],將mlxbd基因定位在Xgwm611和Xgwm577之間。根據側翼標記,進一步縮小候選區域,對應‘中國春’IWGSC RefSeq v1.0基因組(IWGSC,2018)位置是700 631 952~711 234 115,包含高置信度基因157個(TraesCS7B01G432900.1 ~TraesCS7B01G448500.1),低置信度基因191個 (TraesCS7B01G735300LC.1~TraesCS7B01 G754300LC.1),其中含有NBS或LRR抗病結構
域基因9個(表4)。在小麥表達數據庫中,基因TraesCS7B01G440800.1在白粉病菌誘導后下調表達,基因TraesCS7B01G437300.1、TraesCS7 B01G437400.1、TraesCS7B01G441600.1和TraesCS7B01G441700.1在白粉病菌誘導后上調表達(圖4)。
3 討 論
Pm5(Pm5a~Pm5e)是所有抗白粉病位點中唯一一個隱性復等位基因位點。其中Pm5a~Pm5c并沒有進行精細定位及遺傳圖譜構建[22];而Pm5d被定位在染色體7BL遠端區域,有6個微衛星標記與其緊密連鎖,其中側翼標記為Xgwm611和Xgwm577,遺傳距離分別為2.1 cM和2.0 cM[23];Pm5e被Huang等[24]發現由7個分子標記與其緊密連鎖,其中兩個共顯性標記Xgwm783和Xgwm1267相對較近,兩側遺傳距離分別為11.0 cM和6.6 cM。除Pm5復等位基因外,許多重要的抗白粉病基因也被定位在小麥染色體7BL遠端區域上,如mlxbd[20]、mlmz[21]、PmH[22]、PmTm4[25]、PmHYM[26]、PmBYYT[27]和PmSGD[28],其中PmTm4、PmHYM、PmBYYT和PmSGD已被精細定位。已有研究表明,在小麥7BL染色體上Pm5位點可能存在更多復等位基因或抗白粉病基因簇[29]。
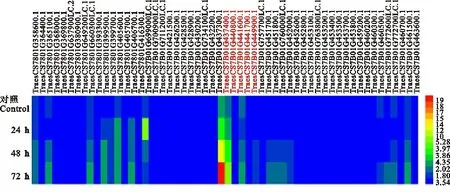
圖4 候選區域內抗病基因在接種Bgt E09后的表達分析Fig.4 Expression analysis of resistance genes (56 genes) in candidate regions after inoculation with Bgt isolate E09

基因名 Gene name染色體位置 Chromosome position功能注釋 Functional annotation TraesCS7B01G735900LC.1chr7B:700 820 349..700 821 167Leucine-rich repeat receptor-like protein TraesCS7B01G740600LC.1chr7B:703 703 155..703 706 321Disease resistance family protein/LRR family protein TraesCS7B01G437300.1chr7B:703 714 206..703 718 796 Disease resistance protein RPM1 TraesCS7B01G437400.1chr7B:703 719 566..703 723 769Disease resistance protein (NBS-LRR class) family TraesCS7B01G440800.1chr7B:706 083 250..706 090 834Disease resistance protein (TIR-NBS-LRR class) family TraesCS7B01G441600.1chr7B:706 808 371..706 811 012NBS-LRR resistance-like protein TraesCS7B01G441700.1chr7B:706 811 897..706 816 722Disease resistance protein (NBS-LRR class) family TraesCS7B01G748500LC.1chr7B:708 846 276..708 855 429Disease resistance protein RPM1 TraesCS7B01G748600LC.1chr7B:708 866 559..708 867 224Disease resistance protein RPM1
隨著SLAF-Seq、BSR-Seq(Bulked segregant RNA-Seq)、GBS (Genotyping-by-sequencing)以及外顯子捕獲等技術的出現,越來越多的SNP標記被開發,并被廣泛應用于小麥各項研究中[5,28,30-31]。本研究基于SLAF-Seq技術在候選區域內開發788個SNP和47個InDel。薛飛等[20]研究表明,‘小白冬麥’和‘唐麥4號’抗譜相同,且都位于Pm5位點附近區域,推測mlxbd和PmTm4可能含有相同的白粉病抗性基因。PmTm4基因[25]最近的側翼標記為WGGC6892和WGGC5746。本研究通過SLAF測序發現,在WGGC6892和WGGC5746之間的SNP和InDel分別為15個和2個,這些SNP和InDel將有助于進一步精細定位抗白粉病基因mlxbd。
隨著新技術和新方法的不斷出現和成熟,二代測序技術的成本急劇下降,一方面加快傳統圖位克隆的進程,另一方面也可以作為圖位克隆的補充技術,將大大促進小麥未知基因的克隆和功能研究。隨著‘中國春’小麥全基因組測序的完成[32],小麥基因組研究已進入一個全新的時代。Thind等[33]利用TACCA(Targeted chromosome based cloningvia long-range assembly)技術快速構建抗葉銹病基因Lr22a精細遺傳圖譜,并獲得4個NLR類型的候選基因,最終結合單倍型及EMS突變體確定目標基因NLR1。Xing等[34]采用抗病基因富集測序技術(RenSeq),合理運用靶序列富集,克隆到Pm21小麥抗白粉病基因。Arora等[35]結合關聯分析和抗病基因捕捉測序,開發AgRenSeq(Associationgenetics with R gene enrichment sequencing)技術,在短時間內同時克隆多個稈銹病(Stem rust,Sr)抗病基因。Steuernagel等[36]利用MutRenSeq方法(結合化學誘變、外顯子捕捉和高通量測序分析)從六倍體小麥中快速克隆兩個稈銹病抗病基因Sr22和Sr45。Snchez-Martín等研發出染色體分離測序技術(MutChromSeq),分別對大麥無蠟質基因(Eceriferum-q,位于2H染色體)和小麥Pm2(位于5D染色體)抗白粉病基因的突變體進行分離測序,驗證了此方法的有效性[37],為快速克隆目標基因奠定基礎。本研究中,通過SLAF-Seq、‘中國春’IWGSC RefSeq v1.0參考基因組(IWGSC,2018)以及小麥表達譜數據庫確定mlxbd基因的候選區間。其中9個基因含有NBS或LRR抗病結構域,4個基因在白粉病菌誘導后表達增強,可能是mlxbd的候選基因。然而,在不同品種之間,不僅所含抗病基因不同,且存在候選基因缺失的情況。因此,進一步精細定位抗病基因以及構建抗白粉病基因所在染色體區域的飽和分子遺傳連鎖圖,將為更好的利用現有抗源,以及深入了解mlxbd基因和Pm5位點的關聯性提供理論 依據。

