桂花OfAP1基因的克隆及表達分析
蔣琦妮,付建新,張 超,董 彬,趙宏波
(浙江農林大學 風景園林與建筑學院,浙江 杭州 311300)
高等植物成花受到自身遺傳因素和外界環境因素的共同影響[1-2]。當植物完成成花誘導,花器官原基開始分化,逐漸形成花萼、花瓣、雄蕊和心皮等組織。在這期間,植物的莖尖分生組織形態發生了很大的變化,同時成花素FT(FLOWERING LOCUS T)的表達量顯著增加并參與調控花芽分化[3]。在模式植物擬南芥Arabidopsis thaliana以及許多物種中已經證實,FT可以通過激活花芽分化組織特異性基因AP1(APETALA1)的表達促使植物完成開花進程[4]。過表達AP1基因,不論在長、短日照下擬南芥都會出現早花表型[5],其突變體ap1則出現明顯的晚花表型[6]。在莖尖分生組織,AP1基因不僅在植物成花誘導的調控網絡中起核心作用,而且也決定花器官的形成。AP1基因是植物ABCDE分化模型中的A類基因,控制第一輪花萼和第二輪花被片組織的形成[7],同時AP1也調控SEP3(SEPALLATA3)與LFY(LEAFY)基因的表達,促進花萼和花瓣的分化[8-9]。另外在擬南芥ap1突變體研究中發現,AP1突變會引起花器官的異常發育,如花萼發育成葉片、花瓣缺失等[10-11]。人們已從多種觀賞植物中分離得到了AP1基因,并且對多個物種的AP1基因進行了功能驗證和分析。超表達AP1及其同源基因都能促進開花,在擬南芥中異源表達百合Lilium longiflorum,蝴蝶蘭Phalaenopsis aphrodite以及山茶Camellia japonica的AP1同源基因都能引起早花現象[12-14]。同時,在擬南芥AP1突變體中異源表達百合,枇杷Eriobotrya japonica以及麻瘋樹Jatropha curcas中的AP1基因,都可以回補其突變體花器官缺陷的性狀[12,15-16]。桂花Osmanthus fragrans是中國十大傳統名花之一,也是常見的園林綠化樹種。按照開花習性不同,可分為秋桂和四季桂,秋桂僅在每年秋季開花,花序常為無總梗的聚傘花序[17];關于其成花過程以及花芽分化和發育報道較少。木本植物桂花成花的機制與草本植物擬南芥等有一定差異。為深入了解桂花的成花機制,有必要分離成花相關基因并展開相關研究。本研究以秋桂品種 ‘堰虹桂’Osmanthus fragrans‘Yanhonggui’為材料,克隆桂花AP1基因(OfAP1),并對其序列進行生物信息學分析,同時運用熒光定量對其組織和時空表達進行分析,以明確OfAP1基因的基本特征和表達模式,為今后桂花的開花分子機理研究提供科學依據。
1 材料與方法
1.1 材料
以盆栽 ‘堰虹桂’為材料,選取相同株齡且生長一致的植株。取桂花不同組織的樣品,包括根、莖、葉、葉芽、花芽、盛開期的花朵;同時根據桂花花芽分化進程取葉芽、花序分化期、花萼花瓣分化期、雌雄蕊分化期以及花開放不同階段圓珠期、鈴梗期、初花期、盛花期和盛花末期的樣品[18],用于基因克隆、組織特異性和時空表達分析。所有材料使用液氮冷凍并于-80℃保存備用。
RNA提取試劑盒(RNAprep pure Plant Kit)購自天根公司(北京)。反轉錄試劑盒、熒光定量試劑盒、膠回收試劑盒、PremixTaq酶、pMD-18T載體和大腸埃希菌Escherichia coliDH5α均購自Takara公司(大連),運用實時熒光定量PCR(Applied Biosystems,Foster City,美國)分析OfAP1基因的組織特異性及時空表達變化。
1.2 方法
1.2.1 RNA提取及反轉錄 按照RNAprep pure Plant Kit試劑盒說明書提取各樣品的總RNA,并參照Reverse Transcriptase M-MLV反轉錄酶說明書合成cDNA的第一鏈后,儲存于-20℃備用。
1.2.2 桂花OfAP1基因的克隆 利用前期轉錄組測序獲得的AP1 Unigene序列,設計特異性引物AP1-F: 5′-TAGAGTGAGAAAATGGGGAGA-3′;AP1-R: 5′-AATACAATCCCTGGCTACTT-3′。 以花芽分化期莖尖RNA反轉錄的cDNA為模板進行PCR擴增,PCR反應體系為:上下游引物各1 μL,cDNA 1 μL,PremixTaqDNA 10 μL和去離子水7 μL。PCR反應條件:95℃預變性5 min;然后進行35個循環,每個循環包括95℃變性30 s,60℃退火30 s,72℃延伸1 min;最后72℃保溫10 min,4℃保存。PCR反應產物經質量分數為1%的瓊脂糖凝膠電泳檢測后回收、純化,與pMD18-T載體連接,轉化大腸埃希菌DH5α感受態細胞,藍白斑篩選陽性克隆,經PCR鑒定后送生工生物工程(上海)股份有限公司測序。1.2.3 序列生物信息學分析 對獲得的基因序列進行生物信息學分析,其中編碼區分析采用美國國家生物技術信息中心(NCBI)在線網站 ORFFinder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html), 編碼蛋白質的保守區段分析采用 NCBI Conserved Domain Search 軟件(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),編碼區蛋白質特征分析采用在線軟件ProtParam(http://web.expasy.org/protparam/)和TMHMM Server v2.0(http://www.cbs.dtu.dk/services/TMHMM/)。多序列比對采用軟件DNAman,系統進化樹構建采用Clustal X+MEGA 7.0軟件并選擇鄰位連接法(neighbor joining method)用Bootstrap法檢驗1 000次。
1.2.4 實時熒光定量PCR 按照SYBRRPremix ExTaqTMⅡ(Tli RNaseH Plus)試劑盒的說明書,檢測桂花OfAP1基因不同組織以及不同時期的表達情況。OfAP1定量引物為qAP1-F:5′-GCAGAAGTGGCTTTGATTTG-3′;qAP1-R:5′-GTTTGCTGGTGACTGAGGTT-3′。同時以 OfACT 為內參qACT-F:5′-CCCAAGGCAAACAGAGAAAAAAT-3′;qACT-R: 5′-ACCCCATCACCAGAATCAAGAA-3′[19]。 qRT-PCR 反應體系: 2×SYBR Premix ExTaqⅡ(Tli RNaseH Plus) 10 μL, 上下游定量引物(10 μmol·L-1)各 0.8 μL, cDNA 2 μL,50×ROX Reference Dye 0.4 μL, 雙蒸水補齊至 20 μL。 qRT-PCR 程序如下: 95 ℃預變性 30 s;95℃變性5 s,60℃退火30 s,72℃延伸1 min,35個循環;72℃延伸10 min。3次生物學重復。反應后根據熔解曲線分析qRT-PCR產物的特異性,并采用2-△△CT法計算相對表達量。
2 結果與分析
2.1 桂花OfAP1基因的克隆
以 ‘堰虹桂’花芽分化期cDNA為模板,擴增得到約750 bp片段(圖1),其開放閱讀框長為720 bp,編碼239個氨基酸(圖2)。該基因片段具有MADS-box基因的保守區,在NCBI上進行BLASTX在線分析,發現該序列與芝麻Sesamum indicum(AIS82596.1),葡萄Vitis vinifera(ACZ26528.1)和油橄欖Olea europaea(XP_022878350.1)等中AP1 基因的同源性最高,命名為OfAP1,并在GenBank注冊,登錄號為MH593222。
2.2 桂花OfAP1基因的氨基酸序列分析
OfAP1蛋白氨基酸序列運用NCBI在線CDD(Conserved Domain Datebase)分析發現,OfAP1具有典型的MEF2_like MADS結構域和K-box結構域,MADS結構域位于N端第2~74位氨基酸,K-box結構域位于第90~168位氨基酸(圖2)。OfAP1蛋白的分子式為C1199H1941N341O368S16,其相對分子質量為27 534.62,理論等電點為8.4。負電荷殘基總數(天冬氨酸Asp+谷氨酸Glu)為32,正電荷殘基總數(精氨酸Arg+賴氨酸Lys)為35。在組成OfAP1蛋白的20種氨基酸中,亮氨酸(Leu)所占比例最高,為11.7%,其次為谷氨酸(Glu),占10.0%,色氨酸(Trp)所占比例最低,為0.8%。預測親水性指數(GRAVY)為-0.665,表明OfAP1具有較好的親水性。
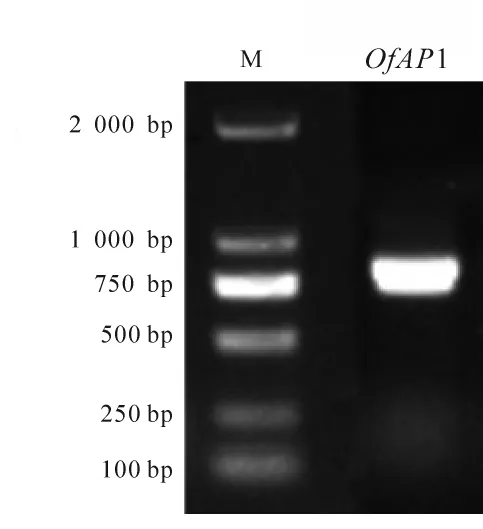
圖1 桂花OfAP1電泳圖Figure 1 PCR products of OfAP1

圖2 桂花OfAP1氨基酸序列比對Figure 2 Comparative analysis of OfAP1 protein sequence
采用GOR4軟件對OfAP1蛋白二級結構預測發現,OfAP1蛋白二級結構由α螺旋、伸展鏈和無規則卷曲組成,其中,α螺旋(Hh)占56.49%,β折疊(Ee)占10.46%,無規則卷曲(Cc)占33.05%。同時采用Phyre 2在線工具對OfAP1蛋白三級結構進行預測,OfAP1有3個典型的α螺旋和2個β折疊(圖3)。NetPhos 2.0 Server預測結果表明:OfAP1蛋白序列中存在11個潛在的磷酸化位點,包括6個Ser位點(22Ser, 61Ser, 74Ser, 87Ser, 110Ser, 121Ser, 135Ser, 150Ser, 216Ser)和 1 個 Thr位點(220Thr)。NetNGlyc 1.0 Server預測結果表明:OfAP1蛋白存在1個潛在的N-糖基化位點,90Asn。
2.3 桂花OfAP1系統進化關系分析
為了分析OfAP1基因與其他物種中AP1基因的系統進化關系,采用MEGA 7.0軟件構建系統進化樹(圖4)。單子葉植物(如短柄草Brachypodium distachyon,水稻Oryza sativa,玉米Zea mays)和雙子葉植物可以明顯地分為2個大類。桂花OfAP1與芝麻,豇豆Vigna unguiculata,枇杷,印加果Plukenetia volubilis,麻瘋樹,葡萄,芍藥Paeonia lactiflora,柑橘Citrus sinensis以及擬南芥中的AP1基因聚在一起,說明這些基因親緣關系較近且功能具有一定的相似性。
2.4 桂花OfAP1基因的組織特異性與時空表達分析
為明確OfAP1基因在桂花不同組織以及花芽分化不同時期的表達情況,對不同組織和器官(根、莖、葉、葉芽、花芽以及花)及不同發育時期的樣品進行熒光定量RT-PCR分析。結果表明:OfAP1基因在花芽中的表達量最高,其次為葉,在莖中的表達比較弱,在根中幾乎不表達(圖5)。同時,與葉芽時期相比,OfAP1基因在分化初期(即花原基的形成以及花萼花瓣分化期)表達量最高,隨后呈下降趨勢(圖6),到花開放階段表達量均較低。
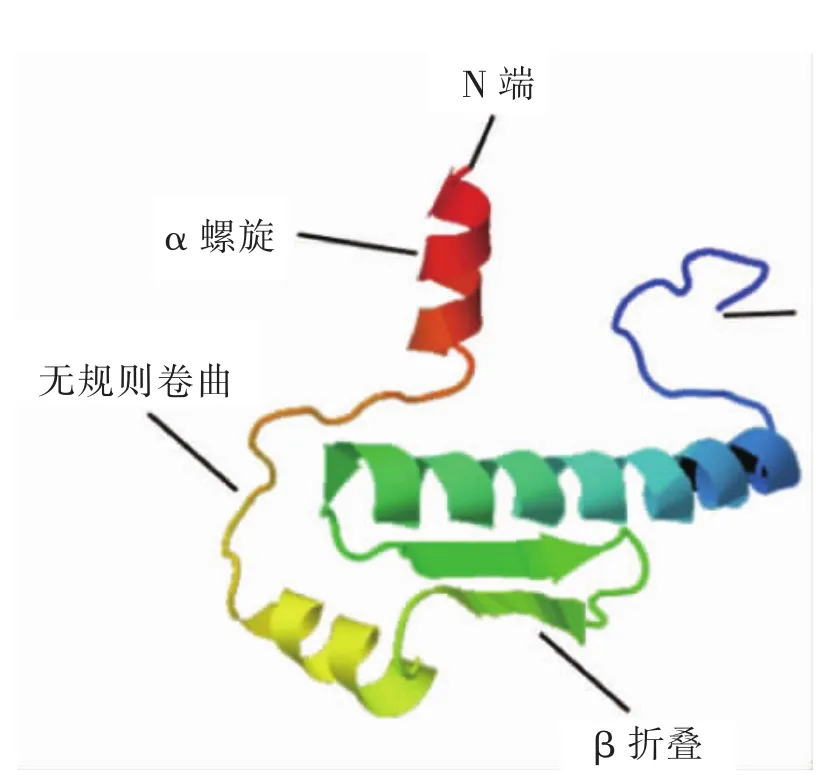
圖3 桂花OfAP1蛋白的三級結構預測Figure 3 Tertiary structure prediction for OfAP1 protein

圖4 桂花OfAP1的系統進化樹Figure 4 Phylogenetic tree based on OfAP1 gene
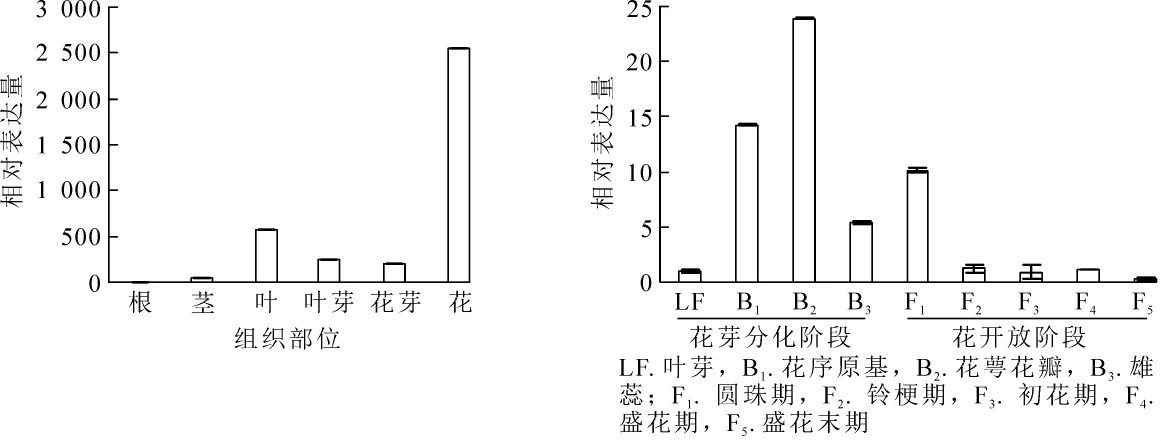
圖5 OfAP1基因在不同組織中的表達量Figure 5 Expression of OfAP1 gene in different tissues
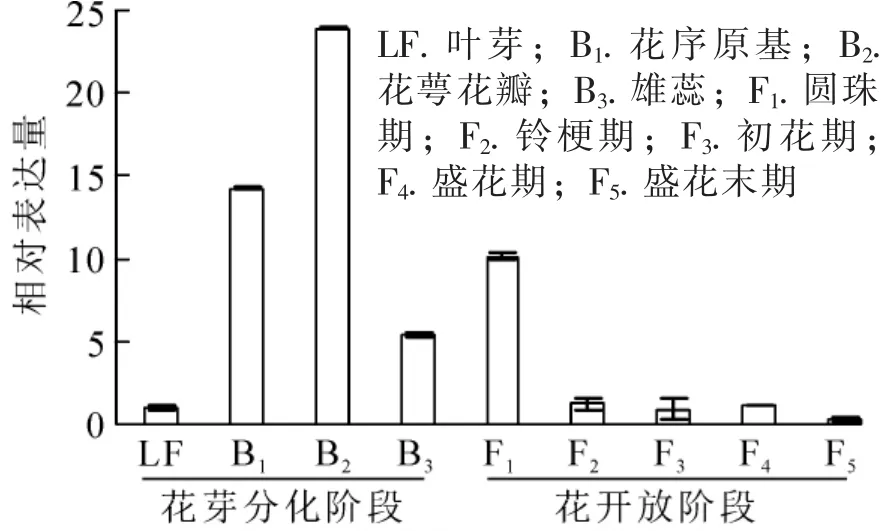
圖6 OfAP1基因在 ‘堰虹桂’成花及花開放過程的表達量Figure 6 Expression pattern of OfAP1 in flowers of different stages
3 討論
AP1及其同源基因已在多個物種中克隆得到,但桂花中的AP1至今沒有相關報道。本研究克隆得到OfAP1基因,發現其與芝麻、葡萄以及油橄欖等AP1基因高度相似,OfAP1氨基酸序列含有高度保守的MEF2_like MADS結構域和次級保守的K-box結構域以及可變C,MADS結構域具有結合DNA、蛋白質二聚化以及與其他因子結合的功能;K-box結構域的蛋白二級結構為3個α螺旋,具有介導蛋白-蛋白之間的相互作用[20]。AP1基因C末端的變化較大,但是不同類的MADS-box基因常含有一些保守的基序(motif),這些基序在蛋白復合體的形成和轉錄激活中起重要作用[21]。
開花是植物重要的發育階段,是多條基因網絡共同調控的復雜過程[2]。AP1基因是植物特有的MIKCC-Type MADS-box基因,既參與花分生組織的形成,又是花器官形成的重要基因,在植物成花中起關鍵調控作用[3]。根據在模式植物擬南芥中的研究,AP1是花分生組織形成的標志物,成花整合基因FT和SOC1能夠激活其表達并使其參與對成花抑制基因TFL1基因的拮抗,促進花分生組織的形成。在花器官發育中,AP1一方面通過促進AP3和PI的表達誘導花瓣和雄蕊的發育,另一方面受到AG基因的抑制作用控制萼片和花瓣的發育[22]。在百合中AP1的同源基因LMADS5/6轉基因擬南芥后發現其能夠造成早花,并且花器官中萼片變成心皮狀,花瓣變成雄蕊狀結構[12];同樣地,在洋桔梗Eustoma grandiflorum中超表達AP1發現:轉基因植株花期提前,部分花瓣出現雄蕊類似的結構,進一步突變體互補實驗表明:AP1基因對花瓣形成,尤其是第2輪花被片的發育有重要作用[23]。在建蘭Cymbidium ensifolium,芍藥以及菊花Chrysanthemum morifolium等中,AP1基因在花芽中的表達量顯著高于其他器官組織,尤其在花芽分化的初期階段[24-26]。在桂花中,OfAP1在花芽中有較高的表達,其在花芽分化初期表達量顯著上調,尤其是成花轉變以及花瓣、花萼分化時期,而當完成花萼花瓣分化后,表達量則顯著下調。這充分說明桂花OfAP1基因參與了桂花成花轉變和花瓣、花萼等器官分化,但其具體作用機制還有待進一步通過轉化實驗加以驗證。

