以細胞系為中心的基因-突變-疾病語義網絡構建研究*
吳 萌 李 姣 侯 麗
(中國醫學科學院醫學信息研究所 北京 100020)
1 引言
語義網廣義上來說是對未來網絡的一個設想,狹義上來說是一種智能網絡,不但能夠理解詞語和概念,而且還能夠理解它們之間的邏輯關系[1]。隨著語義網概念的提出,互聯網逐步從僅包含網頁之間超鏈接的文檔萬維網轉變為描述各種實體之間關系的數據萬維網。基于此,知識圖譜這一概念于2012年5月由谷歌率先提出,其目標在于描述真實世界中存在的各種實體和概念,及實體與概念之間的關聯關系,從而改善搜索結果。同時, 資源描述框架(Resource Description Framework, RDF)及其模式(Resource Description Framework Schema, RDFS)在語義Web中處于核心地位, 是實現Web信息共享和數據交換的基礎。
伴隨生物醫學領域測序技術的飛速發展和精準醫學概念的提出,越來越多的科學研究開始關注于疾病發生的內在復雜機制,以及各個生物醫學實體之間的網絡調控通路和關聯關系,以提供個性化的治療方案。知識圖譜等語義網絡技術為多源異構的生物醫學數據的整合和復雜關系網絡的建模提供新的解決方案,通過利用統一的數據表示標準,為生物醫學數據的檢索、分析、挖掘提供基礎。在癌癥生物學的研究過程中,人類癌癥細胞系作為一種易于獲取、方便使用的生物模型,廣泛應用于探索癌癥的分子特征以及相應的治療反應。由于臨床試驗復雜且昂貴,而借助細胞系進行臨床前實驗有助于極大地提高臨床實驗的成功率。目前,許多項目都致力于為細胞系及其遺傳學和基因組學數據提供系統的整合方案,例如癌癥體細胞突變目錄(Catalogue of Somatic Mutation In Cancer,COSMIC),使用戶在進行生物實驗和藥物測試時可以選擇更合適的癌癥細胞系,也為生物研究提供臨床依據[2]。本研究從NCBI gene、ClinVar、COSMIC、Cellosaurus、OMIM與NCIt 6個數據庫中分別獲取基因、突變、細胞系與疾病及其間的語義關系數據,擬以細胞系數據為核心,構建一個包含基因、突變與疾病數據及其語義關系的RDF語義網絡。旨在對疾病基因組學等相關領域中的生物醫學數據進行建模與整合,以期為進一步發現新的醫學實體語義關系,理解與分析疾病的致病機制提供數據支撐。
2 研究現狀
2.1 語義模型在醫學數據領域的應用
2.1.1 生物醫學語義模型 隨著2015年美國總統奧巴馬提出精準醫學計劃,世界多個國家陸續開始部署精準醫學項目,更多的科學研究開始關注于疾病發生過程中內在的分子機制,而所催生的大量多來源異構的生物醫學數據,迫切需要統一的數據整合方案。語義模型技術為多來源異構的生物醫學數據整合提供方案,并致力于提供一套統一的生物醫學實體表示標準,使機器和人都可以理解,其靈活性、可擴展性以及可對語義關系進行模型等特點非常適用于表示復雜的生物醫學網路數據。如上海曙光醫院構建的中醫藥知識圖譜、醫學系統命名法-臨床術語(Systematized Nomenclature of Medicine-Clinical Terms,SNOMED-CT)和IBM Watson Health等系統[3]。基于鏈接數據,也可以識別出新的語義關系。如Dalleau等對藥物、疾病和基因相關的6個數據庫進行整合與鏈接,構建藥物基因組學相關的RDF格式語義網絡,共包含2 640 793個3元組。基于構建的鏈接數據,分別用兩種基于圖的機器學習的方法——隨機森林和圖核,對藥物與基因是否相關進行預測,從而發現新的藥物-疾病關系[4]。
2.1.2 細胞系相關語義模型 細胞系目前已在許多生物醫學實驗和研究中被廣泛使用。復雜疾病,如癌癥的發生通常開始于一系列體細胞DNA變化所導致的失控的細胞增殖,這些大部分變化指的是突變等特定的DNA序列變化。研究認為細胞從正常狀態轉變到完全的惡性形態的過程,必須積累5~10個體細胞突變,每一種突變都會引發不一樣的細胞功能改變[5]。對癌癥細胞系進行基因組測序,可以發現引發細胞機制發生變化的重要突變,整合并分析腫瘤發生過程中細胞系發生的突變信息,有利于更好地理解腫瘤發生的內在分子機制,從而發現新的治療方案。而現有醫學數據的語義模型中,整合細胞系、突變和疾病等相關實體及其語義關系的語義模型還非常少見。大多數模型只聯系突變和疾病,或細胞系和疾病等實體。如細胞系本體(The Cell Line Ontology,CLO),主要描述細胞系、癌癥、細胞和有機體之間的關聯[6]。COSMIC數據庫整合細胞系、突變和疾病等實體之間的關聯,但是對于疾病的描述沒有采用通用的表示方法,不利于與其他疾病數據庫進行映射,其表示方式沒有采用語義模型。
2.2 關系數據向語義數據的轉化
隨著下一代測序技術的發展,越來越多豐富的組學數據被生產、注釋出來。其大多數存儲形式為關系型數據庫或類似的表格文件形式。而如何將關系型數據的數據資源和語義關系信息轉化為RDF語義網絡格式,成為一種普遍需求。許多項目如Bio2RDF[7],the EBI platform[8],PDBj[9]以及Linked Open Drug Data (LODD)[10]等都致力于推動健康科學數據轉化為統一的鏈接數據形式。其中,Bio2RDF是一個開源的項目,采用語義網技術構建并提供生命科學數據的鏈接數據網絡。Bio2RDF定義一套簡單的規則,為多來源異構的數據集合創建RDF(S)兼容的鏈接數據形式。目前已為clinicaltrials.gov,dbSNP,GenAge等35個數據庫提供RDF鏈接數據結構[7]。R2RML[11]是W3C RDB2RDF工作組于2012年9月發布的一種映射語言,可以定義關系型數據庫與RDF格式數據之間的映射規則,從而將關系型數據轉化為RDF 3元組形式。基于R2RML的常用工具如D2RQ、db2triples、OpenLink Virtuoso等都可以實現關系型數據庫向RDF的轉化。
由此可見,在生物醫學領域,構建不同實體間的語義模型對于數據的整合、復雜網絡的表示及發現新的語義關系等都具有突出作用。而目前的研究多集中在藥物、疾病、癥狀等傳統醫學的關系層面,圍繞基因、突變、細胞系等精準醫學領域實體的語義網絡尚不多見。上述關系數據轉化技術中,對比發現D2RQ有明顯的優勢, 它支持任何關系數據庫的數據轉換、通用性強, 支持靈活的映射配置文件, 提供一種標準的轉換方式生成一個虛擬的RDF (S), 確保數據庫的內容更新便捷[12]。鑒于此,本文采用D2RQ工具將數據資源轉化為RDF格式。
3 研究思路與框架
3.1 語義模型
基因、突變、細胞系與疾病之間存在多種語義關系。對語義關系進行有效以及規范的定義,是語義模型構建的基礎,也為后續語義網絡在文本挖掘等領域中的應用提供潛能。Verspoor等人提出一種人類變異組信息注釋模式,對11種實體類型和關系進行規范。將這種模式應用于一個小型的以腸癌主題的全文預料庫中,通過使用這個模式進行全文注釋,注釋結果的一致性得到顯著提高[13]。本研究參考這種模式以及多種數據庫對語義關系的命名方式,最終確定4種語義類型Gene、Mutation、Cell-line、Disease之間的6種語義關系的定義模式。根據收集的數據庫中的數據形式與數據內容,選擇部分基本信息進行提取,例如基因的類型、突變的位點和疾病的別名等。這樣,每種語義類型都有多種相關屬性進行更為全面的描述。具體語義關系模型設計,見圖1。
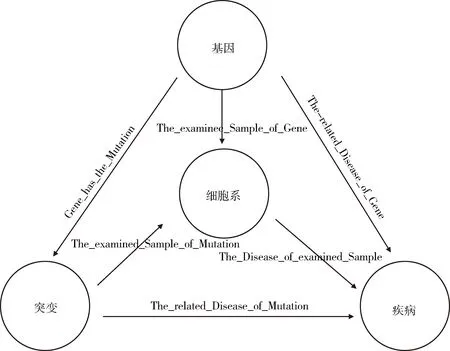
圖1 語義關系模型及數據來源
3.2 實驗流程
根據所設計的語義模型,選取NCBI gene、ClinVar、COSMIC、Cellosaurus、OMIM與NCIt等6個數據庫作為基因、突變、細胞系與疾病等實體數據的來源。通過數據篩選、格式轉化及融合等數據預處理流程,形成待處理數據集,將數據集存入MySQL數據庫中,依據數據特性共存儲為4個實體表,以及6個關系表。其后,利用D2RQ映射工具,根據本研究設計的語義模型,定義關系型數據與RDF數據的映射規則。最后,將關系型數據庫轉化為RDF語義格式并利用D2RQ工具部署本地Web應用,實現對語義網絡的生成、檢索與分析。實驗流程,見圖2。
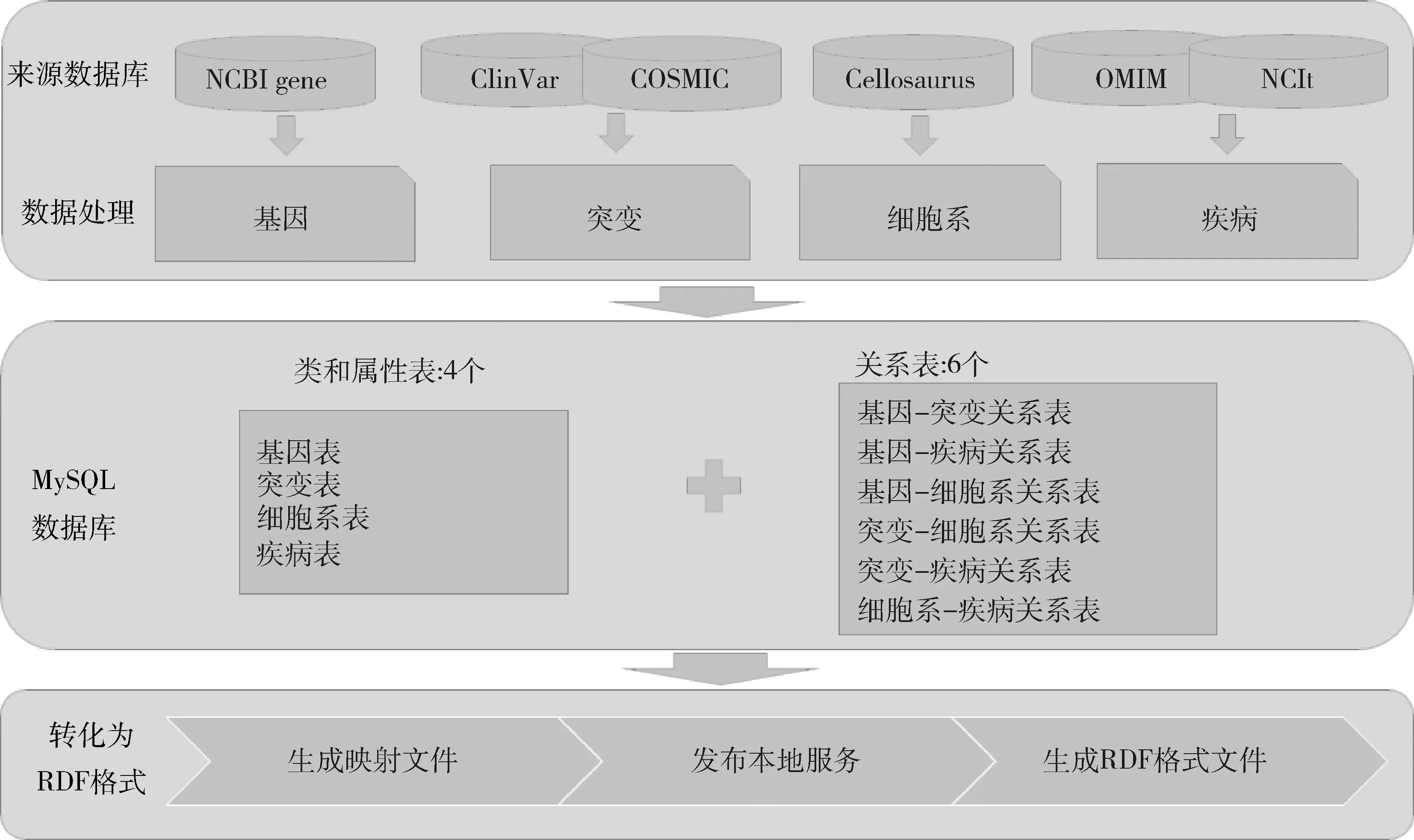
圖2 語義關系模型的構建
4 實驗過程與結果
4.1 數據處理
4.1.1 數據提取 (1)基因數據選自NCBI gene數據庫。NCBI gene數據庫提供了多物種的基因序列信息,包括序列、表達、結構、功能以及引用等信息,基因的唯一標識——Entrez_ID,在美國國家生物技術信息中心(National Center for Biotechnology Information,NCBI)研發的所有數據庫中都是通用的[14]。本研究選擇NCBI gene人類物種的基因數據作為實驗數據的基因部分,共60 195條,包括基因ID、基因名稱、在染色體上的位置和基因類型等信息。以NCBI gene數據庫的Entrez_ID為基因數據的標準標識。(2)突變數據與突變-基因、突變-疾病之間的關系數據選自ClinVar數據庫。ClinVar 是一個公開的數據庫,其中收集與疾病相關的人類遺傳變異[15]。本研究選擇突變概要文件variant_summary.txt中基因組參考序列版本為GRCh38的突變數據,共254 030條,篩選所在基因、突變名稱、突變類型、所在染色體、相關疾病等信息。ClinVar中突變所在的基因使用NCBI gene的Entrez_ID進行標識,可與NCBI gene進行鏈接。突變相關的疾病整合了OMIM 的疾病ID標識,所以也可與OMIM中的疾病鏈接。ClinVar的突變標識以RCV000000000.0.形式表示,在突變對應多個疾病的情況下,一個突變會對應多個突變標識所以本研究自定義突變的唯一標識,并保留RCVaccession的信息。(3)疾病數據與疾病-基因之間的關系數據選自OMIM數據庫。在線人類孟德爾遺傳數據庫(0nline Mendelian Inheritance in Man,OMIM)是一個關于人類基因和表型的權威數據庫,目前每日更新并支持免費獲取[16]。本研究選擇morbidmap.txt文件中7 326條疾病數據,保留疾病名稱、基因名稱、基因的OMIM ID以及基因的位置信息。利用mim2gene.txt中OMIM基因與NCBI基因的映射,獲取疾病-基因之間的關系。由于有些疾病的OMIM ID缺失,所以自定義疾病的唯一標識。(4)細胞系的數據選自Cellosaurus數據庫。Cellosaurus數據庫由瑞士生物信息研究所的團隊在Biocuration 2016大會上提出[17]。是目前整合細胞系信息較為全面的數據庫。下載并處理68 406條細胞系數據為標準格式,篩選細胞系名稱、編碼、別名、相關疾病等信息,自定義細胞系的唯一標識。
4.1.2 數據關聯 基于以上數據處理的步驟,可以得到大部分實驗數據。但疾病-細胞系、突變-細胞系、基因-細胞系的關系是無法直接從已整合的數據庫獲取的,需要引入中間數據庫,才能將這些實體進行關聯。Cellosaurus中存在疾病-細胞系的關系,但是Cellosaurus中的疾病數據使用NCIt[18]的疾病術語進行表示,提供其在NCIt中ID編碼。所以利用NCIt將Cellosaurus細胞系與OMIM疾病進行關聯。參考一體化醫學語言系統(Unified Medical Language System,UMLS)中已對NCIt的疾病術語和OMIM的疾病術語進行整合的信息。利用兩個來源的術語在UMLS數據中是否在同一個概念下進行同義判斷,將NCIt與OMIM的疾病術語進行映射。對于突變-細胞系與基因-細胞系的關系,利用COSMIC數據庫作為中間數據庫來獲取這些信息。COSMIC是世界上最大最全的研究人類體細胞突變對癌癥影響的數據資源[2],其中包含細胞系和原代細胞的基因測序信息和識別的突變信息。本研究重點關注對細胞系與突變和基因的關聯信息的獲取。獲取突變數據與細胞系的關系,先將COSMIC的細胞系與Cellosaurus的細胞系的名稱以及別名進行匹配,再根據突變在染色體中的位置,以及突變的類型,將COSMIC中的突變信息與ClinVar中的突變信息進行映射,以獲得ClinVar突變與Cellosaurus細胞系之間的關聯。而COSMIC中存在NCBI基因與細胞系關系,利用之前COSMIC的細胞系與Cellosaurus的細胞系的映射,可獲得NCBI基因與Cellosaurus細胞系之間的關聯。
4.2 數據映射
4.2.1 數據存儲 本研究采用D2RQ工具將融合的數據資源轉化為RDF格式。D2RQ是一個開源的平臺,提供以虛擬只讀的RDF數據形式訪問關系數據庫的功能。通過D2RQ,可以使用SPARQL語言對關系型數據進行檢索,通過其自帶Web應用瀏覽數據,也可以使用工具獲取生成RDF格式的文件。根據D2RQ工具所需要的輸入文件格式,將基因、突變、細胞系、疾病4種實體類型以及相關屬性存為MySQL數據庫中相應的4張表,6種實體間的關系存為數據庫中相應的6張表。
4.2.2 映射規則 D2RQ提供映射語言來描述關系型數據庫模式向RDFS轉換的映射規則。一個D2RQ映射文件本身就是用Turtle語法編寫的RDF文檔。D2RQ提供一些便利的工具,例如generate-mapping工具可以自動生成一個映射文件mapping.ttl。但是自動生成的映射文件只包含一些基礎的規則設置,更多復雜的映射規則可以參考D2RQ的映射語言[19]。
本研究首先通過聲明一個數據庫為d2rq:Database類來設置數據庫的相關屬性,包括JDBC數據庫的URL,JDBC驅動程序類名,數據庫用戶名密碼等。聲明一個相應的實體為d2rq:ClassMap類來設置類的屬性,包括URI的模式,例如設置gene的URI模式為“gene/@@gene.Gene_ID|urlify@@”,其中gene.Gene_ID為數據庫中gene表的Gene_ID列。利用d2rq:PropertyBridge來設置屬性,例如屬性名稱、屬性值。實體之間的語義關系,利用其中的d2rq:refersToClassMap來定義,例如基因-突變之間的語義關系“Gene_has_the_Mutation”,將兩個實體進行鏈接。
4.3 實驗結果
4.3.1 實驗結果瀏覽與獲取 本研究根據基因、突變、細胞系、疾病4種實體類型以及之間的6種語義關系對應的表格,編寫D2RQ映射語言文件mapping.ttl。利用生成的文件,運行d2r-server工具,啟動D2RQ部署的本地服務http://localhost:2020/。通過這個Web應用,可以對數據進行瀏覽,也可以利用SPARQL執行搜索并設定展示的條目數量。以AKT3基因為例,數據瀏覽結果,見圖3。基因AKT3的信息頁面包含多種信息。其中,該基因相關的突變,用字段“Gene_has_the_mutation”表示;檢測該基因的細胞系樣本,用字段“The_examined_Sample_of_Gene”表示,其值對應的鏈接,可以鏈接到相應的突變和細胞系的信息頁面。其他屬性,例如,基因所在的染色體、基因全名和基因ID等信息也在該頁面中詳細列出。
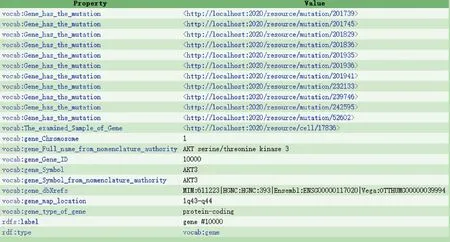
圖3 AKT3基因信息瀏覽頁面
此外,D2RQ也提供通過命令行進行SPARQL語言搜索的功能。通過dump-rdf命令可以將生成的RDF文件導出生成“TURTLE”、“RDF/XML”、“RDF/XML-ABBREV”、“N3”以及 “N-TRIPLE”等多種RDF語法格式。實驗最終構建的語義網絡,共包含基因60 195個,突變254 030個,細胞系68 406個,疾病7 326個,共構建3元組726 236個。其中,基因與突變之間的3元組254 030個,基因與疾病之間的3元組15 477個,基因與細胞系之間的3元組287 342個,細胞系與突變之間的3元組195個,細胞系與疾病之間的3元組36 377個,突變與疾病之間的3元組132 815個。
4.3.2 實驗結果對比分析 對NCBI Gene,ClinVAR,Cellosaurus,COSMIC等幾個數據庫進行分析,對數據類型和數據格式進行調研,與本研究模型進行對比,發現本研究構建的語義關系模型,其細胞系相關的數據類型覆蓋程度更為全面,方便用戶對細胞系及相關信息進行瀏覽和查詢,而不用跨越多個數據庫,緣于已有效地將細胞系相關的數據進行整合。每種數據類型都采用業內通用的數據庫的名稱和標識,提供良好的互操作性,為細胞系的研究提供幫助。相比于NCBI Gene,本研究構建的語義網絡還整合了突變和細胞系的信息,較ClinVar多細胞系信息,比 Cellosaurus數據庫的信息主要多基因和突變的數據,而在數據類型覆蓋層面,COSMIC數據庫同樣覆蓋了基因、突變、細胞系和疾病的信息,但同時本語義網絡在疾病數據的整合方面提供疾病術語命名和編碼的標準,見表1。當然該語義網絡中的許多數據是從其中幾個數據庫中提取并整合的,因此整合更多細胞系相關的新數據、發現更多的語義關系也是本語義網絡應該繼續努力的方向。

表1 本研究模型數據類型覆蓋范圍與其他幾個相關數據庫對比
注:*即COSMIC沒有提供表型標準名稱以及與其他數據庫的映射
本研究通過D2RQ部署的語義網絡的Web應用,目前僅限于內部實驗使用,后期將持續完善細胞系語義網絡瀏覽平臺,發布為外網可訪問的形式并提供多種語義網絡獲取方式。
5 結語
本研究利用基于RDB到RDF映射語言(R2RML)的D2RQ映射工具,對以細胞系數據為中心的包含基因、突變與疾病數據以及語義關系的數據進行建模與整合,分析不同來源生物醫學數據的特性,發現數據融合的有效方法,所構建的語義網絡數據類型覆蓋更為全面,可為用戶提供更加便捷的服務。但是仍有許多不足之處有待改進,今后的工作將從以下內容展開:進一步優化多來源異構數據的融合方法,多方面考慮數據的不同特性,利用實體相似計算方法提高數據的映射成功率。研究突變對于癌癥等復雜疾病帶來的內部細胞機制的變化,豐富實體之間的語義關系。最終將數據來源擴展至文獻、電子病歷等形式,從中挖掘更多的生物醫學實體之間的語義關系,從而對語義網絡進行擴展,提高語義模型的實用性。

