沙特番茄黃化曲葉病毒AC2基因編碼蛋白的生物信息學(xué)分析
朱曉林 魏小紅 王寶強(qiáng) 馮悅 趙耀東
摘?要:該試驗(yàn)旨在通過生物信息學(xué)方法分析沙特番茄黃化曲葉病毒AC2基因編碼的轉(zhuǎn)錄激活蛋白TrAP(transcriptional activator protein)的結(jié)構(gòu)及功能。該文通過在線軟件預(yù)測和分析了番茄中AC2基因編碼蛋白的一級結(jié)構(gòu)、二級結(jié)構(gòu)、磷酸位點(diǎn)、糖基位點(diǎn)、三級結(jié)構(gòu)及其重要的功能;通過最大似然法對不同物種 AC2基因編碼蛋白序列構(gòu)建系統(tǒng)發(fā)育樹,進(jìn)行系統(tǒng)發(fā)育分析。結(jié)果表明:番茄中AC2基因共編碼135個氨基酸,且編碼蛋白屬于不穩(wěn)定的親水性蛋白質(zhì);二級結(jié)構(gòu)以無規(guī)則卷曲折疊為主,比例為58.52%;該蛋白不存在跨膜結(jié)構(gòu)域,屬非跨膜蛋白且不存在信號肽;該蛋白1~129個氨基酸區(qū)域?yàn)殡p生病毒超基因家族 (Gemini_AL2 superfamily) 結(jié)構(gòu)域;系統(tǒng)進(jìn)化情況和植物分類學(xué)基本一致,沙特番茄雙生病毒AC2基因與南非木薯間的親緣關(guān)系最近。該基因編碼蛋白結(jié)構(gòu)與功能的分析為進(jìn)一步研究AC2雙生病毒基因家族奠定了基礎(chǔ),同時為番茄抗病在病蟲害防治方面提供一種可行的思路。
關(guān)鍵詞:沙特番茄黃化曲葉病毒, AC2基因, 轉(zhuǎn)錄激活蛋白, 生物信息學(xué)
中圖分類號:Q943
文獻(xiàn)標(biāo)識碼:A
文章編號:1000-3142(2020)12-1773-08
Abstract:This experiment aimed to analyze the structure and function of the transcriptional activator protein transcriptal activator protein (TrAP) encoded by the AC2 gene of the Saudi tomato yellow leaf curl virus by bioinformatics.In this study, primary structure, secondary structure, phosphate site, glycosylation site, tertiary structure and important functions of the protein encoded by AC2 gene in tomato were predicted and analyzed by online software.Phylogenetic trees were constructed by maximal likelihood method for the encoded protein sequences of AC2 genes from different species for phylogenetic analysis.The results were as follows:The AC2 gene encoded a total of 135 amino acids in tomato, and the encoded protein belonged to an unstable hydrophilic protein; The secondary structure was dominated by random coil folding, with a ratio of 58.52%; The protein did not have a transmembrane domain,?it belonged to non-transmembrane protein and had no signal peptide; The 1-129 amino acid region of this protein was the Gemini_AL2 superfamily domain; The phylogenetics and plant taxonomy were basically the same, and the relationship between Saudi tomato yellow leaf curl virus AC2 gene and the South African cassava is recent.The analysis of the structure and function of the encoded protein provides a basis for further understanding of the AC2 geminivirus gene family, and provides a feasible approach for tomato disease resistance in pest control.
Key words:Saudi tomato yellow leaf curl virus, AC2 gene, transcriptional activator protein,?bioinformatics
番茄(Solanum lycopersicum)果實(shí)營養(yǎng)豐富、風(fēng)味獨(dú)特、富含維生素等,能有效降低消化道癌、前列腺癌等多種疾病發(fā)生的幾率,是深受人們喜愛的蔬菜,在全世界種植廣泛(曹玉鑫等,2018)。隨著種植面積、復(fù)種指數(shù)不斷增加,各種病害也接踵而至。
番茄黃化曲葉病作為蔬菜作物生產(chǎn)中最具毀滅性的病害,其致病因子是黃化曲葉病病毒。屬于雙生病毒科,是一個大型的單鏈DNA病毒家族,它在重要的經(jīng)濟(jì)作物上感染并造成毀滅性疾病(Sung & Coutts, 1996)。該病菌由煙粉虱(Bamesiatabasi)傳播,其病毒可以是單鏈DNA(ssDNA))或二份[(兩個相等)ssDNA稱為DNA A和B](Zerbini et al., 2017)。DNA A編碼互補(bǔ)的4~5個開放閱讀框(ORF)方向(AC1/C1,AC2/C2,AC3/C3,AC4/C4和AC5/C5)和兩個病毒感染的ORF(AV1/V1,AV2/V2)參與反式激活,復(fù)制和反應(yīng)的方向封裝(Hanley et al., 2013)。它們被細(xì)分為九個屬,該病毒屬的成員編碼稱為AC2/C2的致病蛋白,該類轉(zhuǎn)錄激活蛋白是能與啟動子中特定DNA序列結(jié)合并激活基因轉(zhuǎn)錄反應(yīng)的蛋白質(zhì)分子,其相互作用使許多植物蛋白失活并通過C-末端反式激活結(jié)構(gòu)域反式激活許多宿主基因(Babu et al., 2018)。同時TrAP激活葉肉細(xì)胞和原生質(zhì)體中病毒外殼蛋白(CP)和BR1運(yùn)動基因啟動子的表達(dá),并起到抑制植物組織中CP啟動子的作用(Rajeswaran et al., 2007)。謝迎秋等(2001)對棉花曲葉病病毒AC2 功能進(jìn)行了初步探究,揭示了AC2基因編碼的蛋白在寄主植物中反式激活病毒鏈基因啟動子的奇異現(xiàn)象,其為研究病毒與植物間的相互作用的分子機(jī)理提供了新依據(jù)。
目前國內(nèi)在番茄抗黃化曲葉病方面,主要集中于抗病基因的研究,對入侵病毒本身基因方面研究較少,尤其對病毒AC2基因的研究還鮮有報(bào)道,而當(dāng)今控制番茄黃化曲葉病病毒迫在眉睫。因此,完成對AC2 基因編碼的轉(zhuǎn)錄激活蛋白進(jìn)行全面的生物信息學(xué)分析,有助于理解病毒如何成功感染宿主,同時可通過分子手段抑制該基因的表達(dá),從而為以后番茄在防治黃化曲葉病以及抵制抗病方面提供參考價(jià)值。
1?材料與方法
1.1 序列來源
利用NCBI數(shù)據(jù)庫獲取不同物種病毒基因AC2編碼的TrAP氨基酸序列及其他信息。
1.2 蛋白質(zhì)結(jié)構(gòu)分析預(yù)測
1.2.1 一級結(jié)構(gòu)?通過Expasy平臺,根據(jù)Relative mutability參數(shù)、 Zimmerman方案、 Hopp & Woods方案、Bulkiness參數(shù)、Average flexibility參數(shù)、Janin方案預(yù)測蛋白的相對突變性、極性、親水性、松散性、柔韌性、可及性理化性質(zhì)等一級結(jié)構(gòu)。
1.2.2 二級結(jié)構(gòu)?通過法國里昂CNRS的SOPMA 軟件(https://npsa-prabi.ibcp.fr/cgi -bin/npsa_automat.pl? page = npsa_sopma.html)) 預(yù)測該基因編碼蛋白的二級結(jié)構(gòu); 利用CBS 系統(tǒng)中 SignalP5.0 server 平臺(http:/ / www.cbs.dtu.dk/services/SignalP/) 預(yù)測沙特番茄黃化曲葉病毒TrAP蛋白信號肽序列(李玩生等,2010);利用Scratch Predict Protein 在線軟件(徐慶剛等,2010)對該基因編碼產(chǎn)物進(jìn)行二硫鍵結(jié)合的預(yù)測與分析;利用 CBS 系統(tǒng)中TMHMM Server v.2.0平臺(http://www.cbs.dtu.dk/services/TMHMM/)進(jìn)行蛋白跨膜區(qū)域的預(yù)測(Sonnhammer et al., 1998)。
1.2.3 磷酸化位點(diǎn)和糖基化位點(diǎn)預(yù)測?利用 CBS 系統(tǒng)中 NetPhos 3.1平臺(http://www.cbs.dtu.dk/services/NetPhosK)以及NetOGlyc 4.0進(jìn)行蛋白磷酸位點(diǎn)與糖基位點(diǎn)的預(yù)測。
1.2.4 結(jié)構(gòu)域預(yù)測?NCBI中的 CDD 數(shù)據(jù)庫(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) 預(yù)測蛋白質(zhì)功能結(jié)構(gòu)域。
1.2.5 三級結(jié)構(gòu)?通過 SWISSMODEL(Lin et al., 2018)對沙特番茄黃化曲葉病病毒基因AC2編碼蛋白的三維結(jié)構(gòu)進(jìn)行同源建模。
1.3 系統(tǒng)發(fā)育樹的構(gòu)建
通過NCBI對 13個物種AC2基因編碼蛋白序列進(jìn)行同源比對分析,再利用MEGA X(Wang et al., 2017)軟件中的鄰接法(neighbor-joining method,NJ)構(gòu)建系統(tǒng)發(fā)育樹。
2?結(jié)果與分析
2.1 沙特番茄黃化曲葉病毒TrAP氨基酸序列
沙特番茄黃化曲葉病毒AC2基因GenBank登錄號為 NC_022229.1,其編碼的蛋白TrAP氨基酸序列為MQPSSPSTSHCSQVPIKVQHKLAKKKIIRRR
RLDLDCGCSYYLHINCTNHGFTHRGTHHCSSGNEWRFYLGDKQSPLFQDHQPQREATQHEQRHNFNTNPIQSQHQEGVGDSQMFSQLPNLDDLTASDWSFLKSI。對沙特番茄黃化曲葉病毒AC2基因完整編碼蛋白序列進(jìn)行 BLAST 分析,將其與同源性較高的從NCBI里下載的卷心菜( 序列號:NP_620887.1)、南非木薯 (序列號:NP_620664.1)、棉花(序列號:CDO50035.1)、云南煙草 (序列號:NP_722555.1)、甘薯 (序列號:NP_808770.1)、胡椒(序列號:NP_050016.1)、臺灣番茄(序列號:YP_001950236.1)、豆類(序列號:NP_612596.1)、土豆(序列號:NP_808757.1)、海南番茄(序列號:YP_003104749.1)、番木瓜 (序列號:NP_689460.1) 和云南番茄(序列號:YP_008060401.1)等序列構(gòu)建系統(tǒng)發(fā)育樹。
2.2 蛋白結(jié)構(gòu)分析
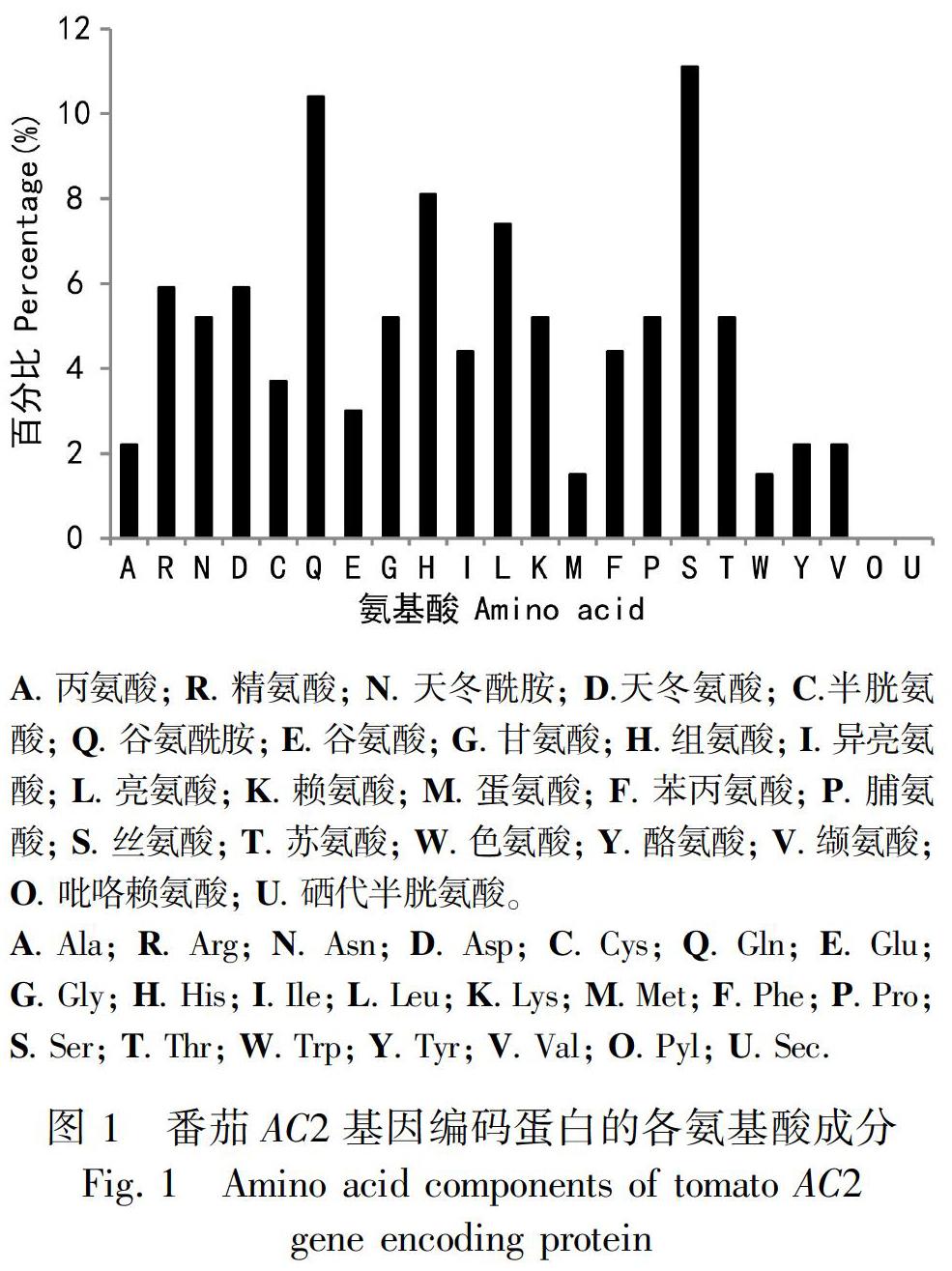
2.2.1 一級結(jié)構(gòu)?預(yù)測結(jié)果顯示,該蛋白共含135個氨基酸,其分子量為15 640.38,等電點(diǎn)為8.67,帶負(fù)電荷氨基酸殘基數(shù)為(Asp + Glu)12個,帶正電荷殘基數(shù)為(Arg + Lys)15個;分子式為C676H1037N211O206S7,原子總數(shù)為2 137個;不穩(wěn)定指數(shù)為61.37;消光系數(shù)為15 470 mol·L-1, 對應(yīng)吸光值是0.989。脂肪族氨基酸指數(shù)為54.89;各個氨基酸組成如圖1所示,其中絲氨酸、谷氨酰胺的比例最高,分別為11.1%、10.4%,蛋氨酸、色氨酸的比例最低,均為1.5%。
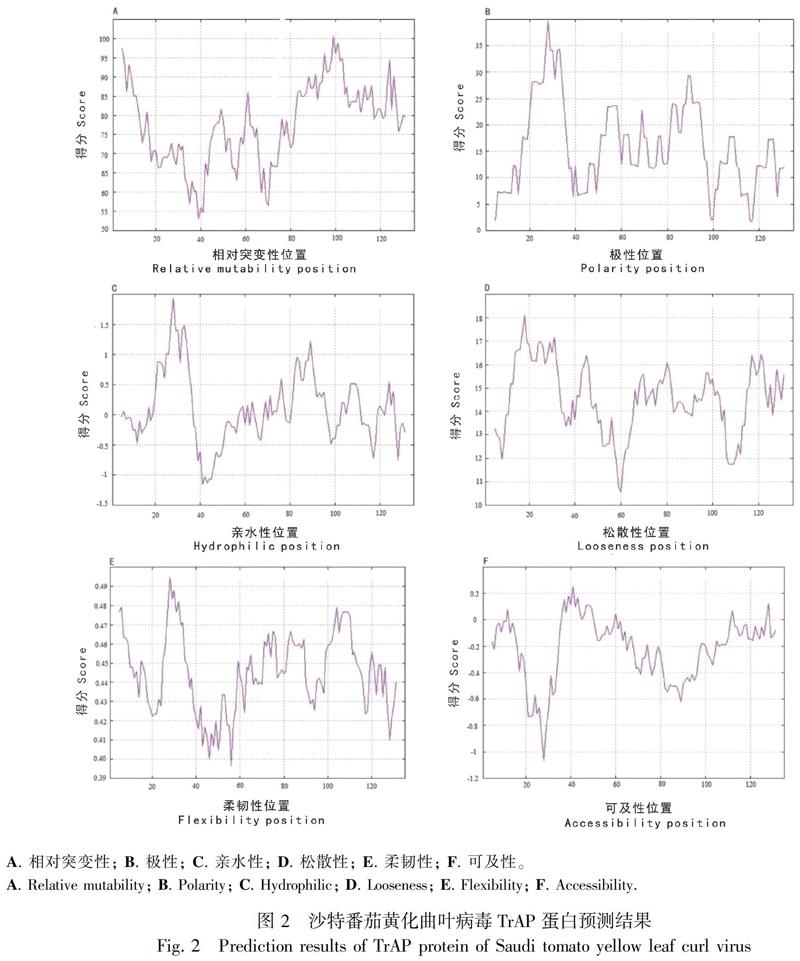
Expasy平臺預(yù)測結(jié)果顯示Relative mutability參數(shù)(圖2:A)得分位于53.000~100.556間,即該蛋白高突變區(qū)位于95~103位氨基酸間;Zimmerman方案(圖2:B)評分位于1.748~39.640間,因此可預(yù)測該蛋白高極性區(qū)位于21~34位氨基酸間;Hopp & Woods方案(圖2:C)評分表明其最大值為1.933,最小值為-1.156,因此該蛋白高親水性區(qū)域位于21~35位氨基酸間;Bulkiness參數(shù)(圖2:D)位于10.574~18.103之間,其蛋白的高松散區(qū)域位于12~33位氨基酸間;Average flexibility參數(shù)(圖2:E)得分位于0.397~0.494間,因此其高柔韌性區(qū)域在25~37位氨基酸之間;Janin方案(圖2:F)評分位于-1.067~0.256間,其高可及性區(qū)位于36~44位氨基酸間。
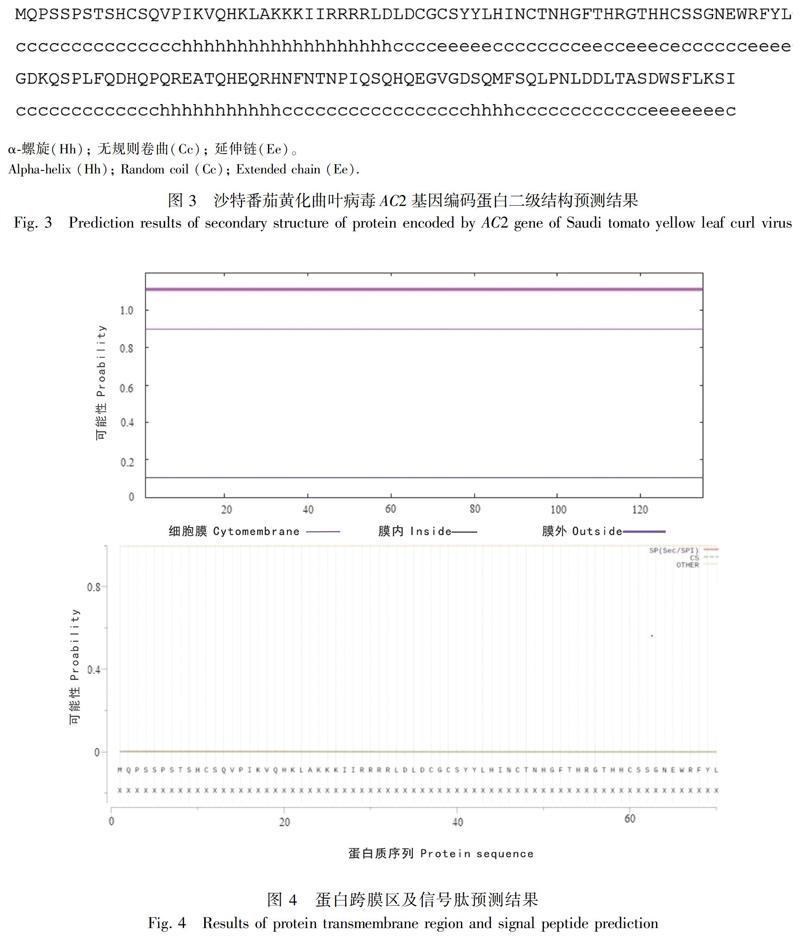
2.2.2 二級結(jié)構(gòu)分析預(yù)測?由圖3可知,無規(guī)則卷曲在整個多肽鏈主鏈骨架上所占比例最大,為58.52%,由79個氨基酸構(gòu)成,且其較為突出的區(qū)域是1~15、71~83、95~111,其次為α-螺旋,為25.19%,由34個氨基酸構(gòu)成,其較突出部位為16~34、84~94,而該氨基酸序列無β-轉(zhuǎn)角與β-折疊,而延伸鏈占16.30%。則該番茄蛋白主要二級結(jié)構(gòu)元件為無規(guī)則卷曲與α-螺旋。按照蛋白質(zhì)二級結(jié)構(gòu)劃分標(biāo)準(zhǔn)來看,沙特番茄黃化曲葉病毒基因編碼產(chǎn)物在結(jié)構(gòu)上屬于混合型(Skolnick & Fetrow, 2000; 曹建等,2012)。
如圖4所示, 經(jīng)預(yù)測, TrAP的1~135位氨基酸均位于細(xì)胞膜表面(圖4:A),由此可以推斷該蛋白無跨膜區(qū)域的形成。同時該蛋白也無信號肽的存在(圖4:B)。運(yùn)用 Scratch Protein Predictor軟件對該蛋白的二硫鍵進(jìn)行預(yù)測,結(jié)果顯示,預(yù)測沙特番茄黃化曲葉病毒TrAP蛋白5個 Cys 中,2個 Cys 形成 1對二硫鍵,預(yù)計(jì)在第37、39、47、60位間形成。
2.2.3 磷酸化位點(diǎn)和糖基化位點(diǎn)預(yù)測?運(yùn)用NetPhos 3.1在線軟件對番茄 TrAP蛋白磷酸化位點(diǎn)進(jìn)行預(yù)測,由圖5可知,該蛋白的氨基酸序列包含24個磷酸化位點(diǎn),且其磷酸化位點(diǎn)分別位于絲氨酸 (Ser)、蘇氨酸 ( Thr)?和酪氨酸 ( Tyr) 殘基上。絲氨酸磷酸化位點(diǎn)15個、蘇氨酸磷酸化位點(diǎn)7個以及酪氨酸磷酸化位點(diǎn)2個,多磷酸化位點(diǎn)在蛋白質(zhì)翻譯后修飾水平上發(fā)揮著重大作用。而NetOGlyc 4.0預(yù)測結(jié)果表明該蛋白含有9個糖基化位點(diǎn)。
2.2.4 結(jié)構(gòu)域預(yù)測?應(yīng)用NCBI 中的CDD 數(shù)據(jù)庫( http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) 預(yù)測蛋白質(zhì)功能結(jié)構(gòu)域。預(yù)測結(jié)果如圖6所示,結(jié)果表明沙特番茄黃化曲葉病病毒基因AC2轉(zhuǎn)錄激活蛋白的1~129 個氨基酸區(qū)域?yàn)殡p生病毒超家族 (Gemini_AL2 superfamily) 結(jié)構(gòu)域,該結(jié)構(gòu)域?qū)υ摼幋a蛋白功能發(fā)揮有極其重要的作用。
2.2.5 三級結(jié)構(gòu)?通過同源建模方法構(gòu)建的沙特番茄黃化曲葉病毒基因AC2編碼蛋白的三級結(jié)構(gòu),結(jié)果如圖7所示。該蛋白結(jié)構(gòu)較為簡單,卷曲鏈與折疊鏈結(jié)構(gòu)較少,蛋白質(zhì)的三級結(jié)構(gòu)對其生物學(xué)功能的發(fā)揮具有極其重要的作用,因此預(yù)測該蛋白功能相對單一。
2.3 AC基因編碼蛋白的系統(tǒng)發(fā)育樹構(gòu)建
從圖8可以看出,在通過系統(tǒng)發(fā)育樹構(gòu)建的13個物種中,沙特番茄黃化曲葉病毒與南非木薯間的親緣關(guān)系最近,其次是棉花、番木瓜、海南番茄、胡椒、云南番茄、臺灣番茄、云南煙草、土豆、豆類、卷心菜、甘薯。
3?討論
煙粉虱作為黃化曲葉病病毒的攜帶者,可操縱植物防御信號。在病蟲害侵染后,植物體內(nèi)主要進(jìn)化出兩種防衛(wèi)反應(yīng)機(jī)制,水平抗性與垂直抗性。植物首先動員水楊酸(SA)依賴的水平抗性防御,其主要針對病原體。垂直抗性是植物在長期抵制病原菌的過程中形成的專一抗性,通過進(jìn)化出專一抗性基因來抵抗病毒(Gill et al., 2019), 而部分番茄品種體內(nèi)不存在抗病基因,因此以病毒基因自身結(jié)構(gòu)作為一個切入點(diǎn),利用RNA干擾沉默病毒AC2基因有助于病害的防御。黃化曲葉病病毒作為危害番茄產(chǎn)量最主要的病害,由雙生病毒引起,其該病毒不僅可作為表達(dá)載體在植物中直接表達(dá)外源基因,還可提供分離的調(diào)控元件,調(diào)控植物體的生理生化過程。研究表明,在AC2基因不存在時, ACMV病毒鏈基因啟動子表達(dá)活性較低,AC2 基因存在時,病毒鏈基因啟動子活性顯著增強(qiáng),從而使病毒高效復(fù)制(Brough et al., 1992)。同時導(dǎo)致AC2激活病毒鏈基因啟動子復(fù)雜性的另一個可能因素是病毒鏈基因啟動子的活性受時空調(diào)控,具有發(fā)育階段特異性和組織特異性(Sunter & Bisaro, 1997) 。而在該病毒入侵植物時,AC2基因編碼的轉(zhuǎn)錄激活蛋白在其病毒自我復(fù)制中發(fā)揮了很重要的作用,已發(fā)現(xiàn)在煙草中AC2基因能產(chǎn)生嚴(yán)重的影響,使煙草屬植物的葉組織發(fā)育不全(Siddiqui et al., 2008)。AC2編碼的蛋白可作為植物體內(nèi)寄主RNA沉默的抑制子,而寄主RNA沉默是植物抵御病毒入侵的有效手段,因此抑制AC2基因編碼產(chǎn)物是防止病毒在植物體內(nèi)傳播的有效途徑。本研究對沙特番茄黃化曲葉病毒基因編碼的TrAP蛋白序列的一級、二級、三級結(jié)構(gòu)以及功能分類等進(jìn)行預(yù)測及分析,該結(jié)果為研究AC2基因及其編碼產(chǎn)物的結(jié)構(gòu)和功能提供了更多的信息,同時對AC2基因進(jìn)行全面的生物信息學(xué)分析,以期通過分子手段抑制其基因的表達(dá),為番茄抗病育種提供可行的思路。
本研究中AC2 基因編碼產(chǎn)物共含135個氨基酸,其中絲氨酸含量最高,該氨基酸含量的高低與蛋白親水性有密切關(guān)聯(lián),同時該蛋白不穩(wěn)定系數(shù)高達(dá)61.37,因此其屬于不穩(wěn)定的親水性蛋白質(zhì)。AC2 基因編碼蛋白的二級結(jié)構(gòu)以α-螺旋和無規(guī)卷曲為主,存在2個二硫鍵,這與該蛋白分子對抗外界因素影響的不穩(wěn)定性有很大關(guān)系。該基因編碼蛋白是一種轉(zhuǎn)錄激活因子,其結(jié)構(gòu)不存在跨膜區(qū),即不屬于定位于生物膜的膜蛋白。該編碼蛋白含有1個雙生病毒超家族結(jié)構(gòu)域,其在細(xì)胞被運(yùn)輸以及結(jié)合嘌呤和嘧啶中起關(guān)鍵的作用,同時在細(xì)胞信號轉(zhuǎn)導(dǎo)中發(fā)揮作用。沙特番茄黃化曲葉病毒基因與南非木薯間的親緣關(guān)系最為密切,其次是棉花、番木瓜、海南番茄、胡椒、云南番茄、臺灣番茄、云南煙草、土豆、豆類、卷心菜、甘薯。由各物種同源性高于 25% 就可判斷其功能上的相似性(Garnier et al., 1996)。 這種同源性一方面體現(xiàn)出各物種間的親緣關(guān)系,另一方面也表明各個物種的AC2基因編碼產(chǎn)物在結(jié)構(gòu)特征中的相對穩(wěn)定性,同時表明AC2基因編碼蛋白在漫長的生物演變過程中更多地受凈化選擇作用的束縛,其保守性較強(qiáng)。
4?結(jié)論
本研究通過生物信息學(xué)方法對沙特番茄黃化曲葉病毒AC2基因編碼的轉(zhuǎn)錄激活蛋白進(jìn)行預(yù)測和分析。結(jié)果表明,沙特番茄黃化曲葉病毒AC2主要編碼病毒轉(zhuǎn)錄激活蛋白,其在病毒入侵番茄的過程中發(fā)揮極其重要的作用,對病毒在植物體內(nèi)的繁殖起關(guān)鍵作用。該編碼蛋白由135個氨基酸組成, 是一種相對不穩(wěn)定的親水性蛋白;二級結(jié)構(gòu)以無規(guī)則卷曲與α-螺旋為主,該蛋白無信號肽與跨膜結(jié)構(gòu)域,但存在二硫鍵;三級結(jié)構(gòu)預(yù)測模型較為簡單;其蛋白1~129 個氨基酸區(qū)域?yàn)殡p生病毒超家族 (Gemini_AL2 superfamily) 結(jié)構(gòu)域。利用鄰接法對AC2基因進(jìn)行系統(tǒng)發(fā)育分析,發(fā)現(xiàn)沙特番茄黃化曲葉病毒AC2與南非木薯間的親緣關(guān)系最近,與卷心菜、甘薯之間的親緣關(guān)系最遠(yuǎn)。
參考文獻(xiàn):
BABU KSD, MANOHARAN P, PANDI G, 2018.Computational studies on begomoviral AC2/C2 proteins[J].Bioinformation, 14:294-303.
BROUGH CL, SUNTER G, GARDINER WE, et al., 1992.Kinetics of tomato golden mosaic virus DNA replication and coat protein promoter activity in Nicotiana tabacum protoplasts[J].Virology, 187:1-9.
CAO J, LUO YZ, HU J, et al., 2012.Polymorphism and bioinformatics analysis of CDS in H-FABP gene in Gannan yak[J].J Gansu Agric Univ, 47 (4) :20-26.[曹建,羅玉柱,胡江,等, 2015.甘南牦牛H-FABP 基因 CDS 區(qū)多態(tài) 性及生物信息學(xué)分析[J].甘肅農(nóng)業(yè)大學(xué)學(xué)報(bào),47(4):20-26.]
CAO YX, CAO HX, WANG P, et al., 2018.Effects of nutrient solution concentration on growth, quality and storability of tomato[J].Food Sci, 39(7):63-70.[曹玉鑫,曹紅霞,王萍,等,2018.營養(yǎng)液濃度對番茄生長、品質(zhì)以及耐貯性的影響[J].食品科學(xué),39(7):63-70.]
GILL U, SCOTT JW, SHEKASTEBAND R, et al., 2019.Ty-6, a major begomovirus resistance gene on chromosome 10, is effective against tomato yellow leaf curl virus and tomato mottle virus[J].Theor Appl Genet, 132:1543-1554.
GARNIER J, GIBRAT JF, ROBSON B,1996.Method for predicting protein secondary structure from amino acid sequence[J].Method Enzymol, 266:540-553.
HANLEY BL, BEJARANO ER, ROBERTSON D, et al., 2013.Geminiviruses masters at redirecting and reprogramming plant processes[J].Nat Rev Microbiol, 11:777-788.
LI YS, LIU L, FENG HY, et al., 2010.Cloning and bioinformatics analysis of porcine T cell receptor beta chain gene[J].J Gansu Agric Univ, 45(3):1-6.[李玩生, 劉磊, 馮海燕, 等, 2010.豬T細(xì)胞受體β 鏈基因的克隆及生物信息學(xué)分析[J].甘肅農(nóng)業(yè)大學(xué)學(xué)報(bào), 45(3):1-6.]
LIN YM, LIN CM, LIN WH, et al., 2018.Mild clinical features of isolated methylmalonic acidemia associated with a novel variant in the MMAA gene in two Chinese siblings[J].Bmc Med Genet, 19:114.
RAJESWARAN R, SUNITHA S, SHIVAPRASAD PV, et al., 2007.The mungbean yellow mosaic begomovirus transcriptional activator protein transactivates the viral promoter-driven transgene and causes toxicity in transgenic tobacco plants[J] .Mol Plant Microbe Interact, 20:1545-1554.
SONNHAMMER EL, HEIJNE G, KROGH A, 1998.Ahidden markov model for predicting transmembrane helices in protein sequences[J].Proc Int ConfIntell Syst Mol Biol, 6:175-182.
SUNG YK, COUTTS RH, 1996.Potato yellow mosaic geminivirus AC2 protein is a sequence non-specific DNA binding protein[J].Febs Lett, 383:51-54.
SUNTER G, BISARO DM, 1997.Regulation of a geminivirus coat protein promoter by AL2 protein ( TrAP):Evidence for activation and depression mechanisms[J].Virology, 232:269-280.
SIDDIQUI SA, SARMIENTO C, TRUVE E, et al., 2008.Phenotypes and functional effects caused ntabacum[J].Mol Plant Microbe Interact, 21:178-187.
SKOLNICK J,?FETROW JS, 2000.From genes to protein structure and function:Novel applications of computational approaches in the genomic era[J].Trends Biotechnol,?18(1): 34-39.
WANG XD, MA DH, HUANG XW, et al., 2017.Complete genome analysis of dengue virus type 3 isolated from the 2013 dengue outbreak in Yunnan, China[J].Virus Res, 238:164-170.
XIE YQ, MENG M, ZHU Z, et al., 2001.Preliminary functional studies on AC2, a novel trans-acting factor from cotton leaf curl virus[J].Bull Bot, 32(5):517-521.[謝迎秋, 孟蒙, 朱禎, 等, 2001.棉花曲葉病毒反式作用因子AC2的功能初探[J].植物學(xué)報(bào), 32(5):517-521.]
XU QG, LU J, ZHENG JZ, et al., 2010.Characterization and bioinformatics analysis of 4SNc-Tudor domain protein of Takifugurubripes[J].J Anhui Agric Sci, 38(3):1163-1166.[徐慶剛, 陸健, 鄭建洲, 等, 2010.河豚 4SNc-Tudor 蛋白的鑒定及生物信息分析[J].安徽農(nóng)業(yè)科學(xué), 38(3):1163-1166.]
ZERBINI FM, BRIDDON RW, IDRIS A, et al., 2017.ICTV virus taxonomy profile:geminiviridae[J].J Gen Virol, 98:131-133.
(責(zé)任編輯?周翠鳴)

