原始小球藻AP2轉錄因子的生物信息學分析
付春 唐易 江納 楊瑤君

摘?要:為了揭示原始小球藻AP2基因家族編碼蛋白的理化特性、分子功能和遺傳進化特征,該文對原始小球藻AP2基因家族的蛋白成員進行了詳細的生物信息學預測和分析,并利用PrtoParam、Pfam 3.20、Protscale等在線工具分析了原始小球藻AP2家族所包含的8個蛋白成員。結果表明:該家族成員含有的氨基酸數為247 (XP_011395646.1)到715 (XP_011401904.1),理論等電點最大為9.39 (XP_011396011.1)、最小為5.81 (XP_011398158.1);家族中各個成員所含有的保守結構域的位置和數量都各不相同;所有蛋白成員均無信號肽,均不包含跨膜螺旋,不具有跨膜區域;蛋白成員中最大親水性值為-3.222 (XP_011398158.1),最大疏水性值為2.333 (XP_011401904.1),且所有成員的平均親疏水性值均小于0;成員中有多個磷酸化位點值遠超標準值0.5,最大磷酸化位點值為0.998;該基因家族的蛋白成員二級結構的組分含量從大到小排序均為無規則卷曲>α-螺旋>延伸鏈>β-轉角,推測α-螺旋和無規則卷曲是其二級結構的主要方式,延伸鏈和β-轉角則分散在所有蛋白成員的氨基酸鏈中;所有成員的三級結構分析中均能觀察到α-螺旋、β-折疊、β-轉角以及N端和C端;系統進化分析結果表明,在其余15個物種中,與原始小球藻親緣關系最遠的是金牛介球菌(Ostreococcus tauri),親緣關系最接近的是小球藻(Chlorella variabilis NC64A)和螺旋孢子蟲(Helicosporidium),較接近的是苦參3號(Picochlorum sp.SENEW3)。該研究較系統地分析了原始小球藻AP2蛋白家族的理化特性、保守基序及系統進化關系,為進一步研究原始小球藻AP2轉錄因子功能和互作關系提供一定參考依據。
關鍵詞:原始小球藻, AP2轉錄因子, 生物信息學
中圖分類號:Q943
文獻標識碼:A
文章編號:1000-3142(2020)12-1800-16
Abstract:In order to reveal the physicochemical properties, molecular functions and genetic evolution characteristics of AP2 gene family encoded proteins in Auxenochlorella protothecoides, in this study, the protein members of AP2 gene family in A.protothecoides are predicted and analyzed in detail by bioinformatics.Eight protein members of the AP2 family of A.protothecoides were analyzed by online tools such as PrtoParam, Pfam 3.20 and Protscale and so on.The results were as follows:Amino acids numbers of the family members were 247(XP_011395646.1) to 715(XP_011401904.1), the maximum theoretical isoelectric point was 9.39(XP_011396011.1), and the minimum was 5.81(XP_011398158.1); The positions and numbers of conserved domains in each member of the family were different; All protein members had no signal peptide, did not contain transmembrane helix, and did not have transmembrane region; The maximum hydrophilicity value of the protein members was -3.222(XP_011398158.1), and the maximum hydrophobicity value was 2.333(XP_011401904.1), and the average hydrophilicity/hydrophobicity values of all members were less than 0; The values of many phosphorylation sites in the members were far beyond the standard value of 0.5, and the maximum value of phosphorylation site was 0.998; The component content of secondary structures of protein members in the gene family was arranged in the order of random coil > alpha-helix > extended strand > β-turn; and it is speculated that alpha-helix and random coil are the main modes of its secondary structure, while extended strand and β-turn are dispersed in the amino acid chain of all protein members; Alpha-helix, β-sheet, β-turn, N-terminal and C-terminal were observed in tertiary structure of all members; Phylogenetic analysis showed that Ostreococcus tauri was the most distant relative to Auxenochlorella protothecoides, the closest relatives were Chlorella variabilis NC64A and Helicosporidium, and the next closest relatives were Picochlorum sp.SENEW3 among the other 15 species.This study systematically analyzed the physicochemical properties, conserved motifs and phylogenetic relationships of AP2 protein family in Auxenochlorella protothecoides.It provides a reference for further study on the function and interaction of AP2 transcription factors in A.protothecoides.
Key words:Auxenochlorella protothecoides, AP2 transcription factor, bioinformatics
原始小球藻(Auxenochlorella protothecoides)是一種生長周期短,易培養,可利用光能光合作用進行自養,又能利用有機碳源進行異養培養積累油脂,高附加值產物豐富的微藻(汪桂林,2013),它被認為是生物柴油最具潛力的原料之一(桂小華,2017)。原始小球藻生長周期短、適應性強、光合效率和含油量高、單位面積產量高,現已被作為生物柴油方面主要能源藻種加以研究開發(汪桂林等,2013)。AP2是植物所特有的轉錄因子家族之一,起到調控植物生長發育的轉錄以及逆境脅迫等生理過程的作用(栗麗和佟少明,2016)。Jofuku(1994)從模式植物擬南芥(Arabidopsis thaliana)中分離了第一個AP2基因,且該基因與花發育有關(張計育等,2012),其后又在原核生物和藍藻中發現了相同的轉錄因子(張麒等,2018),證實該轉錄因子不止只存在于植物中。在不同的植物中含有的AP2/ERF 類轉錄因子基因家族成員數量是不同的(Riccardo et al., 2010),轉錄因子AP2/ERF主要特征是具有高度保守的約60~70個氨基酸組成的AP2結構域(聞可心和劉雪梅,2010),這是植物轉錄因子家族之一,其家族成員均包含保守AP2結構域,人們根據保守結構域的數量和識別成員的不同,通常情況下,將AP2/ERF家族劃分為不同的4個轉錄因子亞家族,分別是AP2、ERF、DREBP、RAV和單獨成員Soloist(張麒等,2018)。轉錄因子AP2基因家族在植物生長發育中起重要作用,它能調控植物的細胞周期、生長發育以及生物和非生物脅迫相關基因的表達(吳麗娟等,2016;郭慧等,2017),也能調控自身生殖器官(花、胚珠和種子)的生長發育(張妍和孫豐賓,2011;王莉莉,2016),而且與其他因子相互協作,參與到復雜的花發育調控網絡(劉楊,2009)。雖然關于藻類AP2基因家族的研究較多,但是未見關于原始小球藻AP2轉錄因子的生物信息學分析的報道。本研究通過分析原始小球藻的AP2轉錄因子編碼蛋白的理化特性、親疏水性、跨膜域、保守結構域、保守基序、亞細胞定位以及AP2轉錄因子在不同物種中的進化起源關系,為進一步研究原始小球藻AP2基因家族的生物學功能奠定一定的理論依據。
1?材料與方法
1.1 材料
從plantTFDB (http://planttfdb.cbi.pku.edu.cn/)數據庫中下載原始小球藻(Auxenochlorella protothecoides)AP2基因家族中所有成員的蛋白序列和巖藻(Bathycoccus prasinos)、萊茵衣藻(Chlamydomonas reinhardtii)、小球藻(Chlorella variabilis NC64A)、膠球藻(Coccomyxa subellipsoidea C-169)、鹽生杜氏藻(Dunaliella salina)、胸狀盤藻(Gonium pectorale)、螺旋孢子蟲(Helicosporidium)、細小微胞藻(Micromonas pusilla CCMP1545)、微單胞藻(Micromonas sp.RCC299)、深藍單殼縫藻(Monoraphidium neglectum)、透明介球菌(Ostreococcus lucimarinus)、介球菌(Ostreococcus sp.RCC809)、金牛介球菌(Ostreococcus tauri)、苦參3號(Picochlorum sp.SENEW3)以及團藻(Volvox carteri)等物種的AP2成員蛋白序列。對原始小球藻的AP2所有成員的蛋白質序列進行詳細的生物信息學分析,且將原始小球藻與巖藻(Bathycoccus prasinos)等16個物種AP2蛋白進行同源序列比對分析并構建進化樹,探究16個物種間的進化起源關系。
1.2 方法
1.2.1 AP2轉錄因子基因的蛋白成員的一級結構和理化性質分析?用在線程序PrtoParam (https://web.expasy.org/protparam/)對原始小球藻AP2轉錄因子蛋白成員的一級結構和理化性質進行分析,包括了蛋白成員氨基酸數目、理論pI、分子量、帶負電氨基酸殘基、帶正電氨基酸殘基、不穩定系數、脂肪指數、親疏水性、分子式、原子總數等方面(韓長志和王娟,2016)。通過在線程序Cell-PLoc 2.0 (http://www.csbio.sjtu.edu.cn/bioinf/ Cell-PLoc-2/)對原始小球藻AP2轉錄因子的8個蛋白成員進行亞細胞定位分析(徐志軍等,2019),參數設置:物種選擇為真核生物。
1.2.2 氨基酸的保守結構域、跨膜結構及信號肽分析?用Pfam 3.20 (http://pfam.xfam.org/)對原始小球藻的蛋白成員進行保守結構域預測(張海燕等,2013),統計分析蛋白成員氨基酸保守結構域的個數及位置。在TMHMM Severv.2.0 (http://www.cbs.dtu.dk/ services/TMHMM/)在線程序中輸入AP2轉錄因子基因蛋白成員,分析跨膜螺旋的數量以及跨膜結構的區域,確定基因蛋白成員是否具有跨膜區域,判斷是否為跨膜蛋白。用SignaIP3.0 Server(http://www.cbs.dtu.dk/services/SignalP-3.0/)對原始小球藻蛋白成員進行信號肽分析(柳燕等,2017)。
1.2.3 氨基酸的親疏水性和磷酸化位點預測分析?用Protscale (https://web.expasy.org/protscale/)在線工具,對該轉錄因子基因蛋白成員進行親疏水性預測分析,確定最大親水值和最大疏水值及其相對應的位置,判斷該基因蛋白成員為親水蛋白還是疏水蛋白。用NetPhos 3.1 Server (http://www.cbs.dtu.dk/services/NetPhos/)在線程序對轉錄因子基因蛋白成員進行潛在磷酸化位點預測分析,并統計磷酸化位點種類和數量。
1.2.4 編碼蛋白的二級結構和三級結構預測分析?用SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在線程序對該轉錄因子蛋白成員的二級結構進行預測分析,統計該蛋白成員中α-螺旋、無規則卷曲、延伸鏈和β-轉角相對應的百分比。通過同源建模法,利用SWISS-MODEL (https://www.swissmodel.expasy.org/)在線工具(Schwede et al., 2003),對該蛋白家族的三級結構進行預測分析,確定其三級結構的組成和構象變化。
1.2.5 保守基序分析?用MEME7.0 (http://meme-suite.org/)在線工具對原始小球藻AP2轉錄因子的蛋白成員進行保守基序motif分析,參數設置:motif數目為10個,其余參數設為默認值。
1.2.6 同源性分析及進化樹分析?在plantTFDB (https://web.expasy.org/protparam/)在線程序中,將原始小球藻的8個成員序列,分別和同為綠藻門下的其余15個物種AP2基因家族的蛋白成員進行同源序列的對比,找到相對應的其余物種的同源序列,并歸納。用MEGA7.0 (Tamura et al., 2011)軟件中的ClustalW功能對原始小球藻AP2轉錄因子所有成員蛋白對應的序列組,分別進行多重比對并鄰接法構建其系統進化樹(李建伏等,2008),對比8個蛋白成員的同源序列的差距,判斷各個物種與原始小球藻之間的親緣關系。
2?結果與分析
2.1 AP2轉錄因子基因成員的一級結構和理化性質分析
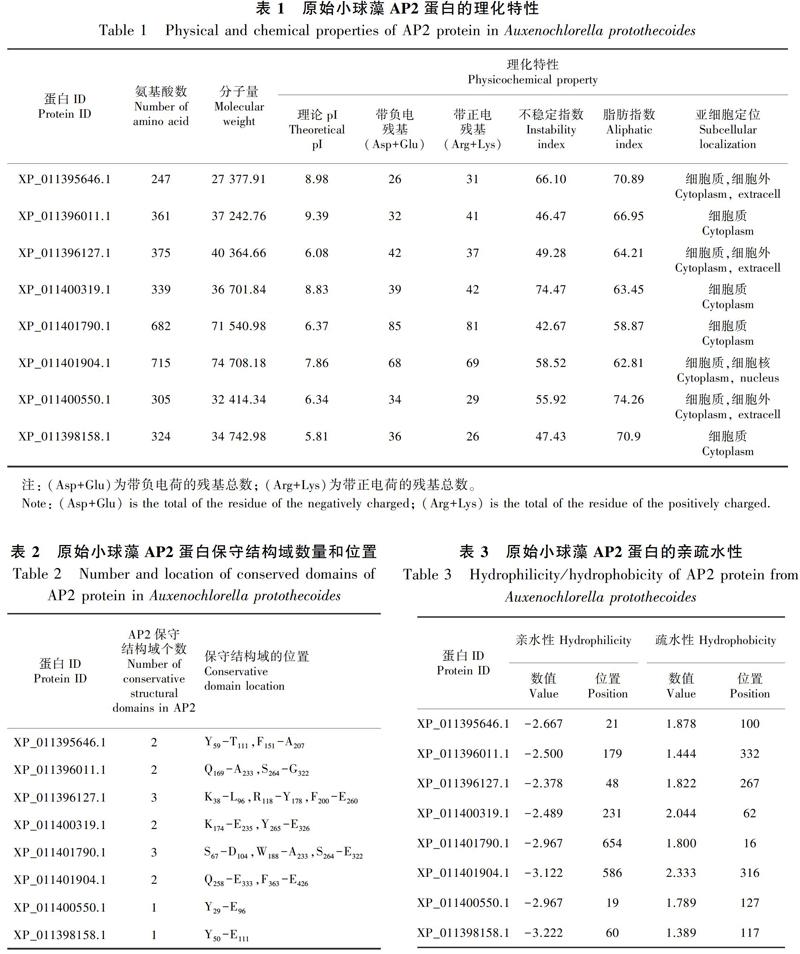
由表1可知,原始小球藻AP2轉錄因子蛋白家族含有8個成員,該蛋白家族氨基酸數目范圍為247~715;等電點范圍為5.81(XP_011398158.1)~9.39(XP_011396011.1),其中,XP_011395646.1、XP_011396011.1、XP_011400319.1和XP_011401904.1,理論等電位點>7,為堿性氨基酸,XP_011396127.1、XP_011401790.1、XP_011400550.1和XP_011398158.1,理論等電位點<7,為酸性氨基酸;該家族成員帶負電氨基酸殘基數目范圍為26(XP_011395646.1)~85(XP_011401790.1);該家族成員帶正點氨基酸殘基數目范圍為26(XP_011398158.1)~81(XP_011401790.1);不穩定指數均大于40(不穩定系數<40時穩定)(夏巧玉,2007),推斷為不穩定蛋白;脂肪指數范圍為58.87(XP_011401790.1)~74.26(XP_011400550.1);原始小球藻AP2蛋白成員進行的亞細胞定位分析中顯示該家族全部成員的亞細胞都可定位于細胞質(Cytoplasm),其中4個蛋白成員:XP_011396011.1、XP_011400319.1、XP_011401790.1和XP_011398158.1的亞細胞只定位于細胞質(表1)。通過氨基酸組成分析可知,在所有蛋白成員中,除了XP_011396011.1(氨基酸的最大含量為甘氨酸15.80%)和XP_011398158.1(氨基酸的最大含量為亮氨酸11.10%)兩個蛋白家族成員外,其余六個蛋白家族成員含量最高的氨基酸均為丙氨酸,丙氨酸含量最高的成員為XP_011400550.1(19.70%),含有丙氨酸數為60,丙氨酸含量最低的成員為XP_011400319.1(11.2%),含有丙氨酸數為38(圖1)。
2.2 氨基酸保守結構域、信號肽和跨膜域結構分析
蛋白成員中,不同基因蛋白成員的AP2保守結構域的數量和位置各不相同,其中XP_011396127.1(K38-L96,R118-Y178,F200-E260)和XP_011401790.1(S67-D104,W188-A233,S264-E322)兩個成員各含3個保守結構域;XP_011398158.1(Y50-E111)和XP_011400550.1(Y29-E96)兩個成員中各自都只含有1個氨基酸保守結構域;余下的XP_011395646.1(Y59-T111,F151-A207)、XP_011396011.1(Q169-A233,S264-G322)、XP_011400319.1(K174-E235,Y265-E326)和XP_011401904.1 (Q258-E333,F363-E426)4個成員,均包含2個保守結構域。保守結構域跨度最大成員是XP_011401904.1(Q258-E333),其氨基酸數量達75個,蛋白成員XP_011401790.1(S67-D104)中保守結構域跨度最小,其氨基酸數量為37個(表2,圖2)。各個蛋白成員的位置和跨度都各不相同,但卻有相似,如XP_011395646.1、XP_011400319.1和XP_011401904.1這3個成員就含有相似之處(圖2)。信號肽分析表明原始小球藻AP2蛋白均不存在信號肽,屬于非分泌型蛋白。TMHMM跨膜域結構分析表明原始小球藻所有蛋白成員均不含跨膜結構域,推斷該蛋白為非跨膜蛋白。
2.3 氨基酸親疏水性預測分析
在這8個蛋白家族成員中,氨基酸疏水性的最大值達到了2.333 (XP_011401904.1),疏水性最小值為1.389 (XP_011398158.1);氨基酸親水性最大值達到了-3.222 (XP_011398158.1),親水性最小值為-2.378 (XP_011396127.1)。該家族成員XP_011401904.1的氨基酸親疏水性差值最大,達到了5.455,對應平均親水指數(GRAVY)為-0.394 5,其第316位氨基酸表現最大疏水性為2.333,第584、585、586、588、589位氨基酸均表現最大親水性為3.122。所有的AP2蛋白家族成員中的平均親水指數均小于0,所以該蛋白親水性更強,推斷該原始小球藻AP2轉錄因子蛋白為親水
2.4 磷酸化位點分析
經分析發現,在閾值為0.5時,原始小球藻AP2轉錄因子蛋白成員中潛在的磷酸化位點最多的是XP_011401790.1 (77),潛在磷酸化位點最少的是XP_011400550.1 (24)。將同種氨基酸對比分析,在整個家族中,潛在位點為絲氨酸(S)數量最多的是XP_011401790.1,達到了49個位點,數量最少的是XP_011395646.1和XP_011400550.1兩個成員,均含有17個位點;潛在位點為蘇氨酸(T)數量最多的是XP_011401790.1,達到了24個位點,數量最少的是XP_011400550.1,僅含有4個位點;潛在位點為絡氨酸(Y)數量最多的是XP_011396011.1和XP_011401904.1,都達到了5,數量最少的是XP_011398158.1,僅1個位點(表4)。在第4個分支XP_011400319.1中,第164位、第218位的蘇氨酸,第142位、第160位絲氨酸以及第268位絡氨酸可能是潛在的磷酸化位點,其中最有可能是潛在磷酸化位點的是第142位和第160位的絲氨酸,其值均已達到0.998,遠超標準值0.5,同時也說明蛋白質可能通過絲氨酸磷酸化位點來實現其功能的調控(表4)。
2.5 原始小球藻AP2轉錄因子編碼蛋白的二級結構和三級結構預測分析
蛋白質的二級結構主要是指蛋白質的多肽鏈中有規則重復的構象 (曹晨和馬堃,2016),在原始小球藻AP2轉錄因子基因蛋白成員中,二級結構中組分含量從大到小排序均為無規則卷曲>α-螺旋>延伸鏈>β-轉角,其中無規則卷曲和α-螺旋的占比較大,推測其空間結構較小,且受到側鏈間相互作用較大的影響(王月,2017)。基因二級結構組分中α-螺旋值最大的是XP_011400550.1,占比達到40.00%,α-螺旋值最小的是XP_011396011.1,其占比為26.04%;該蛋白三級結構組分中延伸鏈占比最大為XP_011400319.1,達16.22%,其延伸鏈最小占比為XP_011396127.1,達10.40%;該組分中β-轉角值為3.64% (XP_011395646.1)~8.64% (XP_011398158.1);其結構組分中無規則卷曲值為40.00% (XP_011400550.1)~54.55% (XP_011401904.1)(表5)。由此可推斷,在原始小球藻AP2基因蛋白成員中α-螺旋和無規則卷曲是二級結構的主要方式,延伸鏈和β-轉角則分散在整個基因蛋白成員的氨基酸鏈中(表5,圖4)。
蛋白質三級結構預測分析表明原始小球藻AP2家族蛋白成員均含有α-螺旋、β-折疊、β-轉角以及成員的N端和C端等結構,其中最主要的結構是α-螺旋,其次是β-折疊。且XP_011398158.1、XP_011400550.1和XP_011398158.1的三級結構高度相似,而XP_011400319.1和XP_011400319.1的三級結構也同樣高度相似(圖5)。
2.6 保守基序MEME分析
通過分析可知,在原始小球藻AP2轉錄因子家族的8個成員中,motif1只存在于XP_011395646.1和XP_011401904.1中;除XP_011400550.1和XP_011398158.1外,其余6個成員中均含有motif2和motif4;所有成員均包含motif3和motif5;motif6存在于XP_011395646.1和XP_011396011.1中;motif7存在于XP_011396127.1和XP_011398158.1中;motif8存在于XP_011396127.1和XP_011401790.1中;motif9存在于XP_011400319.1、XP_011396127.1和XP_011398158.1中。該家族成員可分為四類,其中XP_011395646.1和XP_011396011.1為一類,都含有motif2、motif3、motif4、motif5、motif6和motif10;第二類是XP_011400319.1和XP_011401904.1, 都含有motif3、motif4和motif5;第三類是XP_011396127.1和XP_011401790.1,都含有motif2、motif3、motif4和motif8;第四類是XP_011400550.1和XP_011398158.1, 都含有motif3和motif5。在原始小球藻AP2蛋白成員中,含有的主要是motif3、motif5(圖6,圖7)。
2.7 同源性比對及進化樹分析
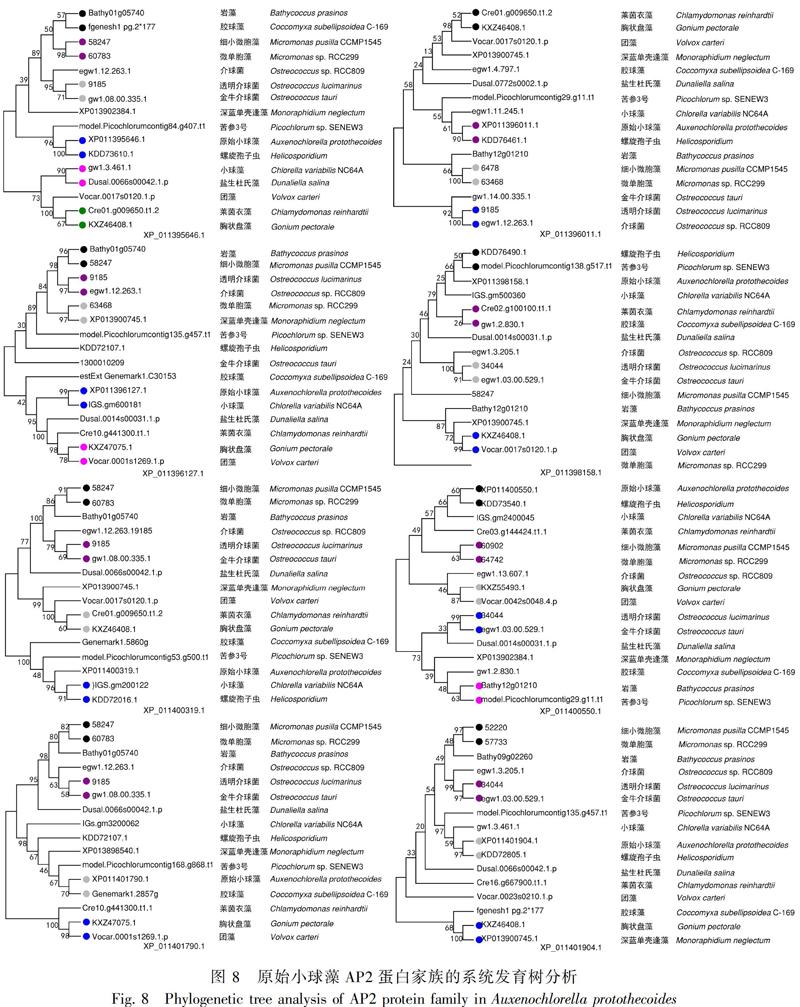
將原始小球藻的8個成員序列分別在plantTFDB在線程序中,與原始小球藻同為綠藻門下的其余15個物種AP2基因家族的蛋白成員進行同源序列的對比,找到相對應的其余物種的同源序列,并分別使用MEGA7.0軟件對8個蛋白成員序列組進行系統進化樹的構建,對比8個蛋白成員的同源序列的差距,判斷各個物種與原始小球藻之間的親緣關系。在XP_011396127.1和XP_011396011.1兩個進化樹中親緣關系最遠的是金牛介球菌,在XP_011395646.1進化樹中最遠的是團藻,在XP_011400319.1進化樹中,最遠的是鹽生杜氏藻,在XP_011401790.1進化樹中,最遠的是萊茵衣藻,在XP_011401904.1進化樹中,最遠的是膠球藻,在XP_011400550.1進化樹中,最遠的是深藍單殼縫藻,在XP_011398158.1進化樹中,最遠的是微單胞藻,根據此推斷,16個物種中與原始小球藻親緣關系最遠的是金牛介球菌;在XP_011395646.1、XP_011396011.1、XP_011400550.1和XP_011401904.1這4個進化樹中都是螺旋孢子蟲與原始小球藻的親緣關系最近,在XP_011396127.1和XP_011398158.1進化樹中,親緣關系最近的是小球藻, 在XP_011401790.1進化樹中, 親緣關系最近的是膠球藻,在XP_011400319.1進化樹中親緣關系最近的是苦參3號。在所有的進化樹中,小球藻與原始小球藻親緣關系在同一大分支中一共有7次,苦參3號有6次,螺旋孢子蟲有7次,膠球藻有3次,鹽生杜氏藻有1次,萊茵衣藻有2次,深藍單殼縫藻、胸狀盤藻和團藻各有1次,根據此推斷,16個物種中與原始小球藻親緣關系最接近的是小球藻和螺旋孢子蟲,較接近的是苦參3號 (圖8) 。
3?討論
通過對原始小球藻AP2基因蛋白成員進行詳細的生物信息學分析后,可較詳細地了解原始小球藻AP2基因蛋白成員的各種特性。原始小球藻中含有8個家族成員,而在擬南芥(Arabidopsis)中為18個(唐美芳,2007),水稻(Oryza sativa)中為26個(施楊等,2013),玉米中為31個(莫曉婷等,2013)。可見,不同物種的AP2家族蛋白成員數量是不一樣的。原始小球藻屬于植物界、小球藻科、真核生物(吳麗娟等,2016),在系統進化中最為原始,該蛋白家族成員含有247到715個氨基酸,在其AP2轉錄因子蛋白成員中,含有多個保守結構域,但是不存在信號肽和跨膜結構域,且AP2蛋白的親和性平均值均小于0,故其為非分泌、非跨膜、親水性型蛋白。在原始小球藻的理化性質分析中,AP2家族成員共編碼3 348個氨基酸,其中同時存在堿性氨基酸和酸性氨基酸,不穩定系數>40,為不穩定蛋白,而在雷公藤(Tripterygium wilfordii)(祝傳書等,2018)、茶樹(Camellia sinensis)(吳致君等,2014)和棗(Ziziphus jujube)(紀晴等,2018)中的編碼氨基酸同樣含有堿性氨基酸和酸性氨基酸、為不穩定蛋白,但茶樹含有的堿性氨基酸略多于酸性氨基酸,且蛋白質殘基為210~290;而棗的酸性氨基酸多于堿性氨基酸,這與本文研究結果相似。在保守結構域的分析中,不同物種的個數及位置不盡相同:原始小球藻和棗(Ziziphus jujube)(紀晴等,2018)的AP2成員中同樣存在3個保守結構域,其中原始小球藻的氨基酸數量跨度最大,達到75,但野生大豆(Glycine soja)(朱延明等,2019)的保守結構域個數只存在兩個,其氨基酸數量跨度較大,達到104(209~313)。在親疏水性分析中,原始小球藻AP2成員蛋白與菠蘿 AcPEPC 蛋白(馬海洋等,2020)和蔓花生PEPC基因家族(涂嘉琦等,2018)相同,所有蛋白成員均為親水性蛋白,磷酸化位點分析中,原始小球藻AP2蛋白家族成員含有多個潛在磷酸化位點,最有可能的磷酸化位點為絲氨酸,在蓖麻(Ricinus communis)APs分析中潛在磷酸化位點同樣為絲氨酸。在原始小球藻的二級結構分析中,其二級結構的組分含量從大到小排序均為無規則卷曲>α-螺旋>延伸鏈>β-轉角,α-螺旋和無規則卷曲是二級結構的主要方式,延伸鏈和β-轉角則分散在所有蛋白成員的氨基酸鏈中,在中國櫻桃(Chinese cherry)(王月,2017)和甘薯(Dioscorea esculenta)(阮先樂等,2017)中的二級結構占比分布和原始小球藻相同,α-螺旋和無規則卷曲占比最大;但在楊樹(Pterocarya stenoptera)(趙金玲等,2015)中,其組分與原始小球藻有所差異,僅以無規則卷曲為主。在三級結構分析中,甘藍與原始小球藻都能明顯觀察到α-螺旋和β-折疊,但原始小球藻三級結構家族成員之間結構差異較大,而甘藍(Brassica oleracea)(郭慧等,2017)和甘薯(Dioscorea esculenta)(阮先樂等,2017)的家族成員三級結構基本相似。在原始小球藻保守基序中,將所有基序一共分為四類,其中所有成員共同含有的是motif3、motif5,這兩個可能是參與原始小球藻細胞生長發育和逆境脅迫等多個生理過程有關的功能基序(栗麗和佟少明,2016)。在原始小球藻AP2蛋白家族中不存在信號肽和跨膜結構域,同樣在沙棘(Hippophae rhamnoides)WRI1轉錄因子(馬倩等,2016)蛋白序列和薔薇科植物的19個DELLA蛋白(宋偉等,2013)序列中也均無信號肽和跨膜結構域,但同時也存在不同,例如:在花生profilin蛋白(肖杰等,2011)序列中,信號肽位于第1~24位蛋白,卻沒有跨膜結構域;在大豆GmANKTM家族(柏錫等,2019)蛋白中存在跨膜結構域;在小麥(Triticum aestivum)F-box蛋白(王俊生等,2013)基因序列中,第94至第115位存在一個可能的跨膜區,且在該蛋白質N端序列不存在信號肽。
目前在植物界對AP2轉錄因子的研究主要在單雙子葉植物上,如:在擬南芥(Arabidopsis)(Jofuku et al.,1994)中發現124個AP2/ERF家族基因;水稻(Oryza sativa)(Nakano, 2006)中發現了139個AP2/ERF家族基因;大豆(Glycine max)中發現了120個AP2/ERF家族基因(Zhang et al., 2008);小蘭嶼蝴蝶蘭(Phalaenopsis equestris)中發現了107個AP2/ERF家族基因等(朱光哲,2016);而在藻類上AP2轉錄因子的研究較少。本研究主要描述的是對原始小球藻AP2所有蛋白成員進行了詳細的生物信息學分析,系統進化分析結果說明了原始小球藻最為原始,與原始小球藻親緣關系最遠的是細小微胞藻ccmp1545 (Micromonas pusilla CCMP1545),親緣關系最近的是團藻(Volvox carteri)。本研究結果為今后進一步研究AP2轉錄因子在原始小球藻生長發育過程中的生理特點和分子功能提供了一定的參考依據。
參考文獻:
BO X, WANG XY, CHEN Y, et al., 2019.Bioinformatics analysis of GmANKTM family abiotic stress in soybean[J].J NE Agric Univ, 50(7):18-27.[柏錫, 王昕奕, 陳云, 等, 2019.大豆GmANKTM家族非生物脅迫生物信息學分析[J].東北農業大學學報, 50(7):18-27.]
CAO C, MA K, 2016.Protein secondary structure assignment[J].Chin J Bioinform, (14):187.[曹晨, 馬堃, 2016.蛋白質二級結構指定[J].生物信息學, (14):187.]
GUI XH, 2017.Culture conditions for oil accumulation and regulation of lipid synthetic metabolism of Chlorella protothecoides[D].Wuhan:Huazhong University of Science and Technology.[桂小華, 2017.原始小球藻產油條件及油脂合成代謝調控的研究[D].武漢:華中科技大學.]
GUO H, JIN SY, LIU H, et al., 2017.In silico cloning and bioinformatics analysis of AP2/ERF gene family from Brassica oleracea[J].Chin Pharm, 20(1):6-10.[郭慧, 金司陽, 劉寒, 等, 2017.甘藍AP2/ERF轉錄因子的克隆和生物信息學分析[J].中國藥師, 20(1):6-10.]
HAN CZ, WANG J, 2016.Bioinformatics analysis of typeⅥ secreted protein in Xanthomonas campestris pv.raphani 756C[J].J Huazhong Agric Univ, 35(4):42-48.[韓長志, 王娟, 2016.黃單胞菌Xanthomonas campestris pv.raphani 756C中Ⅵ型分泌蛋白的生物信息學分析[J].華中農業大學學報, 35(4):42-48.]
JI Q, ZHOU F, ZHOU J, et al., 2018.Whole genome identification and bioinformatics analysis of AP2/EREBP transcription factors of Ziziphus jujube[J].Genom Appl Biol, 37(7):2983-2997.[紀晴, 周凡, 周軍, 等, 2018.棗AP2/EREBP轉錄因子的全基因組鑒定及生物信息學分析[J].基因組學與應用生物學, 37(7):2983-2997.]
JOFUKU KD, DEN BOER BG, VAN MONTAGU M, et al., 1994.Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J].Plant Cell, 6(9):1211-1225.
LI JF, GUO MZ, LIU Y, et al., 2008.An evolutionary tree reconstruction method combining QuartetPuzzling and neighbor joining[J].J Comput Res Dev, 45(11):1965-1973.[李建伏, 郭茂祖, 劉揚, 等, 2008.一種基于QuartetPuzzling和鄰接法的進化樹構建算法[J].計算機研究與發展, 45(11):1965-1973.]
LIU Y, 2009.Evolutionary dynamics of flower development regulatory network[D].Beijing:University of Chinese Academy of Sciences, University of Chinese Academy of Sciences.[劉楊, 2009.花發育調控網絡的進化動態研究[D].北京:中國科學院研究生院,中國科學院大學.]
LIU Y, XIE LY, LAI ZX, et al., 2017.Cloning and bioinformatics analysis of amaAG in Amaranthus tricolor L.[J].Acta Agric Univ Jiangxi, 39(1):168-174.[柳燕, 謝禮洋, 賴鐘雄, 等, 2017.莧菜amaAG基因克隆與生物信息學分析[J].江西農業大學學報, 39(1):168-174.]
MA HY, ZHAO QF, CHEN S, et al., 2020.Bioinformatics analysis of PEPC gene family in pineapple[J].Chin J Trop Crop, 41(1):97-103.[馬海洋, 趙秋芳, 陳曙, 等, 2020.菠蘿PEPC基因家族生物信息學分析[J].熱帶作物學報, 41(1):97-103.]
MA Q, LI JB, RUAN CJ, et al., 2016.Bioinformatics analysis of WRI1 gene in Hippophae rhamnoides[J].Hubei Agric Sci, 55(22).[馬倩, 李景濱, 阮成江, 等, 2016.沙棘WRI1轉錄因子基因的生物信息學分析[J].湖北農業科學, 55(22).]
MO XT, ZHAO J, FAN YL, et al., 2013.Research progress on structure and function of maize transcription factors[J].J Agric Sci Technol Chin, 15(3):7-17.[莫曉婷, 趙軍, 范云六, 等, 2013.玉米轉錄因子結構與功能研究進展[J].中國農業科技導報, 15(3):7-17.]
NAKANO T, 2006.Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J].Plant Physiol, 140(2):411-432.
RICCARDO V, ANDREY Z, JASON A, et al., 2010.The genome of the domesticated apple (Malus domestica Borkh.)[J].Nat Genet, 42(10):833-839.
RUAN XL, ZHANG J, ZHANG FL, et al., 2017.The bioinformatics analysis of AP2 gene family in sweet potato[J].Mol Plant Breed, 15(6):2066-2072.[阮先樂, 張杰, 張福麗, 等, 2017.甘薯AP2基因家族的生物信息學分析[J].分子植物育種, 15(6):2066-2072.]
SCHWEDE T, KOPP J, GUEX N, et al., 2003.SWISS-MODEL:An automated protein homology-modeling server[J].Nucl Acid Res, 31(13):3381-3385.
SHI Y, XU X, LI HY, et al., 2013.Bioinformatics analysis of the expansin gene family in rice[J].Hereditas, 36(8):809-820.[施楊, 徐筱, 李昊陽, 等, 2013.水稻擴展蛋白家族的生物信息學分析[J].遺傳, 36(8):809-820.]
SONG W, LI DL, WANG R, et al., 2013.Bioinformatics analysis of DELLA proteins in Rosaceous plants[J].Chin Agric Sci Bull, 29(19):142-148.[宋偉, 李鼎立, 王然, 等, 2013.薔薇科植物DELLA蛋白的生物信息學分析[J].中國農學通報, 29(19):142-148.]
SU L, TONG SM, 2016.Sequence analysis and translation of AP2/ERF transcription factor gene family in Vitis vinifera[J].Biotechnol World, (4):18-19.[栗麗, 佟少明, 2016.葡萄(Vitis vinifera)AP2/ERF轉錄因子基因家族序列分析[J].生物技術世界, (4):18-19.]
TAMURAK, PETERSON D, PETERSON N, et al., 2011.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J].Mol Biol Evol, 28(10):2731-2739.
TU JQ, GAN L, FENG LL, et al., 2018.Bioinformatics analysis of PEPC gene family in Arachis duranensis[J].J Trop Subtrop Bot, 26(2):107-115.[涂嘉琦, 甘璐, 馮蘭蘭, 等, 2018.蔓花生PEPC基因家族的生物信息學分析[J].熱帶亞熱帶植物學報, 26(2):107-115.]
WANG GH, GUI XH, DENG W, et al., 2013.Effect of “heterotrophic-stress”segmental culture on biomass and lipid content of Chlorella protothecoides[J].Chin Biotechnol, 33(3):99-104.[汪桂林, 桂小華, 鄧偉, 等, 2013.“異養-脅迫”分段培養對原始小球藻生物量和油脂含量影響研究[J].中國生物工程雜志, 33(3):99-104.]
WANG GL, 2013.Two-step cultivation mode for ?Chlorella protothecoides and extraction of high-valued product[D].Wuhan:Huazhong University of Science and Technology.[汪桂林, 2013.原始小球藻分段培養條件優化及高附加值產物提取工藝研究[D].武漢:華中科技大學.]
WANG JS, LI LL, YANG H, et al., 2013.Electronic clone and bioinformatics analysis of F-box protein gene from wheat(Triticum aestivum L.)[J].J Henan Agric Sci,?42(2):15-19.[王俊生, 李俐俐, 楊歡, 等, 2013.小麥F-box蛋白基因的電子克隆和生物信息學分析[J].河南農業科學, 42(2):15-19.]
WANG LL, 2016.Genome-wide investigation of AP2 subfamily in Brassica napus L.and WRI1 in plants[D].Yangling:Northwest A & F University.[王莉莉, 2016.甘藍型油菜AP2亞家族轉錄因子及植物中WRI1的基因組學分析[D].楊凌:西北農林科技大學.]
WANG S, LI Y, LI MJ, et al., 2019.Cloning and bioinformatics analysis of APs gene in Ricinus communis[J].Mol Plant Breed, 17(22):7344-7349.[王雙, 李躍, 李孟建, 等, 2019.蓖麻APs基因克隆及生物信息學分析[J].分子植物育種, 17(22):7344-7349.]
WANG Y, 2017.Study on expression and function of AP2/ERF transcription factor in flower bud of chinese cherry during dormancy release[D].Jinhua:Zhejiang Normal University.[王月, 2017.中國櫻桃AP2/ERF轉錄因子在花芽休眠解除過程的表達與作用研究[D].金華:浙江師范大學.]
WEN KX, LIU XM, 2010.The important role of AP2 functional genes in plant floral development[J].Biotechnol Bull, (2):1-7.[聞可心, 劉雪梅, 2010.AP2功能基因在植物花發育中的重要作用[J].生物技術通報, (2):1-7.]
WU LJ, YU ZL, LIN XZ, et al., 2016.Advances and perspectives of genetic engineering in Chlorella[J].Bull Sci Technol, 32(12):51-56.[吳麗娟, 余志良, 林祥志, 等, 2016.小球藻基因工程的研究進展及應用前景展望[J].科技通報, 32(12) :51-56.]
WU ZJ, LU L, LI XH, et al., 2014.Isolation and expression profiles analysis of AP2/ERF-B3 group transcription factor from Camellia sinensis[J].J Nanjing Agric Univ, 37(4):67-75.[吳致君, 盧莉, 黎星輝, 等, 2014.茶樹AP2/ERF-B3類轉錄因子基因的克隆與表達特性分析[J].南京農業大學學報, 37(4):67-75.]
XIAO J, WU XY, WANG LL, et al., 2011.Bioinformatics of profilin protein from peanut[J].Immunol J, 27(2):158-161.[肖杰, 吳序櫟, 王琳琳, 等, 2011.花生profilin蛋白的生物信息學分析[J].免疫學雜志, 27(2):158-161.]
XIA QY, 2007.Cloning and expression of venom allergen soli1 and soli4 genes in the Solenopsis invicta[D].Chongqing:Southwest University.[夏巧玉, 2007.紅火蟻毒素致敏原soli1、soli4基因的克隆及表達[D].重慶:西南大學.]
XU ZJ, LIU Y, XU L, et al., 2019.Bioinformatics analysis of NF-YB gene family of transcription factor in Zea mays[J].Mol Plant Breed,17(12):3807-3816.[徐志軍, 劉洋, 徐磊, 等, 2019.玉米轉錄因子NF-YB基因家族的生物信息學分析[J].分子植物育種, 17(12):3807-3816.]
YANG L, 2015.Clone and functional analysis of carbonic anhydrase gene of tobacco[D].Chengdu:Sichuan Agricultural University.[楊朗, 2015.煙草碳酸酐酶基因的克隆及其生物信息學分析[D].成都:四川農業大學.]
ZHANG G, CHEN M, CHEN X, et al., 2008.Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean (Glycine max L.)[J].J Exp Bot, 59(15):4095-4107.
ZHANG HY, LI ZT, ZHAO CJ, et al., 2013.Genome-wide analysis of LIM domain-containing protein gene family in maize[J].J Maize Sci, (3):40-47.[張海燕, 李佐同, 趙長江, 等, 2013.玉米LIM結構域蛋白基因家族分析[J].玉米科學, (3):40-47.]
ZHANG JY, WANG QJ, GUO ZR, 2012.Progresses on plant AP2/ERF transcription factors[J].Hereditas, 34(7):835-847.[張計育, 王慶菊, 郭忠仁, 2012.植物AP2/ERF類轉錄因子研究進展[J].遺傳, 34(7):835-847.]
ZHANG Q, CHENG J, LI L, et al., 2018.Research progress on plant AP2/ERF transcription factor family[J].Biotechnol Bull, 34(8):1-7.[張麒, 陳靜, 李俐, 等, 2018.植物AP2/ERF轉錄因子家族的研究進展[J].生物技術通報, 34(8):1-7.]
ZHANG Y, SUN FB, 2011.Function of AP2 in plant flower development[J].Chin Sci Technol Overview, (23):436.[張妍, 孫豐賓, 2011.AP2在植物花發育中的功能簡述[J].中國科技縱橫, (23):436.]
ZHAO JL, YAO WJ, WANG SJ, et al., 2015.AP2/ERF gene family in Populus trichocarpa by bioinformatics[J].J NE For Univ, 43(10):21-29.[趙金玲, 姚文靜, 王升級, 等, 2015.楊樹AP2/ERF轉錄因子家族生物信息學分析[J].東北林業大學學報, 43(10):21-29.]
ZHU CS, LIU Y, CHENG MM, et al., 2018.Cloning and expression analysis of two AP2/ERF transcription factors in Tripterygium wilfordii Hook.f[J].Acta Bot Boreal-Occident Sin, 38(8):43-50.[祝傳書, 劉艷, 陳蒙蒙, 等, 2018.雷公藤AP2/ERF轉錄因子基因的克隆與分析[J].西北植物學報, 38(8):43-50.]
ZHU GZ, 2016.Bioinformatics analysis of AP2/ERF gene family in Phalaenopsis equestris[D].Beijing:Chinese Academy of Forestry.[朱光哲, 2016.小蘭嶼蝴蝶蘭AP2/ERF基因家族生物信息學分析[D].北京:中國林業科學研究院.]
ZHU YM, YU JY, YU Y, et al., 2019.GsRAV3 gene, encoding a member of the AP2/RAV family from Glycine soja, negatively regulates plant ABA sensitivity in Arabidopsis[J].J NE Agric Univ, 50(5):8-18.[朱延明, 于紀洋, 于洋, 等, 2019.野生大豆AP2/RAV亞家族轉錄因子GsRAV3負調控擬南芥對ABA的敏感性[J].東北農業大學學報, (5):8-18.]
(責任編輯?周翠鳴)

