CRISPR/Cas9系統中的脫靶效應及檢測技術研究進展
張晨 雷展 李凱 商穎 許文濤
(1. 昆明理工大學農業與食品學院,昆明 650504;2. 中國農業大學食品科學與營養工程學院 北京食品營養與人類健康高精尖創新中心,北京 100083;3. 農業部農業轉基因生物安全評價(食用)重點實驗室,北京 100083)
CRISPR/Cas9系統是細菌和古生菌免疫病毒或噬菌體侵害的一種獲得性免疫機制,其中sgRNA可特異性識別外源遺傳物質,Cas9蛋白可靶向切割雙鏈DNA。因為組成簡單、特異性好、切割效率高,CRISPR/Cas9系統經改造后成為新一代基因編輯工具并迅速被廣泛應用。在實際應用過程中,CRISPR/Cas9系統優勢凸顯,其快速、準確的基因編輯能力,使通過基因定點突變治療人類遺傳疾病成為可能。然而在基因編輯過程中存在的脫靶效應,嚴重阻礙了CRISPR/Cas9系統的發展。因此,為明確CRISPR/Cas9系統產生的脫靶效應及提高基因編輯效率,本文綜述了CRISPR/Cas9系統的脫靶類型、影響因素、降低策略以及脫靶檢測技術的最新研究進展。
1 CRISPR/Cas9系統簡述
1987年,在研究大腸桿菌堿性磷酸酶(iap)基因功能時,首次發現了串聯間隔重復序列[1]。2002年,這種結構被命名為聚類規則間隔的短回文重復序列,臨近的一組基因被命名為Cas[2]。2007年,證實CRISPR及Cas編碼的蛋白與細菌的獲得性免疫機制有關[3]。2013年,首次利用CRISPR/Cas9系統實現對人和小鼠內源基因的高效編輯[4-5]。
在自然界中,約90%古生菌和40%細菌通過基因組或質粒上存在的CRISPR/Cas系統對外來病毒或噬菌體的侵害進行免疫。當病毒或噬菌體首次將遺傳物質注入細菌內部時,Cas蛋白會將外源遺傳物質上的一小段序列整合到CRISPR序列的5'端,形成對外源遺傳物質的“免疫記憶”。當細菌再次遭遇侵害時,含有外源遺傳物質信息的CRISPR序列轉錄形成sgRNA。sgRNA與Cas蛋白結合,從而特異性切割外源DNA。外源DNA被切斷后沉默,細菌對外來病毒或噬菌體“免疫成功”[6]。
CRISPR/Cas系統存在多種類型,依據其編碼的效應蛋白可以將其分為兩大類(圖1)。第1大類CRISPR/Cas系統的效應蛋白是由含有4-7個Cas蛋白組成的多亞基效應復合物組成。第2大類CRISPR/Cas系統中僅包含單一的多結構域效應蛋白。兩大類系統又被細分為6種類型,其中第1大類包含類型I,III和IV,第2大類包含類型II,V和VI。根據特征蛋白的不同,可區分類型I、II、III。其中Cas3蛋白代表類型I,Cas9蛋白代表類型II,Cas10蛋白代表類型III。類型IV缺乏由Cas1和Cas2蛋白組成的適應模塊;類型V具有預測效應蛋白Cas12a;類型VI是唯一具有毒素特性的靶向RNA的CRISPR/Cas系統[7]。因為效應復合物結構簡單,第2大類CRISPR/Cas系統被改造成為基因編輯工具。其中類型II的CRISPR/Cas9系統是最常見且廣泛應用于各個領域的基因編輯工具[8]。

圖1 CRISPR/Cas系統分類圖[8]
CRISPR/Cas9系統由Cas9蛋白和sgRNA兩部分組成。Cas9含有HNH和RuvC核酸酶結構域,其中HNH結構域切割互補DNA鏈,RuvC結構域負責切割非互補鏈。sgRNA由CRISPR RNA(crRNA)和反式激活RNA(tracrRNA)組成,可識別約20 nt的靶序列。在基因編輯時,Cas9首先與sgRNA形成復合體;然后由Cas9識別特定的前間隔序列鄰近基 序(Protospacer adjacent motif,PAM);sgRNA與靶序列特異性結合,Cas9切割雙鏈DNA,產生雙鏈DNA斷裂(Double-strand DNA breaks,DSB)。最后,通過細胞內部的非同源末端連接(Non-homologous end joining,NHEJ)或同源重組機制(Homologous recombination,HR)對斷裂的DNA引入插入缺失、修復或替換(圖 2)[9]。

圖2 CRISPR/Cas9作用機制及DNA自我修復過程圖[10]
第一代基因編輯技術因為涉及到HR,效率低下且耗時費力。第二代基因編輯技術ZFN和TALEN的出現,基因編輯效率大幅提高。ZFN和TALEN均是合成蛋白,對靶序列切割后可產生黏性突出端DSB。ZFN和TALEN大大加快了基因編輯技術的發展,但在應用過程中,其復雜的蛋白設計、昂貴的成本和較高的難度,仍使基因編輯技術的廣泛應用受限[11]。
在此背景下,第三代基因編輯技術CRISPR/Cas9系統應運而生。同ZFN和TALEN相比,CRISPR/Cas9系統不需要設計復雜的DNA結合蛋白以及DNA結合蛋白與Fok I核酸酶的融合過程。通過軟件就可快速設計sgRNA,并對其進行初步篩選。同時,通過改變sgRNA中的一小段序列,CRISPR/Cas9系統可快速實現對其他基因位點的編輯。由于過程簡單,成本低廉,規模化,高通量,編輯效率高,實驗要求低等特點,CRISPR/Cas9系統迅速被應用到廣泛的研究領域當中[11]。例如醫學領域,CRISPR/Cas9系統已經建立了多種細胞系及動物模型[12];動物科學領域,利用CRISPR/Cas9系統生產更廣泛的基因修改動物[13];植物科學領域,CRISPR/Cas9系統促進作物育種,加速作物改良,增強全球糧食安全等[14]。
2 CRISPR/Cas9系統的脫靶類型
CRISPR/Cas9系統中的脫靶突變主要是由于在復雜的基因組中sgRNA存在數量龐大的脫靶位點所造成的。此外,Cas9識別低頻率的“NAG”模式的PAM序列也會導致脫靶,但PAM序列對錯配容忍度較低[15]。現階段,根據連續堿基不同錯配、間隔堿基不同錯配以及PAM近端遠端不同錯配可以將CRISPR/Cas9系統的脫靶類型簡單分成3類。
對于連續堿基不同錯配,Fu等[16]基于定量人類細胞的增強型綠色熒光蛋白(Enhanced green fluorescent protein,EGFP) 破 壞 試 驗, 評 估 了CRISPR/Cas9系統中sgRNA與DNA界面內連續不同錯配對Cas9活性的影響。結果表明,對于單堿基錯配,sgRNA的5'末端錯配率大于3'末端。但是在sgRNA的3'末端,也會存在一些位置對錯配具有良好的耐受性,并且不同靶序列對sgRNA錯配敏感的特定位置是不同的。雙重錯配實驗顯示出類似于單堿基錯配的實驗結果,而引入3個或更多個錯配則會導致Cas9核酸酶活性顯著喪失。
對于間隔堿基不同錯配,同樣基于定量人類細胞的EGFP破壞試驗,Fu等[16]對不同位置間隔的雙重錯配進行了表征。不同位置間隔的雙重錯配實驗顯示出類似于單堿基錯配的實驗結果,即sgRNA的5'末端錯配率大于3'末端。
對于PAM近端遠端不同錯配,之前的研究表明,靶序列上與PAM序列緊鄰的8-12個堿基決定CRISPR/Cas9系統的切割準確性。這一區域也被形象的稱之為種子區域。單堿基錯配敏感的特定位置集中在靠近PAM序列的5-6堿基,且不同靶標對錯配敏感的位置不同。雙重或多個核苷酸錯配,在靠近PAM序列的5-6堿基區域同樣表現出了對錯配更高的敏感性,且連續錯配比間隔錯配更敏感。這表明靶序列中更靠近PAM序列的5-6個堿基才是決定特異性結合的關鍵因素。此外,對于單一或雙重錯配,即使在靠近PAM序列的5-6堿基區域,有時也會表現出對錯配的良好耐受性[17]。
總之,靶序列對sgRNA的3'末端錯配更敏感,且其特異性是復雜的和靶標依賴性的,同時在sgRNA的3'端發生單一或雙重錯配時,通常是良好耐受的。此外,并非所有5'端的sgRNA/DNA界面錯配都具有良好的耐受性。最近,針對CRISPR/Cas9系統的脫靶,Lin等[18]提出了一種新型脫靶類型“DNA凸起”和“RNA凸起”,如圖3。當脫靶位點較靶序列存在一個或多個額外的堿基(插入),sgRNA與該脫靶位點結合時,脫靶位點上多余的堿基就會形成“DNA凸起”。當脫靶位點較靶序列缺少一個或多個堿基(缺失),sgRNA與該脫靶位點結合時,sgRNA上多余的堿基就會形成“RNA凸起”。通過去除或添加sgRNA上所有可能位置上的單堿基,Lin等[18]模擬了潛在脫靶的單堿基插入或缺失的位點。結果顯示,Cas9在3個區域的靶位點耐受DNA凸起:距離PAM的第7個堿基,sgRNA的5'末端和3'末端;鄰近PAM序列的sgRNA凸起禁止切割。2-5 bp的DNA凸起實驗結果與單堿基DNA凸起的結果類似;大于2 bp的sgRNA凸起比同等大小的DNA凸起耐受性更好,長于5 bp的sgRNA凸起未發現切割活性。
3 脫靶影響因素
雖然CRISPR/Cas9系統已經在多個研究領域中取得了優異成果,但是在實際應用過程中依然存在潛在的脫靶風險。Fu等[16]基于人類細胞實驗表征了CRISPR/Cas9系統的脫靶效應,結果顯示CRISPR/Cas9系統的脫靶率高達66%。為降低CRISPR/Cas9系統的脫靶效應實現其更廣泛的應用,研究人員探究了影響脫靶效應的相關因素。
3.1 PAM序列
PAM序列是區分靶序列與其他DNA序列、位于靶序列3'端、高度保守的一小段序列,其長度一般為2-5 nt。PAM序列存在多種模式,如“NAG”、“NGA”等。不同模式PAM序列的CRISPR/Cas9系統切割效率不同。在CRISPR/Cas9系統中,源自化膿性鏈球菌的SpCas9蛋白PAM序列為典型的“NGG”模式。其基因切割效率最高,N代表任意一種核苷酸,如“AGG”、“CGG”等。在基因編輯過程中,Cas9首先識別PAM序列,待sgRNA與靶序列特異性結合時,才能完成對目的基因的切割。PAM序列是CRISPR/Cas9系統發揮作用的先決條件。Cas9識別錯誤或其他模式低頻PAM序列是產生脫靶的部分原因[19]。

圖3 (A)1 bp插入(DNA凸起)(B)1 bp缺失(RNA凸起)示意圖[18]
3.2 sgRNA
sgRNA由crRNA和tracrRNA組成,其中crRNA負責識別約20 bp的靶序列,tracrRNA能夠指導crRNA與靶序列特異性結合。sgRNA的結構和長度會對脫靶效應或切割效率產生一定影響。在Cas9與PAM序列結合完成后,sgRNA與靶序列特異性結合,Cas9對靶序列進行切割。sgRNA是CRISPR/Cas9系統發揮作用的重要條件。由于CRISPR/Cas9系統是源于細菌等對噬菌體侵害的免疫機制,為保證將外源DNA全部清除,sgRNA本身便具有一定容錯能力。研究表明,sgRNA自身可耐受1-5個堿基錯配[20]。sgRNA與靶序列的錯配是產生脫靶現象的最主要原因,前文所述的“DNA凸起”和“RNA凸起”一定程度上也可以認為是sgRNA與靶序列的錯配。
3.3 其他
除以上所列舉的影響脫靶效應的因素外,還存在一些其他干擾因素,如轉染的細胞類型、染色質的結構、基因組背景影響等。研究發現,CRISPR/Cas9系統在斑馬魚胚胎細胞中的突變是高效的,通過質粒轉染進行基因編輯,其靶向突變率可達86%[21]。但是在人K562細胞中,同樣通過質粒轉染,即使是在sgRNA出現錯配的情況下,CRISPR/Cas9系統仍具有較高活性[16]。這表明CRISPR/Cas9系統存在著一定的細胞特異性。由于DNA在細胞中是以染色體的狀態存在,靶序列以及PAM序列均被包埋在染色體內部,Cas9與染色體親和特性也在一定程度上影響CRISPR/Cas9系統特異性[19]。同時,不同的基因組背景也會對系統的特異性產生一定影響,但具體機制還未被揭示。
4 降低脫靶效應的策略
影響脫靶效應的相關因素被成功揭示之后,一系列減少脫靶效應的新技術被成功創立。目前,減少脫靶效應的方法主要是優化或改進sgRNA、改造Cas9蛋白以及應用SpCas9蛋白類似物等。
4.1 sgRNA合理設計及修飾
sgRNA與靶序列的特異性結合是CRISPR/Cas9系統基因編輯的最關鍵因素。不合理的sgRNA設計會造成特異性降低、脫靶率升高。sgRNA設計應遵從以下原則:(1)最小化sgRNA與其他序列的相似性,且sgRNA與非靶序列存在超過3個錯配;(2)至少2個錯配位于非靶序列的種子區域內;(3)錯配應是連續的或者間隔小于4 nt[22]。同時,實驗表明sgRNA中GC含量≤35%時,sgRNA具有良好的特異性[23]。
在合理設計sgRNA的基礎上,通過對sgRNA的修飾可進一步提高CRISPR/Cas9系統的特異性。Cho等[24]在sgRNA的5'端攜帶了兩個額外的G,結果意外發現系統的特異性大幅提高。Hsu等[25]在靶向人EMX1和PVALB基因時,對tracrRNA尾部進行不同程度的延長,發現sgRNA延長后的基因插入水平比未修飾的sgRNA高5倍。這表明tracrRNA尾部的延長,在一定程度上增強了CRISPR/Cas9系統穩定性。此外,5'端截短1-3 bp的sgRNA仍然顯示類似于全長sgRNA的切割活性[18]。同時,通過對sgRNA進行不同的化學修飾,例如嵌合sgRNA法,也能夠達到降低脫靶效應的效果[26]。
4.2 Cas9突變體
Cas9是CRISPR/Cas9系統中的重要組成部分,最廣泛使用的Cas9是源自化膿性鏈球菌的SpCas9。Cas9含有HNH及RuvC核酸酶結構域,使其中一個或將其全部失活,可分別獲得D10A Cas9(Cas9 D10A mutation,Cas9n)[27]以及喪失核酸酶活性的Cas9(Deactivated Cas9,dCas9)[28]。成對的 Cas9n及dCas9可以代替Cas9,且成倍的提高系統特異性。Cas9蛋白剪切DNA過程,如圖4。
Cas9與DNA的接觸影響Cas9/sgRNA與DNA結合的穩定性以及對錯配的耐受性。對SpCas9中負責與靶序列接觸的關鍵氨基酸殘基的改變,獲得了高保真變體“SpCas9-HF1”[30]。改變 SpCas9與非靶鏈結合的氨基酸獲得了增強型特異性SpCas9變體“eSpCas9”[31]。SpCas9-HF1、eSpCas9 均顯著降低了脫靶效應,且保留了穩健的靶向切割。通過對REC3結構域的突變,Chen等[32]設計了一種新型超精確SpCas9變體“HypaCas9”。HypaCas9具有更優異的特異性,同時具有更高的靶向活性。此外,最近還獲得了高保真度的“evoCas9”[33],擴展PAM兼容性的“xCas9”[34],可以同截短或延長的sgRNA組合使用的“Sniper-Cas9”[35]以及高特異的“Hifi Cas9”[36]。
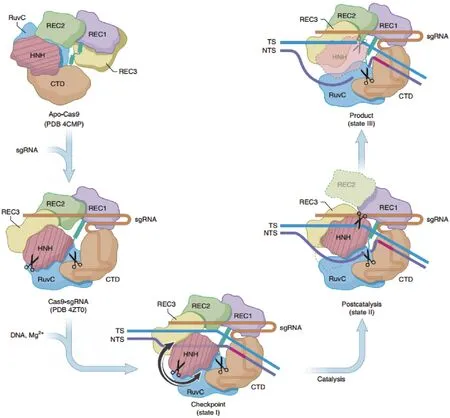
圖4 Cas9蛋白剪切DNA全過程的結構變化[29]
4.3 Cas9類似物
除了Cas9突變體,SpCas9類似物也提供了一種降低脫靶效應的新途徑。通過識別不同的、更復雜的PAM,SpCas9類似物獲得了更高的特異性。SpCas9類似物來源廣泛包括:金黃色葡萄球菌SaCas9[37]、 嗜 熱 鏈 球 菌 St1Cas9[38]、St3Cas9[39]、腦膜炎奈瑟菌 NmCas9[40]、弗朗西斯菌 FnCas9[41]、空腸彎曲桿菌CjCas9[42]以及Cas12a家族中的毛螺旋菌 LbCas12a[43]和氨基酸球菌 AsCas12a[44]。此外,最近還發現BhCas12b能夠表現出比SpCas9更高的特異性[45]。
雖然SpCas9類似物具有較高的特異性,但其較低的靶向性阻礙了其廣泛應用。在眾多類似物中,僅SaCas9、CjCas9存在與SpCas9相當的靶向性。SaCas9識別“NNGRRT”,可與20-24 nt sgRNA組合作用[46]。CjCas9識別“NNNNACAC”和“NNNNRYAC”,與GX22sgRNA共同作用時,顯示了最佳靶向活性[42]。通過設計突變體可以改善SpCas9類似物較低的靶向性。目前,通過對SaCas9進行突變,獲得的“SaCas9-KKH”可以提升靶向性2-4倍[47]。此外,還可以通過多種方法擴展SpCas9類似物的應用,如通過抗CRISPR蛋白與NmCas9的結合,可實現對NmCas9活性的調節[48];通過dCas9與FnCas9共同作用,可提升FnCas9的靶向活性等[49]。
4.4 Cas9/sgRNA濃度改變
酶的特異性和活性強度通常高度依賴于反應條件,高酶濃度下會弱化酶的特異性。Hsu等[25]在靶向人EMX1基因的實驗中,減少轉染細胞的質粒數(編碼Cas9蛋白和sgRNA),結果發現系統的特異性大大增強。這表明通過改變Cas9/sgRNA復合物的濃度實現最佳特異性的可行性。然而Cas9/sgRNA濃度的降低會導致CRISPR/Cas9系統切割效率的降低,所以需要通過實驗確定最佳Cas9/sgRNA濃度。在使用質粒轉染細胞時,通過減少細胞中sgRNA的數量,可直接限制細胞內Cas9/sgRNA復合物濃度。此外,在使用質粒轉染細胞時,采用不同啟動子影響sgRNA轉錄,也能夠降低細胞內Cas9/sgRNA濃度,減少脫靶[50]。
5 脫靶檢測技術
脫靶檢測技術是一系列針對CRISPR/Cas9系統作用機制研發的用于確定CRISPR/Cas9系統基因編輯準確性的檢測工具。脫靶檢測在揭示CRISPR/Cas9系統的脫靶機制以及進一步提高系統靶向性的研究中具有重要作用。
早期的脫靶檢測技術是由軟件預測和測序組成,如Sanger測序、NGS測序、全外顯子組測序等[51-52]。該類技術的原理是針對預測獲知的脫靶位點進行測序,以確定是否在這些位點發生了非特異性結合。Sanger測序法是檢測CRISPR/Cas9系統脫靶的常用方法之一。首先,通過Cas-OFFinder等[53]脫靶預測軟件進行預測,獲得可能的脫靶位點。然后,對預測的脫靶位點進行PCR擴增、測序,從而確定該位點是否發生脫靶突變。該類技術存在明顯的偏向性,其主要針對的是軟件預測的脫靶位點,而軟件預測往往容易造成部分脫靶位點的遺漏。
由易錯的NHEJ修復Cas9誘導的DSB時,往往會發生脫靶突變。檢測Cas9脫靶的最直接方法是跟蹤基因組中的DSB。通過對DSB的標記實現了全基因組無偏脫靶檢測,如IDLVs、BLESS、GUIDE-seq技術等[54-56]。這類技術的原理是通過將特定的雙鏈DNA或生物素接頭整合到DSB中,從而達到檢測目的。GUIDE-seq是無偏檢測脫靶效應的方法之一。首先,需要將特定的雙鏈寡核苷酸(Double stranded oligodeoxynucleotides,dsODN)整合到斷裂位點。然后提取基因組DNA,隨機打斷;對含有dsODN片段的序列進行擴增、富集。最后,測序后分析切割位點,評估脫靶效應[57]。該類技術雖然實現了全基因組無偏檢測,但是其只能檢測斷裂時期的DSB,對于已經修復或者未發生的DSB則不能檢測。
近年來,利用Cas9蛋白能夠在體外消化DNA的特性,使用發夾接頭或生物素等標記DNA片段,開 發 了 Digenome-seq、Circle-seq、SITE-seq等 技術[58-60]。該類技術的原理是利用Cas9體外核酸酶特性,在體外對基因組DNA進行切割,產物經處理后,通過測序或其他手段,實現對脫靶位點的篩選。Digenome-seq是利用Cas9體外消化DNA特性檢測脫靶效應的技術之一。Cas9體外消化基因組DNA、測序;比對具有相同末端的序列,通過軟件分析即可評估脫靶效率[61]。該類技術同樣從全基因組角度實現了無偏檢測,且精度較前兩類技術更高,唯一存在的問題是Cas9在體內和體外發揮作用時可能會存在一定的差異。
此外,脫靶檢測方法還有利用T7EⅠ酶、Surveyor酶等對錯配堿基切割的酶切法;利用dCas9與靶序列和非靶序列結合,結合測序手段的Chipseq技術以及基于染色體易位原理的HTGTS檢測法等。最近,針對各類脫靶檢測方法存在的問題,開發了一種普遍適用的無偏脫靶識別方法DISCOVERSeq。DISCOVER-Seq的優勢在于利用了DNA修復蛋白MRN復合物的亞基MRE11,MRE11與DNA的結合在插入缺失之前就可達到了峰值,結合Chipseq與定制軟件BLENDER,通過軟件得分便可實現對脫靶事件的高度特異性鑒定[62]。DISCOVER-Seq提供了一種定義和量化整個生物體中基因編輯脫靶效應的一般策略,從而為促進體內基因編輯療法的開發提供了藍圖。
6 總結與展望
作為主流基因編輯工具,CRISPR/Cas9的應用大大加快了分子生物學、植物學、生命科學等研究工作。在生物學中,CRISPR/Cas9用于轉錄調節、基因成像以及表觀遺傳學研究,這有助于進一步理解真核基因表達;在植物學中,CRISPR/Cas9加速了作物品種改良工作,縮短了作物育種年限,極大緩解了全球糧食危機;在醫學中,CRISPR/Cas9顯示出治愈困擾人類許久的疑難雜癥的極大潛力,已被廣泛用于細胞及動物模型的創建、藥物設計篩選等,這是CRISPR/Cas9邁向醫學領域的第一步。目前影響脫靶效應因素的研究集中在sgRNA與靶序列的結合以及Cas9對PAM序列的識別,其他諸如鹽離子濃度等對脫靶結合的影響還未被揭示。通過優化/改進sgRNA、突變Cas9和采用Cas9類似物,已成功減少非特異性結合的發生,但脫靶率的降低有時也會導致靶向切割效率的降低。如何在進一步降低脫靶效應的同時保持原有切割效率將成為新的研究重心。此外,伴隨精準測序技術的實現,更為精準的檢測技術也必將不斷推陳出新。隨著脫靶影響因素、降低策略及脫靶檢測技術研究的不斷深入,未來CRISPR/Cas9系統必將會應用在更廣泛的領域造福人類。

