絕對定量分析不同條銹病抗性小麥品種根際細菌群落結構
李菲 李欲軻 洪鯤 張宇斌 湯小辛

摘要:【目的】分析不同條銹病抗性小麥品種根際細菌群落的結構和功能差異,為開發防治小麥條銹病的環境友好方案提供理論基礎和創新思路。【方法】以同一地塊中對條銹病具有不同抗性的4個小麥品種(抗病品種貴農19和華麥1223,感病品種002和I19)為研究材料,采集不同抗性小麥品種根際土壤樣品,采用絕對定量擴增子分析技術,研究抗病和感病小麥品種根際細菌的群落結構和功能。【結果】同一地塊的4個小麥品種條銹病病情指數具有明顯差異。4個小麥品種根際土壤細菌群落結構絕對定量分析表明,感病與抗病小麥品種的根際細菌群落分別聚類成獨立的兩類。感病小麥品種根際細菌群落豐度和多樣性顯著高于抗病小麥品種根際細菌群落(P<0.05)。坐標分析(PCoA)表明,所有小麥品種根際細菌群落按照抗病性不同分為兩組;細菌群落功能分析表明,抗病小麥品種根際細菌群落具有更多的氨基酸和抗生素分泌功能。【結論】條銹病抗性對小麥根際細菌群落的影響超過小麥不同品種間遺傳變異的影響。通過分析和挖掘不同條銹病抗性小麥品種的根際細菌群落,可開發出環境友好的小麥條銹病生物防治菌劑。
關鍵詞: 小麥;條銹病;根際細菌群落;絕對定量
中圖分類號: S435.121.43? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2020)05-1115-07
Abstract:【Objective】The structural and functional differences of rhizosphere bacterial communities of wheat cultivars with different stripe rust resistance levels were analyzed, which provided theoretical basis and innovative ideas for the development of environmentally friendly ways for the control of wheat stripe rust. 【Method】Four wheat cultivars(resistant cultivars Guinong 19 and Huamai 1223, susceptible cultivars 002 and I19) with different resistance levels to stripe rust in the same plot were collected. Rhizosphere soils samples were collected, and absolute quantitative amplicon analysis techniques was used to study the differences in rhizosphere bacterial community structure and function of resistant cultivars and susceptible cultivars. 【Result】There was obvious difference in stripe rust disease index among the four wheat cultivars in the same plot. The absolute quantitative analysis on rhizosphere bacterial community structure of the four wheat cultivars indicated that the rhizosphere bacterial community of resistant cultivars and susceptible cultivars clustered into two groups. Compared with the resistant varieties, the abundance and diversity of rhizosphere bacterial communities of stripe rust susceptible wheat were significantly higher(P<0.05). Coordinate analysis(PCoA) analysis showed that the wheat cultivars could be divided into two groups according to the resistance. Functional analysis showed that the rhizosphere bacterial community of resistant wheat cultivars had more amino acids and antibiotic secretion functions. 【Conclusion】The effect of stripe rust resistance on the rhizosphere bacterial community of wheat cultivars exceed the genetic varia-tion among different wheat cultivars. Through analyzing rhizosphere bacterial community of wheat cultivars with different resistances can develop environmentally friendly biological control agent for wheat stripe rust.
Key words: wheat; stripe rust; rhizosphere bacterial community; absolute quantification
Foundation item: National Natural Science Foundation of China(U1812401)
0 引言
【研究意義】小麥(Triticum spp.)是世界上種植面積最大的糧食作物,2018年種植面積約2.2億ha,產量約7.6億t,全球產量在玉米之后,排名第二(United States Department of Agriculture-Foreign Agricultural Service,2019)。小麥條銹病是影響小麥產量的重要因素,病原菌是擔子菌門條形柄繡菌(Puccinia striiformis f. sp. tritici條形柄銹菌小麥專化型,簡稱Pst)。Pst是專性活體營養的真菌,生長在寄主細胞間隙中,通過吸器從小麥細胞吸取養分。目前防治小麥條銹病主要依靠化學藥劑和種植抗病品種。抗條銹病小麥品種由于對條銹菌具有小種的專化抗性,如過度集中使用,即長期大面積種植單一抗性品種,常會促使條銹病菌生理小種產生定向變異,進而產生新的毒力小種并克服品種的抗性,最后導致新的毒性小種大面積暴發流行,造成小麥產量損失,加大農業生產的風險。因此,開發環境友好的高效小麥條銹病控制方案,是小麥生產可持續發展的迫切需求。【前人研究進展】植物根系吸收水分和無機養分,并分泌各種有機質到土壤中(Hacquard et al.,2015)。植物根際的每克土壤中約含有數十億個微生物,形成數萬個不同的種類,因此,植物根際土壤被認為是一個高度復雜和動態的生態系統(Bulgarelli et al.,2013;Mendes et al.,2013;Phili-ppot et al.,2013)。目前已知植物共生微生物在植物應對病菌侵染中具有重要作用(Berg et al.,2016;Castrillo et al.,2017)。有學者從玉米(Planchamp et al.,2014)等農作物的根際分離篩選出根際菌株,并證明部分菌株可用于防治農作物病害。番茄品系對青枯病菌的不同抗性,部分原因是根際微生物不同所致(Kwak et al.,2018)。研究表明,植物根際促生細菌(Plant growth promoting rhizobacteria,PGPR)能抑制植物根際有害微生物生長(Hu et al.,2018)。此外,PGPR還能誘導植物產生系統抗性,從而提高宿主植物整體的抗病能力(Pieterse et al.,2014)。目前常規16S擴增子測序在分析樣本微生物組成時僅考慮微生物的相對豐度(比例),但微生物的相對豐度只表征了一個樣本中微生物類群的相對比例。以往大多數研究用相對豐度來進行跨樣本微生物豐度的比較,當樣本間總微生物絕對含量存在差異時,可能會得出與事實相反的結論,所以通過一定手段獲得微生物絕對豐度信息能為反映微生物群落結構及其動態變化提供更多有價值的參考信息(Smets et al.,2016)。Tkacz等(2018)比較了多個環境下相對定量和絕對定量擴增子分析的結果,發現會存在某一微生物種類絕對數量上升,而相對數量下降的情況。說明只有絕對定量分析才能反映樣本每種微生物的真實數量變化和各組間樣本的真實差異,絕對定量分析是微生態研究的首選方法(Smets et al.,2016;Tkacz et al.,2018)。【本研究切入點】根際微生物對宿主植株抗病能力的影響已有較多研究,但根際微生物對小麥條銹病抗性的影響目前尚無文獻報道。【擬解決的關鍵問題】采用絕對定量擴增子測序的方法,對在同一地塊中對條銹病表現不同抗性的小麥品系的根際微生物進行比較分析,研究小麥、條銹病菌和根際微生物群落之間的相互作用,為開發小麥條銹病防治的環境友好方案提供理論基礎和創新思路。
1 材料與方法
1. 1 試驗材料
田間試驗于國家小麥育種改良中心花溪實驗站進行。該實驗站位于我國西南部貴州省貴陽市花溪區,屬亞熱帶季風氣候,四季分明,冬無嚴寒、夏無酷暑,最冷月1月氣溫4~10 ℃,最熱月7月氣溫25~28 ℃;雨量充沛,濕度較大,年均降水量1100~1300 mm。小麥條銹病菌在該地區過冬,為翌年在全國流行提供菌源。
本研究選擇同一地塊中對條銹病表現出不同抗性的4個小麥品種,感病和抗病品種各2個,分別為抗病品種貴農19和華麥1223,感病品種002和I19。小麥對條銹病抗感的病情指數使用0~9分評分(Line and Qayoum,1992):高抗[R,0~3分,葉片無銹斑(0分)或很少(3分),無孢子堆(0分)或孢子堆很小(3分)]、中抗(MR,4~5分)、中感(MS,6分)和易感(S,7~9分,9分是指葉片孢子堆大而多,葉片黃色)。灌漿期采集4個小麥品種的根際土壤,每個小麥品種設3個生物學重復,共12個土壤樣品。每個樣品包括10~12株長勢基本一致的小麥根際土壤。盡可能完整地挖出小麥植株的全部根系,輕柔晃動,把浮土甩掉,甩不掉的土壤認定為小麥的根際土壤,立即將樣品帶回實驗室,使用滅菌200目篩對根際土壤進行篩慮。剔除篩后粉末中的細根和石子等,將所得土壤粉末保存于-80 ℃冰箱,待用。
1. 2 試驗方法
將采集的根際土壤樣品送至上海天昊生物技術公司進行絕對定量16S rRNA Miseq的擴增子測序。按照PowerSoil DNA試劑盒說明(MoBio,Carlsbad,USA)提取總基因組DNA。將具有至少4種不同濃度(內標的103、104、105和106個拷貝)的9種不同加標序列添加到樣品DNA庫中。加標序列由選擇的天然16S rRNA基因和人工可變區相同的保守區組成,然后通過擴增細菌16S rRNA基因的V4~V5區域產生細菌擴增子文庫。
1. 3 統計分析
使用TrimGalore 0.5.0(Babraham Bioinformatics,UK)和Mothur1.25.1(https://www.mothur.org/)從raw reads中去除接頭和引物序列。僅使用平均質量分數大于20且無模糊堿基的長度大于200 bp的序列進行隨后的分析。這些序列聚類成操作分類單位(OTU),序列相似性為97%,然后濾出加標序列并對OTUs進行計量。使用Smets等(2016)和Tkacz等(2018)描述的R-3.2.2進行根際微生物絕對定量分析。使用Mothur1.25.1classify.seqs命令和RDP數據庫進行OTUs分類(Cole et al.,2014)。熱圖、坐標分析圖(PCoA)和平均鏈接層次聚類由R-3.2.2執行。
使用PICRUSt軟件將條銹病抗病小麥和感病小麥根際土壤細菌群落的基因家族(功能)豐度表(KEGG)進一步匯總到更高水平的功能分類上。使用Welchs T-test對兩個分組樣本細菌群落KEGG功能條目的豐度進行差異比較,找出兩組中具有顯著差異的KEGG功能通路。
2 結果與分析
2. 1 小麥品種田間條銹病抗性調查結果
4個小麥品種均種植在國家小麥育種改良中心花溪實驗站,數十年來,該地塊種植的小麥均會發生條銹病。田間調查發現,該地塊各小麥品種條銹病病情指數具有明顯差異(圖1)。其中貴農19和華麥1223表現出極強的抗條銹病能力,田間病情指數分值分別為2和1分;002和I19兩個品種則嚴重感病,田間病情指數分值分別為8和9分。采集4個小麥品種的根際土壤進行絕對定量測序分析。
2. 2 小麥根際土壤細菌群落擴增子測序結果
去除模糊、短、低質量的reads和單獨的OTU后,共有3688572個reads用于12個土壤樣品的細菌群落分析。每個樣本生成的OTU數量為3838~4735,并由RDP數據庫進一步分類。基于內部加標標準及其衍生的標準曲線評估16S-seq數據的絕對量(如,R1樣品的標準曲線公式為y=1.0342x-1.5416,其中,y表示以對數計的加入加標序列的拷貝數,x表示以對數觀察到的OUT讀數;所有擬合系數R2>0.99)。
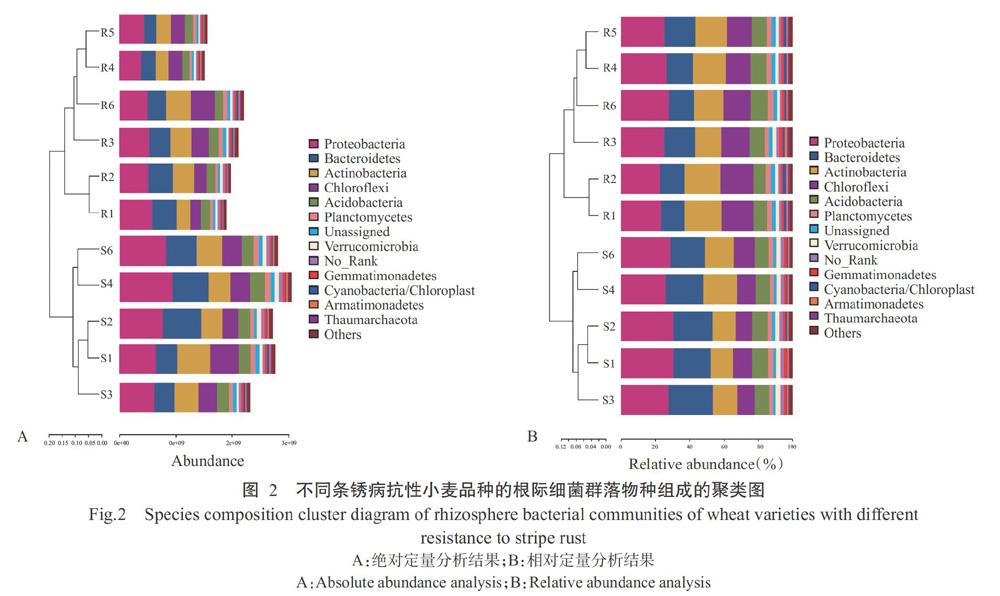
微生物的相對豐度只表征一個樣本中微生物類群的相對比例。與相對定量分析相比,絕對定量分析確定了每個微生物類群的絕對量,有利于不同樣本間的比較。條銹病感病和抗病小麥品種根際土壤樣本細菌群落相對和絕對定量聚類結果見圖2。相對定量分析數據顯示,小麥各品種間聚類較雜亂,感病小麥品種與抗病小麥品種未聚類在一起;絕對定量分析數據顯示,感病小麥品種與抗病小麥品種分成獨立的兩類。
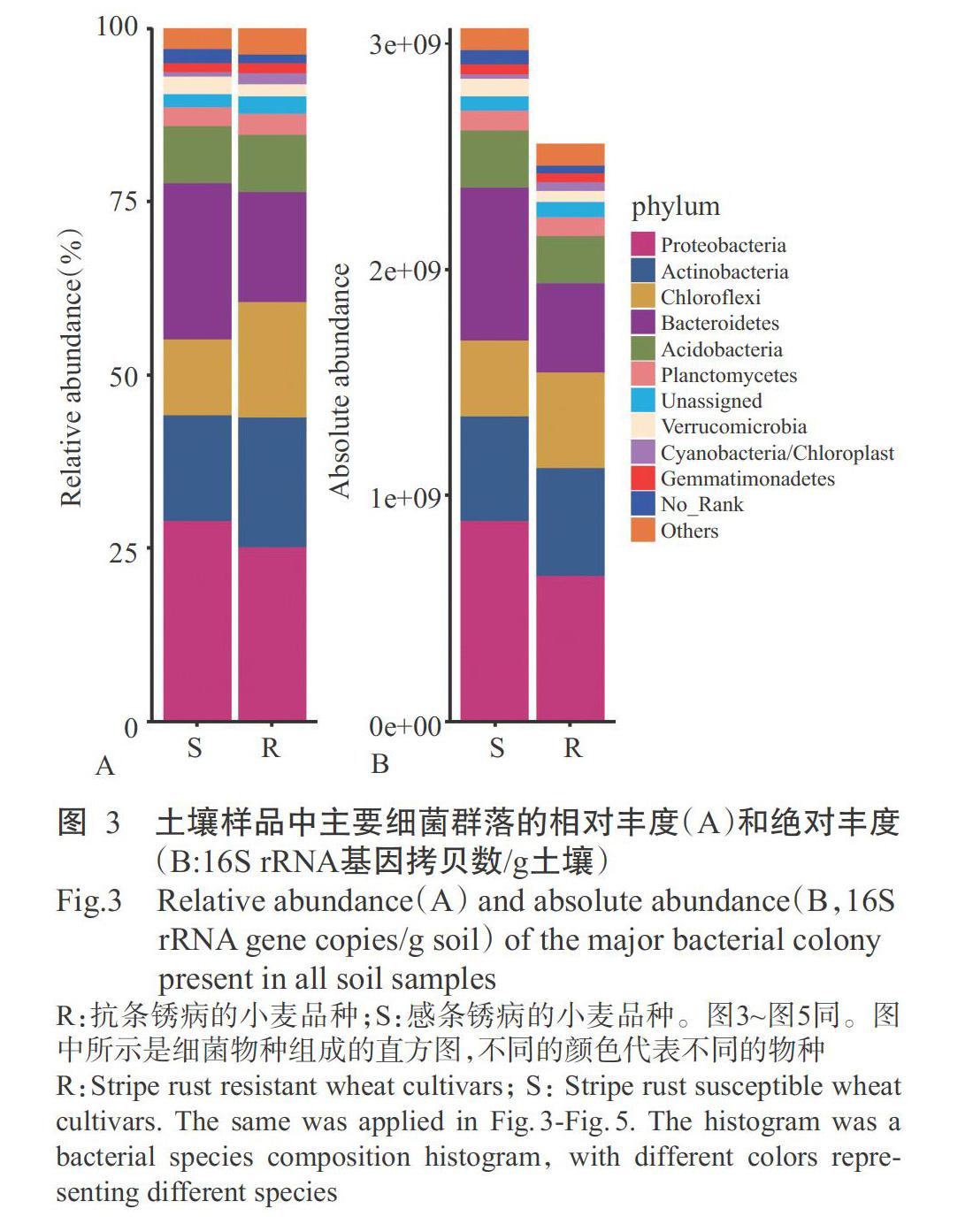
條銹病感病和抗病小麥品種根際土壤樣本細菌群落相對定量和絕對定量比較見圖3。與感病小麥品種(S)相比,抗病小麥品種(R)根際土壤中細菌的總豐度相對較小。絕對定量分析顯示,與抗病小麥品種相比,感病小麥品種根際的變形菌門(Proteobacteria)和擬桿菌門(Bacteroidetes)均顯著升高(P<0.05,下同);相對定量分析顯示,感病小麥根際放線菌門(Actinobacteria)含量低于抗病小麥,但絕對定量分析顯示,兩種小麥的根際細菌群落中Actinobacteria的絕對量無顯著差異(P>0.05,下同)。綜上所述,因絕對定量分析數據能更準確地反映真實情況,使用絕對定量測序數據進行后續分析。
2. 3 小麥根際土壤細菌群落結構分析結果
感病小麥品種和抗病小麥品種的根際細菌群落具有明顯不同的α多樣性指數。與感病小麥品種相比,抗病小麥品種細菌群落OUT數目較少,Chao1、ACE和Shannon指數均低于感病小麥品種,群落多樣性較低;感病小麥品種根際細菌群落具有更多的OUT數目和更高的群落多樣性(圖4)。
根據PCoA(在Weighted UniFrac上計算,圖5-A)和平均連鎖分層聚類(UPGMA,在Bray-Curtis上計算,圖5-B),感病小麥品種與抗病小麥品種根際細菌群落在PCo1(67.98%)水平上明顯分開。感病和抗病小麥品種內部,細菌群落結構的差異不明顯,說明條銹病抗性的差異對小麥植株根際細菌群落結構具有顯著影響,且其影響大于小麥不同品種間基因型差異的影響。
2. 4 抗病相關細菌群落功能分析結果
使用Welchs T-test對感病小麥品種和抗性小麥品種兩組樣本細菌群落KEGG功能條目的豐度進行差異比較,找出兩組中具有顯著差異的功能。通過初步分析,發現134個KEGG(KO) term在感病小麥品種和抗病小麥品種間存在顯著差異。
在抗病小麥品種的根際細菌群落中,多個KEGG功能條目具有更高的豐度值,包括轉運蛋白、鏈霉素的生物合成、纈氨酸、亮氨酸和異亮氨酸的生物合成等。植物病原體相互作用等KEGG途徑功能條目在感病小麥中具有更高的豐度值(圖6)。
3 討論
植物根際土壤是一個高度復雜和動態的生態系統(Bulgarelli et al.,2013;Mendes et al.,2013;Philippot et al.,2013)。已有充分的研究證明生長在鹽、旱、熱、重金屬和病菌、害蟲侵害等不利環境中的植物,其對逆境的適應能力部分甚至全部來自于與其共生的微生物(Zhou et al.,2015;Raza et al.,2016;Wang et al.,2016;Yuan et al.,2016)。溫室和野外試驗均證實,如果去除關鍵的內生菌,某些耐逆境植物不再能夠適應原本生存的不利環境(Redman et al.,2002)。有科學家提出,自然界中植物的表型是植物基因組和根際/體內廣泛存在的微生物協同作用的產物(Vandenkoornhuyse et al.,2015)。
目前對小麥條銹病抗性的研究主要集中在抗病種質和基因的挖掘,忽略了小麥健康的一個重要影響因素——根際微生物。高通量測序技術極大地方便了人們對植物根際微生物群落結構和功能的研究。針對模式植物擬南芥和農作物水稻、玉米、小麥的研究發現,土壤理化性質、植物發育階段和基因型是影響植物根際細菌群落結構的主要因素(Bulga-relli et al.,2012;Lundberg et al.,2012;Mahoney et al.,2017),但對不同小麥品種間根際細菌群落結構的差異尚無研究報道。
對同一地塊不同條銹病抗性小麥品種根際微生物進行比較分析發現,與抗病小麥品種相比,感病小麥品種根際細菌群落豐度增加,多樣性增加。功能分析表明,抗病小麥根際細菌群落具有分泌更多氨基酸(包括纈氨酸、亮氨酸和異亮氨酸等)和抗生素(如鏈霉素)的功能。抗條銹病小麥品種與感條銹病小麥品種的根際細菌群落存在顯著差異,其影響大于小麥品種基因型的影響。因此,進一步挖掘抗病小麥品種根際的有益細菌,提高小麥根際抗生素分泌功能,應可顯著改善小麥植株的抗條銹病能力。這一結果為開發小麥條銹病防治的環境友好方案提供了理論基礎和創新思路。
4 結論
通過采集同一地塊中對條銹病具有不同抗性的4個小麥品種根際細菌群落進行絕對定量擴增子分析,發現條銹病抗性對小麥根際細菌群落的影響超過小麥不同品種間遺傳變異的影響;通過分析和挖掘不同條銹病抗性小麥品種的根際細菌群落,可開發出環境友好的小麥條銹病生物防治菌劑。
參考文獻:
Berg G,Rybakova D,Grube M,K?berl M. 2016. The plant microbiome explored: Implications for experimental botany[J]. Journal of Experimental Botany,67(4):995-1002.
Bulgarelli D,Rott M,Schlaeppi K,Van Themaat E V L,Ahmadinejad N,Assenza F,Rauf P,Huettel B,Reinhardt R,Schmelzer E,Peplies J,Glockner F O,Amann R,Eickhorst T,Schulze L P. 2012. Revealing structure and asse-mbly cues for Arabidopsis root-inhabiting bacterial microbiota[J]. Nature,488(7409):91-95.
Bulgarelli D,Schlaeppi K,Spaepen S,Van Themaat E V,Schulze-Lefert P. 2013. Structure and functions of the bacterial microbiota of plants[J]. Annual Review of Plant Biology,64(1):807-838.
Castrillo G,Teixeira P J,Paredes S H,Law T F,De Lorenzo L,Feltcher M E,Dangl J L. 2017. Root microbiota drive direct integration of phosphate stress and immunity[J]. Nature,543(7646):513-518.
Cole J R,Wang Q,Fish J A,Chai B,Mcgarrell D M,Sun Y,Tiedje J M. 2014. Ribosomal database project: Data and tools for high throughput rRNA analysis[J]. Nucleic Acids Research,42:633-642.
Hacquard S,Garrido-Oter R,Gonzalez A,Spaepen S,Ackermann G,Lebeis S L,Schulze-Lefert P. 2015. Microbiota and host nutrition across plant and animal kingdoms[J]. Cell Host & Microbe,17(5):603-616.
Hu L,Robert C A M,Cadot S,Zhang X,Ye M,Li B,Manzo D,Chervet N,Steinger T,Van Der Heijden M G A,Schlaeppi K,Erb M. 2018. Root exudate metabolites drive plant-soil feedbacks on growth and defense by shaping the rhizosphere microbiota[J]. Nature Communications,doi: 10.1038/s41467-018-05122-7.
Kwak M J,Kong H G,Choi K,Kwon S K,Song J Y,Lee J,Kim J F. 2018. Rhizosphere microbiome structure alters to enable wilt resistance in tomato[J]. Nature Biotechno-logy,36(11):1100-1109.
Line R F,Qayoum A. 1992. Virulence,aggressiveness,evolution and distribution of races of Puccinia striiformis(the cause of stripe of wheat) in North America,1968—1987[J]. Technical Bulletin,1788:44.
Lundberg D S,Lebeis S L,Paredes S H,Yourstone S,Gehring J,Malfatti S A,Dangl J L. 2012. Defining the core Arabidopsis thaliana root microbiome[J]. Nature,488(7409):86-90.
Mahoney A K,Yin C T,Hulbert S H. 2017. Community structure,species variation,and potential functions of rhizosphere-associated bacteria of different winter wheat(Triticum aestivum) cultivars[J]. Frontiers in Plant Science,8:132.
Mendes R,Garbeva P,Raaijmakers J M. 2013. The rhizosphere microbiome: Significance of plant beneficial,plant pathogenic,and human pathogenic microorganisms[J]. Fems Microbiology Reviews,37(5):634-663.
Philippot L,Raaijmakers J M,Lemanceau P,Der Putten W H. 2013. Going back to the roots: The microbial ecology of the rhizosphere[J]. Nature Reviews Microbiology,11(11):789-799.
Pieterse C M J,Zamioudis C,Berendsen R L,Weller D M,Van Wees S C,Bakker P A H M. 2014. Induced systemic resistance by beneficial microbes[J]. Annual Review of Phytopathology,52(1):347-375.
Planchamp C,Glauser G,Mauch-Mani B. 2014. Root inoculation with Pseudomonas putida KT2440 induces transcriptional and metabolic changes and systemic resistance in maize plants[J]. Frontiers in Plant Science,5:719.
Raza W,Ling N,Yang L D,Huang Q W,Shen Q R. 2016. Response of tomato wilt pathogen Ralstonia solanacearum to the volatile organic compounds produced by a biocontrol strain Bacillus amyloliquefaciens SQR-9[J]. Scienti-fic Reports,6(1):24856.
Redman R S,Sheehan K B,Stout R G,Rodriguez R J,Henson J M. 2002. Thermotolerance generated by plant/fungal symbiosis[J]. Science,298(5598):1581.
Smets W,Leff J W,Bradford M A,Mcculley R L,Lebeer S,Fierer N. 2016. A method for simultaneous measurement of soil bacterial abundances and community composition via 16S rRNA gene sequencing[J]. Soil Biology & Biochemistry,96:145-151.
Tkacz A,Hortala M,Poole P S. 2018. Absolute quantitation of microbiota abundance in environmental samples[J]. Microbiome,6(1):1-13.
United States Department of Agriculture-Foreign Agricultural Service. 2019. World Agriculture Production Report 2019. [2019-04-20]. https://www.fas.usda.gov/data/world-agricultural-production.
Vandenkoornhuyse P,Quaiser A,Duhamel M,Van A L,Dufresne A. 2015. The importance of the microbiome of the plant holobiont[J]. New Phytologist,206(4):1196-1206.
Wang J L,Li T,Liu G Y,Smith J M,Zhao Z W. 2016. Unrave-ling the role of dark septate endophyte(DSE) colonizing maize(Zea mays) under cadmium stress: Physiological,cytological and genic aspects[J]. Scientific Reports,6(1):22028.
Yuan Z L,Druzhinina I S,Labbe J,Redman R S,Qin Y,Rodriguez R J,Lin F C. 2016. Specialized microbiome of a halophyte and its role in helping non-host plants to withstand salinity[J]. Scientific Reports,6(10):32467.
Zhou W N,White J F,Soares M A,Torres M S,Zhou Z P,Li H Y. 2015. Diversity of fungi associated with plants growing in geothermal ecosystems and evaluation of their capacities to enhance thermotolerance of host plants[J]. Journal of Plant Interactions,10(1):305-314.
(責任編輯 麻小燕)

