利用CRISPR/Cas9系統編輯擬南芥ILR3基因及功能驗證
李鵬

摘要:擬南芥轉錄因子ILR3(IAA-Leucine Resistant 3)在鐵穩態的調節、葡萄糖異硫氰酸鹽(glucosinolate,簡稱GLS)的生物合成和病原體響應方面起到重要作用。為更深入探索該轉錄因子在植物體內的更多功能,利用YAO基因啟動子驅動Cas9在擬南芥中表達,成功獲得ILR3基因編輯突變體。測序結果及序列分析結果表明,在ILR3編輯擬南芥中,該基因編碼區發生了堿基缺失或插入,導致蛋白ILR3保守結構域丟失。并且,這些基因編輯突變體T2代幼苗與T-DNA插入突變體ilr3-2在缺鐵環境下表現出相同的性狀,進一步驗證ILR3轉錄因子在植物對鐵的吸收及體內平衡的作用,也為深入研究其更多生物學功能奠定了工作基礎。
關鍵詞:擬南芥;AtILR3;YAO啟動子; CRISPR/Cas9;基因編輯
中圖分類號: Q784? 文獻標志碼: A
文章編號:1002-1302(2020)14-0078-05
CRISPR/Cas9(clustered regularly interspersed short palindromic repeats /CRISPR-associated protein 9)系統已被用于基因組編輯,具有很高的準確性,并能高效完成真核基因組中基因敲除(knockout/knockin)和點突變[1],在人類細胞[2-3]、植物[4]、動物[5]、微生物[6]中均得到了很好的應用,并取得了舉世矚目的成果。YAO參與擬南芥的胚胎發生及配子發育,主要表達于分裂能力強的組織中[7];中科院遺傳與發育生物學研究所謝旗研究組通過YAO基因啟動子驅動Cas[STBX]9[STBZ],極大地提高CRISPR/Cas9編輯擬南芥基因組的效率[8]。
AtbHLH105/AtILR3(IAA-Leucine Resistant 3)基因屬于擬南芥bHLH家族Ⅳc亞家族[9],所編碼的蛋白可與其他諸多蛋白協同作用,共同參與植物鐵穩態的調節[10-11]。
筆者所在課題組在長期的研究中發現,ILR3在植物生長發育過程中發揮更多的生物學功能。因此,本研究首次利用YAO基因啟動子驅動的Cas9編輯擬南芥ILR3基因,所獲得的基因編輯株系發生堿基插入和缺失,與T-DNA插入突變體ilr3-2在缺鐵處理時具有相同的表型,與文獻報道結果一致,為研究ILR3在生物體中的更多功能,奠定工作基礎。
1 材料與方法
1.1 材料
本試驗所用擬南芥(Arabidopsis thaliana)野生型為Col-0,突變體ilr3-2(Salk_004997C)購買自網站https://www.arabidopsis.org/,雙元表達載體pYAO-Cas9-SK由中國科學院遺傳與發育生物學研究所謝旗研究組饋贈,大腸桿菌(Escherichia coli) DH5α和根癌農桿菌(Agrobacterium tumefaciens) EHA105由筆者所在實驗室保存。限制性內切酶BamHⅠ購自NEB(北京)公司,質粒提取和膠回收試劑盒購自天根生化科技(北京)有限公司,T載體、DNA連接試劑盒、Taq DNA聚合酶購自寶日醫生物技術(北京)有限公司,其他生化試劑及引物購自生工生物工程(上海)股份有限公司。
1.2 擬南芥AtILR3編輯靶位點的確定
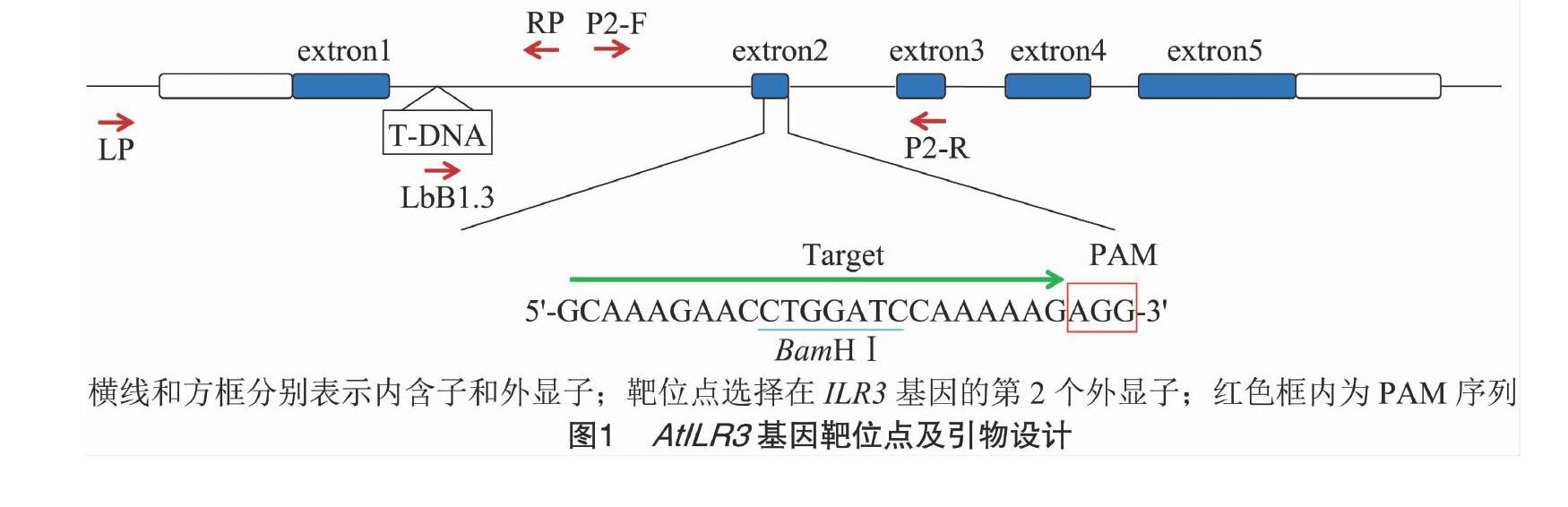
將擬南芥AtILR3基因(At5g54680)提交NCBI檢索,獲得其基因序列及外顯子序列信息,在外顯子區域尋找PAM(protospacer adjacent motif)位點為“NGG”的23 bp序列,且該序列最好帶有酶切位點,并通過擬南芥bHLH家族各成員同源序列比對驗證該靶位點的特異性。針對篩選到的靶位點,在序列5′端添加BsaⅠ限制性內切酶的黏性末端接頭TGATT和AAAC,分別設計引物P1-F和P1-R(表1)。
1.3 gRNA的合成及與載體的連接
將10 μmol/L的P1-F和P1-R各取1 μL,加入到8 μL退火緩沖液(TE+50 mmol/L NaCl)中,混勻后在PCR儀中退火:從95 ℃緩慢降溫至 16 ℃,速率為0.1 ℃/s;取1 μL退火產物,與用 BsaⅠ 酶切過的載體pYAO-Cas9-SK進行連接,體系如下:1 μL 退火產物,1 μL載體,1 μL 10×T4緩沖液,0.5 μL T4連接酶,加ddH2O至10 μL,連接條件為25 ℃反應2 h。采用熱激法轉化大腸桿菌DH5α感受態細胞,經篩選鑒定后測序。將測序正確質粒導入農桿菌感受態EHA105。
1.4 擬南芥遺傳轉化及基因編輯植株的獲得
采用浸花法對擬南芥進行轉化[12],收集轉化后的T0代種子,干燥后,75%乙醇消毒1 min,10%次氯酸鈉消毒10 min,無菌水漂洗5次,均與播種于含潮霉素25 mg/L的1/2MS表面進行抗性篩選,培養條件為22 ℃,16 h光照,8 h黑暗。
2周后,選取有潮霉素抗性的擬南芥幼苗,移栽至土壤基質中生長,同時摘取1張葉片,采用改良的CTAB法提取基因組DNA,并以此DNA為模板,以潮霉素抗性基因序列設計引物HygR-F、HygR-R(表1),進行PCR鑒定。PCR體系:在20 μL反應體系中,Taq DNA聚合酶1.0 U,dNTP各0.25 μmol/L,引物0.5 μmol/L,100 ng模板DNA。PCR反應程序:94 ℃預變性5 min;94 ℃變性30 s,58 ℃退火 30 s,72 ℃延伸1.5 min,循環30次;72 ℃延伸 10 min,4 ℃保存。1%瓊脂糖凝膠電泳后檢測,能完整擴增出潮霉素抗性基因的個體為轉基因擬南芥陽性植株。T1、T2代擬南芥陽性植株采用相同方法鑒定。
根據基因AtILR[STBX]3[STBZ]序列,在靶位點上下游200~300 bp處設計引物P2-F、P2-R(表1、圖1),以T2代轉基因擬南芥陽性植株的基因組DNA為模板,再次進行PCR,反應體系與程序與上述相同,擴增產物為含靶位點的562 bp的片段。電泳后,回收目的片段,取其中一部分進行限制性內切酶處理。將未被切開的PCR片段克隆到T載體,轉化至大腸桿菌中,并對插入片段進行菌落PCR,陽性克隆進行質粒提取并測序。
1.5 AtILR3編輯擬南芥的表型分析
將擬南芥野生型、ilr3-2突變體及獲得的4個株系的ILR3編輯擬南芥T1代幼苗,播種于1/2MS、1/2MS-Fe和1/2MS-Fe+50 μmol/L菲洛嗪(ferrozine,Frz)培養基,2周后,拍照并統計最長根長度。
采用三引物法[13]對ilr[STBX]3-2[STBZ]突變體進行鑒定,引物LP、RP和LbB1.3位點及序列見圖1和表1。
2 結果與分析
2.1 AtILR3編輯靶位點的篩選及載體構建
序列分析結果表明,AtILR3基因包5個外顯子,4個內含子,編碼區全長705 bp,根據CRISPR/Cas9 設計原理,選擇其第2個外顯子上的特異序列為編輯靶序列(圖1),并且該靶位點PAM序列前6 bp包含1個BamHⅠ限制性內切酶位點(GGATCC),方便檢測。
根據靶位點序列合成靶點接頭引物P1-F和P1-R,退火形成靶點接頭后克隆到CRISPR/Cas9 載體pYAO-Cas9-SK[8],測序后將正確質粒導入農桿菌感受態EHA105。
2.2 擬南芥基因編輯植株的獲得及鑒定
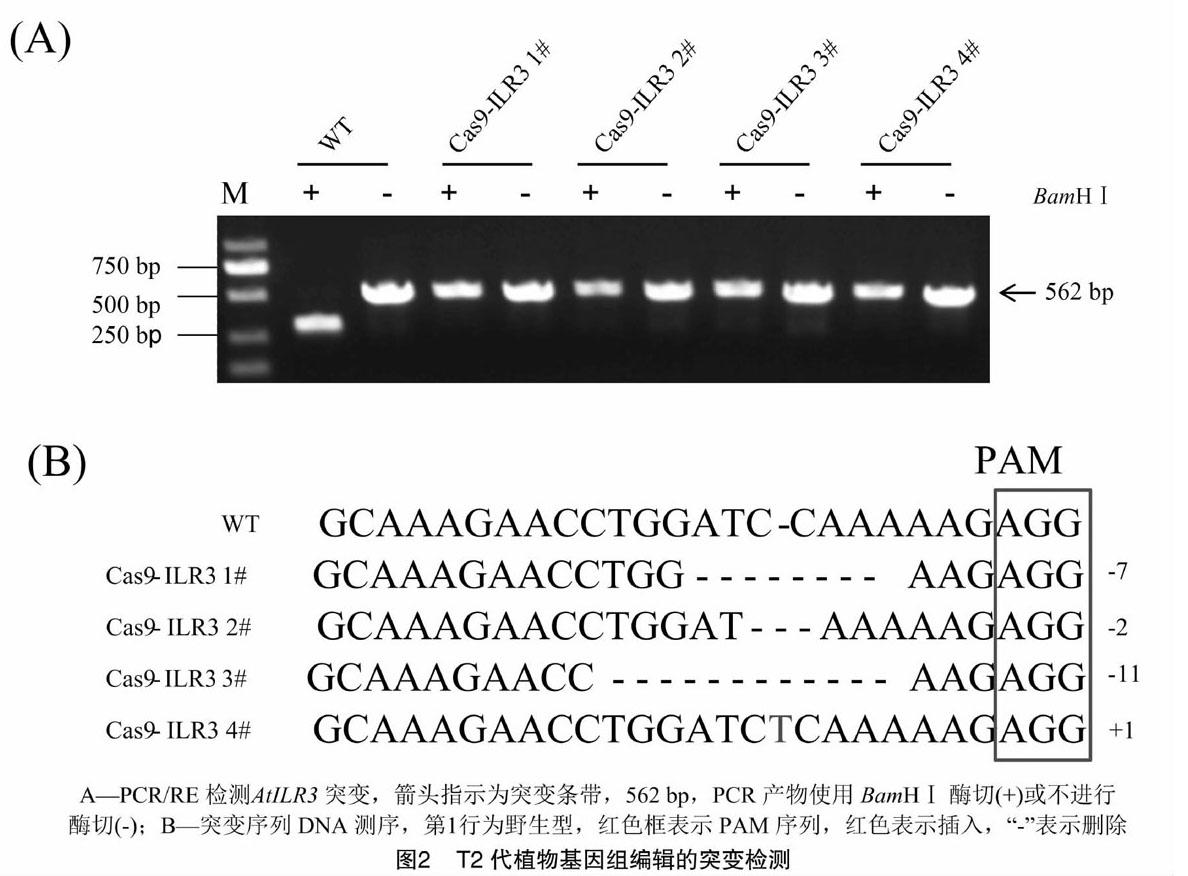
利用引物HygR-F、HygR-R做PCR鑒定得到轉基因陽性苗;利用P2-F、P2-R為引物,陽性苗基因組DNA為模板,擴增出含靶位點的DNA片段;回收此片段,用BamHⅠ酶切鑒定電泳后,獲得4株ILR3基因編輯擬南芥幼苗。如圖2-A所示,野生型的片段被BamHⅠ識別,并完全切斷為2個長度分別為276 、286 bp的片段(由于2個片段大小過于接近,電泳結果只看到1條帶),而突變片段的擴增產物則無法被識別和切割。
將未消化的PCR片段克隆到T載體中,轉化大腸桿菌,并將插入片段進行菌落PCR,陽性單菌落培養過夜后提取質粒進行測序。結果表明,在靶點位置發生不同類型的編輯,在PAM序列前3~7 bp分別產生了插入和缺失突變, 其中缺失突變占多數(圖2-B)。
2.3 AtILR3編輯擬南芥的表型
擬南芥ILR3蛋白在鐵動態平衡的調控過程中發揮著重要作用,突變體在缺鐵環境中出現黃化、發育不良等表型[10-11]。將野生型、ilr[STBX]3-2[STBZ]純合突變體及篩選得到的4個基因編輯T1代幼苗播種于1/2MS、1/2MS-Fe和1/2MS-Fe+50 μmol/L Frz培養基,2周后發現,4個基因編輯株系的幼苗與ilr3-2突變體出現相同的表型,均弱于野生型(圖3-A)。數據顯示,在含有鐵元素的1/2 MS上,6個株系的擬南芥生長狀況基本無差異;在缺鐵培養基上,野生型根系明顯增長,由約1.8 cm增長到約74 cm,而突變體ilr[STBX]3-2[STBZ]和4個基因編輯株系僅增長到約2.7 cm;Frz可以螯合培養基中瓊脂中的微量鐵元素[14],在1/2MS-Fe+50 μmol/L Frz 的培養基上,雖然ilr[STBX]3-2[STBZ]和基因編輯株系最長根長度影響不大,但側根減少,長勢更弱,而野生型影響較小(圖3-B)。
為鑒定T-DNA插入突變體ilr3-2是否為純合子,采用三引物法對10棵突變體單株和野生型進行PCR鑒定。電泳結果(圖3-C)表明,可以證明所用突變體ilr3-2為純合子,以上表型數據可信。
3 討論
CRSPR/Cas9是一種高效且簡便的基因定點編輯技術,該技術具有合成簡單、周期短、操作靈活、效率高等優點,給基因工程帶來技術性革命,在眾多植物基因編輯中已經得到了成功應用,如擬南芥[15]、水稻[16]、玉米[17]、小麥[18]、大豆[19]、矮牽牛[20]、巨桉[21]等。將這一技術應用于擬南芥的遺傳轉化過程中,多采用農桿菌介導的浸花法來進行,因而囊胚是T-DNA的主要靶點[22]。35S和泛素啟動子常用于驅動Cas[STBX]9[STBZ]基因在擬南芥的中表達,前者應用得更多。而已有的研究表明,CaMV 35S啟動子在生殖細胞中活性極低,用CaMV 35S驅動的Cas[STBX]9[STBZ]對擬南芥基因組的編輯效率明顯低于水稻,并且絕大多數T1植株只是體細胞突變,并不能遺傳到T2[23-24]。YAO是一個在細胞分裂旺盛組織優先表達的基因,尤其在胚、胚囊、胚乳以及花粉中有很高的表達量[7];謝旗研究組用該基因的啟動子pYAO代替35S啟動子來啟動Cas9的表達,提高了編輯效率并獲得生殖系突變,且編輯形式更多[8]。因此,本研究也利用啟動子pYAO來驅動Cas[STBX]9[STBZ]的表達,T0共獲得4株基因編輯擬南芥株系。為簡化突變檢測過程,本研究選擇在PAM上游存在BamHⅠ限制性內切酶位點的序列作為靶位點。利用 BamHⅠ 進行突變檢測和測序的結果顯示,4個株系中有3個為缺失突變,1個為插入突變,突變位點位于PAM上游3~7 bp處。由于本試驗中只有不被BamHⅠ消化的PCR產物才會被收集鑒定,因此,存在雖然發生突變,但突變位點不在BamHⅠ內切酶位點的可能,這樣的突變將不會被檢測到。因此本試驗所檢測到的各突變類型的比例可能比真實的突變類型偏小,試驗設計還有待進一步完善。
擬南芥bHLH家族Ⅳc亞家族成員bHLH34/104/105/115是鐵穩態調節的主要因子,其中bLHLH105(ILR3)可與PYE[25]、BTS[25-27]、bHLH34[28]、bHLH104[10]、bHLH115[29]等蛋白協同作用,調控鐵吸收、轉運相關下游基因FIT、ZIF1、NAS4、FRO2/IRT1、bHLH38/39/100/101、MYB10/72[28]的表達;ILR3還和PYE共同參與植物缺鐵脅迫下葡萄糖異硫氰酸鹽(glucosinolate,簡稱GLS)積累的調控網絡[11]。由此可知,ILR3基因在植物生長發育、抵御脅迫過程中發揮著重要作用。ILR3的T-DNA插入突變體ilr3-2已廣泛應用在該基因功能的研究中。本研究發現,ILR3在生長發育過程中具有新的功能,所以利用CRISPR/Cas9技術對ILR3基因進行編輯。為確保突變后的基因完全失去功能,靶位點一般選擇在編碼區,尤其是靠近5′端或處于編碼蛋白功能區之前的序列[30]。因此,本研究選擇對ILR3基因第2個外顯子進行編輯,共獲得4個突變株系,且這些株系在缺鐵情況下與 ilr[STBX]3-2[STBZ] 表現出相同的生長狀態,與文獻報道結果一致,進一步驗證了ILR3在植物生長發育過程中及抗逆性方面所起的重要作用。
參考文獻:
[1]Wu W,Yang Y,Lei H. Progress in the application of CRISPR:from gene to base editing[J]. Medicinal Research Reviews,2019,39(2):665-683.
[2]Strecker J,Jones S,Koopal B,et al. Engineering of CRISPR-Cas12b for human genome editing[J]. Nature Communications,2019,10(1):212.
[3]Li T,Zhu L,Xiao B,et al. CRISPR-Cpf1-mediated genome editing and gene regulation in human cells[J]. Biotechnology Advances,2019,37(1):21-27.
[4]Das A,Sharma N,Prasad M. CRISPR/Cas9:a novel weapon in the arsenal to combat plant diseases[J]. Frontiers in Plant Science,2018,9:2008.
[5]Chao T,Liu Z,Zhang Y,et al. Precise and rapid validation of candidate gene by allele specific knockout with CRISPR/Cas9 in wild mice[J]. Frontiers in Genetics,2019,10:124.
[6]Hidalgo-Cantabrana C,Goh Y J,Barrangou R. Characterization and repurposing of type Ⅰ and type Ⅱ CRISPR-Cas systems in bacteria[J]. Journal of Molecular Biology,2019,431(1):21-33.
[7]Li H J,Liu N Y,Shi D Q,et al. YAO is a nucleolar WD40-repeat protein critical for embryogenesis and gametogenesis in Arabidopsis[J]. BMC Plant Biology,2010,10(1):169.
[8]Yan L,Wei S,Wu Y,et al. High-efficiency genome editing in Arabidopsis using YAO promoter-driven CRISPR/Cas9 system[J]. Molecular Plant,2015,8(12):1820-1823.
[9]Rampey R A,Woodward A W,Hobbs B N,et al. An Arabidopsis basic helix-loop-helix leucine zipper protein modulates metal homeostasis and auxin conjugate responsiveness[J]. Genetics,2006,174(4):1841-1857.
[10]Zhang J,Liu B,Li M,et al. The bHLH transcription factor bHLH104 interacts with IAA-LEUCINE RESISTANT3 and modulates iron homeostasis in Arabidopsis[J]. Plant Cell,2015,27(3):787-805.
[11]Samira R,Li B,Kliebenstein D,et al. The bHLH transcription factor ILR3 modulates multiple stress responses in Arabidopsis[J]. Plant Molecular Biology,2018,97(4/5):297-309.
[12]Davis A M,Hall A,Millar A J,et al. Protocol:streamlined sub-protocols for floral-dip transformation and selection of transformants in Arabidopsis thaliana[J]. Plant Methods,2009,5:3.
[13]李 敏,楊 雙,阮燕曄,等. 擬南芥T-DNA插入突變體atsuo[STBX]3[STBZ]的PCR鑒定[J]. 植物生理學通訊,2006,42(1):91-94.
[14]Berker K I,Ozyurt D,Demirata B,et al. Solid-phase extraction spectrophotometric determination of total antioxidant capacity in antioxidant-poor samples by using the ferric-ferrozine method[J]. Analytical Sciences,2017,33(6):683-689.
[15]Yu Z,Chen Q,Chen W,et al. Multigene editing via CRISPR/Cas9 guided by a single-sgRNA seed in Arabidopsis[J]. Journal of Integrative Plant Biology,2018,60(5):376-381.
[16]Toda E,Koiso N,Takebayashi A,et al. An efficient DNA- and selectable-marker-free genome-editing system using zygotes in rice[J]. Nature Plants,2019,5(4):363-368.
[17]Wang B,Zhu L,Zhao B,et al. Development of a haploid-inducer mediated genome editing system for accelerating maize breeding[J]. Molecular Plant,2019,12(4):597-602.
[18]Liang Z,Chen K,Li T,et al. Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes[J]. Nature Communications,2017,8:14261.
[19]Li C,Nguyen V,Liu J,et al. Mutagenesis of seed storage protein genes in soybean using CRISPR/Cas9[J]. BMC Research Notes,2019,12(1):176.
[20]Sun L,Williams J S,Li S,et al. S-locus F-box proteins are solely responsible for S-RNase-based self-incompatibility of Petunia pollen[J]. Plant Cell,2018,30(12):2959-2972.
[21]李莉梅,歐陽樂軍,尹愛國,等. 1種大片段敲除巨桉細胞分裂素氧化酶基因的CRISPR載體構建[J]. 江蘇農業科學,2018,46(12):19-22.
[22]Ye G N,Stone D,Pang S Z,et al. Arabidopsis ovule is the target for Agrobacterium in planta vacuum infiltration transformation[J]. Plant Journal,1999,19(3):249-257.
[23]Feng Z,Zhang B,Ding W,et al. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research,2013,23(10):1229-1232.[HJ1.9mm]
[24]Ma X,Zhang Q,Zhu Q,et al. A robust CRISPR/Cas9 system for convenient,high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant,2015,8(8):1274-1284.
[25]Long T A,Tsukagoshi H,Busch W,et al. The bHLH transcription factor POPEYE regulates response to iron deficiency in Arabidopsis roots[J]. Plant Cell,2010,22(7):2219-2236.
[26]Selote D,Samira R,Matthiadis A,et al. Iron-binding E3 ligase mediates iron response in plants by targeting basic helix-loop-helix transcription factors[J]. Plant Physiology,2015,167(1):273-286.
[27]Hindt M N,Akmakjian G Z,Pivarski K L,et al. BRUTUS and its paralogs,BTS LIKE1 and BTS LIKE2,encode important negative regulators of the iron deficiency response in Arabidopsis thaliana[J]. Metallomics,2017,9(7):876-890.
[28]Li X,Zhang H,Ai Q,et al. Two bHLH transcription factors,bHLH34 and bHLH104,regulate iron homeostasis in Arabidopsis thaliana[J]. Plant Physiology,2016,170(4):2478-2493.
[29]Liang G,Zhang H,Li X,et al. bHLH transcription factor bHLH115 regulates iron homeostasis in Arabidopsis thaliana[J]. Journal of Experimental Botany,2017,68(7):1743-1755.
[30]孫勤富,劉東曉,林 俐,等. 甘藍型油菜和甘藍CRISPR/Cas9編輯效果的快速檢測[J]. 中國油料作物學報,2018,40(6):737-744.
收稿日期:2019-08-01
基金項目:上海市青年科技英才揚帆計劃(編號:19YF1414800)。
作者簡介:李 鵬(1983—),男,安徽濉溪人,碩士,工程師,主要從事植物抗逆性研究。E-mail:penglee98@163.com。

