CRISPR/Cas9技術在農作物中應用的局限及改進
武林琳 竹夢婕 王咪 李曉萍 劉躍鵬 裴蕾 楊淑巧 許琦 王華 郭文治
摘要 ? ?基因編輯技術是一種可以直接對DNA序列進行穩定、精準改造的技術,其中CRISPR/Cas9技術以簡便、高效、經濟等優勢脫穎而出。CRISPR/Cas9技術被廣泛應用于作物遺傳育種、植物基因改造和農作物品種改良等多個方面,給農作物領域帶來巨大機遇。但機遇與挑戰并存,該技術在實際應用中亦遇到一些困難。因此,本文圍繞CRISPR/Cas9技術的原理、局限及改進方案進行綜合闡述,以期為CRISPR/Cas9技術在農作物中的進一步應用提供理論基礎。
關鍵詞 ? ?基因編輯;CRISPR/Cas9;農作物;局限;脫靶效應;改進手段
中圖分類號 ? ?Q943.2 ? ? ? ?文獻標識碼 ? ?A
文章編號 ? 1007-5739(2020)22-0026-04 ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?開放科學(資源服務)標識碼(OSID)
Abstract ? ?Gene editing technology is a technology that can directly modify the DNA sequence stably and accurately, in which CRISPR/Cas9 technology stands out for the advantages of simplicity, high efficiency and economy. CRISPR/Cas9 technology is widely used in crop genetic breeding, plant genetic modification, crop variety improvement and other aspects, bringing great opportunities to the field of crops. However, there are both opportunities and challenges, and the technology also encounters some difficulties in practical application. Therefore, this paper comprehensively expounded the principle, limitation and improvement scheme of CRISPR/Cas9 technology, in order to provide theoretical basis for the further application of CRISPR/Cas9 technology in crops.
Keywords ? ?gene editing; CRISPR/Cas9; crop; limitation; off-target effect; improvement mean
隨著世界人口的增加,到2050年為全球超過90億的預期人口提供食物是21世紀的主要挑戰之一。Henry等[1]研究結果表明,在耕地擴張不超過地球邊界的情況下,至2050年向全球人口提供充足的食物是可能的,但需要對糧食系統的需求方進行重大改變或推進生物技術。因此,基因編輯技術應運而生。
基因編輯是一種在基因組水平上對DNA序列進行精確敲除、插入和替換,從而實現基因組序列穩定改善的技術[2]。基因編輯技術的原理是利用人工內切核酸酶(SSNs)在目標基因組位置產生DNA雙鏈斷裂(double-strand breaks,DSBs),激活細胞的DNA修復系統,在修復過程中改變DNA序列[2]。DSBs可以通過非同源末端連接(non-homologous end joining,NHEJ)和同源重組(homologous-directed recombination,HDR)進行修復。NHEJ是DNA斷裂后最常見的修復方式,僅能造成DNA序列的隨機改變,而HDR則能夠引入一個與DSBs同源的外源DNA作為修復模板,使基因組DNA產生精確的序列改變。
目前,在農作物中應用的基因編輯技術主要有3種,即鋅指核酸酶(Zinc-finger nucleases,ZFNs)[3]、轉錄激活因子樣效應物核酸酶(Transcription activator-like effector nucleases,TALENs)[4]和成簇規律間隔的短回文重復序列及其相關系統(Clustered regularly interspaced short palindromic repeats/CRISPR-associated 9,CRISPR/Cas9)技術,其中以CRISPR/Cas9最為常用。CRISPR/Cas9技術已被用于作物遺傳育種、植物基因改造、農作物品種改良等多個方面。
1 ? ?CRISPR/Cas9基因編輯技術
能夠識別并特異性切割DNA序列的核酸酶是各種基因編輯技術的特色所在,CRISPR/Cas9技術中的核酸酶為Cas9,通過識別前間區序列鄰近基序(protospacer adjacent motif,PAM)進行DNA雙鏈切割[5]。
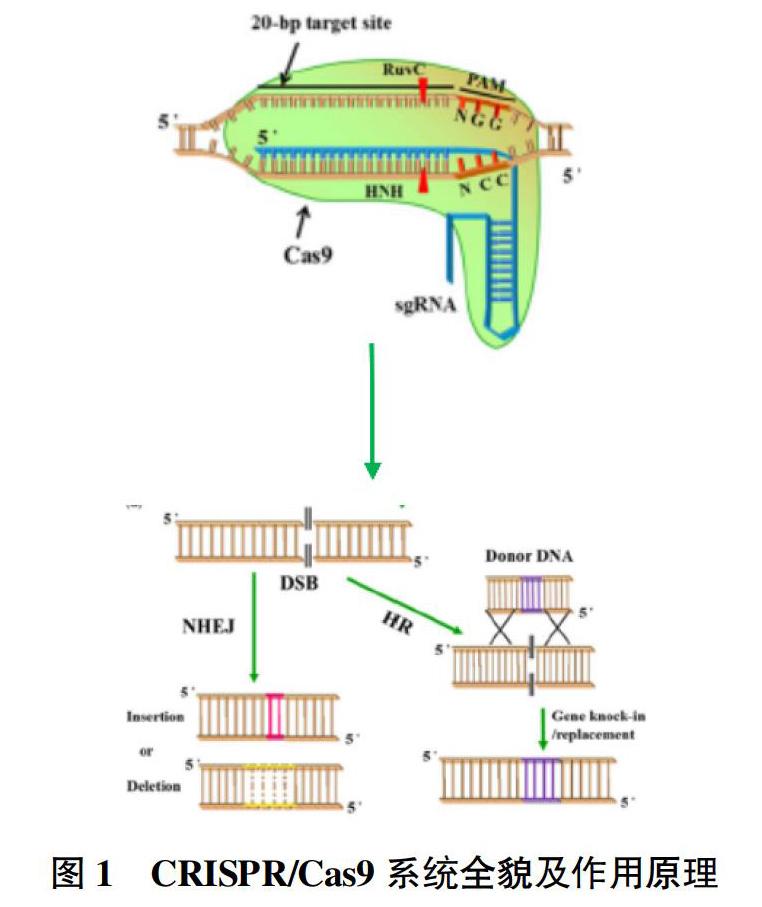
CRISPR廣泛存在于古細菌和細菌中,是細菌進行自我防御的重要機制。當噬菌體侵入細菌后,細菌會將病毒DNA短片段整合到自己的染色體上,之后將該序列轉錄為短RNA,稱為crRNA(CRISPR RNA),然后與另一個稱為tracrRNA(反式激活CRISPR RNA)的RNA形成復合體。這種復合物被稱為“引導RNA(gRNA)”,引導核酸內切酶Cas9至病毒DNA部位定點切割,產生DSBs。當gRNA通過識別PAM序列在噬菌體DNA中找到其匹配序列時,Cas9核酸酶就會裂解病毒DNA,從而抵抗病毒攻擊[6]。
CRISPR/Cas9技術來源于上述細菌獲得性免疫產生機制,該系統主要包括Cas9核酸內切酶和單鏈引導RNA(single guide RNA,sgRNA)。CRISPR/Cas9在sgRNA與Cas9蛋白的特定位點相結合后進入激活狀態;sgRNA識別具有PAM序列的DNA序列,通過前20個核苷酸與目標DNA堿基配對;然后引導Cas9核酸酶至靶切割位點切割DNA,產生DSBs;最后通過NHEJ和HDR進行細胞修復[7](圖1)。
由上可知,CRISPR/Cas9技術只需改變sgRNA的前20個核苷酸序列就可對基因組的不同位點實現靶向編輯,理論上能夠識別任何含PAM序列的DNA片段。其因具有方便、精準、高效、經濟等特點,在農作物改良中被迅速推廣。
2 ? ?CRISPR/Cas9基因編輯技術的局限性
CRISPR/Cas9已被成功用于編輯超過25種植物和100個基因,創造出符合人們要求的主要作物的理想性狀[8-9]。但CRISPR/Cas9技術仍有局限性,如該技術實現基因編輯有賴于PAM序列,在附近無PAM序列的區域無法進行編輯;由于CRISPR/Cas9技術會出現sgRNA與DNA間的錯配,基因組中與靶位點序列相似的序列也會被CRISPR/Cas9識別并切斷,出現脫靶。
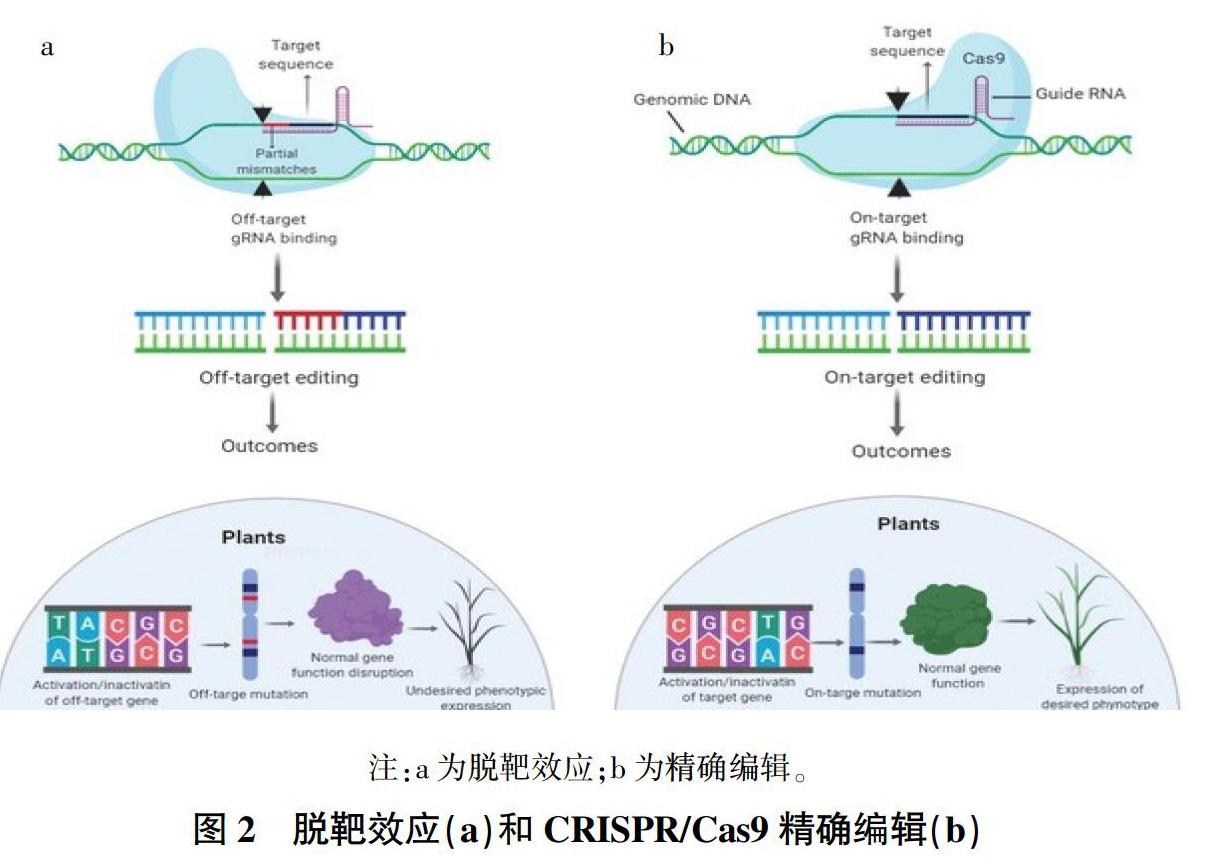
Cas9復合體與非靶標區域結合并啟動切割,稱為脫靶效應[10]。與ZFNs和TALENs相比,CRISPR/Cas9系統更易發生脫靶效應。原因在于Cas9的靶向準確度主要由基因組目標序列附近的20 nt sgRNA和PAM位點決定,單鏈的sgRNA更易產生識別DNA序列的錯配,導致脫靶[11]。
脫靶可能導致染色體重排,造成不完全匹配的基因組位點的破壞[12]。另外,除干擾染色體穩定性外,脫靶效應還可能導致功能基因活性的喪失,從而導致各種信號異常[13](圖2)。潛在的脫靶效應是CRISPR/Cas9在作物應用中備受關注的問題[14],它限制了該技術在作物育種及作物改良中的進一步擴展應用[15]。
3 ? ?對CRISPR/Cas9技術局限性的改進手段
鑒于CRISPR/Cas9技術的強大優勢,科研工作者采取多種策略降低CRISPR/Cas9的脫靶效應。CRISPR/Cas9系統由Cas9核酸內切酶和sgRNA兩部分組成,對降低脫靶效應策略的研究大都集中在兩方面。
3.1 ? ?設計最優sgRNA
設計最優sgRNA,有利于消除基因編輯致命的脫靶現象。為此,首先需要選擇該目標基因型的高質量基因組作參考基因組[16];其次,選擇CRISPR-P 2.0[17]、E-CRISP[18]、Breaking-Cas[19]、CasFinder[20]來設計最優的sgRNA序列以最小化脫靶機會。
3.2 ? ?改進CRISPR/Cas9系統
3.2.1 ? ?CBES和ABES(Cytosine and Adenine Base Edi-tors,BEs,胞嘧啶和腺嘌呤堿基編輯器)。BEs是由催化結構域和DNA靶向模塊組成的嵌合蛋白,具有脫氨腺嘌呤或胞苷的能力。通過靶向sgRNA,將具有脫氨酶結構域的蛋白酶引導至靶標位置,進行切割,同時可將目標部位配對堿基中的C精確轉化為T或將A轉化為G[21-23]。該過程不產生DSBs,大大減小了靶點位置上隨機插入、缺失產生的脫靶效應。
3.2.2 ? ?xCas9和Cas9-NG。xCas9和Cas9-NG是SpCas9的變異體,在提高靶點特異性和擴大靶點范圍方面顯示出巨大的潛力。xCas9-3.7是一個高效的xCas9變體,可針對多達16個候選NGN PAM組合(如NGG、NG、GAA和GAT)進行有針對性地編輯。xCas9已被證明具有最廣泛的PAM識別能力,與SpCas9相比,xCas9表現出更高的DNA特異性,在全基因組范圍內所有NGG PAM的脫靶效應明顯降低[24]。xCas9具有靶向標準NGG PAM的能力,而Cas9-NG是植物基因編輯中識別非標準PAM的首選酶[25]。
3.2.3 ? ?Cas9n(Cas9 Nickase) and dCas9(Dead Cas9)。成對的Cas9n是Cas9的一個突變體,其中Cas9的HNH或RuvC結構域通過引入H840A或D10A改變而失活。成對的Cas9n由2個針對相鄰位點的sgRNA引導至靶標位點進行切割、插入[26]。Cas9n也被用作高頻堿基編輯的靶向模塊[27]。dCas9工作模式與Cas9n非常相似。由于堿基編輯不產生DSBs,Cas9n和dCas9都被用來通過靶向脫氨酶結構域來編輯特定的堿基。
3.2.4 ? ?Tru-gRNAs(Shorter/Truncated Guide RNAs for On-Target Site)。研究表明,gRNA由最初20 bp縮短至17~18 bp,基因編輯效率并未改變,而脫靶位修飾減少[26]。在植物中,在使用組成型啟動子的CRISPR/Cas9系統中使用截短的sgRNA會導致靶上編輯率增高,脫靶效應大大降低[27]。此外,將Tru-gRNAs與Cas9n相結合,進一步減少了脫靶突變[26]。
3.2.5 ? ?Cas9高保真突變體。①SpCas9-HF1(High-Fidelity Engineered Variants of SpCas9)。SpCas9-HF1是一種高保真的變體,旨在減少非特異性DNA相互作用。由于其非凡的準確性,可成為臨床治療的替代品[28]。②eSpCas9(Enhanced Specificity of SpCas9)。eSpCas9減少了脫靶效應并保持了有效的靶位編輯。對于需要高特異性的基因編輯工具(例如臨床醫學),eSpCas9非常實用。可通過合理設計生成的eSpCas9(K848A,K1003A,R1060A)減少與非目標序列的結合,提高基因編輯的特異性[29]。③HypaCas9(Hyper-Accurate Cas9 Variant)。HypaCas9系統能夠在不影響人類細胞目標基因組編輯的情況下實現更高的全基因組保真度。這個系統提供了一種平衡核酸酶激活和目標識別之間關系的改進方法,以實現精確的基因編輯[30]。
4 ? ?總結與展望
CRISPR/Cas9技術的出現為植物基因操作領域帶來了新的機遇。作為用于定點誘變的基因組編輯工具,CRISPR/Cas9滿足了人們的所有期待:高效率、高特異性、價格低廉、操作方便,這些是傳統誘變方法無法企及的。由于人們對CRISPR/Cas9系統了解相對較深,其構建方法已逐漸得到改善,同時研究者們正在嘗試將脫靶效應降至最低。所有這些優點使研究者迫切將其用于植物基因組學研究和作物改良,特別是在與產量、品質以及非生物和生物脅迫耐受性/抗性相關的特征方面。目前,脫靶效應仍是CRISPR/Cas9系統進一步廣泛應用的阻礙,還需要更多的研究來進一步提高靶標的特異性,以避免或減輕脫靶效應。
5 ? ?參考文獻
[1] HENRY R C,ENGSTR?魻M K,OLIN S,et al.Food supply and bioenergy production within the global cropland planetary boundary[J].PLoS One,2018,13(3):194695.
[2] HSU P D,LANDER E S,ZHANG F.Development and appli-cations of CRISPR/Cas9 for genome engineering[J].Cell,2014,157(6):1262-1278.
[3] MILLER J C,HOLMES M C,WANG J,et al.An improved zinc-finger nuelease architecture for highly specific genome editing[J].Nat Biotechnol,2007,25(7):778-785.
[4] CHRISTIAN M,CEMAK T,DOYLE E L,et al.Targeting DNA double-strand breaks with TAL effector nucleases[J].Genetics,2010,186(2):757-761.
[5] KNOTT G J,DOUDNA J A.CRISPR-Cas guides the future of genetic engineering[J].Science,2018,361(6405):866-869.
[6] WIEDENHEFT B,STERNBERG S H,DOUDNA J A.RNA-guided genetic silencing systems in bacteria and archaea[J].Nature,2012,482(7385):331-338.
[7] BAO A,BURRITT D J,CHEN H,et al.The CRISPR/Cas9 system and its applications in crop genome editing[J].Crit Rev Biotechnol,2019,39(3):321-336.
[8] ZHOU X,JACOBS T B,XUE L J,et al.Exploiting SNPs for biallelic CRISPR mutations in the outcrossing woody perennial Populus reveals 4-coumarate:CoA ligase specificity and redundancy[J].New Phytol,2015,208(2):298-301.
[9] MALZAHN A,LOWDER L,QI Y.Plant genome editing with TALEN and CRISPR[J].Cell Biosci,2017,7:21.
[10] ALKAN F,WENZEL A,ANTHON C,et al.CRISPR-Cas9 off-targeting assessment with nucleic acid duplex energy parameters[J].Genome Biol,2018,19(1):177.
[11] TANG X,LIU G,ZHOU J,et al.A large-scale whole-genome sequencing analysis reveals highly specific genome editing by both Cas9 and Cpf1(Cas12a)nucleases in rice[J].Genome Biol.,2018,19(1):84.
[12] YIN J,LIU M,LIU Y,et al.Optimizing genome editing strategy by primer-extension-mediated sequencing[J].Cell Discov,2019,5:18.
[13] MANGHWAR H,LI B,DING X,et al.CRISPR/Cas systems in genome editing:methodologies and tools for sgRNA design,off-target evaluation,and strategies to mitigate off-target effects[J].Adv Sci(Weinh),2020,7(6):1902312.
[14] BORTESI L,FISCHER R.The CRISPR/Cas9 system for plant genome editing and beyond[J].Biotechnol Adv,2015,33(1):41-52.
[15] JOUNG J K.Unwanted mutations:standards needed for gene-editing errors[J].Nature,2015,523(7559):158.
[16] LESSARD S,FRANCIOLI L,ALFOLDI J,et al.Human genetic variation alters CRISPR-Cas9 on-and off-targeting specificity at therapeutically implicated loci[J].Proc Natl Acad Sci U S A,2017,114(52):11257-11266.
[17] LIU H,DING Y,ZHOU Y,et al.CRISPR-P 2.0:an improved CRISPR-Cas9 tool for genome editing in plants[J].Mol Plant,2017,10(3):530-532.
[18] HEIGWER F,KERR G,BOUTROS M.E-CRISP:fast CRISPR target site identification[J].Nat Methods,2014,11(2):122-123.
[19] OLIVEROS J C,FRANCH M,TABAS-MADRID D,et al.Breaking-Cas-interactive design of guide RNAs for CRISPR-Cas experiments for ENSEMBL genomes[J].Nucleic Acids Res,2016,44(W1):W267-W271.
[20] N?魪RON B,M?魪NAGER H,TOUCHON M,et al.MacSyFind-er:a program to mine genomes for molecular systems with an application to CRISPR-Cas systems[J].PLoS One,2014,9(10):110726.
[21] KOMOR A C,KIM Y B,PACKER M S,et al.Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J].Nature,2016,533(7603):420-424.
[22] LI X,WANG Y,LIU Y,et al.Base editing with a Cpf1-cytidine deaminase fusion[J].Nat Biotechnol,2018,36(4):324-327.
[23] GAUDELLI N M,KOMOR A C,REES H A,et al.Program-mable base editing of A·T to G·C in genomic DNA without DNA cleavage[J].Nature,2017,551(7681):464-471.
[24] HU J H,MILLER S M,GEURTS M H,et al.Evolved Cas9 variants with broad PAM compatibility and high DNA specificity[J].Nature,2018,556(7699):57-63.
[25] ZHONG Z,SRETENOVIC S,REN Q,et al.Improving plant genome editing with high-fidelity xCas9 and non-canonical PAM-targeting Cas9-NG[J].Mol Plant,2019,12(7):1027-1036.
[26] RAN FA,HSU PD,LIN CY,et al.Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J].Cell,2013,154(6):1380-1389.
[27] QI L S,LARSON M H,GILBERT L A,et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J].Cell,2013,152(5):1173-1183.
[28] KLEINSTIVER B P,PATTANAYAK V,PREW M S,et al.High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects[J].Nature,2016,529(7587):490-495.
[29] SLAYMAKER I M,GAO L,ZETSCHE B,et al.Rationally engineered Cas9 nucleases with improved specificity[J].Science,2016,351(6268):84-88.
[30] CHEN J S,DAGDAS Y S,KLEINSTIVER B P,et al.Enhanced proofreading governs CRISPR-Cas9 targeting accuracy[J].Nature,2017,550(7676):407-410.

