基于DAE的單細胞RNA測序數據聚類研究
何慧茹 李曉峰 張鑫 柳楠


摘 ?要: 傳統數據降維方法處理單細胞RNA測序數據存在特征提取能力較差、聚類精度較低等問題,有必要引入深度學習方法以提高對復雜數據特征的提取能力。在對數據不進行任何人工篩選的條件下,利用DAE提取表達能力更強的數據特征,分別以K?means和DBSCAN聚類作為DAE的頂層設置形成DAE+K?means和DAE+DBSCAN組合模型,將這兩種深度學習組合模型在Deng數據集上與傳統聚類模型SC3進行對比。與SC3的0.73聚類精度相比,DAE+K?means和DAE+DBSCAN的聚類精度分別達到0.93和0.97,分別提高了0.2和0.24。實驗結果表明,DAE在單細胞聚類領域具有廣闊的應用前景。
關鍵詞: 單細胞聚類; 深度自動編碼器; 深度學習; K?means聚類; DBSCAN聚類; 結果分析
中圖分類號: TN919?34; TP391 ? ? ? ? ? ? ? ? ? 文獻標識碼: A ? ? ? ? ? ? ? ? ? ? ?文章編號: 1004?373X(2020)24?0144?05
Research on single?cell RNA sequencing data clustering based on DAE
HE Huiru, LI Xiaofeng, ZHANG Xin, LIU Nan
(School of Computer Science and Technology, Shandong Jianzhu University, Jinan 250101, China)
Abstract: As the traditional data dimension reduction method in processing single?cell RNA?sequencing data has some problems, such as poor feature extraction ability and low clustering accuracy, it is necessary to introduce the deep learning method to improve the extraction ability of complex data feature. Without any manual screening of data, the deep auto?encoder (DAE) is used to extract data feature with stronger expression ability. The K?Means and DBSCAN clustering is taken as the top?layer setting of DAE respectively to form DAE+K?Means or DAE+DBSCAN combined model, and the two deep learning combined models are compared with the traditional clustering model SC3 on Deng dataset. In comparison with the 0.73 clustering accuracy of SC3, the clustering accuracy of DAE+K?Means and DAE+DBSCAN reaches 0.93 and 0.97, respectively, which is improved by 0.2 and 0.24, respectively. The experimental results show that the DAE has a broad application prospect in the field of single?cell clustering.
Keywords: single cell clustering; DAE; deep learning; K?Means clustering; DBSCAN clustering; results analysis
0 ?引 ?言
基因表達是生物體生命活動的基礎和關鍵步驟,有效測定基因表達水平是研究生物體生長發育、發現新細胞亞群、探究腫瘤成因等生物學、醫學問題的基礎。近年來發展的基于高通量測序的單細胞RNA測序(single?cell RNA?sequencing, scRNA?seq)技術能夠在單個細胞粒度上進行基因表達測序[1],為辨別生物組織中不同類型細胞的基因表達特征和全面揭示細胞之間的異質性提供了強有力的技術支撐。
由于測序手段的限制以及基因表達高度復雜等原因,scRNA?seq數據普遍存在噪聲較大、維度較高、稀疏性較強等特點,僅靠人工難以挖掘出有價值的生物細胞信息。如何有效地處理數據、區分不同細胞亞群成為目前研究的熱點。
近年來國內外學者利用降維和聚類等方法對scRNA?seq數據進行了相關研究。常用的scRNA?seq數據降維方法主要有主成分分析[2?4]、零膨脹因子分析[5]、t?隨機鄰域嵌入[6]等;聚類方法主要有K?means[2,7]、層次聚類[3]等。
針對傳統scRNA?seq數據處理方法存在特征提取能力差、聚類精度低等問題,提出將深度自動編碼器(Deep Auto?Encoder,DAE)引入到scRNA?seq數據處理中。DAE作為數據特征提取常用的深度學習方法之一,在圖像分類[8?9]、短文本特征提取[10]、醫學診斷[11]等方面取得了良好效果。通過DAE對數據進行特征提取,利用得到的低維特征進行細胞聚類,提出DAE+K?means和DAE+DBSCAN兩種組合模型,提高了對scRNA?seq數據的聚類精度。
利用K?means對數據聚類時,首先需要確定聚類數量。本文利用組內平方誤差和(Sum of Squared Error, SSE)來確定最佳聚類數目。對于SSE和聚類數量[k]的二維圖像,圖像中的拐點所對應的聚類數量即為最佳聚類數量。SSE的計算公式為:
[SSE=i=1kx∈Eidist(ei,x)2] ? ? ? ? ? ?(4)
式中:[k]為聚類的數量;[Ei]為第[i]個簇;[ei]為簇[Ei]的聚類中心;[x]為樣本對象。通過計算SSE來確定聚類數量,可以進一步提高K?means的聚類精度。
2.4 ?DBSCAN聚類
DBSCAN(Density?Based Spatial Clustering of Application with Noise)是一種經典的基于密度的聚類算法,該算法可以發現任意形狀的簇,能夠有效識別噪聲點和離群點[13]。該算法需要確定Eps和MinPts兩個參數,其中,Eps是聚類簇的半徑,MinPts 是聚類簇內最少點數。與K?means聚類相比,DBSCAN聚類無需事先確定聚類數目,但是Eps和MinPts兩個參數對DBSCAN聚類的效果影響較大。
2.5 ?模型評估
本文所用數據集帶有實際類別標簽,為了便于與其他研究對比,使用調整蘭德系數(Adjusted Rand Index, ARI)作為聚類結果評價指標。ARI是一個通用的聚類評價指標,可以用來評估不同模型的聚類精度。ARI的取值范圍為[-1,1],值越大代表聚類效果越好。ARI的計算公式如下:
[ARI=RI-E(RI)max(RI)-E(RI)] ? ? ? ? ? (5)
式中:RI表示蘭德系數(Rand Index,RI);[E(RI)]表示RI的期望。RI的計算公式如下:
[RI=R+WR+M+D+W] ? ? ? ? ? ? (6)
式中:[R]為被聚在一類的2個對象被正確分類的樣本點對數;[W]為不應該聚在一類的2個對象被正確分開的樣本點對數;[M]為不應該聚在一類的樣本被放在一類的樣本點對數;[D]為應該聚在一類的樣本被錯誤分開的樣本點對數。
3 ?算例分析
3.1 ?數據集介紹
本文采用2014年Deng等人一組實驗數據[14]。該數據包含268個細胞,每個細胞在22 431個基因上的測序結果,具有維度高、稀疏性大,并且同一基因在不同細胞上的表達值差異較大等特點。
3.2 ?DAE模型搭建
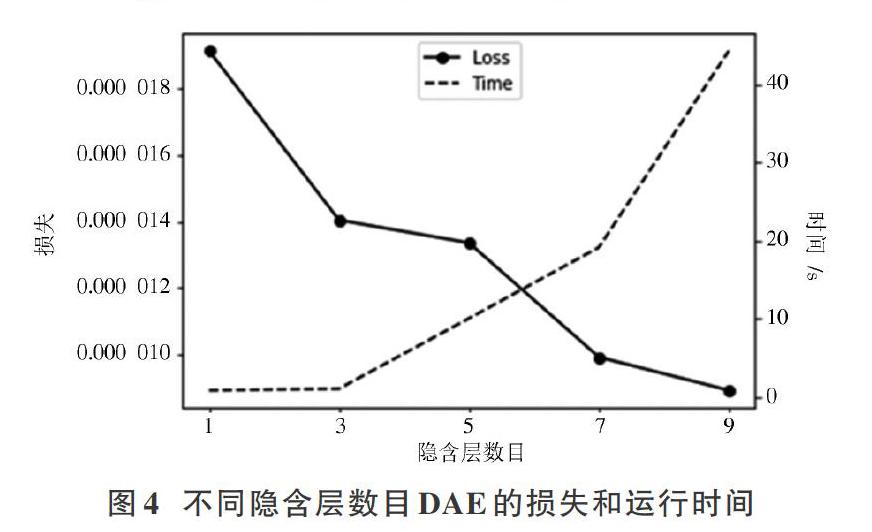
本文DAE模型基于TensorFlow的Keras庫實現。通過大量的實驗,最終構建的DAE網絡結構為22 431?100?6?100?22 431。其中,網絡輸入層和輸出層的神經元個數均為22 431,3個隱含層的神經元個數分別為100,6和100。本文在實驗階段分別嘗試了隱含層數目分別為1,3,5,7,9的情況,不同隱含層數目的損失和運行時間,如圖4所示。實線代表訓練損失,虛線代表網絡訓練需要的時間,以s為單位。當隱含層數目低于3個時,訓練速度較快但訓練損失相對較高,訓練效果不理想;當隱含層數目大于3個時,訓練損失相對較低但訓練時間相對較長;當隱含層數目為3,5時,兩者的訓練損失相差不大,但隱含層數目為5時的訓練速度明顯變慢。因此選擇隱含層數目為3的DAE進行對比實驗。
3.3 ?實驗結果分析
實驗數據樣本總數為268個,每個樣本的屬性個數為22 431維。對原始的scRNA?seq數據正則化處理之后,按照7∶3的比例隨機分成訓練集和測試集,利用訓練數據進行模型訓練,基于訓練好的DAE模型對測試數據進行降維。然后分別使用K?means和DBSCAN兩種聚類函數對DAE降維之后的測試數據進行聚類,與使用傳統降維方法SC3的聚類結果進行對比。
1) K?means聚類結果分析
利用SSE確定聚類簇數,測試集聚類簇數的確定過程如圖5所示。隨著簇數的增大,每一個類別中樣本數量越來越少,簇內距離越來越小,SSE值越來越小。但并不是簇數越多聚類效果越好,當SSE減小幅度緩慢時,即使進一步增大聚類簇數量也不能增強聚類效果。
由圖5可知,拐點為[k]=4,可以確定K?means的最佳聚類簇數為4。
為了進一步驗證SSE的結果,設計了4組對比實驗,分別計算簇數為3,4,5和6時的聚類精度,如表1所示。由于K?means的聚類中心是隨機產生的,為了排除一次性極端情況,本文在固定了1組網絡參數及聚類簇數的情況下進行了40次實驗,并取40次實驗輸出的平均值作為最終結果。
由表1可知,簇數的選擇對聚類性能有很大的影響。當簇數為4時,聚類性能最好,ARI為0.93,進一步證明了SSE確定數據聚類簇數目的有效性。
2) DBSCAN聚類結果分析
首先通過固定MinPts=3、改變Eps,進行了5組實驗,確定Eps的取值,實驗結果分析如表2所示。
由表2可知,當固定MinPts=3,Eps取值為0.33時,聚類性能最好,ARI為0.97。
最后在Eps=0.33的條件下進行了5組實驗來確定最優MinPts值。實驗結果分析如表3所示。
由表3可知,當固定Eps=0.33時,MinPts≤4時,聚類性能均為0.97,但當MinPts>4時,聚類性能變壞。
通過上述實驗可知,當Eps=0.33,MinPts=4時聚類性能最好,ARI為0.97。
3.4 ?聚類精度對比
3種模型的聚類精度如表4所示。
由表4可知,針對當前測試集,DAE+DBSCAN組合模型聚類性能相對較好,ARI為0.97;DAE+K?means組合模型的聚類性能略有下降,ARI為0.93。兩者差距為0.04,差距相對較小,進一步說明了2種模型在處理scRNA?seq數據中具有較高的可用性。但是隨著數據量的增加,K?means在聚類過程中會消耗更多的時間,而DBSCAN在對大數據聚類時效率更高。因此,針對不同的訓練集選擇合適的訓練模型還需根據任務的時間敏感度來決定。另一方面,DAE降維之后的數據聚類精度明顯優于直接利用SC3進行聚類的精度,這表明,在對scRNA?seq數據不進行篩選的情況下,利用深度學習的DAE模型提取基因表達信息更利于后續的單細胞聚類。2種組合模型的聚類精度較SC3都有所提升,最高提升0.24,進一步證明了深度學習在scRNA?seq數據處理方面的優越性。
4 ?結 ?論
特征的有效提取是影響聚類效果的主要因素,而深度學習強大的特征提取能力可以獲得后續細胞聚類所需的有效基因表達信息。目前,將深度學習技術應用于單細胞RNA測序數據聚類的研究相對較少,本文引入深度學習方法以提高對復雜數據特征的提取能力。實驗結果表明,DAE+DBASCN的聚類精度更高。得到這一結果的主要原因是原有方法并未對噪聲數據以及異常數據進行數據預處理,而DBSCAN算法能剔除噪聲、對噪聲數據不敏感,K?means算法對噪聲以及異常數據較敏感,因此K?means聚類精度略低于DBSCAN聚類精度。并且DBSCAN在對大數據進行聚類時效率更高,因而在數據規模較大且含有噪聲的聚類任務中,DAE+DBSCAN組合模型的效率相對較高。通過實驗對比進一步驗證了深度學習的DAE算法在單細胞聚類領域具有較好的應用前景。
參考文獻
[1] BUETTNER F, PRATANWANICH N, MCCARTHY D J, et al. F?scLVM: scalable and versatile factor analysis for single?cell RNA?seq [J]. Genome biology, 2017, 18(1): 212?224.
[2] KISELEV V Y, KIRSCHNER K, SCHAUB M T, et al. SC3: consensus clustering of single?cell RNA?seq data [J]. Nature methods, 2017, 9(3): 384?395.
[3] ZURAUSKIENE, JUSTINA, YAU C. PcaReduce: hierarchical clustering of single cell transcriptional profiles [J]. BMC bioinformatics, 2016, 17(1): 140?150.
[4] SHIN J, BERG D, ZHU Y, et al. Single?cell RNA?seq with waterfall reveals molecular cascades underlying adult neurogenesis [J]. Cell stem cell, 2015, 17(3): 360?372.
[5] PIERSON E, YAU C. ZIFA: dimensionality reduction for zero?inflated single?cell gene expression analysis [J]. Genome biology, 2015, 16(1): 241?250.
[6] ZEISEL A, SIMONE C, PETER L, et al. Cell types in the mouse cortex and hippocampus revealed by single?cell RNA?seq [J]. Science, 2015, 347(6226): 1138?1142.
[7] GRUN D, LYUBIMOVA A, KESTER L, et al. Single?cell messenger RNA sequencing reveals rare intestinal cell types [J]. Nature, 2015, 56: 251?255.
[8] SU Y C, LI J, PLAZA A, et al. Deep auto?encoder network for hyperspectral image unmixing [C]// 2018 IEEE International Geoscience and Remote Sensing Symposium. Valencia: IEEE, 2019: 4309?4321.
[9] 宮浩,張秀再,胡敬鋒.一種基于深度學習的遙感圖像分類及農田識別方法[J].現代電子技術,2019,42(8):179?182.
[10] 譚夢婕,呂鑫,陶飛飛.基于多特征融合的財經新聞話題檢測研究[J].計算機工程,2019,45(3):293?299.
[11] RONG W G, NIE Y F, OUYANG Y X, et al. Auto?encoder based bagging architecture for sentiment analysis [J]. Journal of visual languages & computing, 2014, 25(6): 840?849.
[12] 謝娟英,王艷娥.最小方差優化初始聚類中心的K?means算法[J].計算機工程,2014,40(8):205?211.
[13] 胡健,朱海灣,毛伊敏.基于自適應蜂群優化的DBSCAN聚類算法[J].計算機工程與應用,2019,55(14):105?114.
[14] DENG Q, RAMSKOLD D, REINIUS B, et al. Single?cell RNA?seq reveals dynamic, random monoallelic gene expression in mammalian cells [J]. Science, 2014, 343: 193?196.
作者簡介:何慧茹(1995—),女,山東濟寧人,碩士研究生,研究方向為計算生物、深度學習。
李曉峰(1971—),男,山東臨沂人,博士,教授,碩士生導師,研究方向為計算生物、數據挖掘、深度學習。

