宜昌臘腸與恩施臘腸細菌多樣性的比較分析
周亞澳 黃怡 王韻博 郭壯 張振東 李楊坤 侯強川
摘 要:采用Illumina MiSeq高通量測序技術對宜昌臘腸細菌多樣性進行解析,并與之前報道的恩施臘腸細菌群落結構進行比較分析。結果表明:宜昌臘腸在門水平主要由厚壁菌門(Firmicutes,64.93%)、變形菌門(Proteobacteria,32.12%)和放線菌門(Actinobacteria,1.14%)組成;在屬水平上,主要由葡萄球菌屬(Staphylococcus,16.57%)、乳酸桿菌屬(Lactobacillus,16.22%)、魏斯氏菌屬(Weissella,14.67%)和明串珠菌屬(Leuconostoc,11.43%)等組成。宜昌臘腸細菌豐度顯著低于恩施臘腸(P<0.05),且2 個地區臘腸的細菌菌群結構存在較大差異。基因功能預測結果表明,宜昌臘腸中細菌在碳水化合物代謝和多糖的生物合成與代謝等方面更加旺盛,這些差異與臘腸中乳酸桿菌的相對豐度存在顯著相關性。
關鍵詞:臘腸;高通量測序;微生物群落結構;功能預測
Abstract: The bacterial diversity in Yichang sausage was analyzed by Illumina MiSeq high-throughput sequencing technique and compared with that previously reported for Enshi sausage. The results showed that the bacterial community in Yichang sausages were mainly composed of Firmicutes (64.93%), Proteobacteria (32.12%) and Actinobacteria (1.14%) at the phylum level. At the genus level, it was mainly composed of Staphylococcus (16.57%), Lactobacillus (16.22%), Weissella (14.67%), and Leuconostoc (11.43%). The abundance of bacteria in Yichang sausages was significantly lower than that in Enshi sausage (P < 0.05). Besides, there were great differences in the bacterial structure between the two sausages. The results of gene function prediction showed that bacteria in Yichang sausages were more vigorous in carbohydrate metabolism, and polysaccharide biosynthesis and metabolism, and these differences were significantly correlated with the relative abundance of Lactobacillus in sausages.
Keywords: sausages; high throughput sequencing; microbial community structure; function prediction
DOI:10.7506/rlyj1001-8123-20200711-169
中圖分類號:TS251.5? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼:A 文章編號:1001-8123(2020)10-0008-06
臘腸作為我國一種傳統的發酵肉制品,擁有悠久的制作歷史,現廣泛分布于我國華東、華中和華南等地區[1]。臘腸的制作主要以豬肉為原料,同時輔以食鹽、八角、花椒和白酒等調味料進行腌制,灌入腸衣后進行晾曬發酵[2]。因其風味獨特、口感醇厚而深受廣大消費者的喜愛[3]。
宜昌市位于湖北省西南部,地處長江上游與中游結合部,其氣候為臘腸的發酵提供了有利條件。同時宜昌市是一個多民族雜居的城市,這在一定程度促進了傳統臘腸制作工藝的融合發展。傳統臘腸的制作環境較為粗獷,不同的地理環境和氣候條件可能會賦予臘腸不同的微生物群系[4]。目前,一些學者針對不同品質、原料及發酵和貯藏階段臘腸微生物群落組成和差異開展一些研究,滕安國等[5]發現乳桿菌屬(Lactobacillus)和明串珠菌屬(Leuconostoc)為正常臘腸和腸衣中的優勢菌屬,而臘腸發黏變質后微生物組成發生變化,菌群多樣性降低;劉長建等[6]從臘腸中分離得到多株具有降膽固醇能力的乳酸菌,其中干酪乳桿菌(Lactobacillus casei)的降膽固醇效果最好;田甜等[7]發現在廣式臘腸發酵過程中添加復合發酵劑能加快發酵進程,并降低亞硝酸鹽含量。Fougy等[8]發現減少臘腸中的食鹽添加量會導致腐敗菌生長和細菌多樣性下降;Li Xinfu等[9]通過高通量測序技術解析真空包裝臘腸貯藏過程中微生物群落的變化。此外,越來越多的研究表明,臘腸的發酵菌群直接影響著發酵的進程和產品品質,郭壯等[10]研究發現臘腸中的乳酸菌對產品發酵成熟和風味品質的形成具有至關重要的作用;黃金枝[11]研究發酵菌種與廣式臘腸產品品質的關系,發現戊糖片球菌(Pediococcus pentosaceus)對廣式臘腸發酵品質的影響大于漢遜德巴利酵母(Saccharomyces)和植物乳桿菌(Lactobacillus plantarum)。上述研究的結果表明,臘腸品質與其細菌組成之間存在密切聯系。然而,目前鮮有研究報道宜昌地區臘腸細菌多樣性及其與其他地區臘腸菌群結構的差異,這在一定程度上限制了宜昌地區臘腸品質的提升。
近年來,伴隨分子生物學的發展,越來越多的基于非培養的微生物檢測方法和技術被廣泛應用于發酵食品微生物檢測中。其中,Illumina MiSeq高通量測序技術因其檢測速率快、通量大和結果可信度高等優點[12-13],在海洋、土壤、發酵食品和腸道等環境樣品微生物多樣性的研究中得到廣泛應用[14-15]。鄧風等[16]采用聚合酶鏈式反應(polymerase chain reaction,PCR)-變性梯度凝膠電泳與Illumina MiSeq高通量測序技術相結合的方法研究恩施地區臘腸中的細菌群落結構,發現恩施臘腸樣品中含有大量的共有細菌類群,優勢細菌屬主要包括環絲菌屬(Brochothrix)、葡萄球菌(Staphylococcus)、嗜冷桿菌(Psychrobacter)等,同時恩施臘腸所有測序序列中無法鑒定到屬水平的僅為13.72%。該研究結果進一步表明Illumina MiSeq高通量測序可以作為解析臘腸細菌群落結構的有效技術手段。
本研究采用Illumina MiSeq高通量測序技術對采集自宜昌地區的臘腸細菌多樣性進行解析。此外,通過整合恩施臘腸測序數據[17],結合多元統計學分析和功能預測等手段比較分析2 個地區臘腸細菌菌群結構和功能的差異。以期進一步豐富人們對不同地區臘腸細菌群落結構的認識。
1 材料與方法
1.1 材料與試劑
臘腸采集自湖北省宜昌市五峰土家族自治縣菜市場,共計9 份,編號記為YC1~YC9,所有樣品均為手工制作,且滿足以下標準:1)臘腸產品在宜昌地區生產制作完成;2)臘腸使用豬肉作為制作原料;3)臘腸產品無霉變、無異味且無明顯雜質存在。樣品采集時使用無菌手術刀切取約100 g裝入無菌采樣袋,密封后置于采樣箱中,低溫冷藏運回實驗室進行后續操作。
338F/806R引物 武漢天一輝遠生物科技有限公司;DNeasy Mericon Food Kit 德國Qiagen公司;FastPfu Fly DNA Polymerase、5×TransStart? FastPfu緩沖液和dNTPs Mix 北京全式金生物技術有限公司。
1.2 儀器與設備
Vetiri梯度基因擴增儀 美國AB SCIEX公司;ND-2000C微量紫外分光光度計 美國Nano Drop公司;Illumina MiSeq PE300高通量測序平臺 美國Illumina公司;R960機架式服務器 美國Dell公司;UVPCDS8000凝膠成像分析系統、DCode? System 美國Bio-Rad公司;CR21N型高速離心機 日本日立金屬株式會社。
1.3 方法
1.3.1 樣品預處理及微生物宏基因組DNA提取
將采集的臘腸切碎后,稱取5 g加入到45 mL生理鹽水中,使用拍擊器拍擊5 min,400 r/min離心8 min后取上清液,于12 000 r/min離心8 min,取沉淀備用[16]。使用基因組提取試劑盒提取沉淀物宏基因組DNA。
1.3.2 PCR擴增和Illumina MiSeq測序
使用338F/806R引物(帶有7 個堿基核苷酸標簽)對細菌16S rRNA V3~V4區進行PCR擴增,擴增條件參照王玉榮等[17]方法。使用1.00%的瓊脂糖凝膠電泳和紫外分光光度計對擴增產物的純度和濃度進行檢驗,將合格DNA產物送至上海美吉生物醫藥科技有限公司進行測序。
1.3.3 生物信息學分析
依照郭壯等[18]研究中序列質控所約束的參數對下機序列進行質量控制,并依照核苷酸標簽進行序列的分配,將質控后的序列分配到不同的樣本中。參照Yang Chengcong等[19]的方法,使用QIIME V1.9.1平臺對宜昌和恩施臘腸中的細菌組成和多樣性進行解析。具體的生物學分析過程如下:1)采用PyNAST軟件對序列數據進行比對和校準[20];2)使用UCLUST分別通過100%和97%相似度對校準后的序列進行劃分[21],分別得到非冗余數據集和操作分類單元(operational taxonomic units,OTU);3)使用ChimeraSlayer軟件識別OTU中的嵌合體序列[22],并對相應的嵌合體序列進行刪除;4)挑選每個OTU中的代表性序列分別使用Greengene(Version 13.8)[23]、RDP(Relace 11.5)[24]和Sliva(Version 132)數據庫[25]進行同源性比對,確定各OTU的分類學地位,使用內部腳本整合3 個數據庫的注釋結果,確定各OTU的最終分類學地位;5)使用FastTree軟件構建基于OTU代表性序列的系統發育樹[26];6)依據最大序列數計算每個臘腸樣本的α多樣性指數(Chao 1指數、Shannon指數、Observed species指數和Simpson指數);7)依據最小序列數計算樣品間的β多樣性。
使用PICRUSt(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)軟件對臘腸中細菌微生物的基因功能進行預測[27],并參照京都基因與基因組百科全書(Kyoto Encyclopedia of Genes and Genomes,KEGG)數據庫進行基因功能注釋。
1.3.4 多元統計學分析
使用非參數的Mann-Whitney秩和檢驗對不同地區臘腸樣品菌群組成進行差異顯著性分析;使用LefSe軟件分析并基于LDA Effect Size判別組間豐度有顯著差異的微生物;基于Pearson相關性分析臘腸中優勢菌屬和差異代謝通路之間的相關性。使用R軟件(v3.5.0)、STAMP軟件和Origin 2019b軟件進行數據可視化處理。
2 結果與分析
2.1 宜昌臘腸序列豐富度和多樣性分析
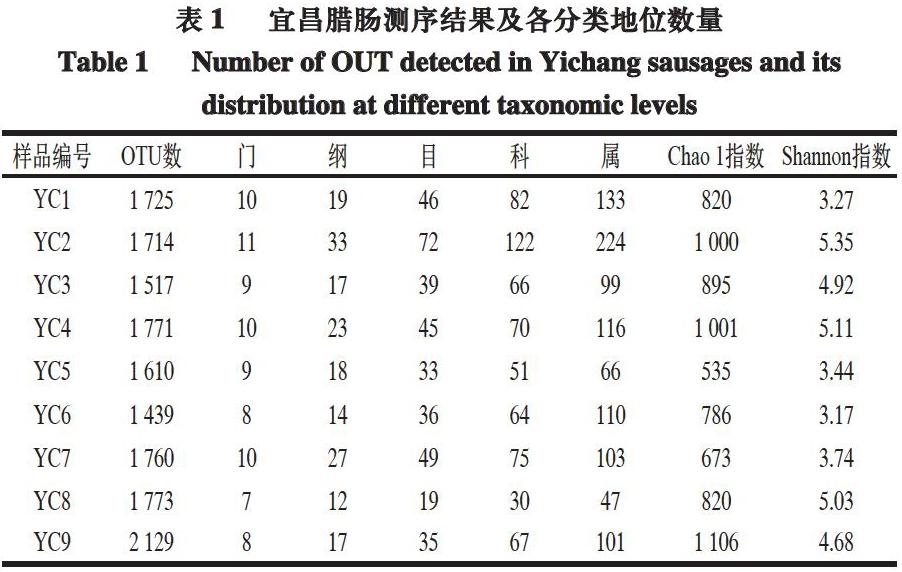
宜昌臘腸所有樣品共測得序列427 570 條,每個樣品的平均測序量為26 009 條(12 215~40 828,標準偏差3 192),上述序列在97%相似度劃分得到5 330 個非嵌合體OTU,每個樣本平均OUT數為931。
由表1可知,YC9臘腸的OTU數和Chao 1指數最大,而YC2臘腸的Shannon指數最大。通過對OTU數、Chao 1指數和Shannon指數的變異系數進行計算發現,宜昌臘腸在α多樣性指數上存在較大的組內差異。相關研究顯示,Chao 1指數和Shannon指數常分別用來評估環境中微生物的豐度和多樣性[28]。表明YC9臘腸中微生物豐度最高,而YC2臘腸的微生物多樣性最高,且宜昌臘腸中的微生物豐度和多樣性存在較大差異。
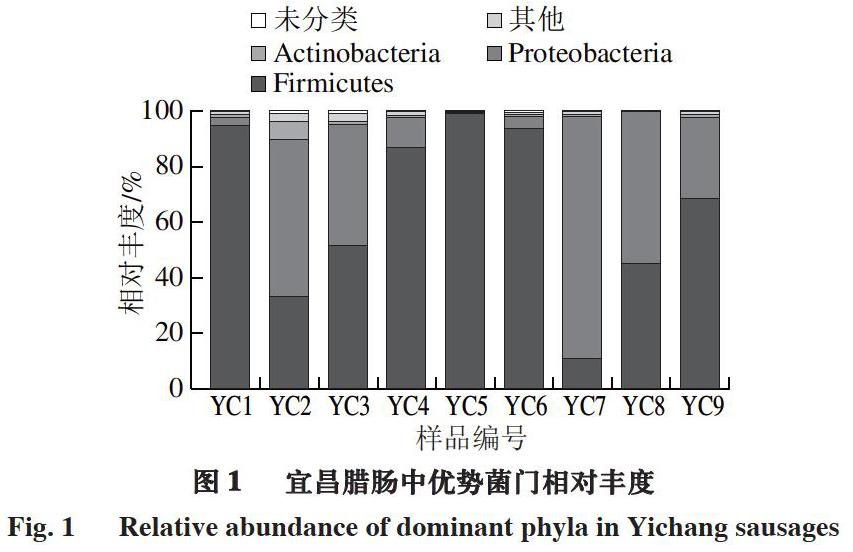
進一步分析宜昌臘腸中的細菌菌落組成,所有樣品共注釋得到15 個菌門。由圖1可知,宜昌臘腸中優勢菌門(平均相對豐度大于1.00%)共有3 個,分別為厚壁菌門(Firmicutes)、變形菌門(Proteobacteria)和放線菌門(Actinobacteria),平均相對豐度分別為64.93%、32.12%和1.14%。可見,宜昌臘腸中的主要菌門為厚壁菌門和變形菌門,這與鄧風等[16]研究得到的恩施臘腸細菌群落結構結果基本一致。
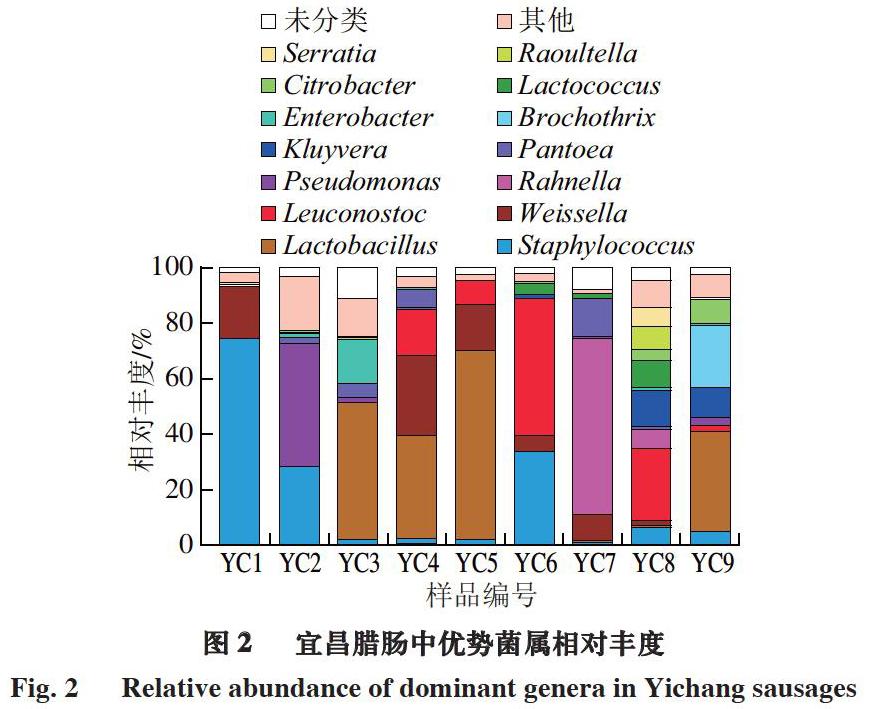
本研究進一步在屬水平對宜昌臘腸細菌群落結構進行分析,由圖2可知,宜昌臘腸中優勢菌屬(平均相對豐度大于1.00%)共有14 個,其中葡萄球菌屬(Staphylococcus,16.57%)、乳酸桿菌屬(Lactobacillus,16.22%)、魏斯氏菌屬(Weissella,14.67%)、明串珠菌屬(Leuconostoc,11.43%)和環絲菌屬(Brochothrix,2.54%)隸屬于厚壁菌門;拉恩菌屬(Rahnella,7.91%)、假單胞菌屬(Pseudomonas,5.74%)、泛菌屬(Pantoea,3.16%)、克呂沃爾氏菌屬(Kluyvera,2.76%)、腸桿菌屬(Enterobacter,2.21%)、乳球菌屬(Lactococcus,1.67%)、檸檬酸桿菌屬(Citrobacter,1.43%)、拉烏爾菌屬(Raoultella,1.21%)和沙雷菌屬(Serratia,1.14%)隸屬于變形菌門。可見,宜昌臘腸中相對豐度最高的是葡萄球菌屬和乳酸桿菌屬,這與恩施臘腸中的優勢菌屬不盡相同,后者優勢菌屬除葡萄球菌屬和乳酸桿菌屬外,還包括環絲菌屬,三者在樣品中的平均相對豐度分別為9.79%、2.80%和38.34%[16]。宜昌臘腸中細菌主要以乳酸菌為主,其平均相對豐度接近60%(主要以乳酸桿菌屬、魏斯氏菌屬和明串珠菌屬為主)。乳酸菌作為發酵肉制品中常見的發酵菌種,在一定程度上決定著發酵制品最終品質。值得注意的是,本研究在宜昌臘腸中檢測到大量的機會性致病微生物如葡萄球菌屬和沙雷菌屬等,提示需要進一步規范化臘腸制作過程的安全管理和質量控制。
2.2 宜昌和恩施臘腸中細菌群落結構的比較分析
由圖3可知,宜昌臘腸中細菌的Chao 1指數極顯著低于恩施臘腸(P<0.01),Observed species指數顯著低于恩施臘腸(P<0.05),Shannon指數低于恩施臘腸,且差異不顯著,而Simpson指數略高于恩施臘腸。Chao 1指數和Observed species指數常用于評估環境樣本中微生物的豐度,Shannon指數和Simpson指數常用于評估環境中微生物的多樣性[28]。可見,宜昌臘腸中微生物的豐度明顯低于恩施臘腸,而多樣性沒有明顯差異。
本研究進一步通過基于非加權和加權UniFrac距離的主坐標分析比較2 個地區臘腸菌群的組間差異。由基于非加權UniFrac距離的主坐標分析結果(圖4A)可知,宜昌和恩施臘腸的微生物菌群在空間排布上呈現明顯的分離趨勢,宜昌臘腸和恩施臘腸分別位于X軸的正方向和負方向,多元方差分析結果也表明二者的微生物群落結構差異顯著(P<0.05);而基于加權UniFrac距離的分析結果(圖4B)表明,雖然2 個地區的臘腸樣品整體存在分離趨勢,但部分樣品在空間排布上出現一定的重疊。上述結果表明,宜昌和恩施臘腸中優勢菌群組成較為相似,但二者均含有較多獨特的低豐度菌群,這部分菌群對臘腸的整體菌群結構產生較大影響。
為揭示宜昌和恩施臘腸的菌群結構差異,通過LefSe分析比較兩地區臘腸存在顯著差異的菌群。由圖5可知,共有7 個細菌類群(判別得分>3)在2 組臘腸樣品中存在顯著差異(P<0.05),其主要為果膠桿菌屬(Pectobacterium)、腸桿菌屬和假單胞菌屬,值得注意的是,甄別出的所有差異菌均在恩施臘腸中的相對豐度顯著較高(P<0.05)。相關報道顯示,腸桿菌屬和假單胞菌屬廣泛存在于傳統發酵食品中,能夠有效促進發酵制品的發酵進程,其中假單胞菌屬與發酵制品的顏色密切相關,且對酪胺和組胺的產生具有一定的影響[29]。同時,發酵制品中的菌群結構與地理環境、制作工藝和原材料等有直接的關系,不同菌群結構可能是造成不同地區臘腸色澤、滋味和風味差異的主要原因之一。通過對不同地區發酵制品中微生物多樣性進行解析和菌株的收集及保藏,將對傳統發酵食品的工業化和個性化生產具有重要意思。
2.3 PICRUSt功能預測
本研究利用PICRUSt軟件對宜昌和恩施臘腸中細菌類群的基因功能進行預測,使用KEGG數據庫對預測基因進行功能注釋,在此基礎上對差異代謝通路進行分析。Z值是用于衡量樣本均值偏離整體均值的方差倍數,當用于差異代謝通路查找時,Z值越大,則相應的代謝通路在兩組間的差異越大。本研究選擇Z值>1.6(95%置信區間)[30],同時代謝通路中的基因在某一地區樣品高表達概率>80%作為判定2 個地區差異代謝通路的條件。
由圖6A可知,宜昌和恩施臘腸中的細菌菌群在4 條代謝通路上存在顯著差異,且所有差異代謝通路均隸屬于與代謝相關的一級功能層。其中與碳水化合物和多糖生物合成及代謝的相關基因在宜昌臘腸微生物中顯著富集,而與能量代謝和氨基酸代謝的相關基因在恩施臘腸微生物中顯著富集。由圖6B可知,魏斯氏菌屬與氨基酸代謝呈顯著負相關(P<0.05),而乳酸桿菌屬與多糖生物合成及代謝呈顯著正相關(P<0.05),與碳水化合物代謝呈極顯著正相關(P<0.01)。可見,宜昌地區臘腸中微生物對碳水化合物的代謝更為旺盛,并且這與臘腸中的乳酸桿菌密切相關。乳酸桿菌屬對碳水化合物的利用效率受到環境的溫度、濕度等因素的強烈影響[31]。宜昌地區相較恩施地區空氣濕度更低,且平均溫度更高,上述環境因素均有利于臘腸中乳酸桿菌的生長和代謝,這可能是宜昌臘腸中微生物碳水化合物代謝相關基因富集的原因之一。亮氨酸和異亮氨酸均為人體的必需氨基酸,由于人體無法合成,因此必須從飲食中攝取。恩施臘腸中微生物亮氨酸和異亮氨酸生物合成通路更為活躍,提示該地區生產的臘腸中這2 種必需氨基酸的含量可能會更為豐富。
3 結 論
本研究采用Illumina MiSeq高通量測序技術對湖北宜昌臘腸中的細菌多樣性進行解析,并和恩施臘腸進行比較分析。結果表明,宜昌臘腸中的細菌主要隸屬于厚壁菌門、變形菌門和放線菌門,總相對豐度98.19%;而優勢菌屬主要以葡萄球菌屬、乳酸桿菌屬、魏斯氏菌屬和明串珠菌屬等為主。宜昌臘腸中的微生物豐度顯著低于恩施臘腸,且果膠桿菌屬、腸桿菌屬和假單胞菌屬等菌屬在宜昌地區的相對豐度顯著低于恩施地區。功能預測結果表明,宜昌和恩施地區臘腸與代謝相關的通路存在差異顯著,宜昌臘腸中細菌碳水化合物代謝和多糖生物合成與代謝更加旺盛,這些差異可能與2 個地區的氣候不同存在一定關系。
參考文獻:
[1] 龍強, 聶乾忠, 劉成國. 發酵肉制品功能性發酵劑研究現狀[J]. 食品科學, 2016, 37(17): 263-269. DOI:10.7506/spkx1002-6630-201617044.
[2] 楊江, 楊成聰, 凌霞, 等. 基于電子鼻和氣相色譜-質譜聯用技術評價襄陽地區臘腸風味品質[J]. 肉類研究, 2018, 32(8): 46-50. DOI:10.7506/rlyj1001-8123-201808008.
[3] 劉琨毅, 王琪, 王衛, 等. 茶多酚對低鹽中式臘腸防腐保鮮的影響[J]. 肉類研究, 2018, 32(3): 34-39. DOI:10.7506/rlyj1001-8123-201803007.
[4] 杜莎, 李柯, 黃晴, 等. 人工發酵劑對湘西臘腸微生物菌群和理化特性的影響[J]. 現代食品科技, 2017, 33(4): 241-247. DOI:10.13982/j.mfst.1673-9078.2017.4.037.
[5] 滕安國, 齊曉娜, 張芹, 等. 高通量測序技術分析天然腸衣香腸的菌群組成[J]. 食品研究與開發, 2016, 37(11): 113-116. DOI:10.3969/j.issn.1005-6521.2016.11.027.
[6] 劉長建, 蔣本國, 姜波, 等. 臘腸中降膽固醇乳酸菌的篩選及鑒定[J]. 中國釀造, 2011, 30(8): 35-37. DOI:10.3969/j.issn.0254-5071.2011.08.011.
[7] 田甜, 詹銳琪, 張雅琳, 等. 微生物發酵技術對廣味香腸安全性提升的研究[J]. 中國調味品, 2019, 44(12): 26-30. DOI:10.3969/j.issn.1000-9973.2019.12.006.
[8] FOUGY L, DESMONTS M, COEURET G, et al. Reducing salt in raw pork sausages increases spoilage and correlates with reduced bacterial diversity[J]. Applied and Environmental Microbiology, 2016, 82(13): 3928-3939. DOI:10.1128/aem.00323-16.
[9] LI Xinfu, LI Cong, YE Hua, et al. Changes in the microbial communities in vacuum-packaged smoked bacon during storage[J]. Food Microbiology, 2019, 77(2): 26-37. DOI:10.1016/j.fm.2018.08.007.
[10] 郭壯, 王玉榮, 葛東穎, 等. 臘腸發酵過程中細菌多樣性評價及其對風味的影響[J].食品科學, 2020. DOI:10.7506/spkx1002-6630-20191121-255.
[11] 黃金枝. 發酵廣式臘腸加工菌種優選及品質變化研究[D]. 南昌: 江西農業大學, 2015.
[12] LIANG Huipeng, YIN Liguo, ZHANG Yahao, et al. Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai, a traditional Chinese fermented vegetable food, as assessed by Illumina MiSeq sequencing, DGGE and qPCR assay[J]. Annals of Microbiology, 2018, 68(2): 111-122. DOI:10.1007/s13213-017-1321-z.
[13] SCHIRMER M, IJAZ U Z, DAMORE R, et al. Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J]. Nucleic Acids Research, 2015, 43(6): e37. DOI:10.1093/nar/gku1341.
[14] WALSH A M, FIONA C, KIERAN K, et al. Microbial succession and flavor production in the fermented dairy beverage kefir[J]. mSystems, 2016, 1(5): 2379-5077. DOI:10.1128/mSystems.00052-16.
[15] HE Guoqing, LIU Tongjie, ASADIQ F, et al. Insights into the microbial diversity and community dynamics of Chinese traditional fermented foods from using high-throughput sequencing approaches[J]. Journal of Zhejiang University-Science B, 2017, 18(4): 289-302. DOI:10.1631/jzus.b1600148.
[16] 鄧風, 王玉榮, 尚雪嬌, 等. 恩施地區臘腸的細菌多樣性[J]. 肉類研究, 2018, 32(9): 18-22. DOI:10.7506/rlyj1001-8123-201809004.
[17] 王玉榮, 沈馨, 董蘊, 等. 鲊廣椒細菌多樣性評價及其對風味的影響[J]. 食品與機械, 2018, 34(4): 25-30. DOI:10.13652/j.issn.1003-5788.2018.04.005.
[18] 郭壯, 葛東穎, 尚雪嬌, 等. 退化和正常窖泥微生物多樣性的比較分析[J]. 食品工業科技, 2018, 39(22): 93-98. DOI:0.13386/j.issn1002-0306.2018.22.018.
[19] YANG Chengcong, ZHAO Feiyan, HOU Qiangchuan, et al. PacBio sequencing reveals bacterial community diversity in cheeses collected from different regions[J]. Journal of Dairy Science, 2020, 103(2): 1238-1249. DOI:10.3168/jds.2019-17496.
[20] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al. PyNAST: a flexible tool for aligning sequences to a template alignment[J]. Bioinformatics, 2010, 26(2): 266-267. DOI:10.1093/bioinformatics/btp636.
[21] EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10: 996. DOI:10.1038/nmeth.2604.
[22] EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381.
[23] DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006, 72(7): 5069-5072. DOI:10.1128/aem.03006-05.
[24] MAIDAK B L, COLE J R, LILBURN T G, et al. The RDP (ribosomal database project) continues[J]. Nucleic Acids Research, 2000, 28(1): 173-174. DOI:10.1093/nar/28.1.173.
[25] MONIKA B, HUSON D H. SILVA, RDP, Greengenes, NCBI and OTT- how do these taxonomies compare?[J]. BMC Genomics, 2017, 18(Suppl 2): 114. DOI:10.1186/s12864-017-3501-4.
[26] PRICE MORGAN N, DEHAL PARAMVIR S, ARKIN ADAM P. FastTree: computing large minimum evolution trees with profiles instead of a distance matrix[J]. Molecular Biology and Evolution, 2009, 26(7): 1641-1650. DOI:10.1093/molbev/msp077.
[27] WILKINSON T J, HUWS S A, EDWARDS J E, et al. CowPI: a rumen microbiome focussed version of the PICRUSt functional inference software[J]. Frontiers in Microbiology, 2018, 9(5): 1095. DOI:10.3389/fmicb.2018.01095.
[28] 姚國強. 傳統發酵乳中細菌多樣性及其功能基因研究[D]. 呼和浩特: 內蒙古農業大學, 2017.
[29] XIE Mengxi, AN Feiyu, ZHAO Yue, et al. Metagenomic analysis of bacterial community structure and functions during the fermentation of da-jiang, a Chinese traditional fermented food[J]. LWT-Food Science and Technology, 2020, 30(4): 109450. DOI:10.1016/j.lwt.2020.109450.
[30] STADLMAYR A, FENG Qiang, HUBER-SCH?NAUER U, et al. Gut microbiome development along the colorectal adenoma-carcinoma sequence[J]. Nature Communications, 2015, 6: 6528. DOI:10.1038/ncomms7528.
[31] 張筠, 孟祥晨. 乳酸菌的脅迫應答及其對碳水化合物代謝的影響[J].?中國食品學報, 2017, 17(6): 145-151. DOI:10.16429/j.1009-7848.2017.06.020

