楸樹葉綠體基因組密碼子偏性分析
李鳳 辛靜 辛雅萱 肖遙 屈亞亞 王軍輝 麻文俊 辛培堯

摘要:【目的】分析楸樹葉綠體基因組密碼子使用偏性,為楸樹乃至梓屬植物葉綠體基因組學、物種進化特征及遺傳多樣性分析等研究提供理論依據。【方法】利用Codon W 1.4.2及EMBOSS expiorer的cusp和chips軟件對51個楸樹葉綠體基因的編碼區(CDS)序列進行密碼子相關參數、中性繪圖、ENC-plot和PR2-plot等分析,并通過構建高、低表達偏性基因庫,以ENC值和RSCU值為參考標準,最終篩選最優密碼子。【結果】51個基因密碼子的平均GCall含量為38.48%,密碼子不同位置GC含量為:GC1(47.03%)>GC2(39.93%)>GC3(28.09%),說明第3位的GC并非均勻分布;ENC為34.93~56.50,平均為46.87。GC2與GC1呈極顯著正相關(P<0.01),而GC3與GC1和GC2無顯著正相關(P>0.05);ENC與GCall、GC1、GC3和N均呈顯著正相關(P<0.05,下同);密碼子數(N)與GC3和ENC呈顯著正相關。RSCU>1的密碼子共有30個,其中,以A或U結尾的密碼子有29個。中性繪圖分析結果顯示,GC3與GC12相關系數為0.0188,二者無顯著相關性,回歸系數為0.143。ENC-plot分析結果顯示,大部分基因偏離了ENC預期標準曲線,少數分布在預期曲線附近;有7個密碼子分布在GC3含量-0.05~0.15區間,其余區間共有44個密碼子,而這44個基因離ENC標準曲線較遠。PR2-plot分析結果顯示,落在中線上的基因較少,平面圖右下方的基因分布較多,表明在各堿基使用頻率方面存在偏性,即T>A,G>C。各堿基使用頻率存在偏性(T>A,G>C),說明基因組密碼子的偏性更多受選擇效應的影響。最終篩選出7個為最優密碼子UUU、CUU、CCU、AAA、GAU、CGA和GGU。【結論】楸樹葉綠體基因組密碼子偏性較弱,偏好使用A或U結尾的密碼子,主要受選擇壓力的影響,受突變等其他因素影響較小。
關鍵詞: 楸樹;葉綠體基因組;密碼子偏好性;最優密碼子
中圖分類號:S792.99? ? ? ? ? ? ? ? ? ? ? ? ? ? 文獻標志碼: A 文章編號:2095-1191(2021)10-2735-09
Abstract:【Objective】Analyzed the codon usage bias of Catalpa bungei C. A. Mey. chloroplast genome to provide theoretical basis for the research of C. bungei and even Catalpa chloroplast genomics,species evolution characteristics and genetic diversity analysis. 【Method】Codon correlation parameters,neutral plots,ENC-plot and PR2-plot were analyzed for 51 chloroplast gene coding regions (CDS) sequences of C. bungei by Codon W 1.4.2 and cusp and chips of EMBOSS expiorer. And through the constitutive high and low expression-biased gene library, ENC value and RSCU value were used as reference criteria to screen the optimal codon. 【Result】The average GCall content of 51 gene codons was 38.48%,and GC content at different positions of codons was as follows: GC1(47.03%)>GC2(39.93%)>GC3(28.09%),indicating that GC at the third position was not evenly distributed. ENC ranged from 34.93 to 56.50,with an average of 46.87. GC2 was extremely significantly positively correlated with GC1 (P<0.01),while GC3 was not significantly positively correlated with GC1 and GC2 (P>0.05).? ENC was significantly positively correlated with GCall,GC1,GC3 and N (P<0.05,the same below).? The codon number (N) was positively correlated with GC3 and ENC. There were 30 codons with RSCU>1,among which 29 codons ended in A or U. Neutral plot analysis results showed that the correlation coefficient between GC3 and GC12 was 0.0188,and there was no significant correlation between them,and the regression coefficient was 0.143. ENC-plot analysis showed that most of the genes deviated from the ENC expected standard curve,and a few were distri-buted near the expected curve. There were 7 codons distributed in the range of GC3 content from -0.05 to 0.15,and there were 44 codons in the other regions,which were far from the ENC standard curve. PR2-plot analysis results showed that fewer genes fell on the middle line,and more genes were distributed in the lower right part of the graph,indicating that there was a bias in the use frequency of each base,namely T>A,G>C.There was bias (T>A,G>C) in the use frequency of each base,indicating that the bias of the genome codon was more influenced by the selection effect. Finally,7 optimal codons were selected as UUU,CUU,CCU,AAA,GAU,CGA and GGU. 【Conclusion】The chloroplast genomes of C. bungei show weak codon bias,preferring to use codons ending in A or U,which is mainly affected by selection pressure and less affected by mutation and other factors.
Key words:Catalpa bungei C. A. Mey.; chloroplast genome; codon bias; optimal codons
Foundation item: Fundamental Research Funds for the Central Public Welfare Research Institutes(CAFYBB2017 ZA001-8)
0 引言
【研究意義】楸樹(Catalpa bungei C. A. Mey.)是紫葳科(Bignoniaceae)梓屬(Catalpa Scop)落葉喬木,原產我國,分布范圍較廣,主產地為黃河流域和長江流域,素有“木王”之美稱,其材質不翹不裂,用途廣泛,樹體高大挺拔,樹姿優美,集用材、觀賞、環保和醫用價值等優點于一身,是我國特有的優質、藥用和園林觀賞樹種(吳麗華等,2010)。葉綠體基因組不同于細胞核中的基因組DNA,通常是雙鏈環狀結構,不與蛋白質結合,擁有一套完整的復制、轉錄和翻譯系統(辛雅萱等,2020),其結構保守,并包含了長、短單拷貝區(LSC和SSC)和2個反向重復區(IR)共4個大片段,這些特性有利于分析葉綠體基因組。目前葉綠體基因組已廣泛用于分子進化、生物多樣性分析、DNA條形碼、系統發育和遺傳表達等研究領域(倪梁紅等,2015;范李強等,2018)。研究表明,同義密碼子在無任何自然選擇和基因突變等壓力影響的情況下以同樣的概率出現,但如果受到影響則會導致同義密碼子的偏性使用,被稱為密碼子使用偏性(Codon usage bias,CUB),該現象廣泛存在于生物中(吳憲明等,2007;吳妙麗等,2019;王艷等,2021)。研究葉綠體密碼子偏性可為葉綠體基因工程相關研究打下基礎(季凱凱等,2020)。因此,分析楸樹葉綠體基因組密碼子使用偏性,確定密碼子的使用模式及其形成因素,對楸樹乃至梓屬植物葉綠體基因組學、物種進化特征及遺傳多樣性分析等研究具有重要意義。【前人研究進展】大量研究證實,不同物種的葉綠體基因組密碼子偏性因物種而不同(原曉龍等,2021),但相同物種或者親緣關系較近的物種之間的密碼子使用偏性相似(王英等,2020)。葉綠體基因組密碼子使用偏性影響的因素除了受選擇壓力和自然壓力外,tRNA豐度(Moriyama and Powell,1997)、mRNA二級結構(Gu et al.,2004)、外源基因堿基突變(Hershberg and Petrov,2008)及在寄主中的表達水平(武立偉等,2020)、蛋白表達效率和準確性(Lee et al.,2009;Brandls and Hughes,2016)等均會影響密碼子使用偏性的產生。目前分析密碼子的使用偏性、篩選最優密碼子及通過優化密碼子來提高外源基因在宿主中的表達水平方面的研究較多(Stephen et al.,2013;Deng et al.,2020),如余桂容等(2015)通過密碼子優化提高蘇云金芽胞桿菌(Bt)cry1Ah基因在轉基因玉米中的表達水平;王超等(2020)對朝倉花椒(Zanthoxylum piperi-tum DC.var. inerme Makino)基因的密碼子使用偏性進行分析,確定花椒基因使用偏性較高的密碼子,通過優化密碼子的方式提高花椒基因在物種內的表達水平;柳燕杰等(2020)通過分析美國紅梣(Fraxinus pennsylvanica Marsh)葉綠體基因組密碼子使用模式及影響密碼子使用偏性的因素,確定其密碼子偏性和最優密碼子,從而預測基因的表達水平,確定未知基因的位置和改良外源基因。隨著葉綠體的基因工程在優良品種繁育和種質資源保護中的作用日益凸顯,且大量物種的葉綠體基因組序列相繼公布,對不同物種的葉綠體基因組的研究日益增多(梁璇等,2021)。近年來,對葉綠體基因組密碼子偏性已在酸棗(Ziziphus jujuba)(胡曉艷等,2019)、巴山木竹(Arundinaria fargesii)(吳妙麗等,2019)、印度簕竹(Bambusa bambos)(吳妙麗等,2019)、杜梨(Pyrus betulifolia)(辛雅萱等,2020)等多種植物中進行研究。【本研究切入點】我國楸樹在優良新品系的選擇(麻文俊等,2012)、抗旱脅迫的評價(董蕾等,2014)及規模化繁育(孟路等,2020)等方面的研究已較成熟。雖然楸樹葉綠體基因組的測序現已完成,但未見有關楸樹密碼子偏性的研究報道。【擬解決的關鍵問題】利用Codon W 1.4.2及EMBOSS expiorer的cusp和chips軟件對楸樹葉綠體基因組密碼子的使用偏性進行相關分析,從而確定最優密碼子及密碼子偏性的主要影響因素,旨在為楸樹的遺傳變異及基因組學的研究提供強有力的分子理論依據。
1 材料和方法
1. 1 序列的獲取和篩選
從NCBI數據庫(http//www.ncbi.nlm.gov/)中下載楸樹葉綠體基因組序列(GenBank登錄號為MT610898,序列全長158238 bp),經篩選共獲得84個基因的編碼區(CDS)序列。為了降低因序列較短而造成的誤差,從84個基因序列中去除重復序列、長度小于300 bp的序列及內部含有終止密碼子和不完整的序列,最終獲得起始密碼子為ATG,終止密碼子為TAA、TGA和TAG的51個基因序列,用于后續序列分析。
1. 2 密碼子相關參數計算
通過Codon W 1.4.2分析51個基因序列,并獲取其相對同義密碼子使用度(RSCU)(即某一特定密碼子所編碼氨基酸時的所有密碼子相對使用次數),把RSCU作為判斷密碼子是否存在偏性的標準:當RSCU=1時,證明該密碼子使用上無偏性;RSCU>1時,則表明其使用頻率相對較高,存在偏性(牛元等,2018;胡振民等,2020)。利用EMBOSS expiorer中的cusp軟件分析不同基因序列的GC1、GC2、GC3(分別代表密碼子第1位、第2位和第3位的GC含量)和GCall(密碼子總GC含量)含量;并用EMBOSS expiorer中的chips軟件分析有效密碼子數(ENC)(即密碼子非均衡使用的偏好程度),ENC的取值范圍為20~61(馮瑞云等,2019)。最后,使用SPSS 21.0作相關分析。
1. 3 中性繪圖分析
根據不同基因密碼子的GC3含量和GC12含量[即GC12=(GC1+GC2)/2]進行中性繪圖,從而分析判斷影響密碼子偏性形成的因素(王鵬良等,2018)。若GC3與GC12相關性強,則說明突變壓力對密碼子3個位置的堿基的使用影響較大;若GC1、GC2與GC3相關性弱,則表明選擇壓力對密碼子使用影響較大(秦政等,2018;惠小涵等,2020)。
1. 4 ENC-plot繪圖分析
根據不同基因密碼子的GC含量和ENC,利用ENC-plot制作散點圖,并計算ENC的標準曲線,公式為:ENC=[29GC32+(1?GC3)2]+GC3+2,從而分析突變壓力在密碼子使用過程中的作用(尹為治等,2020)。如果各基因分布在ENC預期曲線的周圍,說明葉綠體基因組密碼子的使用偏性受突變壓力的影響較大,而如果各基因分布在ENC預期曲線的下方或偏離預期曲線,則表明葉綠體基因組密碼子的偏性受到選擇壓力的影響較大(王鵬良等,2018)。
1. 5 PR2-plot繪圖分析
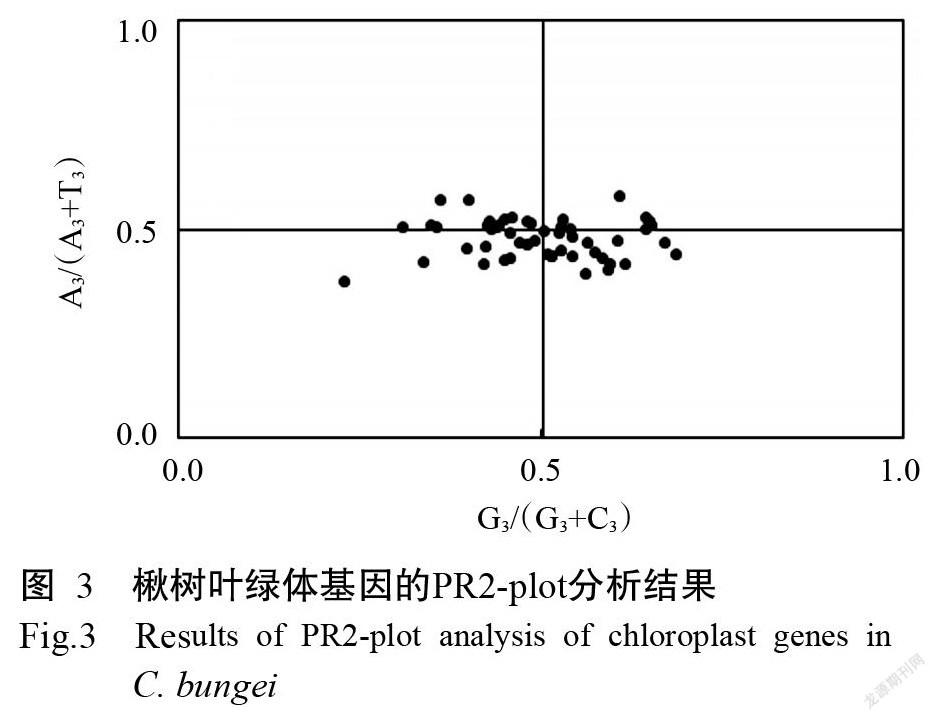
為了探究不同基因密碼子第3位堿基的嘌呤與嘧啶之間的突變壓力是否平衡,即A、T與G、C之間均衡分布的情況,對由同義密碼子編碼的氨基酸第3位上A、T、C、G 4種堿基組成情況進行PR2-plot繪圖分析(金剛,2019),以確定楸樹葉綠體基因組密碼子偏性的影響因素。其中,以G3/(G3+C3)、A3/(A3+T3)分別為橫、縱坐標,PR2偏倚圖的中心(0.5,0.5)表示A=T且C=G(宋輝等,2015)。其余點由中心點向該點發出的矢量表明基因的偏倚程度及方向(陸奇豐等,2020)。
1. 6 最優密碼子分析
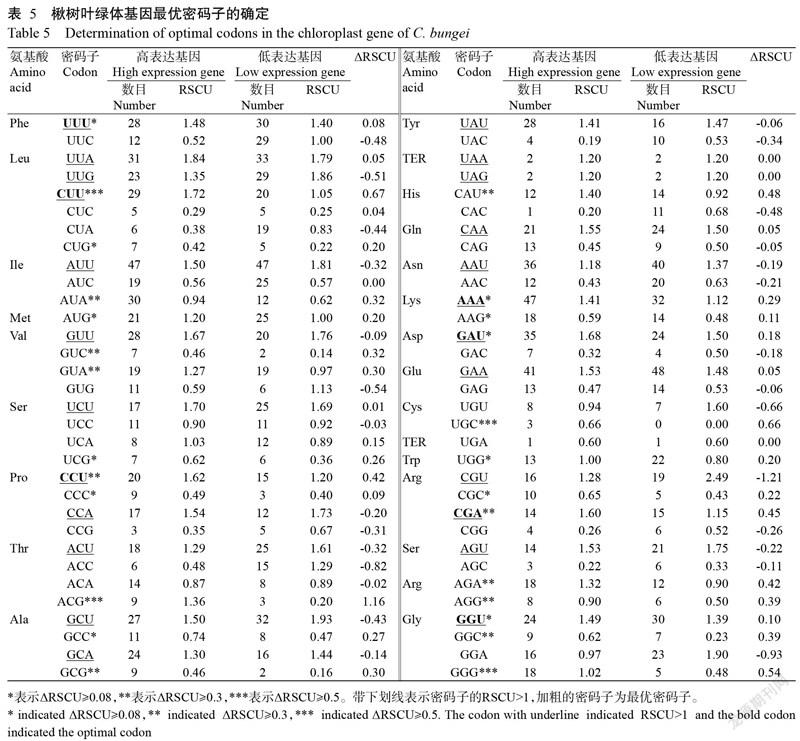
為了選出楸樹葉綠體基因組中的最優密碼子,利用Codon W 1.4.2計算每個密碼子的RSCU,把RSCU>1的密碼子定義為高頻密碼子。以5個基因的ENC作為偏性標準,對其進行從小到大排序,取前后兩端10%基因(前后各5個基因)構建高、低表達偏性基因庫。兩組間的差值ΔRSCU≥0.08(ΔRSCU=RSCU高表達- RSCU低表達)的密碼子作為高表達優越密碼子,最終將同時滿足RSCU>1和ΔRSCU≥0.08的密碼子作為楸樹葉綠體基因組的最優密碼子(胡莎莎等,2016)。
2 結果與分析
2. 1 密碼子堿基組成及偏好性分析
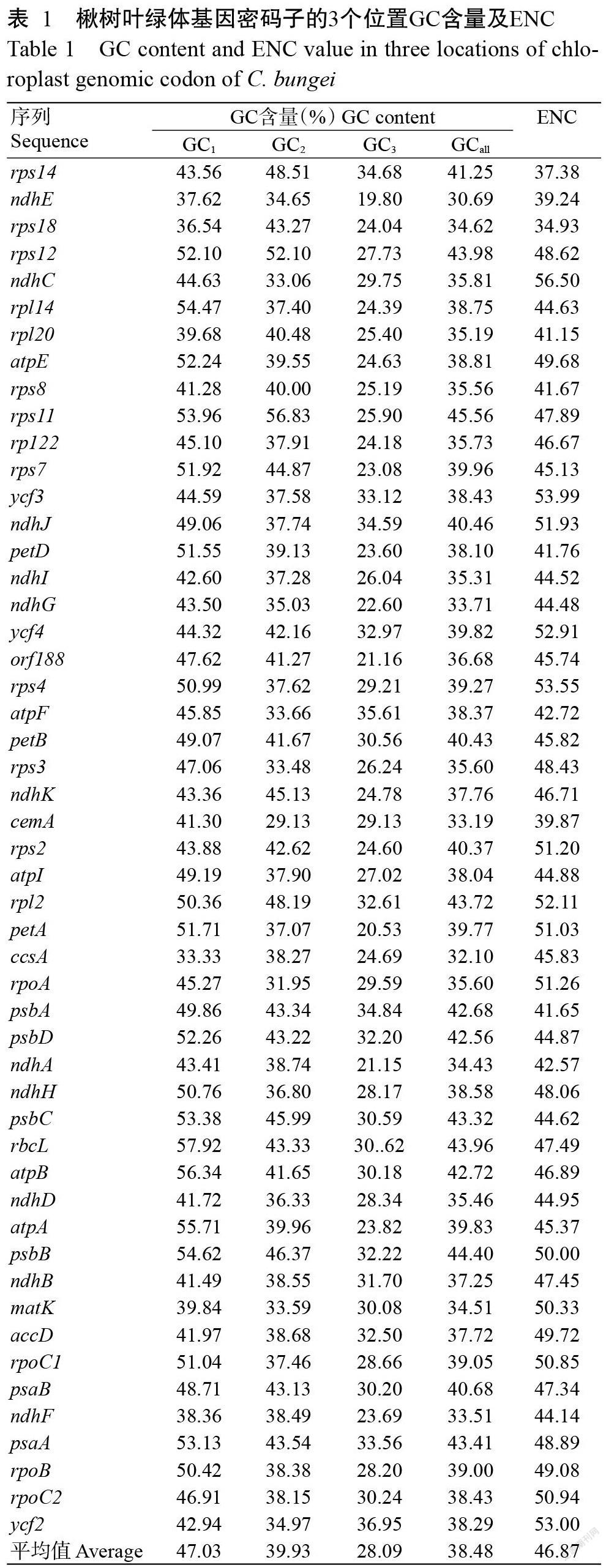
由表1可知,51個基因密碼子的平均GCall含量為38.48%,平均GC1、GC2和GC3含量分別為47.03%、39.93%和28.09%,即GC3含量明顯低于GC1和GC2含量,說明第3位的GC并非均勻分布,分布趨勢為GC1>GC2>GC3。可見,密碼子第3位以A或U(T)結尾的頻率高于以G或C結尾的頻率。51個基因的ENC為34.93~56.50,平均為46.87,說明楸樹葉綠體基因組的密碼子偏性較弱。
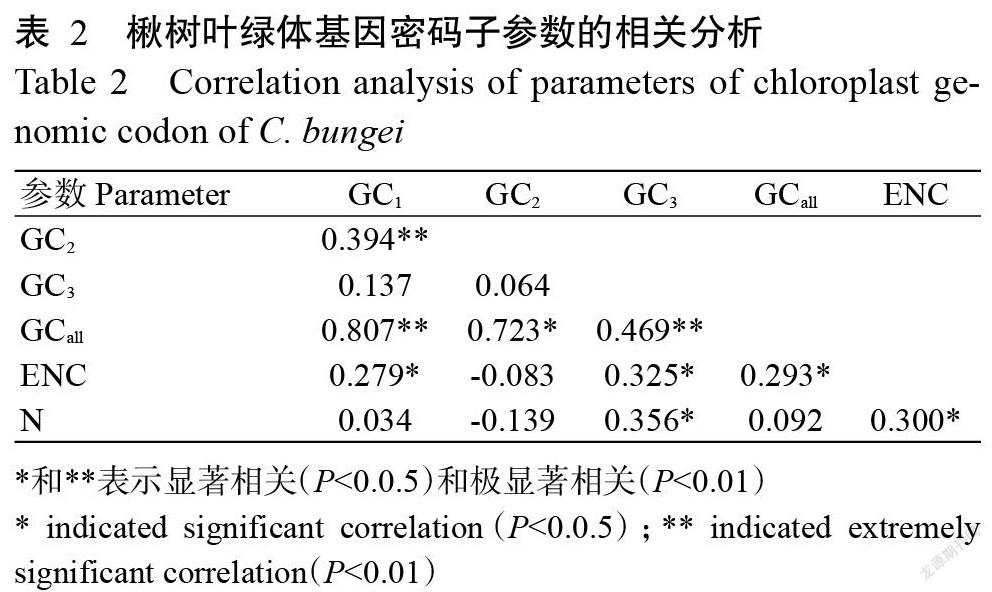
對楸樹葉綠體基因的密碼子數量(N)、ENC和各密碼子的GC含量進行相關分析,結果如表2所示。GC2與GC1呈極顯著正相關(P<0.01,下同),而GC3與GC1和GC2無顯著正相關(P>0.05,下同),說明密碼子的第1位與第2位的堿基相似度較高,但二者與第3位的堿基相似度較低;GCall與GC1和GC3均呈極顯著正相關,與GC2呈顯著正相關(P<0.05,下同);ENC除了與GC2無顯著負相關外,與GCall、GC1、GC3和N均呈顯著正相關,說明密碼子3個位置的堿基組成對ENC的影響有所不同,其中,第1位和第3位堿基組成對密碼子的偏性有影響。密碼子數(N)除了與GC3和ENC呈顯著正相關外,與其他數值均無顯著相關。
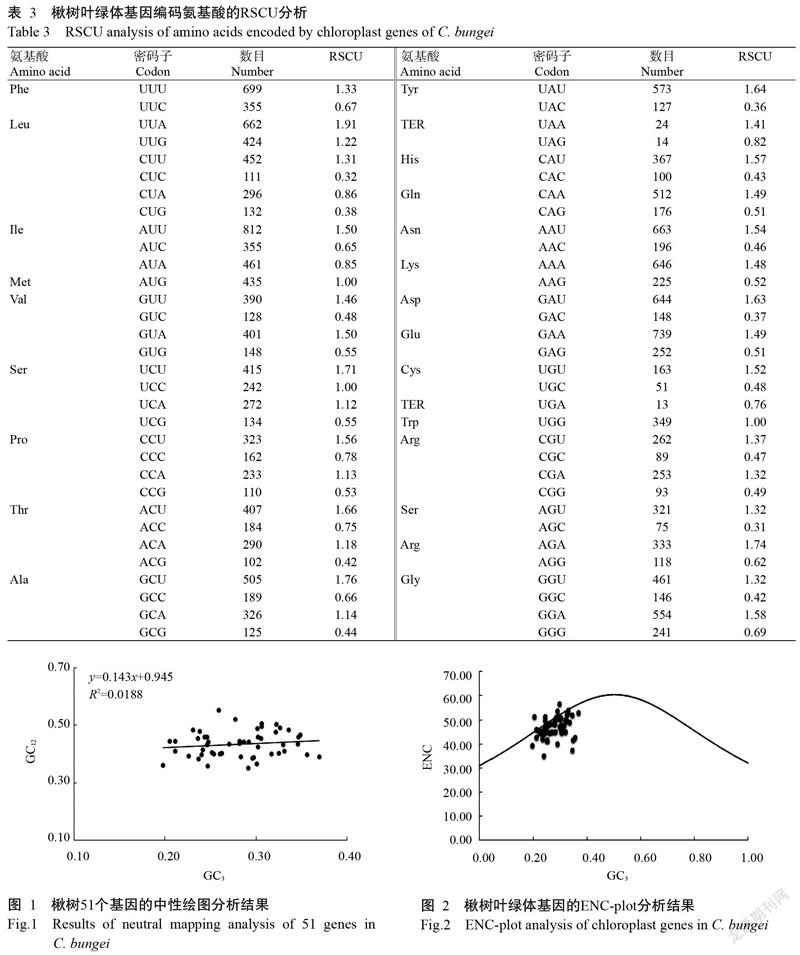
由表3可知,RSCU>1的密碼子共有30個,其中,以U(16個)或A(13個)結尾的密碼子數量明顯多于以G(1個)結尾的密碼子,說明以A或U結尾的密碼子是楸樹葉綠體基因組偏好的密碼子。
2. 2 中性繪圖分析結果
由圖1可知,GC12含量為0.20~0.37;GC3含量為0.35~0.55,二者的相關系數和回歸系數分別為0.0188和0.143,表明在密碼子的第1、2位堿基與第3位堿基組成相關性較弱,楸樹葉綠體基因組編碼基因保守性較高,密碼子偏好性受選擇壓力的影響較大。
2. 3 ENC-plot繪圖分析結果
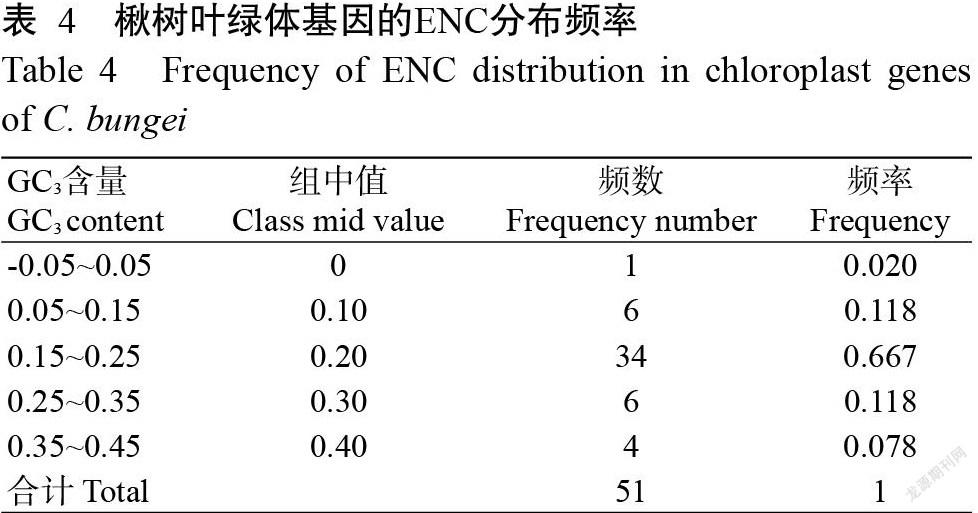
由圖2可知,在51個基因中,大部分基因偏離了ENC預期標準曲線,少數分布在預期曲線附近,說明楸樹葉綠體基因組的偏性受選擇壓力的影響較大,受基因突變壓力的影響較小;位于標準曲線上方的基因密碼子偏性較弱,而位于標準曲線下方的基因密碼子偏性較強。由不同GC3含量區間分布的密碼子數量(表4)可知,有7個密碼子分布在-0.05~0.15區間,其余區間共有44個密碼子,而這44個基因距離ENC標準曲線較遠(圖2),表明楸樹葉綠體基因組密碼子的使用偏性主要受選擇壓力的影響較大,而受其他因素(如堿基突變等)的影響相對較小。
2. 4 PR2-plot繪圖分析結果
由圖3可知,51個基因在PR2平面圖的4個部分并非均勻分布,且落在中線上的基因較少,平面圖右下方的基因分布較多,表明各堿基使用頻率存在偏性,即T>A,G>C,說明選擇壓力不是影響楸樹葉綠體基因組密碼子偏性的唯一因素,突變等其他因素也對楸樹葉綠體基因組的密碼子偏性產生一定的影響。
2. 5 最優密碼子的篩選
從51個楸樹葉綠體基因選取中5個高表達基因(petA、ndhC、rps2、atpE和rps4)和5個低表達基因(cemA、atpF、psbA、rps18和rps14),然后利用Codon W 1.4.2計算出兩組基因同義密碼子的RSCU,再計算出ΔRSCU,結果(表5)表明,高頻密碼子有24個(RSCU≥1),高表達優越密碼子有12個(ΔRSCU≥0.08,用*表示),ΔRSCU≥0.30(用**表示)的密碼子有10個,ΔRSCU≥0.50(用***表示)密碼子有4個。同時滿足高頻(ΔRSCU≥0.08)和高表達(RSCU≥1)的密碼子有7個,確定為最優密碼子,分別是UUU、CUU、CCU、AAA、GAU、CGA和GGU,其中5個以U結尾,2個以A結尾。
3 討論
GC3是密碼子偏性的一個重要判斷指標。本研究對楸樹葉綠體基因密碼子3個位置的GC含量進行分析,結果表明,GC1和GC2含量顯著高于GC3含量,說明楸樹葉綠體基因組偏好使用U或A結尾的密碼子,RSCU分析結果也證實該結論。在糜子(Panicum miliaceum L.)(劉慧等,2017)、普通油茶(Camellia oleifera Abel)(王鵬良等,2018)和西南樺(Betula alnoides Hamilton)(原曉龍等,2020b)等植物的葉綠體基因組偏性的研究也發現,這些植物偏好使用A或U結尾的密碼子。本研究最終篩選出的7個最優密碼子,均以U或A結尾,與杜梨(Pyrus betulifolia Bge.)(辛雅萱等,2020)的最優密碼子研究結果一致。
選擇壓力是引起密碼子使用偏性的主要因素,突變等其他方面的因素也發揮一定作用(趙森等,2020)。本研究發現,楸樹葉綠體基因的ENC為34.93~56.50,平均為46.87,說明楸樹葉綠體基因密碼子偏性較弱,與大花香水月季(Rosa odorata var. gigantean Rehd. & Wils)(牛元等,2018)、酸棗[Zizi-phus jujube var. spinosa(Bunge) Hu ex H. F. Chow](胡曉艷等,2019)、灰毛漿果楝[Cipadessa cinerascens(Pellegr.) H. -M.](原曉龍等,2020c)等植物的葉綠體密碼子使用偏性研究結果一致,由此推測葉綠體基因組結構的保守性是導致密碼子使用偏性的主要因素之一。結合相關分析和中性繪圖分析結果發現,堿基突變也是影響楸樹葉綠體基因組偏性的主要因素之一,與羅火林等(2016)研究結論一致。本研究的PR2-plot和ENC-plot分析結果均表明,楸樹葉綠體基因組密碼子的使用偏性主要受選擇壓力的影響較大,而受其他因素(如堿基突變等)的影響相對較小,與云南藍果樹(Nyssa yunnanensis W. C. Yin)(原曉龍等,2020a)、灰毛漿果楝(C. cinerascens (Pellegr.) H. -M.)(原曉龍等,2020b)和西南樺(Betula alnoides Hamilton)(原曉龍等,2020c)等研究結果一致,但與水稻(Oryza sativa Linn.)(劉慶坡和薛慶中,2004)研究結果不一致。結論存在差異可能因為基因表達程度和密碼子堿基組成不同所引起。由此可知,物種的葉綠體基因組密碼子使用偏性的影響因素眾多,且不同物種的影響因素各不相同(傅建敏等,2017)。今后應基于本研究結果對楸樹葉綠體功能基因的進化產生是否受選擇壓力影響進行深入研究。
4 結論
楸樹葉綠體基因組密碼子偏性較弱,偏好使用A或U結尾的密碼子,主要受選擇壓力的影響,受突變等其他因素影響較弱。
參考文獻:
董蕾,陳博,李吉躍,王軍輝,謝堃. 2014. 5個楸樹無性系抗旱性的綜合評價[J]. 中南林業科技大學學報,34(4):30-35. [Dong L,Chen B,Li J Y,Wang J H,Xie K. 2014. Drought resistance evaluation of 5 Catalpa bungei clones[J]. Journal of Central South University of Forestry & Technology,34(4):30-35.] doi:10.3969/j.issn.1673-923X. 2014.04.007.
范李強,胡歡,鄭洪蕾,王田靜,王亞玲,馬濤,毛康珊. 2018. 響葉楊(楊屬)葉綠體基因組測序與比較分析[J]. 四川大學學報(自然科學版),55(1):165-171. [Fan L Q,Hu H,Zheng H L,Wang T J,Wang Y L,Ma T,Mao K S. 2018. Complete sequence and comparative analysis of the chloroplast genome of the Chinese aspen(Populus adenopoda,Salicaceae)[J]. Journal of Sichuan University(Naiural Seienee Edtion),55(1):165-171.] doi:10.3969/j.issn.0490-6756.2018.01.027.
馮瑞云,梅超,王慧杰,雷夢林,田翔,侯雅靜. 2019. 籽粒莧葉綠體基因組密碼子偏好性分析[J]. 中國草地學報,41(4):8-15. [Feng R Y,Mei C,Wang H J,Lei M L,Tian X,Hou Y J. 2019. Analysis of codon usage in the chloroplast genome of grain amaranth(Amaranthus hypochondriacus L.)[J]. Chinese Journal of Grassland,41(4):8-15.] doi:10.16742/j.zgcdxb.20180227.
傅建敏,索玉靜,劉慧敏,譚曉風. 2017. 柿屬植物葉綠體蛋白質編碼基因密碼子用法[J]. 經濟林研究,35(2):38-44. [Fu J M,Suo Y J,Liu H M,Tan X F. 2017. Analysis on codon usage in the chloroplast protein-coding genes of Diospyros spp.[J]. Nonwood Forest Research,35(2):38-44.] doi:10.14067/j.cnki. 1003-8981.2017.02.006.
胡莎莎,羅洪,吳琦,姚慧鵬. 2016. 苦蕎葉綠體基因組密碼子偏愛性分析[J]. 分子植物育種,14(2):309-317. [Hu S S,Luo H,Wu Q,Yao H P. 2016. Analysis of codon bias of chloroplast genome of tartary buckwheat[J]. Molecular Plant Breeding,14(2):309-317.] doi:10.13271/j.mpb. 014.000309.
胡曉艷,許艷秋,韓有志,杜淑輝. 2019. 酸棗葉綠體基因組密碼子使用偏性性分析[J]. 森林與環境學報,19(6):621-628. [Hu X Y,Xu Y Q,Han Y Z,Du S H. 2019. Codon usage bias analysis of the chloroplast genome of Ziziphus jujuba var. spinosa[J]. Journal of Forest and Environment,19(6):621-628.] doi: 10.13324/j.cnki.jfcf.2019.06.009.
胡振民,萬青,李歡,李榮林,王楓,楊亦揚. 2019. 茶樹CsNRT1.1基因密碼子使用特性分析[J]. 江蘇農業學報,35(4):896-903. [Hu Z M,Wan Q,Li H,Li R L,Wang F,Yang Y Y. 2019. Analysis of codon usage features of CsNRT1.1 gene in Camellia sinensis[J]. Jiangsu Journal of Agricultural Sciences,35(4):896-903.] doi:10.3969/j.issn. 1000-4440.2019.04.021.
惠小涵,程婷婷,柯衛東,郭宏波. 2020. 蓮藕PPO基因密碼子偏好性特征分析[J]. 江蘇農業學報,36(2):438-446. [Hui X H,Cheng T T,Ke W D,Guo H B. 2020. Analysis on codon preference of PPO gene in lotus root[J]. Jiangsu Journal of Agricultural Sciences,36(2):438-446.] doi:10.3969 /j.issn.1000-4440.2020.02.026.
季凱凱,宋希強,陳春國,李革,謝尚淺. 2020. 木蘭科葉綠體基因組的密碼子使用特征分析[J]. 中國農業科技導報,22(11):52-62. [Ji K K,Song X Q,Chen C G,Li G,Xie S Q. 2020. Codon usage profiling of chloroplast genome in Magnoliaceae[J]. Journal of Agricultural Science and Technology,22(11):52-62.] doi:10.13304/j.nykjdb.2019. 0937.
金剛,王麗萍,龍凌云,吳鳳,唐玉娟,覃劍鋒,危丹妮,黃秋偉,蘇文潘. 2019. 普通野生稻線粒體蛋白質編碼基因密碼子使用偏好性的分析[J]. 植物科學學報,37(2):188-197. [Jin G,Wang L P,Long L Y,Wu F,Tang Y J,Qin J F,Wei D N,Huang Q W,Su W P. 2019. Analysis of codon usage bias in the mitochondrial protein-coding genes of Oryza rufipogon[J]. Plant Science Journal,37(2):188-197.] doi:10.11913/PSJ.2095-0837.2019.20188.
梁璇,李鵬,許冬梅,賈小云,王文斌. 2021. 紫蘇葉綠體全基因組特征分析[J]. 山西農業科學,49(3):265-272. [Liang X,Li P,Xu D M,Jia X Y,Wang W B. 2021. Characteristics analysis of whole chloroplast genome in Perilla frustescens[J]. Journal of Shanxi Agricultural Sciences,49(3):265-272.] doi:10.3969/j.issn.1002-2481. 2021.03.02.
劉慧,王夢醒,岳文杰,邢光偉,葛靈巧,聶小軍,宋衛寧. 2017. 糜子葉綠體基因組密碼子使用偏性的分析[J]. 植物科學學報,35(3):362-367. [Liu H,Wang M X,Yue W J,Xing G W,Ge L Q,Nie X J,Song W N. 2017. Analysis of codon usage in the chloroplast genome of broomcorn millet(Panicum miliaceum L.)[J]. Plant Scien-ce Journal,35(3):362-367.] doi:10.11913/PSJ.2095-0837.2017.30362.
劉慶坡,薛慶中. 2004. 粳稻葉綠體基因組的密碼子用法[J]. 作物學報,30(12):1220-1224. [Liu Q P,Xue Q Z. 2004. Codon usage in the chloroplast genome of rice(Oryza sativa L ssp japonica)[J]. Acta Agronomica Sinica,30(12):1220-1224.] doi:10.3321/j.issn:0496-3490. 2004.12.008.
柳燕杰,田旭平,李倩. 2020. 美國紅梣葉綠體基因組密碼子偏好性分析[J]. 江蘇農業科學,48(15):83-88. [Liu Y J,Tian X P,Li Q. 2020. Codon usage bias analysis of the chloroplast genome of Fraxinus pennsylvanica[J]. Jiangsu Agricultural Sciences,48(15):83-88.] doi:10. 15889/ j. issn. 1002 - 1302. 2020. 15.014.
陸奇豐,駱文華,黃至歡. 2020. 兩種梧桐葉綠體基因組密碼子使用偏性分析[J]. 廣西植物,40(2):173-183. [Lu Q F,Luo W H,Huang Z H. 2020. Codon usage bias of chloroplast genome from two species of Firmiana Marsili[J]. Guihaia,40(2):173-183.] doi:10.11931/guihaia.gxzw 201811035.
羅火林,張亞楠,羅麗萍,熊冬金,楊柏云. 2016. 蘭科植物FT基因的密碼子偏性分析[J]. 分子植物育種,14(1):51-58. [Luo H L,Zhang Y N,Luo L P,Xiong D J,Yang B Y. 2016. The codon usage bias of FLOWERING LOCUS T gene(FT) in Orchidaceae[J]. Molecular Plant Bree-ding,14(1):51-58.] doi:10.13271/j.mpb.014.000051.
麻文俊,張守攻,王軍輝,董菊蘭. 2012. 楸樹無性系苗期N素利用差異和高產無性系選擇[J]. 林業科學,48(10):157-162. [Ma W J,Zhang S G,Wang J H,Dong J L. 2012. Variation of nitrogen utilization among Catalpa bungei clones at nursery stage and high-yield clones selection[J]. Scientia Silvae Sinicae,48(10):157-162.] doi:10.11707/ j.1001-7488.20121025.
孟路,劉勇,王小平,李進宇,賀國鑫,薛敦孟,邢麗霞,李世安. 2020. 4個楸樹品種組培快繁能力的比較研究[J]. 中南林業科技大學學報,40(1):82-88. [Meng L,Liu Y,Wang X P,Li J Y,He G X,Xue D M,Xing L X,Li S A. 2020. Comparison study on rapid reproductive capacity of four cultivars of Catalpa bungei in tissue culture[J]. Journal of Central South University of Forestry & Technology,40(1):82-88.] doi:10.14067/j.cnki.1673-923x.2020. 01.010.
倪梁紅,趙志禮,米瑪. 2015. 藥用植物葉綠體基因組研究進展[J].中藥材,38(9):1990-1994. [Ni L H,Zhao Z L,Mi M.2015. Research progress on chloroplast genomes of medicinal plants[J]. Journal of Chinese Medicinal Materials,38(9):1990-1994.] doi:10.13863/j.issn1001-4454. 2015.09.046.
牛元,徐瓊,王崳德,戴立蘭,莊健,趙亞蘭. 2018. 大花香水月季葉綠體基因組密碼子使用偏性分析[J]. 西北林學院學報,33(3):123-130. [Niu Y,Xu Q,Wang Y D,Dai L L,Zhuang J,Zhao Y L. 2018. An analysis on codon usage bias of chloroplast genome of Rosa odorata var. gigantean[J]. Journal of Northwest Forestry University,33(3):123- 130.] doi:10.3969/j.issn.1001-7461.2018.03.19.
秦政,鄭永杰,桂麗靜,謝谷艾,伍艷芳. 2018. 樟樹葉綠體基因組密碼子偏好性分析[J]. 廣西植物,38(10):1346-1355. [Qin Z,Zheng Y J,Gui L J,Xie G A,Wu Y F. 2018. Codon usage bias analysis of chloroplast genome of camphora tree(Cinnamomum camphora)[J]. Guihaia,38(10):1346-1355.] doi:10.11931/guihaia.gxzw201805 023.
宋輝,王鵬飛,馬登超,夏晗,趙傳志,張燁,趙術珍. 2015. 蒺藜苜蓿WRKY轉錄因子密碼子使用偏好性分析[J]. 農業生物技術學報,23(2):203-212.[Song H,Wang P F,Ma D C,Xia H,Zhao C Z,Zhang Y,Zhao S Z. 2015. Analysis of codon usage bias of WRKY transcription factors in Medicago truncatula[J]. Journal of Agricultural Biotechnology,23(2):203-212.] doi:10.3969/j.issn.1674-7968.2015.02.007.
王超,趙懿琛,趙德剛. 2020. 朝倉花椒轉錄組密碼子偏好性分析[J]. 分子植物育種,18(21):6989-6996. [Wang C,Zhao Y C,Zhao D G. 2020. Preference analysis of transcriptome codon in Zanthoxylum piperitum DC. var. inerme Makino[J]. Molecular Plant Breeding,18(21):6989-6996.] doi:10.1016/S0309.1740(99)00142-4.
王鵬良,楊利平,吳紅英,農有良,吳雙成,肖玉菲,覃子海,王華宇,劉海龍. 2018. 普通油茶葉綠體基因組密碼子偏好性分析[J]. 廣西植物,38(2):135-144. [Wang P L,Yang L P,Wu H Y,Nong Y L,Wu S C,Xiao Y F,Qin Z H,Wang H Y,Liu H L. 2018. Condon preference of chloroplast genome in Camellia oleifera[J]. Guihaia,38(2):135-144.] doi:10.11931/guihaia.gxzw201708001.
王艷,趙懿琛,趙德剛. 2021. 杜仲基因密碼子使用模式分析[J]. 廣西植物,41(2):274-282. [Wang Y,Zhao Y C,Zhao D G. 2021. Analysis of codon usage pattern of Eucommia ulmoides[J]. Guihaia,41(2):274-282.] doi:10. 11931/guihaia.gxzw201910008.
王英,張太奎,張孟偉,趙玉潔,袁偉程,劉翠玉,苑兆和. 2020. 石榴等陸生植物Actin1基因密碼子偏向性與進化分析[J]. 分子植物育種,18(6):1799-1807. [Wang Y,Zhang T Q,Zhang M W,Zhao Y J,Yuan W C,Liu C Y,Yuan Z H. 2020. Codon usage bias and evolution analyses of Actin1 genes from Punica granatum and other land plants[J]. Molecular Plant Breecling,18(6):1799-1807.] doi:10. 13271/j.mpb.018.001799.
吳麗華,王軍輝,林娟. 2010. 楸樹植物資源的研究概況[J]. 上海交通大學學報(農業科學版),28(1):91-96. [Wu L H,Wang J H,Lin J. 2010. A survey of the studies on the resources of Catalpa bungei[J]. Journal of Shanghai Jiaotong University(Agricultural Science),28(1):91-96.] doi:10.3969/j.issn.1671-9964.2010.01.0017.
吳妙麗,陳世品,陳輝. 2019. 竹亞科葉綠體基因組的密碼子使用偏性分析[J]. 森林與環境學報,39(1):9-14. [Wu M L,Chen S P,Chen H. 2019. Condon preference of chloroplast genome of Bambusoideae[J]. Journal of Forest and Environment,39(1):9-14.] doi:10.13324/j.cnki.jfcf.2019.01.002.
吳憲明,吳松鋒,任大明,朱云平,賀福初. 2007. 密碼子偏性的分析方法及相關研究進展[J]. 遺傳,(4):420-426. [Wu X M,Wu S F,Ren D M,Zhu Y P,He F C. 2007. The analysis method and progress in the study of codon bias[J]. Hereditas(Beijing),(4):420-426.] doi:10.3321/j.issn:0253-9772.2007.04.006.
武立偉,崔英賢,聶麗萍,徐志超,王瑀,宋經元,焦連魁,姚輝. 2020. 細莖石斛葉綠體全基因組序列特征及系統發育分析[J]. 藥學學報,55(5):302-312. [Wu L W,Cui Y X,Nie L P,Xu Z C,Wang Y,Song J Y,Jiao L K,Yao H. 2020. The characteristics of complete chloroplast genome sequence and phylogenetic analysis of Dendrobium moniliforme[J]. Acta Pharmaceutica Sinica,55(5):302-312.] doi:10.16438/j.0513-4870.2019-0940.
辛雅萱,董章宏,瞿紹宏,劉成,葉鵬,辛培堯. 2020. 杜梨葉綠體基因組密碼子偏性分析[J]. 河北農業大學學報,43(6):51-59. [Xin Y X,Dong Z H,Qu S H,Liu C,Ye P,Xin P Y. 2020. Analysis on codon usage bias of chloroplast genome in Pyrus betulifolia Bge[J]. Journal of Hebei Agricultural University,43(6):51-59.] doi:10.13320/j.cnki.jauh.2020.0112.
尹為治,方正,黃良鴻,龍文興,李佳靈. 2020. 海南2種龍腦香科植物葉綠體基因組密碼子偏好性分析[J].林業調查規劃,45(6):19-26. [Yin W Z,Fang Z,Huang L H,Long W X,Li J L. 2020. Codon usage bias in chloroplast genomes of two species of Dipterocarpaceae in Hainan[J]. Forest Inventory and Planning,45(6):19-26.] doi:10. 3969/j.issn.1671-3168.2020.06.005.
余桂容,曹穎,杜文平,宋軍,陳謙,徐利遠. 2015. 抗蟲耐草甘膦雙價基因表達載體的構建及玉米轉化[J]. 西南農業學報,28(2):475-479. [Yu G R,Cao Y,Du W P,Song J,Chen Q,Xu L Y. 2015. Construction of binary vector obtaining insect & herbicide resistance genes and transformation of maize[J]. Southwest China Journal of Agricultural Sciences,28(2):475-479.] doi:10.16213/j.cnki.scjas.2015.02.006.
原曉龍,康洪梅,王毅. 2020a. 云南藍果樹葉綠體基因組密碼子偏好性分析[J]. 西北林學院學報,35(4):26-31. [Yuan X L,Kang H M,Wang Y. 2020a. Codon usage bias analysis of chloroplast genome in Nyssa yunnanensis[J]. Journal of Northwest Forestry University,35(4):26-31.] doi:10.3969/j.issn.1001-7461.2020.04.05.
原曉龍,李云琴,王毅,張勁峰. 2020b. 西南樺葉綠體基因組密碼子偏好性分析[J]. 基因組學與應用生物學,39(12):5758-5764. [Yuan X L,Li Y Q,Wang Y,Zhang J F. 2020b. Analysis of codon usage in the chloroplast genome of Betula alnoides[J]. Genomics and Applied Bio-logy,39(12):5758-5764.] doi:10.13417/j.gab.039.005758.
原曉龍,王毅,張勁峰. 2020c. 灰毛漿果楝葉綠體基因組密碼子使用特征分析[J]. 福建林學院學報,40(2):195-202. [Yuan X L,Wang Y,Zhang J F. 2020c. Characterization of codon usage in Cipadessa cinerascens chloroplast genome[J]. Journal of Fujian College of Forestry,40(2):195-202.] doi:10.13324/j.cnki.jfcf.2020.02.013.
原曉龍,劉音,康洪梅,陳中華,李云琴,王毅. 2021. 蒜頭果葉綠體基因組密碼子偏好性分析[J]. 西南林業大學學報(自然科學),41(3):15-22. [Yuan X L,Liu Y,Kang H M,Chen Z H,Li Y Q,Wang Y. 2021. Analysis of codon usage bias in chloroplast genome of Malania oleifera[J]. Journal of Southwest Forestry University,41(3):15-22.] doi:10.11929/j.swfu.202001047.
趙森,鄧力華,陳芬. 2020. 秋茄葉綠體基因組密碼子使用偏好性分析[J]. 森林與環境學報,40(5):534-541. [Zhao S,Deng L H,Chen F. 2020. Codon usage bias of chloroplast genome in Kandelia obovata[J]. Journal of Forest and Environment,40(5):534-541.] doi:10.13324/j.cnki.jfcf.2020.05.011.
Brandls G,Hughes D. 2016. The selective advantage of synonymous codon usage bias in Salmonella[J]. PLoS Genetics,12(3):e1005926. doi:10.1371/journal.pgen.10059 26.
Deng Y,Hedayioglu F L,Kalfon J,Chu D,Haar T. 2020. Hidden patterns of codon usage bias across kingdoms[J]. Journal of the Royal Society Interface,17(163):20190819.] doi:10.1098/rsif.2019.0819.
Gu W J,Zhou T,Ma J M,Sun X, Lu Z H. 2004. The relationship between synonymous codon usage and protein structure in Escherichia coli and Homo sapiens[J]. Biosystems,73(2):89-97. doi:10.1016/j.biosystems.2003. 10.001.
Hershberg R,Petrov D A. 2008. Selection on codon bias[J]. Annual Review of Genetics,42(1):287-299. doi:10.1021/ cm0010349.
Lee S F,Li Y J,Halperin S A. 2009. Overcoming codon-usage bias in heterologous protein expression in Streptococcus gordonii[J]. Microbiology,155(11):3581-3588. doi:10.1099/mic.0.030064-0.
Moriyama E N,Powell J R. 1997. Codon usage bias and tRNA abundance in Drosophila[J]. Journal of Molecular Evolution,45(5):514-523. doi:10.1007/PL00006256.
Stephen J S,Jeremy D V,Robert M,Li F L,John M,Laszlo Z. 2013. Expression of chicken parvovirus VP2 in chicken embryo fibroblasts requires codon optimization for production of naked DNA and vectored Meleagrid herpesvirus type 1 vaccines[J]. Virus Genes,47(2):259-267. doi:10.1007/s11262-013-0944-9.
(責任編輯 陳 燕)

