胃癌組織環狀RNA表達譜分析
楊青茹,王華,周松峰,申紅星
(1. 江蘇大學醫學院, 江蘇 鎮江 212013; 2. 南通大學醫學院, 江蘇 南通 226019)
胃癌是全球常見的惡性腫瘤,也是導致死亡的第二大主要原因[1]。胃癌的致病因素很多,如EB病毒感染、幽門螺桿菌感染等[2]。胃癌的發生不僅影響蛋白代謝異常[3],也涉及微小RNA(microRNA,miRNA)、長鏈非編碼RNA(lncRNA)和環狀RNA(circluar RNA, circRNA)等非編碼RNA的異常表達。CircRNAs自2013年發現其生物學功能以來,逐漸成為研究的熱點,研究顯示circRNAs參與癌癥等多種疾病的發生發展過程[4]。CircRNAs分子是一類不具有5′末端帽子和3′末端poly(A)尾巴,并通過共價鍵形成環形的非編碼RNA分子,具有一定的組織、時序和疾病特異性,并發揮調節轉錄、翻譯和表達水平等方面的生物學功能[5-6]。已有研究表明,circ-HuR異位表達在體內外均能抑制胃癌細胞的生長、侵襲和轉移[7]。部分circRNAs分子含miRNAs應答元件(miRNA response element,MRE),可充當競爭性內源RNA(competing endogenous RNA,ceRNA),與miRNAs結合,發揮miRNAs海綿的作用,上調靶基因表達水平;也可以翻譯成蛋白質來發揮作用[8]。CeRNA調控在癌癥進展中發揮關鍵作用,如circRNA_LARP4通過抑制miR-424-5p和調控LATS1的表達,抑制胃癌細胞的增殖和侵襲[9];circPDSS1通過海綿miR-186-5p和調節NEK2促進胃癌進展[10];circRNA_100782通過海綿吸附miR-574-3p,抑制Rb表達,促進胃癌的增殖和轉移[11]。
雖然多個報道顯示circRNAs參與調節胃癌的發生發展,但是非編碼RNA尤其是circRNAs和miRNAs在胃癌中的詳細作用機制依然不清楚。本研究通過分析胃癌相關的多個circRNAs的測序數據,旨在揭示circRNAs-miRNAs-mRNAs網絡通過生物進程、細胞組分和分子功能3個方面參與胃癌發生發展的機制。
1 材料與方法
1.1 差異表達circRNAs的獲得
使用美國國家生物技術信息中心(NCBI)的GEO數據庫(http://www.ncbi.nlm.nih.gov/geo/geo2r/)和在線分析工具R語言分析(http://www.dooccn.com/r/)GSE100170、GPL19978(GSE83521和GSE89143)、GSE78092和STAD(未上傳)數據庫中差異表達的circRNAs。
1.2 篩選circRNAs靶向的miRNAs及其驗證
使用Starbase數據庫(http://bioinfo.au.tsinghua.edu.cn/software-database)預測circRNAs的靶向miRNAs,根據CLIP-Data≥2的篩選條件,篩選circRNAs下游的miRNAs,并利用GEO數據庫(GSE23734)作為下游miRNAs準確性的驗證,其結果以韋恩圖呈現(http://bioinformatics.psb.ugent.be/webtools/Venn/)。
1.3 篩選miRNAs靶向的mRNAs
使用TargetScan數據庫(http://www.TargetScan.org)預測miRNAs下游的mRNAs,其篩選標準為Total context+ score≤-0.75。
1.4 GO分析、KEGG分析和ceRNA相互作用網絡
基因本體論(gene ontology,GO)分析包括生物進程、細胞組分和分子功能3個方面[12]。通過GO分析和KEGG分析驗證mRNAs的功能。通過Cytoscape2.0軟件(http://www.cytoscape.org)分析circRNAs-miRNAs-mRNAs三者之間的相互作用關系。
1.5 靶基因相關疾病
利用比較毒物基因組學數據庫(The comparative toxicogenomics database)分析靶基因導致的相關性疾病,網址為https://ctdbase.org/,篩選標準為P<0.05[13]。
2 結果
2.1 預測差異表達circRNAs的下游靶miRNAs以及驗證
本文所使用的數據庫以及篩選流程圖如圖1所示。從GEO數據庫以及STAD中總共獲得了83 748個circRNAs,按照Log2(fold change,FC)>1,P<0.05的篩選標準獲得734個差異表達的circRNAs(表1)。將circRNAs注釋后,利用韋恩網站處理,共獲得59個基因(表2),其韋恩圖如圖2A所示。排除各種因素(沒有針對性的miRNAs,并且無法同時在兩個或多個數據庫中找到)發現17個差異表達的circRNAs,見表3。用Starbase數據庫預測17個circRNAs的下游靶miRNAs,得到 354個miRNAs。以GEO數據庫中的數據集GSE23739 (共80對標本,癌組和癌旁組各占一半)作為參考,進一步驗證354個miRNAs與胃癌的關系。根據P<0.05的篩選標準,從GSE23739數據庫獲得512個miRNAs,并與預測到的354個miRNAs作韋恩圖(圖2B),獲得46個共同的miRNAs(表4)。由于miR-483-3p沒有查詢到靶基因,最終選取45個miRNAs用于下一步分析。為驗證癌組和癌旁組中差異性表達的circRNAs,用R語言作為運行程序繪制熱圖。結果如圖2所示,分別顯示4個數據庫GPL19978(圖2C)、GSE78092(圖2D)、GSE100170(圖2E)和STAD(圖2F)的circRNAs的差異表達。
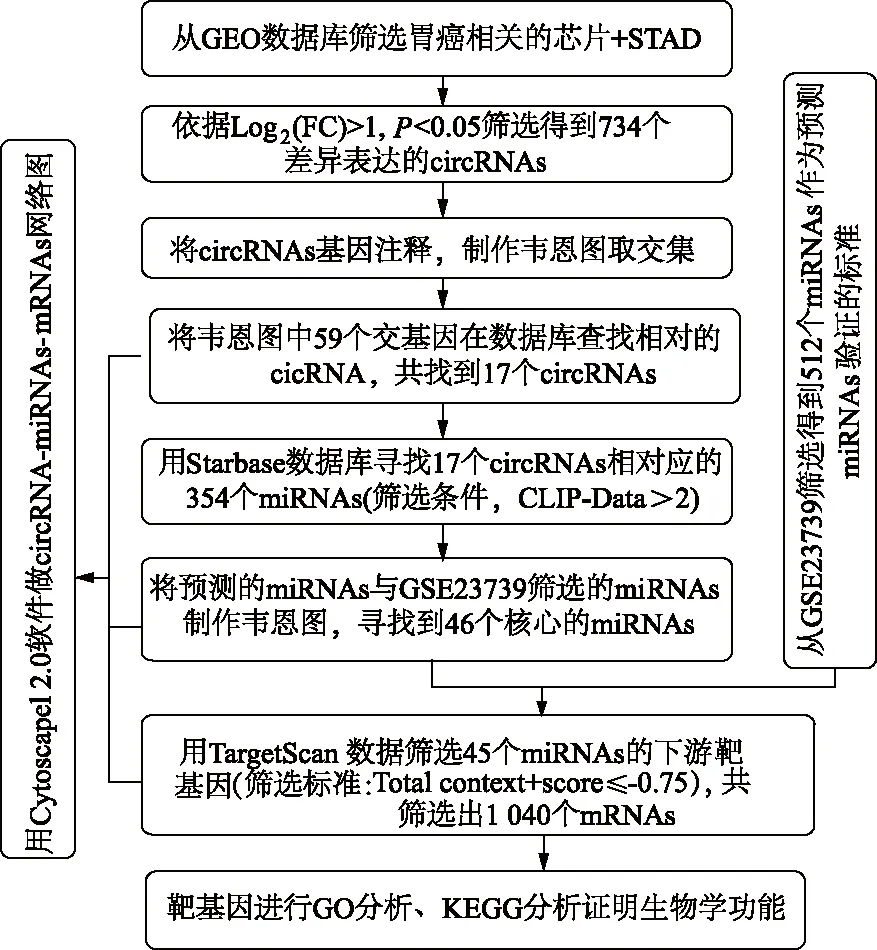
圖1 差異表達circRNAs篩選技術路線圖
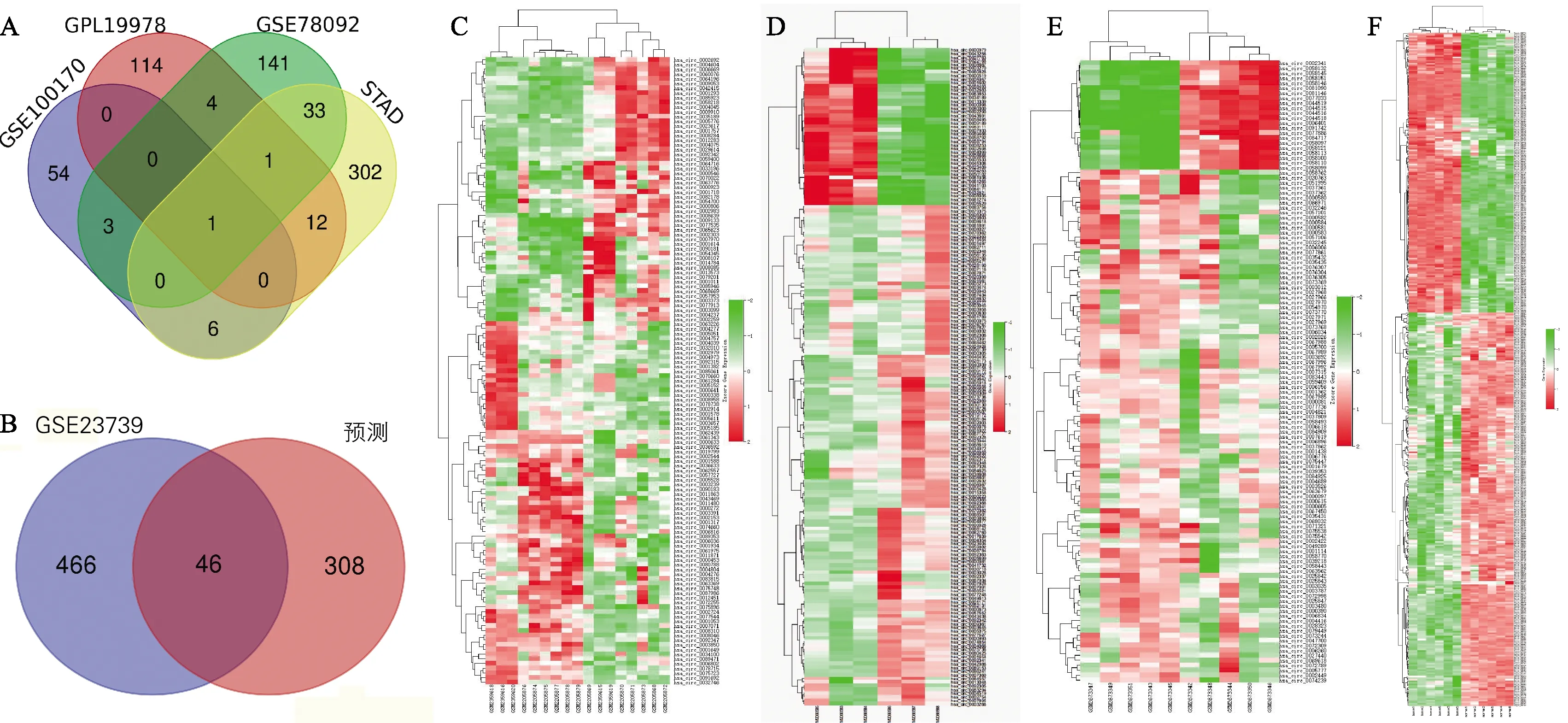
A:4個基因芯片數據(GSE78092,GSE100170,GPL19978,STAD)的韋恩圖;B:GSE23739和預測數據的交集,篩選出46個miRNAs;C:GPL19978(癌組與癌旁組的比例為8 ∶9);D:GSE78092(癌組與癌旁組的比例為3 ∶3);E:GSE100170(癌組與癌旁組的比例為5 ∶5);F:STAD(癌組與癌旁組比例為6 ∶6)圖2 差異表達circRNAs的篩選韋恩圖以及所選數據庫差異表達circRNAs的熱圖
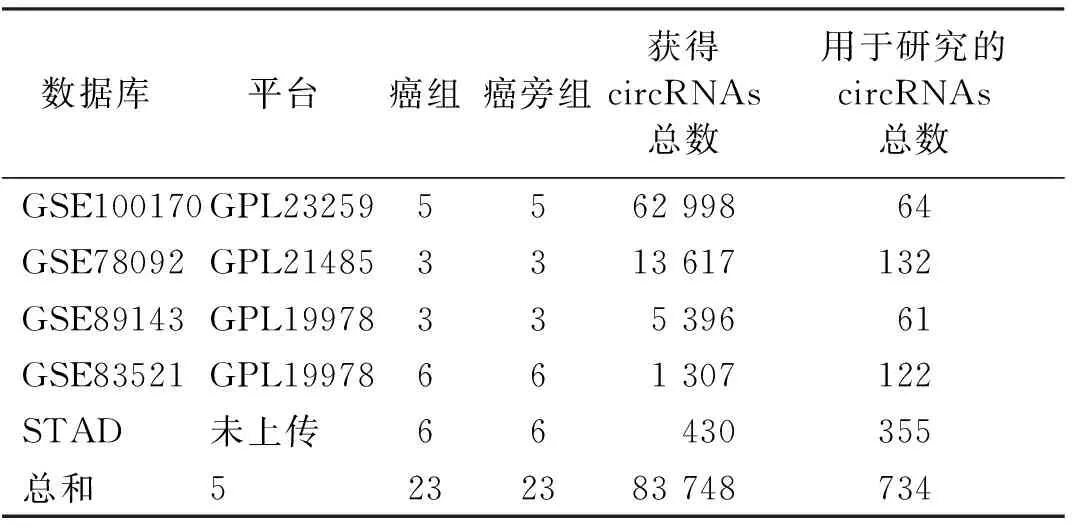
表1 有關數據源的特定信息

表3 差異表達的circRNAs的P值以及倍數變化的對數值

表4 CircRNAs靶向的經過篩選的下游miRNAs
2.2 靶基因的GO分析、KEGG分析以及ceRNA網絡調控
利用TargetScan數據庫,在Total context+score≤-0.75的篩選條件下,篩選出miRNAs下游的靶基因有1 040個。利用Cytoscape 2.0軟件將本研究所分析的17個circRNAs、45個miRNAs以及1 040個靶基因進行ceRNA網絡調控分析(圖3A),結果表明,某些miRNAs(hsa-miR-28-5p和hsa-miR-331-3p)可以結合多個circRNAs,說明其在胃癌的發生發展中發揮核心調節作用。通過GO分析研究核心miRNAs相對應的靶基因,結果表明其主要在蛋白質泛素化,蛋白質轉移、定位,病毒的轉移以及tRNA的核輸出等生物學進程中發揮作用(圖3B)。為進一步研究circRNAs與胃癌的相關性,通過KEGG分析深入研究circRNAs相關靶基因的富集通路,由圖3C可見,其富集通路的途徑主要是通過囊泡轉運中的SNARE相互作用(hsa04130)、氧化磷酸化(hsa00190)、MAPK信號通路(hsa04010)和N-糖基生物合成(hsa00510)來實現。而調控轉錄的生物過程主要是轉錄RNA代謝、細胞內信號傳導級聯和蛋白質定位。
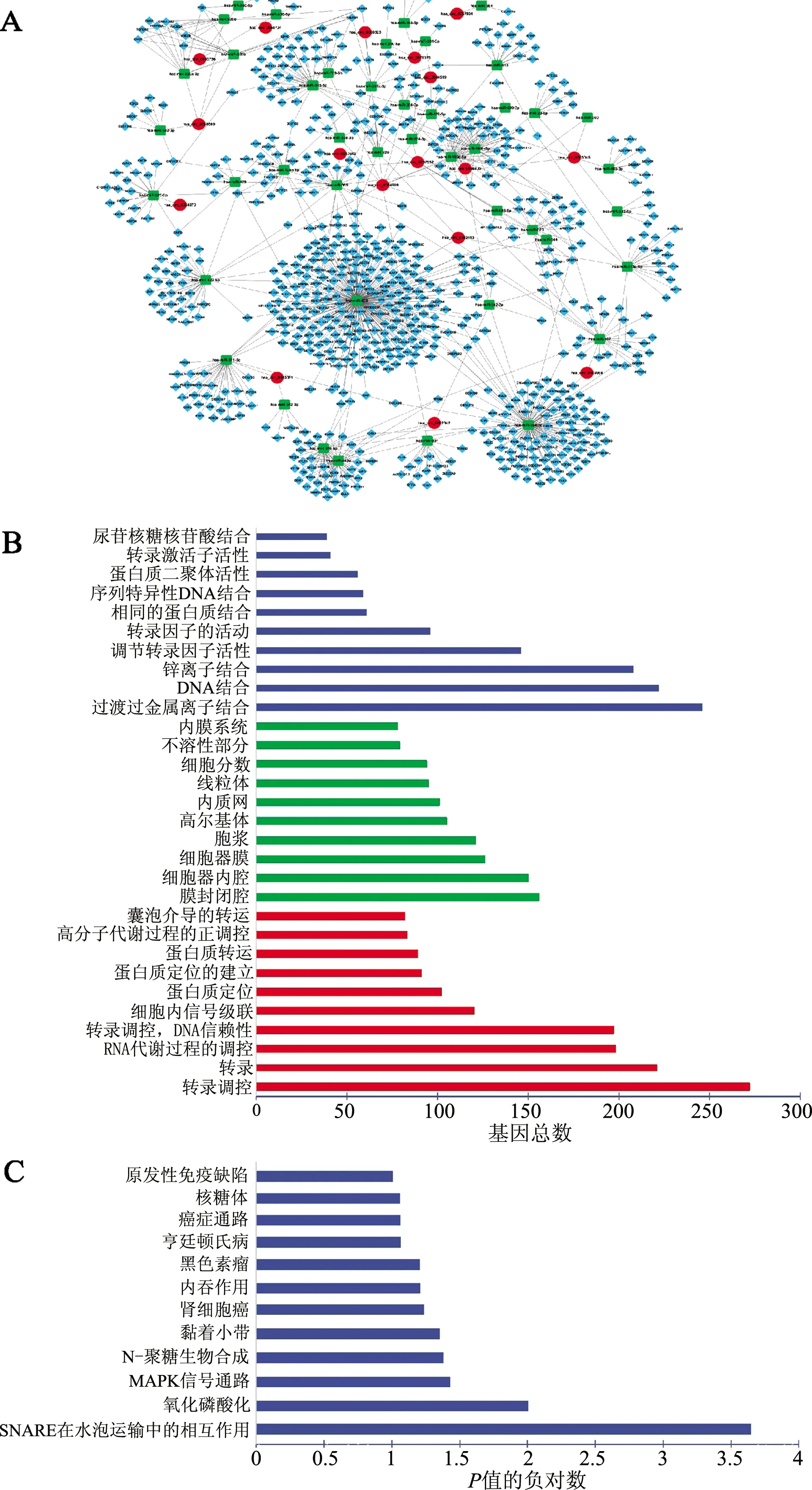
A:用Cytoscape2.0軟件分析circRNAs-miRNAs-mRNAs的ceRNA關系,紅色:circRNAs,綠色:miRNAs,藍色:mRNAs; B:GO分析,紅色:生物進程,綠色:細胞組分,藍色:分子功能;C:靶基因的KEGG分析圖3 靶基因的互作網絡圖以及靶基因GO分析和KEGG分析
由表5可見,miRNAs下游富集的核心基因相關的疾病包括腫瘤、組織學類型的腫瘤、腺癌、皮膚和結締組織疾病、白血病和急性骨髓白血病等,而與其相關程度最大的疾病仍是腫瘤,為我們研究胃癌提供了許多參考依據。CircRNAs在腫瘤中的作用機制研究較多,其中胃癌內的ceRNA網絡調控機制為本課題組的研究重點,為后續研究胃癌相關的circRNAs、miRNAs以及mRNAs奠定了基礎。

表5 核心基因相關疾病 (前10個)
3 討論
本研究利用生物信息學方法發現胃癌差異表達的circRNAs(如hsa_circ_0092342、hsa_circ_0054970和hsa_circ_0084909)可作為胃癌診斷標志物以及發揮ceRNA調控作用,影響胃癌的發生發展。結果表明,hsa_circ_0092342可通過hsa-miR-654-3p靶向CLVS1/PGBD1發揮促進或抑制胃癌的作用;hsa_circ_0054970可通過hsa-miR-525-5p靶向TMEM65對胃癌發揮作用。KEGG分析結果表明,靶基因發揮的主要作用是轉錄調控、RNA代謝過程、細胞內信號級聯和蛋白質定位等。此項研究的意義是發現差異circRNAs表達以及ceRNA調控網絡的變化,尤其是差異表達特別明顯的幾個circRNAs對胃癌的診斷作用。目前為止,研究者已發現胃癌的各種生物標志物,例如腦表達X連鎖4( brain-expressed X-linked 4 ,BEX4),煙酰胺N-甲基轉移酶等[14-15]。多項研究已證實ceRNA調控機制,如circRNA_100269在胃癌中下調,并通過靶向miR-630抑制腫瘤細胞生長;circRNA_001569通過吸附miR-145在胃癌中促進細胞增殖;干擾circRNA_0001947表達可抑制胃癌細胞增殖并誘導其凋亡[16-18]。在胃癌組織中發現大量的miRNAs,高表達miR-125b-5p的胃癌患者生存期明顯縮短,證明miR-125b-5p能促進胃癌的發生發展[19]。本研究中的核心miRNAs為miR-28-5p和miR-331-3p,其中miR-28-5p在胃癌中尚無相關報道,但在鼻咽癌中發現其影響鼻咽癌細胞的增殖、凋亡、遷移和侵襲[20];而miR-331-3p在許多癌癥研究中都有報道,如circCACTIN通過吸附miR-331-3p和調節轉化生長因子β受體1(TGFBR1)的表達促進胃癌進展[21],circ_0038646通過miR-331-3p/GRIK3促進結直腸癌細胞增殖和遷移[22]。
在胃癌中已發現許多circRNAs,部分存在于真核細胞的胞質中,少部分內含子來源的circRNAs存在于細胞核內。若circRNAs主要存在胞質中,則作為miRNAs海綿發揮作用,進而解除miRNAs對其靶基因的抑制作用;若定位于細胞核內,則可充當RNA結合蛋白發揮作用[23]。就目前的研究而言,多數circRNAs主要是通過ceRNA調控機制促進或者抑制胃癌的增殖和遷移,但還有很多尚未知悉的生物學功能,其在疾病診斷治療的研究還不深入,需要進一步求證。今后應深入研究circRNAs在胃癌診斷、治療方面的作用機制,充分了解其生物學功能,使其能更廣泛地應用于研究領域。

