基于K-shell的超網絡關鍵節點識別方法
周麗娜,李發旭,鞏云超,胡 楓
(青海師范大學 a.計算機學院;b.青海省藏文信息處理與機器翻譯重點實驗室;c.藏語智能信息處理及應用國家重點實驗室,西寧 810008)
0 引言




1 相關工作
1.1 超網絡相關概念

1.2 超網絡的拓撲指標

1.3 K-shell算法
K-shell算法是圖算法中的一種經典算法,用以計算每個節點的核數。該算法將網絡劃分為從核心到邊緣的不同層次,具體劃分過程為:首先,將網絡中度為1的節點及其連邊刪除;其次,刪除后網絡中將出現新的度為1的節點,繼續刪除新出現的度為1節點及其連邊;最后,重復上述操作直到網絡中不再新出現度為1的節點為止。此時所有被刪除的節點構成第一層,即1-shell,節點的ks值為1。以此類推,進一步得到更高的殼,直至網絡中的所有節點都被賦予ks值。圖1為3-shell分解過程示例圖。

圖1 3-shell分解示例圖
2 基于超網絡的K-shell算法
2.1 K-shell算法在超網絡中的基本思想及分解過程
在超網絡中,節點超度表示包含該節點的超邊數,通常認為超度大的節點重要性高,但超度僅是衡量節點重要性的局部性指標,忽略了超網絡全局信息對節點重要性的影響。本文將基于復雜網絡位置思想的K-shell指標擴展到超網絡中,提出超網絡的K-shell分解算法。算法步驟為:1)刪除超網絡中超度為1的所有節點,刪除超度為1的節點后若網絡中存在超邊超度為1的超邊,則刪除此超邊;重復此過程,直到網絡中不存在超度為1的節點及超邊,所有被刪除的節點ks值為1;2)重復上述操作,刪除超網絡中所有超度值不大于2的節點和超度為1的超邊,此時,所有被刪除節點的ks值為2;3)以此類推,直到超網絡中所有節點都被賦予ks值。
如圖2所示超網絡的3-shell分解過程示意圖,圖2b~圖2d為1-shell、2-shell和3-shell分解圖。

圖2 超網絡的3-shell分解過程示例圖
圖2a為圖1a中添加超邊以后的超網絡圖,在圖1a中刪除ks值為1的節點后,其余節點的度值均大于1。繼續刪除度值為2的節點,即刪除節點5以及與它相連的兩條邊,則節點1與節點2的度值均變為3。與復雜網絡不同,如圖2a所示,刪除超網絡中節點7和節點8后,所在超邊的超度變為1,依據超網絡的K-shell算法刪除這條超邊。刪除節點9和節點10及其所在的超邊后,繼續刪除網絡中超度變為1的節點6,刪除該節點后,超邊的超度為2,因此保留該超邊。綜上所述,復雜網絡中刪除一個節點時與之相連的邊同時刪除;而超網絡中,只有當超邊中只剩一個節點,即超邊超度為1時,才刪除此超邊。
分析圖2a中,超度最大的節點4、節點14,其超度值為5,由K-shell算法得到節點4的ks值為3,節點14的ks值為1。表明節點14位于網絡的邊緣位置,雖然超度值最大但并不是重要節點。因此,利用超度刻畫超網絡中的重要節點并不完全準確。但由K-shell算法最終分解得到多個重要節點,導致節點的排序結果過于粗糙,如圖2d所示。為了解決此問題,本文進行了改進。
2.2 改進的超網絡K-shell算法
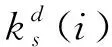
(1)
其中,dH(i)表示節點i的超度,ks(i)表示節點i的ks值。


表1 不同指標對示例超網絡的排序結果
3 實證分析
3.1 示例超網絡分析
為了驗證本文算法的有效性和正確性。對圖2示例網絡進行分析,通過刪除節點形成的子網絡數目及子圖規模大小反映算法的正確性。當刪除節點后生成的子圖數量越多、子圖最大規模越小時,關鍵節點識別算法的準確性越高。表2為按照節點重要性排序逐一刪除節點后得到的子圖數及子圖最大規模。圖3以刪除節點數為橫坐標,以刪除節點后得到的子圖數與子圖規模為縱坐標,展示不同指標之間的差異。

表2 節點被刪除后的子圖數及子圖最大規模

圖3 節點被刪除后子圖數與子圖規模變化情況
3.2 數據來源
蛋白復合物數據來自數據庫CORUM(Comprehensive Resource of Mammalian Protein Complexes)[33]。該數據庫包含2 314個人類蛋白質節點以及它們之間相互作用形成的1 342個復合物超邊。這些復合物大多由3~4個蛋白質組成。此外,最大的復合物包含143個蛋白質,而最小的復合物僅有一個蛋白質。在蛋白質復合物超網絡中,節點的超度表示包含該蛋白質的復合物的個數,也是判斷蛋白質是否是關鍵蛋白的重要依據。
3.3 實驗結果分析
將超網絡中的K-shell算法應用于蛋白復合物超網絡,從而得到所有蛋白質節點的K-shell值與超度的關系,結果如圖4所示。

圖4 超度與K-shell值的散點圖
圖4中橫軸表示K-shell值,處于同一豎線上的蛋白質具有相同的ks值。縱軸的超度表示該蛋白質復合物的數目。此超網絡中,超度值小于10的節點,ks值大部分小于5,占總節點的80%左右,說明大多數蛋白質構成的復合物數量較少。節點360與節點361的ks值為29,處于網絡的中心位置,是網絡的關鍵節點。文獻[34]以介數和節點度為中心測度分析蛋白質在網絡中的位置,提出關鍵蛋白傾向于位于網絡的中心位置。ks值越高代表節點越趨于網絡的中心位置。蛋白復合物超網絡中節點360與361的ks值最大、處于網絡的中心位置,為蛋白復合物超網絡中的關鍵蛋白。由CORUM數據庫可知,這兩個蛋白質結合在一起參與生成的復合物數量占大多數。在生物學中,HDAC蛋白對細胞染色體的結構修飾和基因表達調控發揮著重要的作用,是具有條件依賴性的必需基因,而節點360的蛋白ID是HDAC1,節點361的蛋白ID是HDAC2。由此可知,超網絡的K-shell算法能較為準確地識別網絡中的關鍵節點。
由圖4知,ks值小的節點與其超度值呈線性分布,剩余節點分布則較為分散。例如,節點71的超度值大于節點497,但節點71的ks值卻小于節點497的ks值。節點437的超度值僅次于節點360,但是它們的ks值相差甚大;節點437的ks值為11,位于超網絡拓撲結構的中間位置,因此,蛋白復合物超網絡中不存在節點超度值大,卻處于網絡邊緣位置的節點。節點792,920和921的超度值與ks值均為21,但比較超度與ks值的節點排名相差較大,如節點792,根據超度值排名位于第14位,根據ks值排名位于第6位。若將上述蛋白質移除,至少有21種復合物無法形成,從而影響細胞的生物學功能,甚至使生物體無法存活,因此這些蛋白質是構成此類復合物的必需蛋白質。





圖6 ks值與超度與排名之差

4 結論


