毛竹硝態氮轉運蛋白家族PeNPFs基因鑒定及其表達特性分析
袁婷婷,朱成磊,楊克彬,宋新章,高志民*
(1. 國際竹藤中心竹藤資源基因科學與基因產業化研究所,國家林業和草原局/北京市共建竹藤科學與技術重點實驗室,北京 100102;2. 浙江農林大學,浙江 杭州 311300)
氮是影響植物生長發育的重要礦質營養元素,也是葉綠體、核酸、蛋白質以及很多次生代謝產物的重要組成成分[1]。大多數陸生植物以硝態氮作為主要氮源,并以主動運輸的方式從外部環境吸收硝態氮,運輸到其它組織器官中,供植物生長發育[2]。植物體內的硝態氮轉運體( NRTs)負責從土壤中吸收硝態氮、植物體內硝態氮的運輸和細胞內硝態氮再分配,如NRT1、NRT2和NRT3等[3-4]。植物在進化過程中形成2類不同的轉運蛋白,即高親和力轉運蛋白/系統( HATS)和低親和力轉運蛋白/系統(LATS),它們又分別分為組成型(cHATS和cLATS)和誘導型(iHATS和iLATS)轉運系統。擬南芥(Arabidopsis thaliana)AtNRT1.1(CHL1)是高等植物中第1個被克隆的硝態氮轉運蛋白基因,它的突變導致硝態氮的吸收減少[5],之后又在水稻(Oryza sativaL.)、玉米(Zea maysL.)和小麥(Triticum aestivumL.)中獲得了多種同源基因[6-8]。由于NRT1和PTR (Peptide transporter)成員序列同源性高,且能轉運種類廣泛的底物,它們被統一命名為NRT1/PTR FAMILY,即NPF家族[9]。
植物NPF蛋白可運輸硝態氮、多肽和脫落酸(ABA)等多種底物,是一個功能廣泛的家族,可分為8個亞家族[9-10]。研究表明,在擬南芥中AtNPF4.6負責硝態氮的吸收,它是一個組成型表達的低親和轉運蛋白,也可轉運ABA;AtNPF7.3介導硝態氮的木質部裝載[11];AtNPF1.2的作用是負責木質部到韌皮部的轉移,將硝酸鹽重新分配到正在發育的葉片中[12]。水稻NPF8.9負責硝酸鹽的吸收,有2個不同剪接形式的轉錄本(NPF8.9a和NPF8.9b),它們過表達可增加植株地上干質量;OsNPF6.5作用于硝態氮的吸收和地上地下硝態氮的轉運[13]。玉米中ZmNPF6.6是一個高親和硝酸鹽特異轉運蛋白,而ZmNPF6.4是一個低親和轉運蛋白,同時具有轉入和轉出硝酸鹽的雙向轉運活性[14]。NPF廣泛存在于多種植物中,且成員眾多,如擬南芥NPF家族有53個成員,水稻NPF家族有93個成員[13],之后在黃瓜(Cucumis sativusL.)、毛果楊(Populus trichocarpaTorr. & Gray)、蘋果(Malus domesticaBorkh.)、甘蔗(Saccharum officinarumL.)中相繼鑒定了多個NPF成員[15-18],而竹子中NPFs的相關研究尚屬空白。
竹子是禾本科(Gramineae)竹亞科(Bambusoideae)植物,其中,毛竹(Phyllostachys edulis(Carrière) J.Houz.)是最具特色的竹種之一,具有生長快、材性好、筍材兩用、固碳能力強等特點[19-20]。速生的特點,使得毛竹必須從外界吸收大量的氮素供其快速生長所需[21],因此,生產中毛竹林施用氮肥非常普遍。在一定范圍內(250 kg·hm-2),隨著氮肥施用量增加,毛竹冬筍、春筍和竹材的產量均呈增加的趨勢[22];然而,毛竹如何吸收利用氮素仍不清楚,其分子調節機制及對外界脅迫的響應機制等均有待深入研究。NPF家族成員為氮素的重要運輸者,因此,開展毛竹NPF基因的鑒定與表達模式研究對于揭示其功能具有重要意義。本研究以毛竹為研究對象,利用已有基因組數據[23]對其中NPF家族基因成員進行全面分析,包括基因啟動子、基因結構、編碼蛋白理化性質、保守結構域、系統進化等,并利用毛竹轉錄組數據分析NPF基因的組織表達特異性,以及非生物脅迫冷和干旱處理、激素GA3和NAA處理后的表達模式,以期為深入研究毛竹中NPF基因的功能奠定基礎,為探索毛竹快速生長發育期的氮素吸收利用機制提供科學依據。
1 材料與方法
1.1 毛竹NPF基因序列獲取
分別從 Rice Genome Annotation Project(http://rice.plantbiology.msu.edu/)和Tair (https://www.arabidopsis.org/index.jsp)數據庫下載水稻和擬南芥NPF基因的CDS和氨基酸序列,作為誘餌序列在毛竹基因組數據庫BambooGDB (http://bamboo.bamboogdb.org/)中進行BlastP、BlastN (e-value=1e-10)比對,查找并獲取其中NPF同源基因的候選序列。對獲得的候選序列基于SMART數據庫(http://smart.embl-heidelberg.de)和PFAM數據庫(http://pfam.xfam.org)逐條進行蛋白保守結構域的鑒定和分析,僅保留具有編碼完整保守結構域的序列,并進行基因命名(PeNPFs)。
1.2 基于NPF同源序列的系統進化分析
為分析毛竹NPF成員的進化關系,預測其潛在的功能,使用MEGA 7.0軟件的Clustal W功能對毛竹、水稻和擬南芥的NPF蛋白序列進行同源比對分析,使用鄰接法(Neighbor-joining)構建系統進化樹,bootstrap試驗重復1 000次[24],其它參數設置為默認值。
1.3 毛竹NPF基因結構及其編碼蛋白序列分析
使用Gene Structure Display Server 2.0 (http://gsds.cbi.pku.edu.cn/)在線工具繪制PeNPFs的基因結構圖,利用Plant CARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在線平臺分析其啟動子序列所含作用元件,用ProtParam (http://web.expasy.org/prot-param/)對毛竹NPF基因編碼蛋白質的基本理化特性進行分析,使用MEME Version 5.1.0 (http://meme-suite.org/tools/meme)分析毛竹NPF蛋白的保守基序,使用Plant-mPLoc算法(http://www.csbio.sjtu.edu.cn)預測PeNPFs的亞細胞位置。
1.4 組織表達特異性分析
依據本實驗室前期毛竹7個不同組織(葉、早花期花序、盛花期花序、根、鞭、20 cm筍和50 cm筍)的轉錄組數據[23],用不同組織中NPF基因的RPKM (Reads Per Kilo-bases Per Million Reads)值表示基因的表達豐度,為有效地可視化展示基因表達量,對RPKM的值取以2為底的對數即Log2(RPKM)作為基因的表達量[25]。采用TBtools的熱圖工具[26]制作熱圖展示基因的表達情況,顏色由藍色到紅色逐步變化表示基因的表達豐度數值遞增。
1.5 獲得不同脅迫與激素處理后的轉錄組數據
根據文獻查找,從NCBI數據庫(https://www.ncbi.nlm.nih.gov/sra/)下載獲取毛竹非生物脅迫和激素處理的轉錄組數據。冷和干旱處理的轉錄組數據源自2年生毛竹,5株的離體枝條分別進行冷(0℃和無光條件)和干旱(20℃和相對濕度50%條件)處理2 h后,每個處理分別選取10片未展葉進行轉錄組測序,其RNA-seq數據登錄號為SRS-1759772[27];GA3處理的轉錄組數據均源自毛竹實生苗,1個月的毛竹實生苗分別用水和100 μmol·L-1的GA3噴灑處理后4 h取整株進行轉錄組測序,其對照和處理的RNA-seq數據登錄號分別為SRS256-6465~SRS2566467、SRS2566468~SRS2566470[28];NAA處理的轉錄組數據均源自1個月的毛竹實生苗,用5 μmol·L-1的NAA噴灑處理,間隔1 h處理1次,4 h后取根系進行轉錄組測序,其對照和處理的RNA-seq數據登錄號分別為SRS2294012~SRS2294014、SRS2294015~SRS2294017[29];上述處理取樣時均采用3次生物學重復。從這些轉錄組數據的基因注釋中比對查找,篩選出對應毛竹NPF基因的FPKM值,用于表達分析。
1.6 不同處理后基因的表達模式分析
利用獲取的冷、干旱、GA3和NAA處理毛竹及其對照中NPF基因的FPKM值,比較分析處理前后的基因表達變化情況。為有效地可視化展示基因表達量,用FPKM+1的值取以2為底的對數即Log2(FPKM+1)作為基因的表達量[30]。對所有表達量數據用TBtools軟件繪制表達譜熱圖,分析不同基因的表達情況,同時基于基因FPKM值對具有顯著差異(p< 0.05)表達的基因繪制點線圖。
2 結果分析
2.1 毛竹NPF家族基因成員鑒定與系統進化分析
植物NPF蛋白通常含有PTR2保守結構域,通過比對分析,在毛竹基因組中共鑒定到具有編碼完整PTR2的候選基因27個,其編碼蛋白均含有保守的跨膜結構域,符合MFS (Major Facilitator Superfamily)超家族的NPF家族的特征[31]。根據候選基因在毛竹Scaffold中的位置以及同源基因的名稱,依次命名為:PeNPF1.1~PeNPF8.8。
為確定PeNPFs與其它物種中已鑒定的NPF蛋白之間的進化關系,預測其潛在功能,對毛竹、水稻和擬南芥NPF的氨基酸序列進行了多重序列比對,并利用MEGA7.0軟件構建了系統進化樹。結果表明:所有毛竹、水稻和擬南芥的NPFs被聚類到8個亞家族(S1~S8),PeNPFs成員分布在除S3之外的其它7個亞家族中,每個亞家族中成員數量不等,其中,亞家族S1、S4、S6分別只有1個成員,S8中的成員最多為8個,其余亞家族分別有3~7個成員不等(圖1)。

圖1 基于毛竹、水稻和擬南芥NPF氨基酸序列構建的系統進化樹Fig.1 Phylogenetic tree constructed based on amino acid sequence of NPFs from moso bamboo, rice and Arabidopsis
進一步分析發現,PeNPFs半數成員被聚類在S7 (7個)和S8 (8個)亞家族,而雙子葉植物擬南芥在這2個亞家族的成員分別只有3個和5個,在S5亞家族中,水稻和擬南芥NPF成員分別達到17個和16個,但僅包含6個毛竹成員。雖然NPF家族成員在功能上具有相似性,但不同亞家族成員在功能上也存在一定的差異,如AtNPF1.1和AtNPF1.2負責硝酸鹽向嫩葉的分配[32],而OsNPF 2.4主要在表皮、木質部薄壁細胞和韌皮部的伴胞中表達[33]。另外,所有PeNPFs均優先與水稻NPF聚類在一起,說明在功能上PeNPFs或許與水稻等單子葉植物的同源基因相似。
2.2 PeNPFs基因結構及啟動子分析
基因結構分析表明,PeNPFs均有內含子,數量為2~5個,單個內含子在基因組上的跨度為33 bp(PeNPF7.6的第5個內含子)~3 489 bp (PeNPF2.2的第2個內含子),而單個外顯子在基因組上的跨度為36 bp (PeNPF7.3的第1個外顯子)~1 601 bp(PeNPF7.3第3個外顯子) (圖2)。PeNPFs各個成員之間的內含子和外顯子的數量以及所在的位置存在一定的差異,這可能是影響其轉錄表達的主要原因之一。
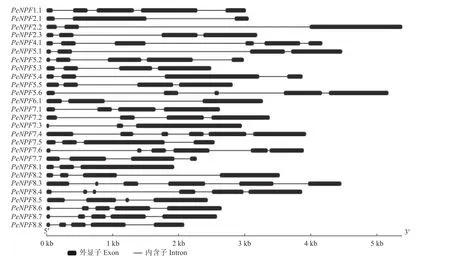
圖2 PeNPFs基因結構Fig.2 Gene structure of PeNPFs
為更好地了解PeNPFs對外界環境的響應和受內在調控因子調節的情況,對PeNPFs的起始密碼子ATG上游2 000 bp的序列所含順式作用元件和應答元件進行分析。結果表明:所有序列中均含有啟動子必需的核心元件TATA-box和CAAT-box。27個PeNPFs的啟動子序列中均包含多種參與基因表達調控的順式作用元件和外界刺激的應答元件,如低溫響應元件LTR,干旱響應元件MBS和TCrich repeats,赤霉素響應元件TATC-box、P-box和GARE-motif,生長素響應元件TGA-element、AuxRR-core和AuxRE,茉莉酸甲酯響應元件CGTCA-motif和TGACG-motif,脫落酸響應元件ABRE等(圖3)。由此表明,PeNPFs的轉錄表達可能會受到非生物脅迫和激素的影響。
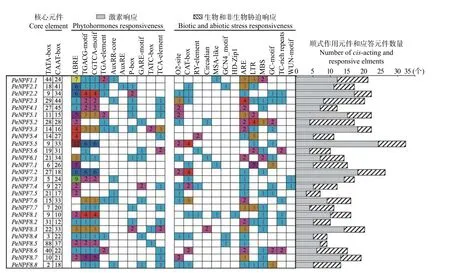
圖3 毛竹PeNPFs基因啟動子響應元件種類統計Fig.3 Statistics of response elements in the promoter of PeNPFs
2.3 PeNPFs的保守基序分析
保守基序分析表明:在毛竹PeNPFs序列中共獲得10個保守基序,分別命名為motif 1~motif 10,其中,motif 1、motif 2和motif 4是27個PeNPFs共有的高度保守基序,而其它基序則在部分序列中缺失。27個PeNPFs中有15個成員含有10個motif,其它12個成員缺失1~3個motif,如PeNPF2.1、PeNPF4.1和PeNPF8.8缺少motif 3,PeNPF8.2缺少motif 5,PeNPF2.1缺少motif 6,PeNPF2.1、PeNPF7.5、PeNPF7.7、PeNPF8.2、PeNPF8.3和PeNPF8.8缺 少motif 7,PeNPF5.2、PeNPF5.5缺少motif 8,PeNPF7.6缺少motif 9,PeNPF5.1、PeNPF5.2、PeNPF5.3、PeNPF5.5缺 少motif 10(圖4A)。
另外,各motif在氨基酸組成的偏好性方面有一定相似性,如motif 1、motif 2和motif 4高度保守,均富含疏水性氨基酸,但各motif在氨基酸組成的偏好性方面也存在一定的差異,如motif 1富含亮氨酸,motif 2富含絲氨酸,motif 4富含甘氨酸(圖4B)。
2.4 編碼蛋白理化性質和亞細胞定位分析
PeNPFs編碼氨基酸序列最長的為761 aa(PeNPF7.4),最短的為452 aa (PeNPF8.8),蛋白分子量介于49.56~82.08 kDa之間,理論等電點介于5.17~9.85之間,大部分是中性或堿性蛋白。親疏水性分析顯示,PeNPFs的親水性值(GRAVY)為0.063~0.607,推測它們屬于親水性蛋白。亞細胞定位預測顯示,所有PeNPFs均定位于細胞膜或液泡中,其中,有8個成員在細胞膜和液泡中均有定位(表1)。
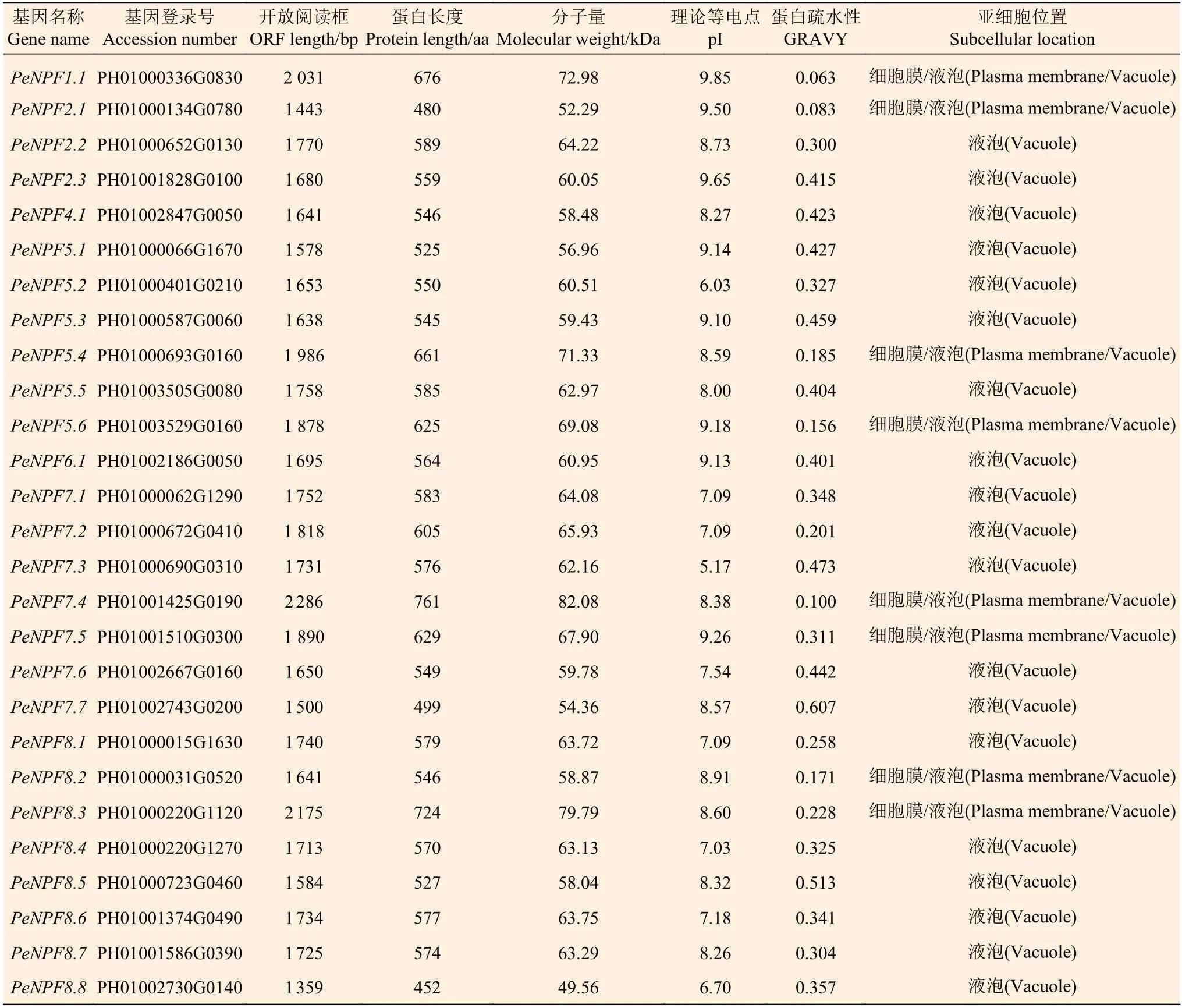
表1 PeNPFs編碼蛋白的基本特征Table 1 Basic characteristics of the proteins encoded by PeNPFs
2.5 PeNPFs的組織表達模式分析
利用毛竹7個不同組織的轉錄組測序(RNASeq)表達譜數據,對PeNPFs的組織表達特異性進行分析。結果(圖5)表明:在不同組織中,PeNPFs表達模式存在一定差異,其中,PeNPF5.3和PeNPF7.6在7個組織中均檢測到表達,且在7個組織中的表達量變化不大,呈組成型表達模式,它們可能與毛竹整個生長發育過程密切相關;而其它PeNPFs成員至少在1個組織中檢測到表達,但各成員的表達豐度差異較大,如在葉片中PeNPF2.3、PeNPF7.5、PeNPF7.7和PeNPF8.7的表達豐度較高;PeNPF-2.1、PeNPF5.6、PeNPF6.1、PeNPF7.1、PeNPF7.2和PeNPF8.2在早花期花序中表達豐度較高;PeNPF-1.1、PeNPF5.2、PeNPF5.6、PeNPF6.1和PeNPF8.1在盛花期花序中表達豐度較高。一些PeNPFs成員表現出組織特異性,如PeNPF4.1主要在竹鞭中表達;PeNPF5.1和PeNPF7.4主要在根中表達;PeNPF8.8主要在20 cm和50 cm高度筍中表達。PeNPFs在葉、花序、鞭和根等不同組織中的表達差異,表明它們在這些組織的生長發育中發揮著不同的功能。
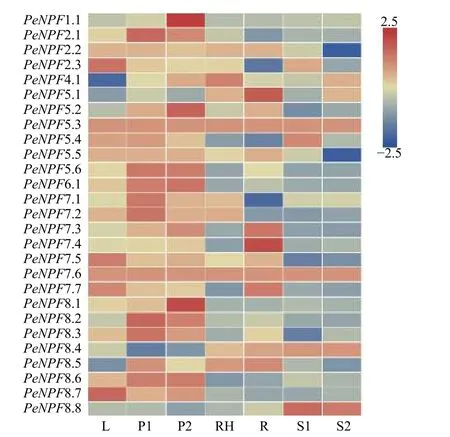
圖5 PeNPFs在毛竹7個組織中的表達分析Fig.5 Expression analysis of PeNPFs in seven tissues of moso bamboo
2.6 毛竹PeNPFs響應非生物脅迫和激素脅迫的表達分析
啟動子調控元件分析表明:PeNPFs的表達可能會受到逆境脅迫的影響,因此,本文利用已發表的轉錄組數據,對冷和干旱處理下毛竹葉片中PeNPFs的表達進行分析。結果表明:在冷和干旱處理的毛竹葉片轉錄組數據中獲得了18個PeNPFs的FPKM值,進一步分析發現它們的表達存在著一定的差異。與處理前(CK)相比,冷處理2 h后共有9個PeNPFs呈下調表達趨勢,9個PeNPFs呈現上調表達趨勢(圖6A);其中,下調表達的基因中有6個(PeNPF2.3、PeNPF5.3、PeNPF5.4、PeNPF7.2、PeNPF7.3和PeNPF8.7)呈顯著下調(p< 0.05),而在上調表達的基因中僅有3個基因(PeNPF5.2、PeNPF7.1和PeNPF8.1)表現為顯著上調(p< 0.05) (圖6B)。在干旱處理2 h后共有15個基因呈現下調表達趨勢,僅有3個基因呈現上調表達趨勢(圖6C);其中下調表達的基因中有6個(PeNPF5.3、PeNPF7.2、PeNPF7.3、PeNPF8.1、PeNPF8.7和PeNPF8.8)呈現顯著下調(p< 0.05),而上調基因的表達差異均不顯著(圖6D)。綜合分析發現,4個基因(PeNPF5.3、PeNPF7.2、PeNPF7.3和PeNPF8.7)在冷和干旱處理后均表現出顯著下調,這與其啟動子序列中存在與低溫和干旱響應調控元件(圖2)相一致,推測它們可能在低溫和干旱響應中發揮著重要作用。
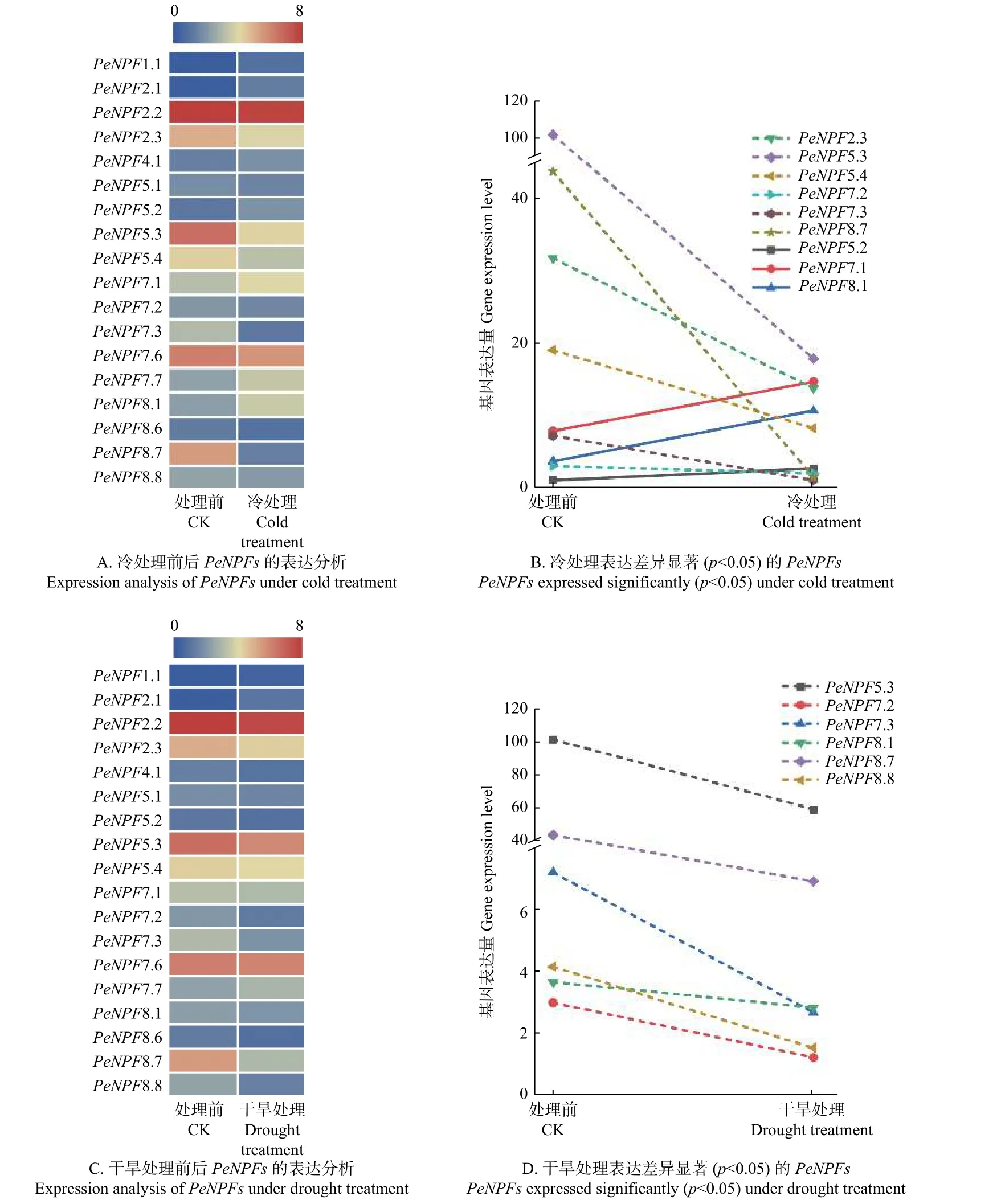
圖6 冷和干旱處理下PeNPFs的表達分析Fig.6 Expression analysis of PeNPFs in moso bamboo under cold and drought treatments
另外,NPF基因的表達還受到激素的影響[34],本文對GA3和NAA處理毛竹幼苗的轉錄組數據中PeNPFs基因的表達模式進行分析。結果表明:在轉錄組數據中獲得了18個PeNPFs的FPKM值,分析發現它們的表達在處理前后存在一定的差異。與處理前(CK)相比,18個PeNPFs在100 μmol·L-1GA3處理4 h后,各有9個基因呈現上調或下調表達趨勢(圖7A),其中,上調表達的5個基因(PeNPF4.1、PeNPF5.3、PeNPF5.4、PeNPF7.1和PeNPF7.6),下調表達的3個基因(PeNPF2.1、PeNPF5.1和PeNPF7.3)的表達差異顯著(p< 0.05)(圖7B);在5 μmol·L-1NAA處理4 h后,7個基因呈上調表達趨勢,11個基因呈現下調表達趨勢(圖7C),其中,PeNPF7.1和PeNPF7.3表現為顯著上調,而PeNPF2.2、PeNPF5.2和PeNPF7.6等基因表現為顯著下調(p< 0.05) (圖7D)。對比分析發現,PeNPF7.3和PeNPF7.6在2種激素處理后呈相反的表達變化,而PeNPF7.1的表達趨勢相同,推測這些基因在響應激素方面可能發揮著不同的功能。
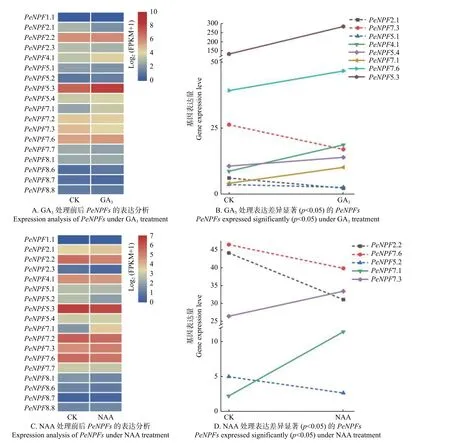
圖7 GA3和NAA處理下PeNPFs的表達分析Fig.7 Expression analysis of PeNPFs in moso bamboo treated with GA3 and NAA
3 討論
NPF蛋白是MFS超家族中成員數量最多的家族之一,這些蛋白質廣泛分布于真核生物和原核生物中,它對于植物、動物、酵母和細菌的氮代謝具有重要作用。本研究從毛竹基因組數據庫中,鑒定到27個NPF成員,這些成員均含有PTR2保守結構域及跨膜結構域,這可能是NPF家族成員具有吸收和轉運硝態氮的基礎。同一個亞家族內的部分NPF成員間的基因結構以及理化性質和亞細胞定位高度相似,如PeNPF8.6和PeNPF8.7的序列的相似性為86.83%,氨基酸長度相差3個,相對分子量、亞細胞位置、基因結構、所含保守基序以及在7個組織中的表達模式相似。另外,部分NPF成員的基因結構、理化性質和亞細胞定位存在較大差異,如毛竹PeNPF5.1和PeNPF5.4序列的同源性為31.19%,氨基酸長度相差136個,二者的分子量分別為56.96 kDa和71.33 kDa,且二者在7個組織中的表達模式相反,這可能是由于非保守區的差異造成的,同時各成員序列非保守區的差異可能導致各個成員的功能具有多樣性[35]。
從毛竹基因組數據庫中,鑒定了27個PeNPFs成員,分別屬于7個亞家族,但沒有鑒定到S3亞家族成員。系統進化分析發現,在其它物種(如水稻)中存在S3亞家族成員,推測本研究中沒有鑒定到S3亞家族成員的原因可能是所用的是毛竹基因組草圖,毛竹可能存在S3亞家族成員,作者將進一步用最新的毛竹基因組進行分析。同時,通過物種進化關系分析發現,聚類在同一個亞家族的成員可能具有相似的功能。在水稻和擬南芥中,S1、S2、S5、S6和S7亞家族成員的主要功能為轉運硝態氮,如OsNPF2.2負責根到地上部分硝態氮的運輸[36],小麥TaNPF6.3負責硝態氮的吸收并調控非根系組織的硝態氮的分配[37],AtNPF7.2和AtNPF 7.3分別負責硝態氮在木質部的卸載和裝載[38],推測毛竹在這5個分枝的成員可能也具有硝態氮的轉運功能。另外,研究表明,AtNPF2.4和AtNPF2.5負責根中Cl-的轉運[39],AtNPF5.2在脅迫條件下可以轉運二肽和三肽,AtNPF7.3還具有轉運K+的功能[40],所以,也不排除相同亞家族中毛竹PeNPFs也具有一些其它功能。基因在不同組織的表達模式是基因功能的重要體現,轉錄組表達譜表明,PeNPFs在毛竹各組織的功能存在差異,多數基因在葉片和花序中的表達量較高,在根和筍中的表達量相對較低。3個毛竹NPF基因具有組織特異性,PeNPF1.1只在盛花期花序中表達,PeNPF5.1只在根中表達,PeNPF8.8只在筍中表達,表明它們可能分別在盛花期花序、根以及筍中發揮著重要作用;而它們分別屬于不同的亞家族,說明各亞家族之間功能有一定的差異。擬南芥S8是小肽轉運體,AtNPF8.1和AtNPF8.2能夠轉運多肽,同時AtNPF8.3在開花和種子發育過程中將小肽轉出液泡,而毛竹S8成員中多個成員在花序發育時期的表達量較高,說明它們也可能參與毛竹開花發育時期小肽的轉運。
真核生物基因的表達受到多層次調控,轉錄因子能夠通過與基因的啟動子特定區域結合從而調控基因的轉錄表達,因此,對基因的啟動子所含的調控元件進行分析對于基因功能的預測具有重要意義。本研究發現,在PeNPFs中發現大量與逆境脅迫及激素響應相關的調控元件,這些元件已經被證實在植物逆境脅迫[41]及植物生長與果實成熟等方面均發揮著重要作用。當植物受到生物或非生物脅迫時,NPF基因通過上調或下調表達發揮其功能。冷處理后,18個PeNPFs中有9個PeNPFs的表達量發生了顯著變化(上調或下調),說明毛竹在一定程度通過PeNPFs表達量變化來響應冷處理;干旱處理后,18個PeNPFs中有15個基因的表達量下調,其中,6個與處理前差異顯著,這與干旱處理能顯著誘導小麥NPF基因表達量下調的結果一致[34]。在GA3處理4 h后18個PeNPFs均發生變化,其中,PeNPF4.1、PeNPF5.3、PeNPF5.4、PeNPF7.1和PeNPF7.6的表達量顯著上升,推測它們可能受GA3的誘導并通過表達量變化進行響應,這與施用GA3能夠增加ZmNRT2.1/2.2的表達水平相似[42];NAA處理后,毛竹有5個基因發生顯著上調或下調,推測毛竹S7亞家族可能參與毛竹響應NAA過程。毛竹中PeNPFs不同成員對GA3和NAA處理呈現不同程度、甚至相反的表達變化,表明各成員功能的差異,這與水稻中OsPTRs在GA3和NAA處理后的表達類似[4]。PeNPFs在毛竹中的具體功能和作用機制需要進一步實驗驗證。
4 結論
本研究從毛竹基因組中共鑒定出27個PeNPFs基因,所有成員編碼的蛋白均包含完整的PTR2保守結構域,根據其蛋白序列差異分為7個亞家族,各亞家族成員之間的分子特征和組織表達特異性均存在著一定的差異,部分成員的表達表現為組織特異性或組成型表達。在PeNPFs啟動子序列中鑒定出多種與非生物脅迫和激素響應相關的調控元件,冷、干旱以及GA3和NAA處理后毛竹的轉錄組表達譜證明PeNPFs參與了不同程度的非生物脅迫和激素的應答,研究結果為揭示PeNPFs的生物學功能提供了重要依據。但對于PeNPFs的研究尚在起步階段,其在毛竹中的具體功能尚需展開大量的實驗進行驗證。

