豬精子發生候選基因PYGO2的分離、表達和亞細胞定位研究
趙 筱,張 霞,范 俐,楊 忠,霍金龍,曾日彬,劉麗仙,霍海龍*
(1.云南農業職業技術學院,昆明 650212;2.呂梁學院生命科學系,呂梁 033001;3.云南省農業科學院糧食作物研究所,昆明 650205;4.云南生物制藥有限公司,昆明 650503;5.云南農業大學動物科學技術學院,昆明 650201)
PYGO2(pygopus family PHD finger 2),也叫PYGOPUS2或PYGOPUSHomolog2,是PYGOPUS家族成員,在果蠅中被首次鑒定為Wnt信號通路中β-連環蛋白/TCF復合物的必需轉錄共激活因子,它作為一種DNA結合蛋白以Wnt依賴性和非依賴性途徑發揮功能[1]。Wnt信號通路是一個復雜的蛋白質相互作用網絡,該通路參與細胞(包括干細胞)的增殖分化過程,其功能涉及胚胎發育、組織穩態維持和癌癥發生等方面,也參與骨骼發育、骨代謝、細胞分化、極性和凋亡等生理過程[2]。該通路與細胞間的旁分泌或細胞自身的自分泌通信相關,在動物中高度保守,共序列在果蠅、小鼠、大鼠、牛、羊、狗、人類中都非常類似[3]。PYGO2能與甲基化的組蛋白結合,招募RNA聚合酶Ⅱ,進而激活靶基因轉錄[4-5],促進組氨酸乙酰化,并調控染色質濃縮[6]。PYGO2能影響細胞的侵染和轉移,能通過Wnt/β-catenin信號通路促進血管的滲透[7],在腦、眼睛、毛囊、皮膚、肺、腎、乳腺、胰腺、卵巢、食道以及生殖腺等組織中廣泛表達[8]。PYGO2調節錯誤或靶向缺失會導致機體嚴重的發育障礙,甚至會引發多種疾病,包括乳腺癌[9]、神經膠質瘤[10]、卵巢癌[11]、肝癌[12]、食管癌[13]、結腸癌[14]、肺癌[15]、肥胖以及糖尿病[16]等。PYGO2能通過Wnt依賴性機制調節基因轉錄、細胞分化及精子發生[6, 12]。睪丸組織中的PYGO2主要在染色質重塑的長形精子細胞中表達,是促進精子成熟的正調控因子,也因此被稱為染色質重塑因子,它可以通過調節組蛋白甲基化和乙酰化而參與染色質重塑過程并引發主動轉錄[17]。另外,PYGO2可擔當CTNNB1、HNMT、TMPRSS11D(HAT)與染色質之間的支架蛋白,并在CTNNB1的核保留中起重要作用[4]。PYGO2在睪丸中以PHD依賴性方式與染色質結合,其具有染色質重塑的睪丸特異性,錯誤調節會導致精子在生成過程中發生障礙[17]。敲除PYGO2基因的雄性小鼠,其精蛋白的表達明顯減弱,導致精子缺陷、數量減少或無精子,進而引起小鼠發育異常和不育[4]。在人上,PYGO2編碼區突變會導致蛋白空間結構破壞并誘發特發性少精子癥和無精子癥[18],PYGO2還參與生殖細胞中不依賴于HAT的調節機制,不僅與染色質結合,而且在睪丸中參與了長形精子細胞的染色質重塑[19]。最近發現,PYGO2在雄激素依賴性和非雄激素依賴性PrCa細胞系的過度表達可能導致前列腺癌加重甚至轉移[20],故PYGO2還可作為前列腺切除術后PSA進展的潛在診斷標記[21]。
近幾十年來,Y染色體微缺失、囊性纖維化、精索靜脈曲張、克萊氏綜合征等導致精子數量減少,男性不育癥成比例增加[22]。但導致不育的原因較為復雜,特發性少精子癥和無精子癥是多病因引發的,其具體發病機制尚未被完全闡明,因此,研究人員一直致力于尋找引發這些疾病的根本原因[23]。基于PYGO2基因在精子形成過程中的重要作用,本研究以版納微型豬近交系(BMI)為研究材料,克隆豬PYGO2基因,并進行了一系列蛋白質生物信息學功能分析,利用qPCR和亞細胞定位確定其表達,為PYGO2在豬精子生成方面的功能研究奠定基礎。
1 材料與方法
1.1 試驗動物和試劑
版納微型豬近交系10月齡公豬3頭,體況良好,來自昆明原種豬場,屠宰取睪丸和其他組織樣品,試驗所需試劑均購自大連TaKaRa公司。
1.2 基因擴增及序列測定
根據GenBank豬PYGO2 mRNA序列(NM_001185175)設計PYGO2-clone引物(表1),以BMI睪丸cDNA為模板擴增PYGO2基因全長編碼區。反應體系25 μL:2×GC Buffer 12.5 μL,2.5 mmol·L-1dNTP mix 2.5 μL,10 μmol·L-1PYGO2-clone上、下游引物各1 μL,20 ng·μL-1cDNA 1.5 μL,Ex Taq酶0.5 μL,H2O 6 μL。擴增程序:95 ℃ 5 min;95 ℃ 30 s,59 ℃ 30 s,72 ℃ 80 s,35個循環;72 ℃ 10 min。擴增產物送昆明碩擎生物公司測序。

表1 PYGO2基因引物信息
1.3 mRNA表達分析
以睪丸組織cDNA為模板制作標準曲線,利用引物PYGO2-qPCR(表1)檢測PYGO2在BMI豬15種組織的mRNA表達,同時以GAPDH(表1)為內標進行校正,采用2-ΔΔCt方法進行數據分析。
1.4 亞細胞定位檢測
根據上述已獲得的PYGO2基因CDS序列及pEGFP-C1綠色熒光蛋白表達載體上酶切位點的分布情況,設計PYGO2-ex引物(表1)再次擴增豬PYGO2基因。將產物與pMD18T克隆載體連接,然后將重組載體pMD18T-PYGO2與pEGFP-C1載體同時雙酶切(EcoRⅠ、BamHⅠ),獲得真核表達重組質粒pEGFP-C1-PYGO2,轉染豬睪丸ST細胞系。用紅色的Mito Tracker染色ST細胞線粒體,用藍色的Hoechst 33342染色ST細胞細胞核,顯微鏡下檢測PYGO2蛋白在ST細胞中的表達定位情況。
1.5 生物信息學分析
利用Lasergene7.1校對測序的PYGO2序列,用ProtParam程序預測PYGO2蛋白質的分子量、分子式、等電點、正負電荷殘基數;PYGO2蛋白質的二級結構、疏水結構和功能位點分別使用SOPMA、ProtScale和Prosite預測;用MEGA7.0分析7個哺乳動物PYGO2氨基酸序列同源性并構建進化樹;用String進行蛋白互作網絡分析。
2 結 果
2.1 PYGO2基因及氨基酸結構信息
利用PYGO2-clone引物擴增PYGO2基因的完整編碼區(CDS)及部分非編碼區(UTR),獲得1 229 bp 長的產物(圖1A)。PYGO2基因CDS序列長1 221 bp,編碼406個氨基酸(圖1B),含有PHD_PYGO2保守結構域(圖1B,1C,1D)。PYGO2定位于豬(Sscrofa11.1)4號染色體,全長4 268 bp,Ensemble網站分析發現有4個外顯子和3個內含子(圖1D)。
2.2 PYGO2蛋白質序列及結構分析
豬PYGO2蛋白質分子量41.19 ku,分子式C1826H2775N507O545S20,等電點6.89,負電荷殘基和正電荷殘基均為26。PYGO2蛋白質406個氨基酸的二級結構中無規則卷曲占比最大,包含307個氨基酸;α螺旋次之,有57個氨基酸;延伸鏈28個氨基酸;β轉角最少,有14個氨基酸。蛋白質的第379位氨基酸具有最大疏水值1.778,第43位氨基酸處具有最小疏水值-3.389,N端和C端均疏水。含有酶磷酸化、酰胺化等活性位點。
2.3 PYGO2多物種氨基酸序列同源性分析
7個哺乳動物PYGO2氨基酸序列比較結果發現,豬PYGO2(AVB77243.1)與牛Cattle(NP_001095712)、綿羊Sheep(XP_014962310)、小鼠Mouse(NP_001280697)、大鼠Rat(NP_001099917)、人Human(NP_612157)和黑猩猩Chimpanzee(XP_008968530)相似性分別為98.3%、97.8%、97.5%、97.3%、97.3%、97.3%,7個 物種間僅有24個不同氨基酸位點(圖2A),構建的NJ樹表明,牛與綿羊聚為一小支,黑猩猩與人聚為一小支,然后兩者與豬聚為一大支;大鼠與小鼠聚為一支(圖2B)。
2.4 PYGO2蛋白互作網絡分析
蛋白互作網絡分析顯示,豬PYGO2與9個蛋白可能存在相互作用,包括BCL9、CTNNB1、UCK1、JUP、LEF1、CBY1、DKK1、WDR5、ENSSSCG00000015829,其中與BCL9、WDR5、CTNNB1、UCK1、JUP、LEF1相互作用較為緊密,與BCL9蛋白相互作用最為緊密(圖3)。
2.5 PYGO2的多組織熒光定量表達
PYGO2 mRNA在被檢測的15個組織中均有不同程度表達,在尿道球腺、前列腺、睪丸、精囊腺、附睪等生殖腺中表達相對較高,在十二指腸、結腸和肺中也有一定表達,在其余組織表達相對較低(圖4)。

柱上不同大寫字母表示差異顯著
2.6 PYGO2亞細胞定位分析
亞細胞定位結果表明,PYGO2蛋白主要定位在豬睪丸ST細胞系的細胞核內,也有少量分布于細胞質,與PSORT網站預測的94.1%定位在細胞核一致(圖5)。
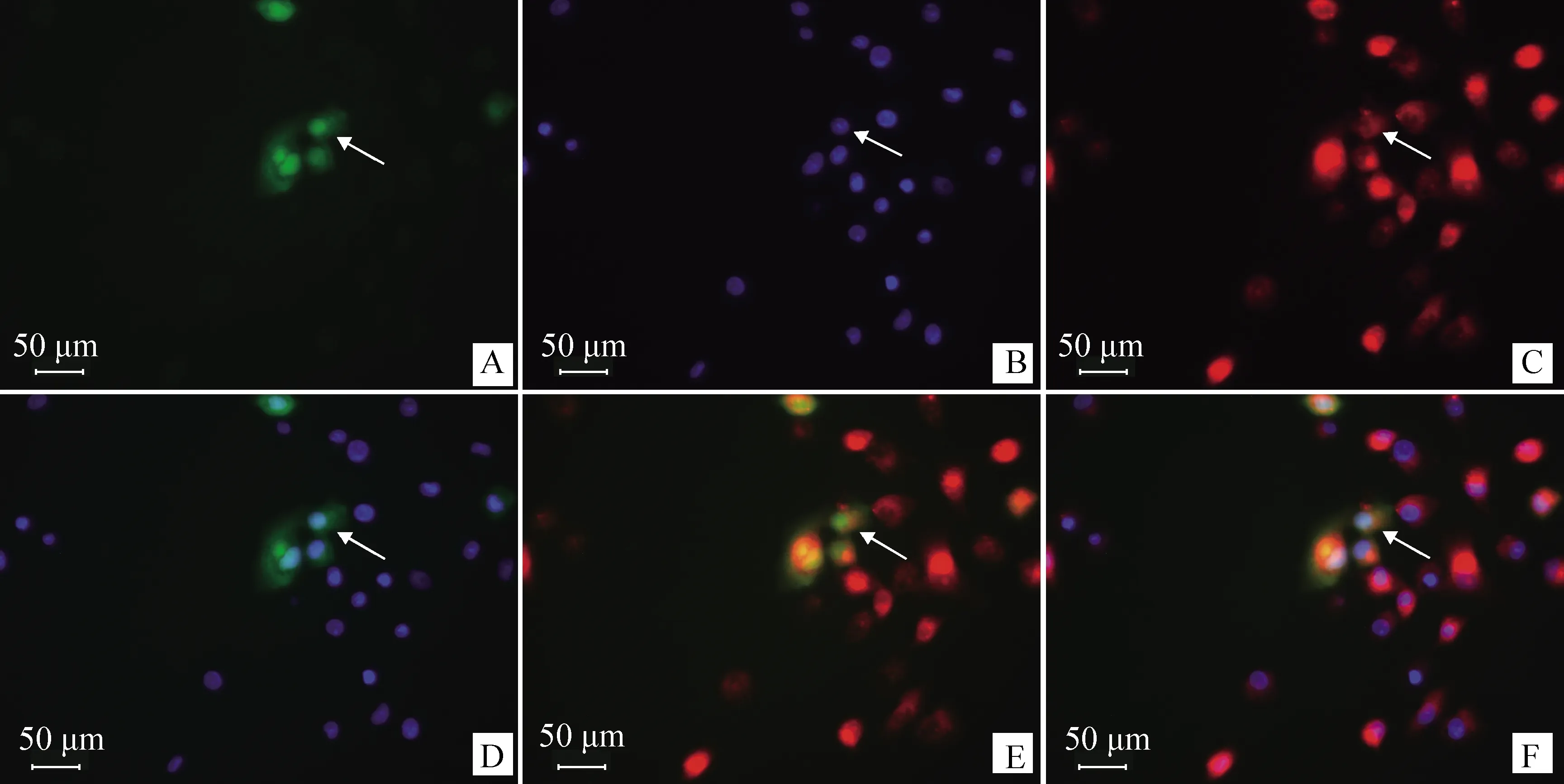
箭頭所示:A.綠色熒光蛋白;B.藍色細胞核;C.紅色線粒體;D.融合的綠色熒光蛋白與細胞核;E.融合的綠色熒光蛋白與線粒體;F.融合的綠色熒光蛋白、細胞核和線粒體
3 討 論
本研究通過BMI睪丸cDNA擴增獲得了PYGO2基因序列1 229 bp(圖1A),包括編碼序列1 221 bp,編碼406個氨基酸(圖1B、1C),序列已提交GenBank,獲得基因和蛋白質登錄號分別為KY644518和AVB77243.1。豬PYGO蛋白C端包含PHD鋅指結構域(圖1B、1C、1D),在329~382位共53個氨基酸。PHD的長度一般多為50~80個氨基酸,其手指基序類似于金屬結合RING域和FYVE域,以單個手指的形式出現,在物種進化中高度保守,常以兩個或三個為簇,也可以和其他結構域共同參與基因表達調控[3, 24]。攜帶PHD結構域的蛋白質具有“讀碼器”功能,能將染色質重塑與基因轉錄的變化聯系起來[25],但PYGO2在睪丸中體現的是與染色質結合的結構蛋白的功能,并非經典的轉錄因子功能,PYGO2-A342E突變會導致小鼠睪丸特異性缺陷,但組織形態沒有明顯異常,表明PYGO2在睪丸中結合的是染色質[26]。Liang等[8]在哺乳動物中發現,PHD可與組蛋白H3上被甲基化的賴氨酸結合,參與組蛋白修飾因子的募集及組蛋白甲基化,并通過Wnt通路的靶基因招募β-catenin來激活轉錄,去除PHD結構域后,PYGO2的轉錄活性降低了80%。但果蠅的PHD結構域攜帶了一個氨基酸突變,使其無法結合H3K4me,這可能是PHD結構域在物種進化上的一個例外[26]。

A.PYGO2基因RT-PCR產物:M.DNA相對分子質量標準;1.PCR產物。B.PYGO2基因編碼序列及氨基酸序列:雙下劃線表示起始密碼子,單下劃線為保守結構域PHD_PYGO2(329~382位氨基酸),星號表示終止密碼子;上一行字母為核酸序列,對應的下一行為其編碼的氨基酸序列。C.PYGO2蛋白質保守結構域。D.PYGO2所處染色體位置、cDNA序列和蛋白質保守結構域
PYGO2主要在Wnt/β-連環蛋白信號傳導通路中發揮重要功能,也是Wnt和Akt兩個信號通路下游的共同節點,可通過翻譯后修飾調節正常組織和腫瘤組織中細胞的生長和分化[27]。在豬PYGO2蛋白質也發現了一些磷酸化和酰胺化位點,這為進一步研究該基因翻譯后修飾的功能奠定了基礎。氨基酸序列比對發現,豬PYGO2與牛、綿羊、小鼠、大鼠、人和黑猩猩等哺乳動物序列相似度均大于97%,7個 物種間僅有24個差異氨基酸位點(圖2A),表明PYGO2在物種進化上高度保守。

圖2 豬和其他6個物種PYGO2氨基酸序列差異位點(A)及分子系統進化樹(B)
蛋白互作網絡分析發現,PYGO2與BCL9、CTNNB1、UCK1、JUP、LEF1、CBY1、DKK1、WDR5、ENSSSCG00000015829等9個蛋白存在相互作用(圖3)。其中,ENSSSCG00000015829還未注釋基因名字;BCL9和PYGO2的相互作用最為明顯,能對Wnt/β-Catenin信號通路起正調節作用,是基因轉錄的關鍵銜接蛋白[28],PYGO2-BCL9復合物可作為結合染色質的連接因子,增強Wnt通路的特異性[29]。CTNNB1(也稱為β-catenin)在Wnt-β-catenin信號傳導途徑中起關鍵作用,不僅與多種腫瘤的發生相關,也在動物生殖生理中發揮重要功能,如在山羊中,它的可變剪接與轉錄和生理過程相關,26 bp片段的缺失和初產仔數相關[30]。UCK1是基于KLHL2的E3連接酶的直接生理底物,能催化尿苷磷酸化為尿苷5′-單磷酸,和KLHL2形成二元復合物,處于嘧啶-核苷酸生物合成的挽救途徑,在細胞凋亡中發揮重要作用,是腫瘤識別的分子標記[31]。JUP能與SOX4一起調控Wnt信號,與睪丸生殖細胞腫瘤相關[32]。LEF1是轉導Wnt3a信號的主要轉錄因子,是癌癥預后的生物標志物[33],特別在前列腺癌方面,LEF1與PYGO2一樣,是引發和維持癌變的信號通路和機制中心,可以調節雄激素活性和其受體的表達,是理想的治療靶標[34]。CBY1參與β-catenin的組成型激活,該蛋白下調能促進β-catenin在細胞核區域的滯留和轉錄激活,能影響干細胞未折疊蛋白的應激反應和引發細胞自噬[35]。DKK1分泌蛋白可通過誘導細胞凋亡和阻止細胞增殖來抑制Wnt/β-catenin通路,DKK1失調與阿爾茨海默病、骨形成和癌癥的發病有關,也可以作為這些病癥的生物標志物,DKK1同樣也是癌癥免疫療法的理想靶標[36]。WDR5能靶定組蛋白H3,在H3K4三甲基化和MLL復合物的形成過程中發揮重要功能,WDR5也是胚胎干細胞多種復合物的組成部分[37]。以上8個與PYGO2互作蛋白的發現為進一步深入研究PYGO2的功能提供了方向。
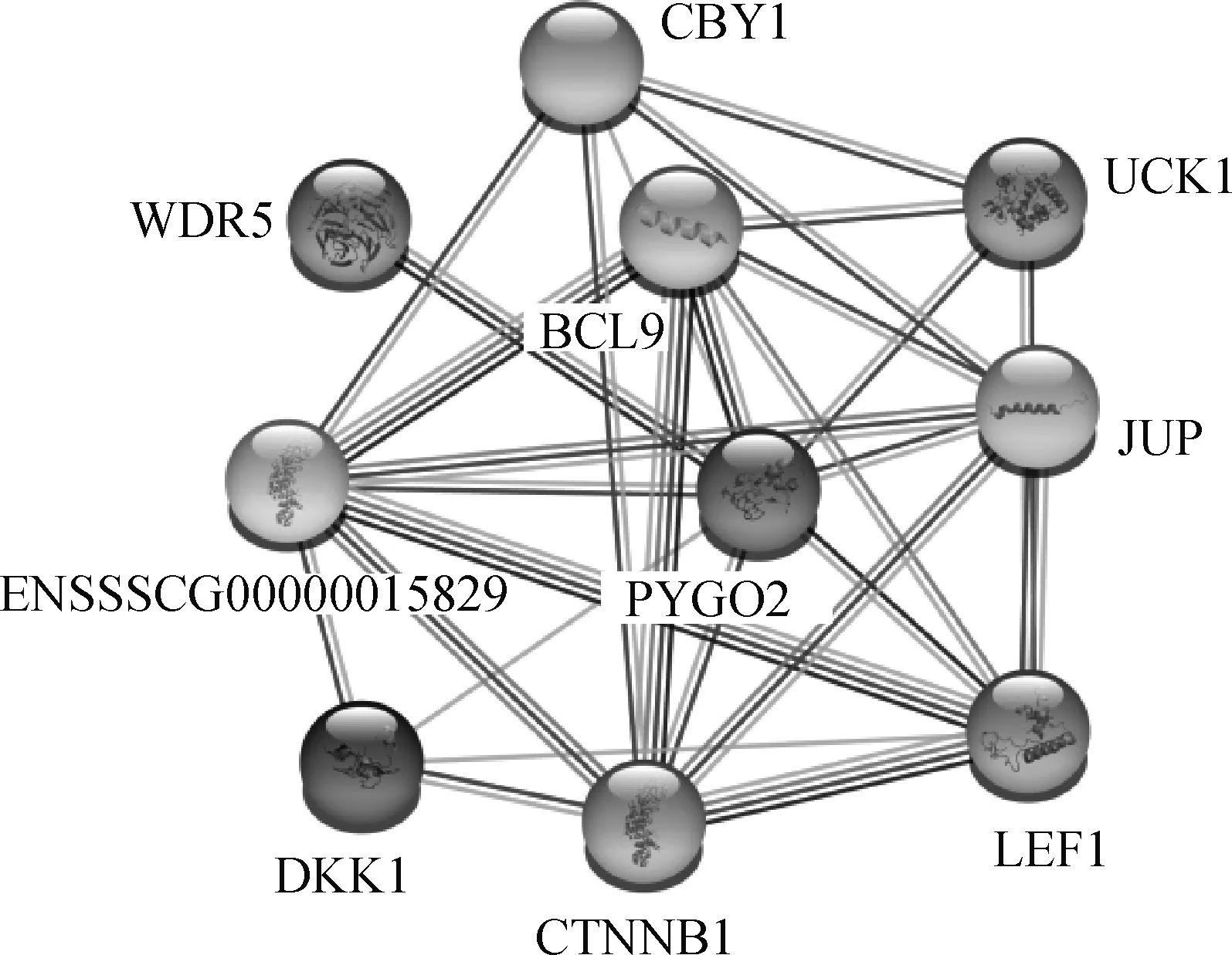
線表示蛋白質之間作用的置信度,線越多置信度越高
豬PYGO2 mRNA在被檢測的15個組織中均有表達,但主要在性腺和副性腺中相對高表達,這說明PYGO2在生殖腺中發揮重要功能;在副性腺特別是尿道球腺中表達最高(圖4),說明該基因在豬中也可能參與交配的性興奮過程,并促進了一些蛋白酶、唾液酸和氨基糖類物質的分泌。小鼠中的研究表明,PYGO2蛋白可以在多種組織、多個系統中廣泛表達,由于該基因參與Wnt信號,故只有在Wnt信號激活后才能維持這些組織的發育和形態發生[38]。PYGO2在睪丸中的表達主要限制在生精過程中染色質重塑發生階段的長形精子細胞中,降低PYGO2的表達會阻滯精子發生,小鼠可以存活到成年,但是雄性生殖細胞會發生核致密化,最終導致雄性不育[4]。進一步,本研究的亞細胞定位結果顯示,PYGO2蛋白主要在細胞核內表達,這與在人[38]和小鼠[26]中的研究結果一致。
4 結 論
本研究使用RT-PCR方法獲得了BMIPYGO2基因的CDS序列;利用生物信息學分析方法研究了該基因的結構、蛋白質的功能和特性,構建了系統進化樹及蛋白質互作網絡;通過qPCR獲得了PYGO2多組織mRNA表達譜;通過綠色熒光表達載體pEGFP-C1獲得了亞細胞定位。本研究可為PYGO2基因在版納微型豬近交系中的功能研究奠定基礎,也可為其他哺乳動物特別是人類男性不育病理原因的深入挖掘提供基礎資料。

