基于WGCNA分析和風險評分建模的骨肉瘤轉移標志物篩選和預后研究*
仲 濤 朱英會 仲偉俍 王志堅 于英楠△ 田 康
【提 要】 目的 應用WGCNA分析篩選骨肉瘤轉移標志物,通過風險評分建立骨內瘤預后的預測模型,為評估骨內瘤轉移和預后提供方法指導。方法 基于GSE14359數據集(n=20),WGCNA法確定骨內瘤轉移模塊中的中心基因;獲取TARGET公共數據庫骨內瘤樣本(n=86),在單因素Kaplan-Meier預后分析基礎上,Cox多因素計算風險評分構建評估患者生存的預測模型。結果 中心基因HLA-DRA和FLI1與骨內瘤轉移密切相關(P<0.05);風險評分=0.149×G0S2-0.572×ARHGDIB+0.048×CD74+0.242×HLA-DMA-0.473×MGAT1-0.813×PLD3+0.230×EPAS1,有較好的預后評估能力(P<0.001;HR=2.72)。結論 WGCNA分析能夠有效篩選OS轉移標志物,風險評分能識別更靈敏的預后模型。
骨肉瘤(Osteosarcoma,OS)是骨骼系統中一種侵襲性惡性腫瘤,其病情發展迅速且預后不良,已經成為兒童和青少年的主要致死性疾病[1]。雖然近年來的新輔助化療不斷發展,手術切除技術不斷進步[2],但是由于缺少早期篩查標記物,約20%OS患者在診斷時就已經出現轉移,特別是肺轉移[3],因此其5年生存率仍然沒有得到顯著提升[4]。因此,尋找潛在的生物標志物來評估OS的轉移和預后具有重要的臨床應用價值。
WGCNA分析[5-6],即加權基因共表達網絡分析,首先通過計算基因之間的表達相關性,將具有相似表達模式的基因聚類到一個模塊中并篩選出中心基因,然后再分析該模塊與樣本特征(包括臨床病理參數和治療方法等)之間的相關性。目前,由于骨肉瘤的異質性,基于小樣本的差異表達分析的骨肉瘤轉移生物標志物往往敏感度和特異度較低,因而缺乏臨床應用價值[7]。WGCNA則從復雜的多樣本轉錄組數據中快速地提取出與轉移相關的模塊及基因,在比較模塊內連接性和基因重要性基礎上,獲得適用性更廣泛的生物標志物[8-9]。風險評分建模基于Cox回歸賦予多個基因的風險系數后計算個體預后風險高低[10],并分析其與患者生存的相關性,多項研究證實多基因模型預后評估的準確性往往高于單一基因[11-13]。
本研究將WGCNA分析用于骨肉瘤轉移標志物的篩選;采用風險評分建模進行預后模型的構建,同時繪制ROC曲線評估多基因模型的準確性。
材料與方法
1.數據來源
本研究收集的數據分別下載自GEO和TARGET數據庫。其中,GEO來源的GSE14359數據集包括2例正常樣本和18例OS組織,后者中有8例是轉移瘤;GSE32981包括5例未轉移OS及11例轉移瘤樣本。從TARGET數據庫獲取的86例OS患者中,有57例生存,29例死亡,中位生存時間為1323(0~5840)天。
2.統計分析方法
(1)基因差異表達分析:使用R語言limma包分析差異表達基因,并繪制火山圖進行可視化。
(2)加權基因共表達網絡分析(WGCNA):通過計算尺度獨立性(R2)和平均連通性以確定表征基因符合無尺度分布的軟閾值,Pearson法選定與“轉移”相關的模塊及其中心基因。
(3)預后分析:風險評分是通過多因素Cox回歸計算風險系數得到的[14],繪制受試者工作特征曲線(ROC曲線)來表征預后模型的靈敏度;log-rank單因素Kaplan-Meier法用于分析單個指標或者風險評分與OS預后的相關性[15]。
結 果
1.識別OS樣本構成的模塊
分析GSE14359數據差異基因發現,相比于正常樣本,OS中有上調基因1108個,下調基因1419個(圖1A)。進一步WGCNA分析顯示,通過尺度獨立性和平均連通性比較發現基因間聯系軟閾值為5后(圖1B),繪制聚類樹狀圖得到了29個關鍵模塊(圖1C)。
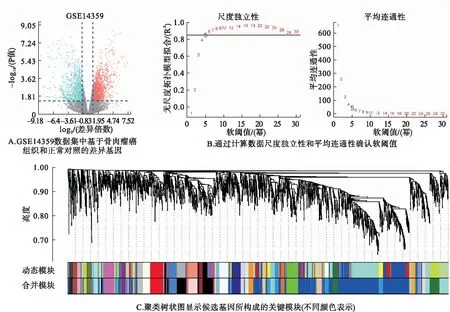
圖1 WGCNA法基于差異基因確定的軟閾值并識別共表達網絡模塊
2.確定OS轉移相關的中心基因
深入分析發現綠色、藍色和棕色模塊與骨轉移密切相關(圖2A);為了進一步驗證模塊中參與調控OS轉移的關鍵基因(n=48),將其與OS轉移和非轉移的差異基因取交集,結果發現只有HLA-DRA和FLI1在OS轉移過程中表達失調(圖2B)。
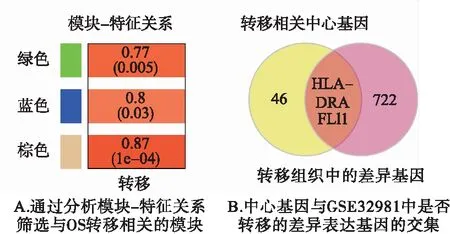
圖2 篩選與OS轉移相關的中心基因
3.分析中心基因與OS患者預后的相關性
通過單因素Kaplan-Meier法分析48個中心基因與OS患者預后的相關性發現,PLD3、ARHGDIB、G0S2、MGAT1、CD74、HLA-DMA高表達時,患者生存時間延長;而EPAS1高表達時,患者預后不良(圖3),ROC曲線分別評估它們,預測預后1、3、5年的靈敏度和特異度(圖4)。
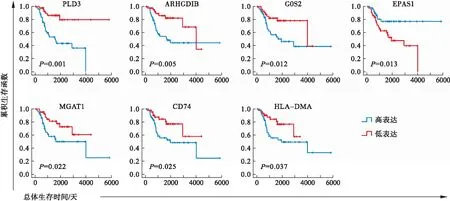
圖3 中心基因和OS預后的Kaplan-Meier曲線

圖4 中心基因預測預后的ROC曲線
4.構建OS患者預后評估最佳模型
將上述7個基因納入多因素Cox回歸分析后得到預后模型:風險評分=0.149×G0S2-0.572×ARHGDIB+0.048×CD74+0.242×HLA-DMA-0.473×MGAT1-0.813×PLD3+0.230×EPAS1(圖5A)。ROC曲線顯示該模型具有較好的準確性(AUC>0.7)(圖5B)。此外,相比于單因素,七基因組合與預后的相關性更顯著,提示更強的評估能力(P<0.001;HR=2.72),見圖5C。
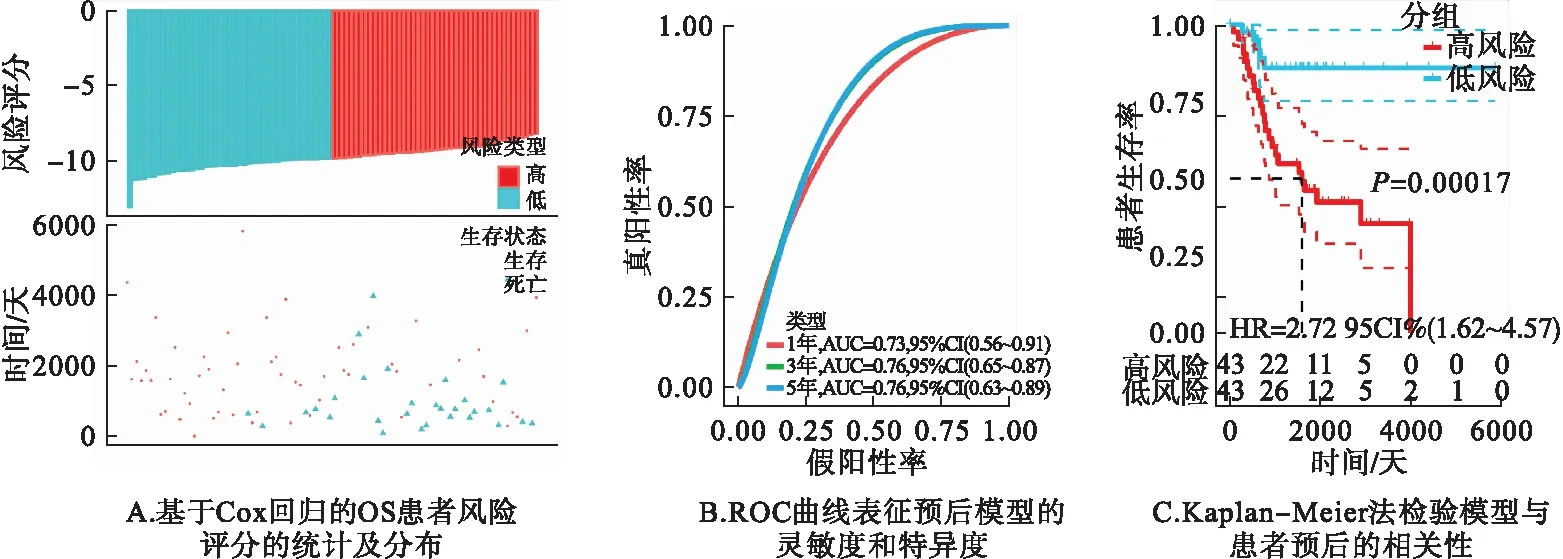
圖5 通過計算風險評分構建預測OS患者生存的高效模型
討 論
OS是最常見的原發性骨惡性腫瘤,在年輕患者中表現出高度侵襲性和早期轉移性[3]。為了實現OS的及時診斷和治療,尋找潛在的生物標志物已經成為目前亟待完成的任務。本研究基于GSE14359和GSE32981數據集,采用WGCNA法篩選出2個與OS轉移顯著相關的中心基因(HLA-DRA和FLI1);進一步分析TATGET數據庫發現,通過Cox回歸風險評分得到的“七基因預后模型”有更好的評估能力。
在本研究中,通過WGCNA法篩選得到三個與OS轉移特性密切相關的模塊中的48個中心基因(P<0.05)。新近研究發現,Tian等同樣基于OS樣本的差異表達基因通過WGCNA法篩選出能分型轉移的SVM過濾器[16];此外,Zhang等使用WGCNA法分析GSE21257數據來確定區分OS轉移的預測標記[17];Wang等則通過分析收集的52例OS樣本的RNA-seq結果[18],采用WGCNA法模塊性狀分析得到其與轉移正相關在內的關鍵模塊及中心基因,為骨肉瘤的分子機制提供見解。值得注意地是,為了精確篩選預測因子,本研究進一步通過交集GSE32981中轉移與否樣本的差異基因,最終確認HLA-DRA和FLI1參與OS轉移調控。由此,本研究在WGCNA法的基礎上進行深入過濾,對篩選OS轉移潛在生物標志物方法進行了優化。
本研究基于單因素Kaplan-Meier法探討OS轉移相關的中心基因對患者預后的影響,結果發現,PLD3、ARHGDIB、G0S2、MGAT1、EPAS1、CD74及HLA-DMA參與了患者生存時間的調節(P<0.05)。為了構建出更好的預后標記,本研究進一步通過計算七個基因的風險系數賦予每個患者相應的預后評分。結果發現,七基因模型的預后評估效能明顯提升(P<0.001;HR=2.72)。有研究報道,Niu等發現EGR1、CXCL10、MYC和CXCR4均可作為OS預后的潛在生物標志物[19];Nakka等的結果也表明miR-21 和miR-221在OS中的表達差異對患者預后具有重要意義[20]。然而,以上證據只提供了單個基因的生存評估作用,本研究則與Liu等[11]基于Cox回歸模型發現兩基因(PML-EPB41)模型比單獨基因具有更好的預后預測價值相似。本研究通過計算風險評分將患者分為高或低風險組,并證實高或低風險組患者生存時間差異更顯著,從而進一步證實多基因模型能更有效地評估OS預后。

