基于GBS技術構建四倍體雜交冰草超高密度遺傳連鎖圖譜
楊東升,于 卓,于肖夏,李佳奇,李景偉,吳國芳,盧倩倩
(內蒙古農業大學農學院,內蒙古呼和浩特 010019)
冰草屬(Agropyron)為小麥族多年生及禾本科草本植物,主要分布于歐亞大陸的溫帶和溫寒帶草原區,共有10個種,其中中國有5個種。細胞遺傳學分析表明,冰草屬有二倍體(2n=2x=14,PP)、四倍體(2n=4x=28,PPPP)和六倍體(2n=6x=42,PPPPPP)3個染色體倍數水平,自然界中以二倍體種和四倍體種占多數,六倍體種較為少見[1]。該屬植物根系較發達,具沙套,抗旱、抗寒性強,適應性廣,是我國北方天然草地改良和人工草地建植的優質飼草[2],同時也是小麥、黑麥等麥類作物抗逆性遠緣雜交改良的寶貴基因資源[3-4]。目前有關冰草的形態學、細胞學、分子生物學等方面已有研究[5-7],但關于冰草高密度和超高密度遺傳作圖方面的研究報道較少。Zhang等[8]以冰草Z2098×Z1842的113個F1分離單株為作圖群體,利用特定長度擴增片段測序技術(specific length amplified fragment sequencing,SLAF-seq)[9]開發SNP標記,首次構建了含 1 032個SNP標記、平均圖距為1.5 cM的二倍體冰草高密度分子遺傳連鎖圖譜,其覆蓋基因組長度為907.8 cM。本課題組前期將產于內蒙古草原抗寒抗旱性強的二倍體野生蒙古冰草(A.mongolicumKeng,2n=2x=14,PP)與引自美國的優質高產的二倍體栽培品種航道冰草(A.cristatumcv. Fairway,2n=2x=14,PP)進行人工授粉雜交,成功獲得種間雜種F1植株,進而用秋水仙堿溶液對F1種子進行染色體加倍,最終得到了遺傳穩定的四倍體雜交冰草[10]。為構建四倍體雜交冰草的高密度分子遺傳連鎖圖譜,通過人工套袋自交獲得了四倍體雜交冰草F2代的347個分離單株,以此為作圖群體,利用AFLP(amplified fragment length polymorphism)和SSR(simple sequence repeats)第一代分子標記技術,首次構建了含766個分子標記(581個AFLP和185個SSR)、平均圖距為3.82 cM的四倍體雜交冰草高密度遺傳連鎖圖譜,其覆蓋基因組長度 2 574.3 cM[11]。
為構建超高密度(平均圖距<0.5 cM)四倍體雜交冰草的分子遺傳連鎖圖譜,本研究以四倍體雜交冰草F2代的115個分離單株及其親本蒙古冰草、航道冰草為遺傳作圖材料,利用基因分型技術(genotyping-by-sequencing,GBS)技術[12],構建首個超高密度的四倍體雜交冰草遺傳連鎖圖譜,以期為深入開展冰草產量、品質及抗旱、抗寒等重要性狀的QTL精細定位、功能基因圖位克隆和分子標記輔助育種研究奠定基礎。
1 材料與方法
1.1 供試材料及試驗地概況
試驗材料為四倍體雜交冰草F2代的115個分離的無性系單株及其二倍體親本蒙古冰草(♀)和航道冰草(♂),種植于呼和浩特市內蒙古農業大學農場的多年生牧草材料圃內,株距25 cm,行距45 cm。該試驗地位于111°42′ E、45°57′ N,海拔1 063 m,年降水量約400 mm無霜期約145 d,土質為沙壤土,pH值在7.8~8.2之間,肥力中等,具灌水條件。生長期內,視生長狀況適時適量灌水施肥。
1.2 冰草基因組DNA的提取及其質量檢測
于2019年5月10日冰草拔節初期,分別剪取四倍體雜交冰草的115個F2分離單株及其親本的幼嫩葉片約1 g,裝入自封袋并迅速放入冰盒中帶回實驗室。每個材料準確稱取0.15 g,剪碎后混勻,置研缽中加適量液態氮充分研磨成細粉狀后,用北京TIANGEN公司生產的植物基因組試劑盒提取DNA,具體方法按照說明書的方法 進行。
用1.2%的瓊脂糖凝膠電泳檢測DNA的純度,用超微量分光光度計(NanoDrop ND-2000,USA)測定DNA的濃度,將符合試驗要求的各樣品DNA稀釋至80 ng·μL-1,置于-40 ℃冰箱內保存備用。
1.3 GBS文庫的構建與測序
供試材料基因組DNA測序采用GBS方法,由北京安諾優達基因科技有限公司完成。具體步驟如下:
(1)用多種限制性內切酶對四倍體雜交冰草F2群體分離單株及其親本的基因組DNA進行酶切,根據酶切試驗結果選出最適合的限制性內切酶進行DNA酶切。在內切酶兩端分別加上P1和P2接頭,其中P1接頭含有PCR擴增所需的引物序列、Illumina測序引物結合位點序列和區分不同樣品的短標簽序列,混合不同接頭的樣品,然后進行PCR擴增。
(2)對質量檢測合格的DNA樣品,用GBS建庫方式構建長度范圍在300~500 bp之間的pair-end文庫。
(3)對PCR產物進行純化,并對構建好的文庫進行質檢,用Illumina HiSeq PE150平臺進行高通量測序。
1.4 SNP標簽的篩選與過濾
對各樣品中的標簽(Tag)進行比對和歸類,按照每類Tag的深度信息由大到小排序,獲得所有個體的Tag頻數表及雜合位點信息。綜合考慮個體間的Tag頻數表信息,尋找個體之間單堿基差異信息并進行篩選和過濾SNP標記,要求Tag完整度>80%,雜合率<5%,等位基因型頻率>5%。最后過濾掉偏分離(P<0.05)的SNP標記,保留多態性好、高質量的SNP標記,用于構建冰草超高密度遺傳連鎖圖譜。
1.5 SNP分子遺傳連鎖圖譜的構建
采用遺傳作圖軟件Join Map 4.0,參考李佳奇等[13]的方法構建四倍體雜交冰草超高密度分子遺傳連鎖圖譜,略作修改。具體步驟如下:
(1)打開軟件,執行New Project命令新建一個文件,點擊Load Date命令導入數據。
(2)運行LOD Grouping(tree)命令,生成樹狀圖,選擇14個LOD≥5.0的連鎖群,打開圖譜分組命令(create groups for mapping),獲得標記不同的連鎖群。
(3)先用Calculate Map命令構建出各個連鎖群SNP圖譜,然后用Map Chart 2.3繪制出含14個連鎖群的超高密度(平均圖距<0.5 cM)分子遺傳連鎖圖譜。
2 結果與分析
2.1 冰草GBS測序數據分析結果
對四倍體雜交冰草F2代115個單株及其親本的基因組DNA測序結果見表1。共獲得 3 989 607 400 reads和584 895 503 397 bases,reads質量值≥30的堿基(Q30,可識別堿基的正確率為99.9%)平均占比為94.14%,GC堿基含量平均占比為43.13%。說明測序質量和文庫構建質量均較高,滿足后續試驗要求。
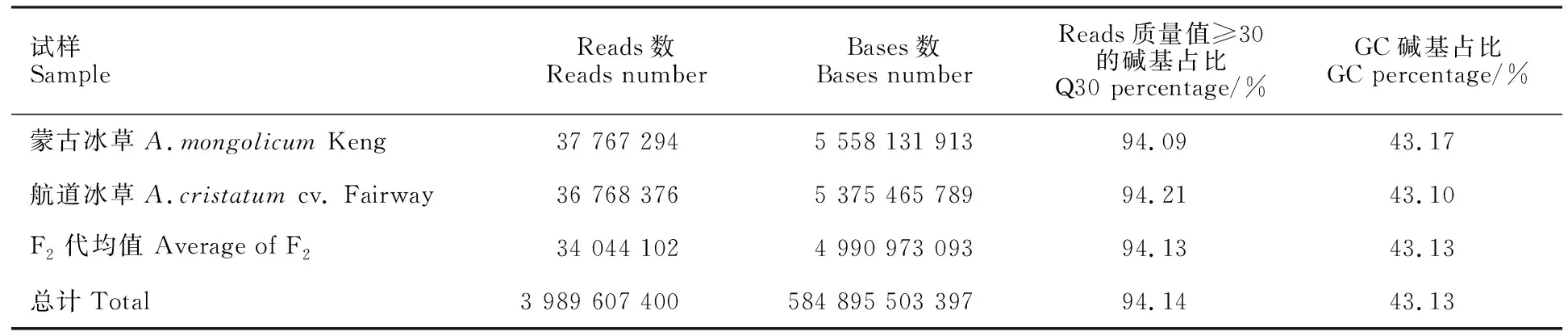
表1 GBS測序結果統計Table 1 Statistics of GBS sequencing results
2.2 冰草的基因分型與SNP過濾
各樣品的基因分型數據見表2。共鑒定出 98 695個高可信度的多態性SNP標記,其中有 34 105個SNP標記具有多態性。在34 105個候選標記中,過濾掉F2分離群體的低覆蓋率序列(覆蓋率<80%),剩余13 164個標記。經偏分離過濾后,最終得到5 035個可用于遺傳作圖的高質量SNP標記。

表2 不同基因分離類型的SNP標記數Table 2 Number of SNP markers in different types of gene segregation patterns
2.3 四倍體雜交冰草超高密度遺傳連鎖圖譜的構建
按照軟件Join Map 4.0操作程序,將GBS測序分析獲得的5 035個高質量SNP標記分成14個連鎖群(LG1~LG14),構建出一張超高密度四倍體雜交冰草分子遺傳連鎖圖譜(圖1)。統計發現(表3),該圖譜覆蓋基因組長度2 148.621 cM,各連鎖群的長度范圍在68.959~280.309 cM之間,平均長度為153.473 cM,以連鎖群LG12最長,LG6最短。5 035個SNP標記不均勻分布在LG1~LG14連鎖群上,每個連鎖群的SNP標記數目在152~767之間,其中LG9的標記數目最多,LG5的標記數目最少,平均標記數目約為360個;各連鎖群標記間的平均間距在0.120~1.304 cM之間,圖譜SNP標記平均間距為0.427 cM,為目前所報道的冰草超高密度分子遺傳連鎖 圖譜。

表3 四倍體雜交冰草遺傳連鎖圖譜特征Table 3 Basic characteristics of genetic map of tetraploid hybrid crested wheatgrass
另外,從圖1中的連鎖群LG6和LG7看出,在同一位點上有多個標記,稱為共位點標記,這種現象在植物超高密度遺傳連鎖圖譜上常有出現[14-15],其實際應用價值尚待深入研究。

圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass

續圖1 四倍體雜交冰草超高密度遺傳連鎖圖譜Continued Fig.1 Ultra-high density genetic linkage map of tetraploid hybrid crested wheatgrass
3 討 論
3.1 遺傳作圖群體的選擇
選擇合適的作圖群體是構建植物遺傳連鎖圖譜的重要基礎,而親本的選配會直接影響作圖群體的分離程度及構建圖譜的難易程度,所以盡可能選擇親緣關系較遠的品種作親本,通常利用F2、BC(backcross)、DH(doubled haploid)、RIL(recombinant inbred lines)或NIL(near isogenic lines)群體來構建遺傳連鎖圖譜[16]。其中,F2群體是由2個高度純合的親本雜交產生的F1代進行自交后得到的分離群體,具有成本低、時間短、遺傳信息量大的優點,是目前應用較廣泛的遺傳作圖群體,如谷子[17]、棉花[18]、芝麻[19]、菠菜[20]和高丹草[21]。
本研究以蒙古冰草×航道冰草雜交后經套袋自交獲得的四倍體雜交冰草F2代115個單株為材料,獲得了5 035個高質量的SNP標記,表明該作圖群體的基因組DNA中含有豐富的SNP多態性標記。原因可能是親本材料分別為內蒙古草原的野生蒙古冰草和美國的航道冰草,它們生長的自然環境條件、形態特征和生物學特性等差異很大[22],其種間雜交F2代的性狀分離明顯而引起的,同時亦說明F2代性狀分離越明顯的材料越適合進行植物的遺傳作圖研究。
3.2 GBS技術與SNP標記的開發應用
構建高質量的超高密度分子遺傳連鎖圖譜是植物產量、品質、抗性等重要性狀QTL精細定位及分子標記輔助選擇等研究的基礎[23-24],標記數目越多、密度越大,遺傳作圖就越精確、實用。GBS技術是在第二代測序技術的基礎上,高效開發的植物基因組SNP分子標記新技術,目前已成功應用于小麥[25]、玉米[26]、水稻[27]等多種重要作物的SNP標記開發及高密度分子遺傳圖譜的構建中。該項技術的最大優點在于構建遺傳圖譜過程中不受參考基因組的限制[28],無參考基因組的植物亦可進行基因分型和大規模地開發SNP標記。而用傳統的RFLP[29]、AFLP[30]及SSR[31]等分子標記構建的植物遺傳連鎖圖譜,往往因標記數目有限而出現較大的連鎖空隙或較多的連鎖斷點,且這些方法在基于凝膠電泳統計條帶時易造成人工誤差。另外,這些方法作圖耗時、成本高,且需要選擇大群體[32],難以滿足后續QTL精細定位等研究的需求[33]。用GBS技術開發的新型SNP標記可以彌補這些傳統分子標記的不足。
冰草還未有全基因組測序數據,為無參考基因組植物。本研究利用GBS技術對四倍體雜交冰草F2分離群體及其親本蒙古冰草和航道冰草基因組DNA首次進行測序分析,成功地獲得了3 989 607 400 reads,經過完整度和偏分離過濾及SNP基因分型共得到5 035個高質量的SNP標記,進而利用Join Map 4.0作圖軟件首次構建了四倍體冰草超高密度遺傳連鎖圖譜,其含有14個連鎖群,覆蓋基因組長度為2 148.621 cM,標記間的平均間距為0.427 cM,是目前飼草中分子標記數目最多、密度最高的遺傳連鎖圖譜,因此,通過GBS技術大規模開發的SNP標記,能有效增加冰草各連鎖群的標記數目,提高遺傳連鎖圖譜的整體密度和飽和度,這為冰草的圖譜構建提供了最新方法,也為后續從正向遺傳學角度深入開展冰草品質、抗寒、抗旱等優異性狀基因的圖位克隆及其功能研究奠定了堅實基礎。

