基于R-shiny的臨床數據分析應用
崔怡丹 魏 婷 孔偉名 俞章盛,△
【提 要】 目的 針對臨床數據變量類型多、數據結構復雜的特征,基于R-shiny開發交互式分析臨床數據并展示結果的應用。方法 基于R-shiny與R語言,搭建應用的UI(用戶界面)和Server(后臺功能),將相應代碼封裝并部署在網頁上。以UK Biobank乳腺癌數據為例,對其臨床及基因數據進行分析,查看R-shiny應用的分析效果。結果 構建出能夠交互式分析臨床數據的R-shiny應用,可供用戶上傳臨床數據、生存型數據及基因數據進行分析。將UK Biobank數據作為示例進行分析,得到了具有臨床與生物學意義的結果。結論 將R-shiny與臨床數據分析結合,為使用者提供了分析臨床數據的簡便方式,并為UK Biobank數據提供新的交互式展示分析平臺。
R語言是免費開源的編程語言[1],適用于各種計算機系統,具有強大的統計分析和圖形繪制功能。R-shiny是RStudio公司基于R語言推出的Web開發工具[2],借助R語言強大的數據分析與圖形繪制功能,幫助熟悉R語言的編程者開發以瀏覽器為依托、具有交互功能的應用。使用者無需學習R語言,僅通過鼠標點擊的方式就可以輕松使用shiny應用。常見的shiny應用包含UI(用戶界面)和Server(后臺功能)兩部分,其中UI的功能對應B/S架構中的B(Browser瀏覽器)端[3],Server對應S(Server應用)端。UI包括sidebarPanel(側邊欄,顯示選項)與mainPanel(主頁面,展示結果),負責展示用戶界面布局并輸出后臺運算結果;Server負責抓取UI動態并在后臺進行相應運算。通過UI與Server配合,能夠實時對用戶在UI頁面的指令進行反饋。本應用使用shiny的navbarPage框架作為UI的架構基礎,運用HTML與Javascript對其外觀與功能進行完善。臨床數據分析使用R語言實現后臺運算,基因分析通過R調用plink[4]進行分析并將結果展示在shiny應用中。
UK Biobank是一項由英國國家衛生局支持的前瞻性開放獲取隊列研究[5]。該項目招募了英國各地超過500000位參與者,采集參與者的表型與遺傳信息,包括生理指標、生活方式、生物學測量、血液尿液、身體及大腦圖像、全基因組測序等數據[6]。基于UK Biobank數據的研究已較為廣泛,包括對腎病[7]、甲狀腺疾病[8]等的臨床與基因數據研究。世界衛生組織發布的《2020世界癌癥報告》指出[9],乳腺癌是全球女性患病率最高的癌癥,同時也是全球女性癌癥死亡的主要原因。尋找乳腺癌易感及影響乳腺癌患者生存時長的臨床與基因因素是亟待解決的問題。因此,本研究選用UK Biobank乳腺癌數據作為shiny應用示例,展示本研究開發的臨床數據分析應用,同時探究影響乳腺癌患病及預后的風險因素。
針對臨床與基因數據特點,以R語言與shiny為基礎開發臨床數據分析應用,可交互式分析數據并展示結果,為不熟悉R語言與plink的使用者提供易上手的數據分析應用,為UK Biobank數據提供展示平臺,為臨床數據分析應用的開發提供新思路。
關鍵問題及解決方案
本應用包含四個功能:數據上傳與描述性統計、回歸分析、生存分析與基因分析。針對以下五個關鍵問題,我們探索出了相應解決方案。
1.如何架構UI能夠兼顧功能多樣與界面簡潔?
臨床與基因數據涉及多個分析方向,若顯示在同一頁面,難免會紛雜不清晰。比較多種shiny架構后,我們在navbarPage架構的基礎上,用navbarMenu與tabPanel進行擴充。navbarPage是shiny中較為常用的架構之一,點擊頁面頂部橫條的不同名稱顯示不同頁面。navbarMenu是navbarPage架構中的可選功能,點擊頂部橫條的名稱顯示下拉式菜單,點擊菜單中的名稱顯示不同頁面。tabPanel是shiny中常用的基礎架構,點擊shiny應用主頁面上方的不同標簽名稱顯示不同頁面。子功能較多且相互獨立的模塊通過navbarMenu展示,子功能具有一定關聯性的模塊通過tabPanel展示。同時,使用HTML與JavaScript語句對UI進行完善,例如通過HTML調整字體大小及添加使用說明,通過JavaScript控制輸出圖片的顯示與隱藏等功能。
2.如何處理臨床數據的變量類型?
R語言讀取csv、Excel、txt格式文件時會將變量讀取為連續型,或通過stringsAsFactors選項將所有變量讀取為分類型,這顯然不符合臨床數據特征。針對這一問題,首先將數據讀取為連續型,利用reactive函數與observe函數,在選擇框中顯示數據集變量名,使用者通過點擊選擇可將相應變量修改為分類型。
3.回歸模型類型如何判斷?
根據結局變量的不同類型,常見的回歸模型包括線性回歸模型(結局變量為連續型)和logistic回歸模型(結局變量為分類型)。為簡化操作,直接根據使用者選擇的結局變量類型判斷回歸模型類型。若結局變量為連續型,glm函數的family參數設置為Gaussian;若結局變量為分類型,family參數設置為Binomial。
4.連續型變量如何進行log rank檢驗?
生存分析模塊的log rank檢驗可以比較不同亞組生存分布。分類型變量可以直接根據變量水平區分亞組,但對于連續型變量,需要先規定其分組依據。為方便使用者,我們選用滑動條作為依托:當檢驗變量為連續型時,應用會顯示一條閾值與該變量取值范圍相等的滑動條,使用者拖動滑塊,以滑塊的取值對該連續型變量進行分組,并對兩組的生存分布進行log rank檢驗。
5.數據量較大的基因數據如何分析?
一些臨床研究包含基因數據,但基因數據量較大,如果在shiny中直接運算,上傳數據會占用較長時間。因此,考慮使用shiny調用plink軟件在本地運算,在shiny中展示本地儲存的運算結果與日志。首先在后臺通過bigsnpr::download_plink()語句在本地目標路徑中安裝plink軟件,后臺通過system(“plink-xxxx”)等語句調用plink進行文件格式轉換、主成分分析、GWAS等運算操作。
shiny應用界面與功能介紹
本應用包含五個模塊:應用介紹、數據上傳及描述、回歸分析、生存分析與基因分析,涵蓋了臨床研究數據分析的主要步驟。
“數據上傳及描述”模塊使用tabPanel顯示可選功能,通過點擊主頁面上方的標簽選擇數據上傳、圖像描述及描述性統計。在數據上傳頁面可以選擇內置的UK Biobank數據,也可上傳格式為csv、Excel、txt的文件并設置變量類型。可通過三種方式對數據進行簡要描述:前十行預覽、數據結構、匯總描述(分別對應R語言head、str、summary函數)。還可以繪制變量的描述性圖像:連續型變量繪制條形圖,分類型變量繪制餅圖。描述性統計頁面展示tableone包輸出的描述性表格,可以描述總體數據,也可分亞組進行對比描述。數據上傳及描述頁面見圖1。
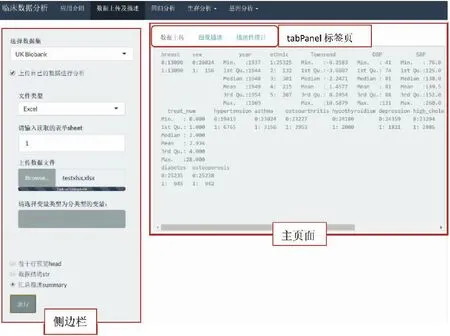
圖1 shiny應用的數據上傳及描述頁面
“回歸分析”模塊根據選擇的結局變量類型擬合線性回歸或logistic回歸模型,可以選擇數據集中的任意變量作為模型自變量。若模型自變量較多,可通過添加懲罰項進行變量選擇,可選的懲罰方式包括SCAD、MCP和Lasso三種。回歸分析模塊頁面展示見圖2。

圖2 shiny應用的回歸分析頁面
“生存分析”模塊包括Kaplan-Meier圖像、生存模型、log rank檢驗三部分。Kaplan-Meier圖像通過ggplot2包繪制,需要依次選擇生存時間與狀態。生存模型包括Cox比例風險模型與可加風險模型兩種,都可進行模型擬合與變量選擇,并在主頁面顯示模型的參數估計與統計量。圖3展示了navbarMenu與Cox比例風險模型頁面。log rank檢驗可根據使用者選擇的變量進行分組并比較亞組間的生存分布,連續型變量會根據滑動條的取值進行分組。圖4展示了log rank檢驗變量為連續型時的頁面。
“基因分析”模塊相對獨立于其他三個模塊,主要功能包括數據類型轉換、主成分分析、單因素及多因素GWAS分析三個功能。數據轉化是將常見的ped/map或vcf格式文件轉化為運算速度更快的二進制bed文件。主成分分析的功能是通過plink計算基因數據的十個主成分,讀取計算結果繪制主成分圖并展示在主頁面。單因素GWAS分析直接計算表型與基因數據關聯程度的P值;多因素GWAS分析計算有臨床數據影響時表型與基因數據關聯程度的P值。若基因數據較多,可勾選“計算矯正相關系數”以避免多重檢驗誤差。圖5展示基因GWAS分析頁面。
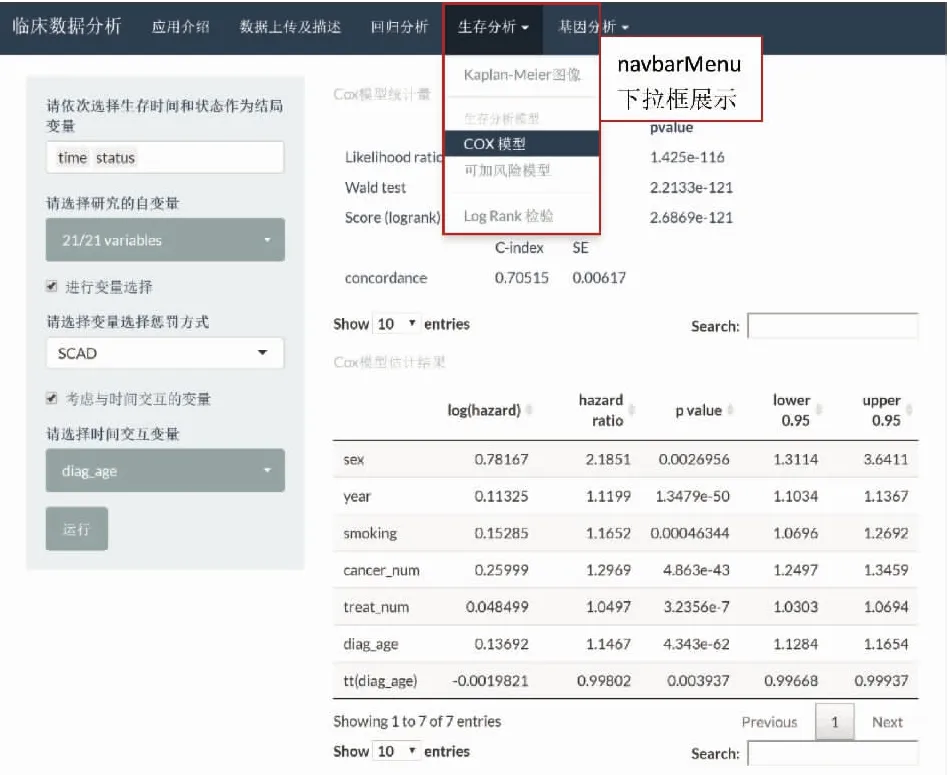
圖3 shiny應用的生存分析頁面
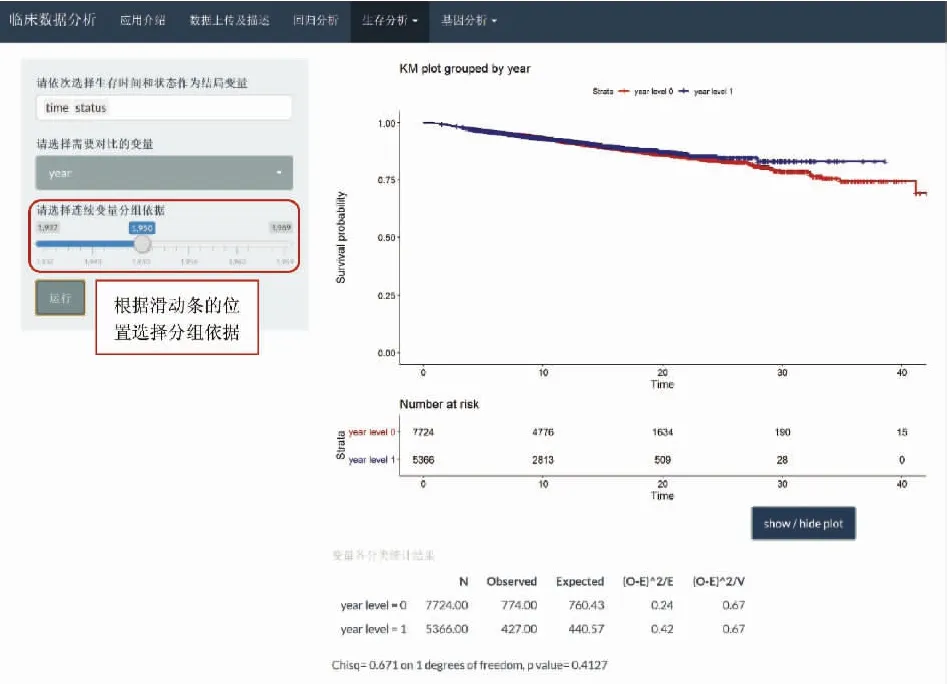
圖4 shiny應用的log rank檢驗頁面

圖5 shiny應用的GWAS分析頁面
部分應用示例結果展示
1.生存分析:Cox比例風險模型
點擊頁面上方“生存分析”按鈕進入Cox比例風險模型頁面。選擇生存時間和狀態后,將剩余變量選為模型自變量,勾選“變量選擇”選項,選擇SCAD懲罰,得到分析擬合結果見表1。性別、吸煙、確診年齡等對乳腺癌患者生存的影響與臨床研究一致[10-12]。
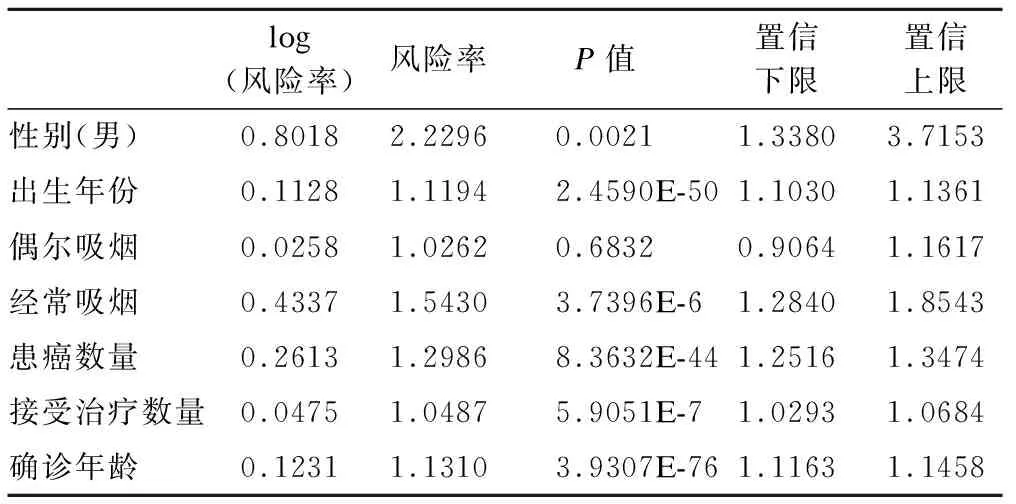
表1 生存分析Cox比例風險模型分析結果
2.基因分析
(1)主成分分析
點擊基因分析按鈕進入主成分分析頁面,粘貼基因數據所在文件路徑,計算主成分。之后根據計算結果繪制第一和第二主成分圖,如圖6,不同顏色代表不同人種。可以看出相同人種之間存在一定相關性,不同人種之間差異較顯著。
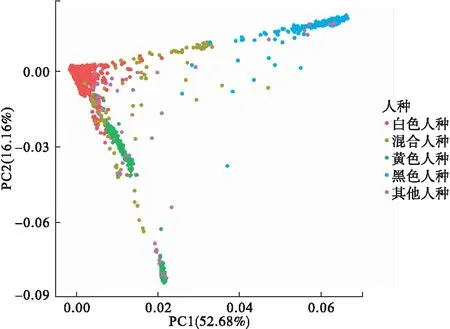
圖6 第一主成分與第二主成分示意圖
(2)單因素GWAS分析
點擊進入GWAS分析,選擇需要分析的文件名并勾選“計算矯正相關系數”選項,選取Bonferroni矯正。得到39個顯著位點,其中23個有研究支持,包括保護性和危害性SNP。例如rs3803662,一項關于乳腺癌患者的隊列研究表明其突變與乳腺癌易感風險增高顯著相關(OR=1.15)[13],這與我們的GWAS分析結果一致(OR=1.20)。具體結果見表2,其中A1為次等位基因,A2為參考基因。
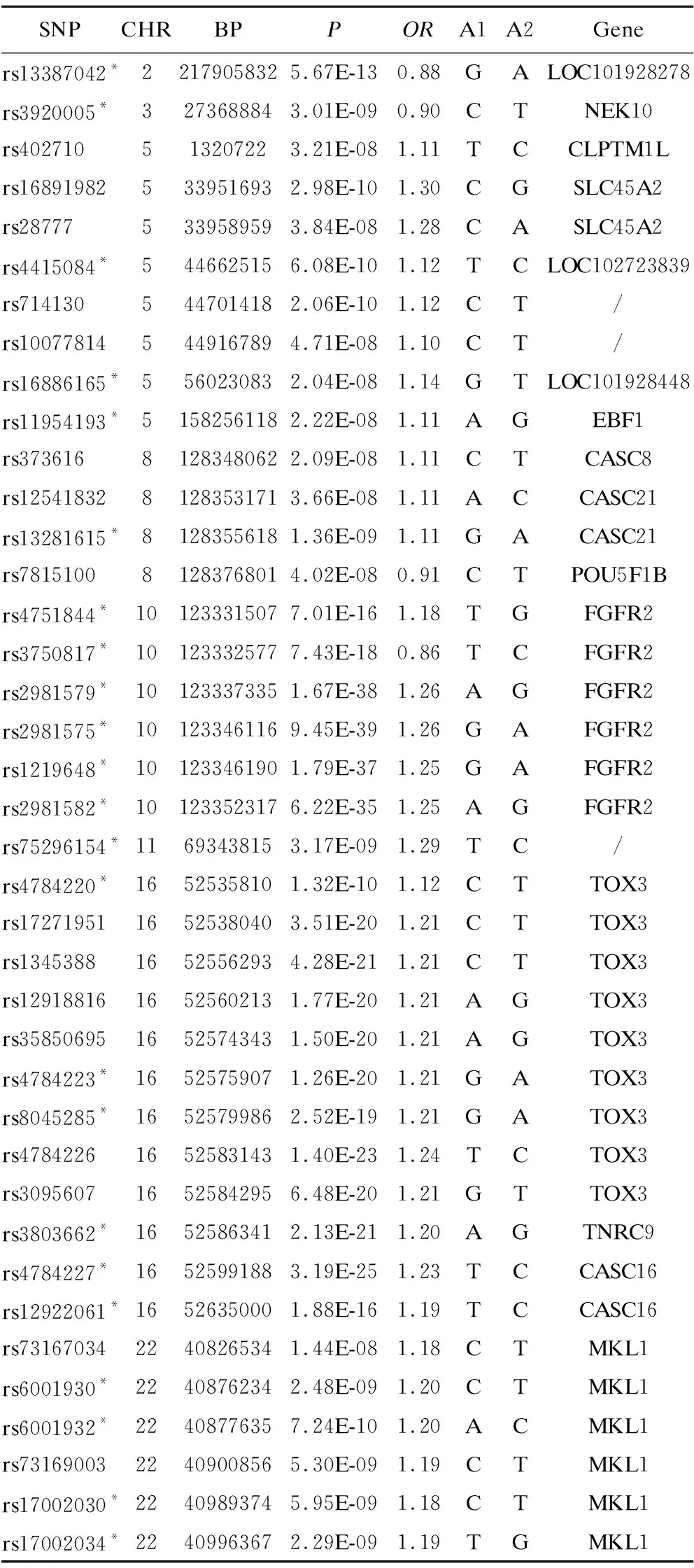
表2 GWAS分析影響罹患乳腺癌的SNP位點
討 論
將臨床數據分析與R-shiny結合,通過鼠標點擊的方式選擇感興趣的分析方法及模型,并交互式地展示相應分析結果,方便不熟悉R語言的使用者輕松使用R語言的分析統計、圖形繪制功能。本研究開發的應用能夠完成常見的分析方法,如描述性統計、回歸分析、生存分析、基因分析等;可以繪制常用的圖形,如餅圖、直方圖、Kaplan-Meier圖像等。在后續的研究中會對本應用繼續改進,完善統計繪圖功能,增強應用頁面使用的友好性、便捷性、交互性。期望本應用能夠協助不熟悉R語言的醫療工作人員進行數據分析,為臨床數據分析相關應用的開發提供新思路,同時為UK Biobank數據提供新的可視化分析統計平臺。

