小麥肉桂醇脫氫酶基因(TaCAD1)全長cDNA序列及生物信息學分析
盧雯瑩 葛昌斌 曹燕燕 黃杰 喬冀良 張振永 王君 齊雙麗 廖平安









摘 要:小麥(Triticum aestivum)是世界三大主糧之一,倒伏嚴重制約了小麥產量的提高。小麥肉桂醇脫氫酶基因(TaCAD1)是小麥木質素合成的關鍵基因,小麥體內木質素含量越高,莖稈強度越高,抗倒伏能力隨之增強。為了探明小麥肉桂醇脫氫酶基因的生物學功能,對TaCAD1基因編碼的氨基酸序列進行生物信息學分析。結果表明:TaCAD1基因cDNA序列全長1311bp,包含1個長度為1083bp的開放閱讀框(ORF),編碼360個氨基酸。TaCAD1蛋白等電點(pI)為5.74,分子量為38625.45 Da,其不穩定系數為23.91,屬于穩定蛋白質。推導的TaCAD1蛋白為疏水性蛋白,不存在跨膜區,可能位于細胞質內,該蛋白的二級結構元件主要為無規卷曲。TaCAD1蛋白在進化過程中較為保守,與節節麥、大麥、高羊茅、黑麥草、短花藥野生稻CAD蛋白的一致性達94.81%。研究結果為進一步探究TaCAD1在小麥抗倒伏過程中的作用及小麥抗倒伏的分子育種提供參考。
關鍵詞:小麥;肉桂醇脫氫酶;木質素;抗倒伏;生物信息學分析
中圖分類號 S512 文獻標識碼 A 文章編號 1007-7731(2022)11-0016-05
Full-length cDNA Sequence and Bioinformatics Analysis of Cinnamyl Alcohol Delivering Enzyme Gene (TaCAD1) in Triticum aestivum
LU Wenying? ?GE Changbin? ?CAO Yanyan? ?HUANG Jie? ?QIAO Jiliang? ?ZHANG Zhenyong? ?WANG Jun
QI Shuangli? ?LIAO Pingan
(Luohe Academy of Agricultural Science, Luohe 462300, China)
Abstract: Wheat(Triticum aestivum) is one of the world's three major staple foods, and wheat lodging has severely restricted the increase in wheat production. The wheat cinnamyl alcohol dehydrogenase gene (TaCAD1) is a key gene for wheat lignin synthesis. The higher the lignin content in wheat, the higher the stalk strength and the enhanced lodging resistance. In order to ascertain the biological function of wheat cinnamyl alcohol dehydrogenase gene, this paper conducts bioinformatics analysis on the amino acid sequence encoded by TaCAD1 gene. Bioinformatics analysis showed that the full-length cDNA sequence of TaCAD1 was 1311 bp, including an open reading frame (ORF) of 1083 bp in length, encoding 360 amino acids. The isoelectric point (pI) of TaCAD1 protein is 5.74, the molecular weight is 38,625.45 Da, and its instability coefficient is 23.91, which is a stable protein. The deduced TaCAD1 protein is a hydrophobic protein, there is no transmembrane region, and may be located in the cytoplasm. The secondary structure of the protein is mainly random coils. The TaCAD1 protein is relatively conservative in the evolutionary process, and it is 94.81% consistent with the CAD protein of Jiejie wheat, barley, tall fescue, ryegrass, and short anther wild rice. This study provides new ideas for further exploring the role of TaCAD1 in the process of wheat lodging resistance and molecular breeding of wheat lodging resistance.
Key words: Wheat; Cinnamyl alcohol dehydrogenase; Lignin; Lodging resistance; Bioinformatics Analysis
小麥作為三大主要糧食作物之一,在世界廣泛種植,其播種面積和產量在各類糧食作物中均居首位,目前世界1/3的人口是以小麥為主食。我國是重要的小麥生產大國之一,小麥產量、消費量居于世界前列。近年來,我國小麥生產發展迅速,但倒伏與高產間的矛盾日益突出。小麥發生倒伏的主要位置是莖稈[1],嚴重倒伏的小麥種植區域可能導致產量減少80%左右[2]。
木質素是一類復雜的酚類聚合體,在植物體內所占比重較高,是構成植物次生細胞壁的重要成分之一。木質素能夠通過自身的交織作用加厚細胞壁,進而加強植物莖稈強度,起到抗壓作用[3]。木質素在小麥體內含量的多少與小麥莖稈的抗倒伏特性存在一定關系[4]。Welton的研究表明,若減弱小麥莖稈的機械強度,小麥倒伏容易發生,且小麥體中木質素含量隨之降低[5];若小麥莖稈機械強度增加,小麥抗倒伏能力增強,小麥體中木質素含量相應升高[6]。
木質素的沉積主要發生在細胞生長完成、次生壁加厚的特定時期[7]。其在植物體內的生物合成過程復雜[8],大致分為三部分:莽草酸途徑、苯丙烷途徑和木質素的特異合成途徑[9]。肉桂醇脫氨酶(Cinnamyl alcohol dehydrogenase,CAD)催化肉桂醛還原成相應的肉桂醇,是木質素合成途徑最后一步的關鍵酶[10]。Ma[11]從小麥中分離出1個可以編碼CAD基因的cDNA——TaCAD1,TaCAD2和TaCAD4可能不具有酶活性[12]。TaCAD1基因在莖稈中高表達,葉片中表達量次之,根中幾乎不表達;抗倒伏小麥品種mRNA的表達豐度、酶活性均高于不抗倒伏的小麥品種[11]。
1 材料和方法
1.1 小麥肉桂醇脫氫酶基因的cDNA序列搜索 搜索GenBank中注冊的小麥CAD基因(TaCAD1)的全長cDNA序列(GU563724),將其開放閱讀框翻譯成相應的氨基酸序列用于后續生物信息學分析。
1.2 TaCAD1蛋白的生物信息學分析 使用ExPASy服務器中ProtParam在線軟件(http://web.expasy.org/protparam/)得到小麥肉桂醇脫氫酶TaCAD1的等電點、不穩定系數、平均疏水性和分子量等理化性質;ProtScale在線軟件(http://web.expasy.org/protscale/)預測氨基酸序列親/疏水性預測圖;通過SignalP-5.0軟件(http://www.cbs.dtu.dk/services/SignalP-5.0/)對小麥肉桂醇脫氫酶進行相關信號肽的預測與分析;應用在線軟件工具PSORT II Prediction(https://psort.hgc.jp/form2.html)預測亞細胞定位;用ExPASy服務器中TMHMM Server v. 2.0在線工具(http://www.cbs.dtu.dk/services/TMHMM-2.0/)獲得小麥肉桂醇脫氫酶蛋白的跨膜結構域;使用NPS軟件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)預測蛋白質二級結構。利用DNAMAN 8.0軟件,對TaCAD1蛋白的氨基酸序列與其他同源物種進行同源性比對分析,在MEGA 6.0軟件的支持下,構建TaCAD1與不同植物CAD蛋白序列的系統進化樹。運用SWISS-MODEL(http://swissmodel.expasy.org/)在線軟件工具,對TaCAD1蛋白進行三級結構建模。在NCBI官方網站(http://www.ncbi.nlm.nih.gov/)中能夠預測保守結構域的數據庫(Conserved Domain Database,CDD)的幫助下,分析TaCAD1蛋白序列是否具有典型的CAD基因家族蛋白的保守結構域。
2 結果與分析
2.1 小麥TaCAD1基因cDNA序列的ORF 小麥TaCAD1基因的cDNA序列全長1311 bp,包含1個長度為1083bp的開放閱讀框(ORF),可編碼360個氨基酸(圖1)。
2.2 TaCAD1蛋白一級結構及理化性質 用ExPASy服務器中ProtParam得到TaCAD1蛋白序列的物理化學性質,見表1。表1顯示,小麥肉桂醇脫氫酶編碼360個氨基酸,分子量為38625.45 Da;等電點為5.74,判斷為堿性蛋白;不穩定系數Ⅱ為23.91,小于40,分類為穩定蛋白。該蛋白質中共包含20種氨基酸,含量最高的為纈氨酸(Val),所占比例為13.1%。
用ExPASy服務器中ProtScale在線軟件獲得氨基酸序列親/疏水性預測圖(圖5),結果表明TaCAD1蛋白的親水性平均系數(GRAVY)為0.011,多肽鏈中具有最低分值(-2.211)的是位于第216位的賴氨酸(K),親水性最強;具有最高分值(2.700)的是位于第95位的甘氨酸(G),疏水性最強。疏水氨基酸(正值)稍多于親水氨基酸(負值),推測小麥肉桂醇脫氫酶基因編碼氨基酸序列對應的蛋白質為疏水性蛋白。
2.3 TaCAD1蛋白亞細胞定位 使用應用在線軟件工具PSORT II Prediction預測TaCAD1蛋白亞細胞定位的結果(表2)顯示:該蛋白質定位于細胞質的概率為0565,位于細胞核的概率為0.130,位于線粒體基質的概率為0.130。因此,推斷該蛋白存在于細胞質的可能性最大。
2.4 TaCAD1蛋白的信號肽和跨膜結構域分析 應用SignalP-5.0軟件預測小麥肉桂醇脫氫酶基因所編碼的蛋白質的信號肽,結果如圖3,該蛋白信號肽預測值為0.037,表明TaCAD1蛋白不存在信號肽區域,推測其可能為非分泌型蛋白。
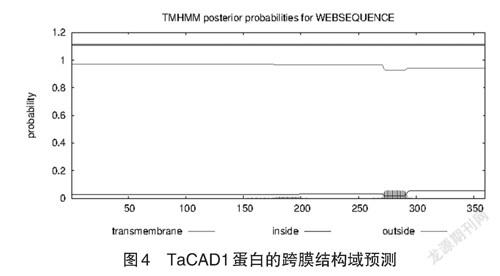
TMHMM Serverv. 2.0在線工具輸入氨基酸序列預測小麥肉桂醇脫氫酶蛋白的跨膜結構域的結果顯示(圖4),跨膜結構域預測圖無峰值,證明TaCAD1蛋白不存在跨膜結構域,可能為非跨膜蛋白,且主要位于膜外區域。綜合上述信號肽分析和跨膜結構域分析,推測該蛋白既不屬于分泌蛋白,也不能認為是膜蛋白。該結論與亞細胞定位分析所顯示的TaCAD1蛋白可能位于細胞質中的預測結果相吻合。
2.5 TaCAD1蛋白的二級結構預測 使用在線軟件NPS預測蛋白質的二級結構(表3,圖5),結果顯示TaCAD1主要由α-螺旋(Alpha helix)、延伸鏈(Extended strand)、無規卷曲(Random coil)3種結構組成,其中無規卷曲是TaCAD1蛋白二級結構最主要的結構元件,占53.61%;α-螺旋和延伸鏈分別占23.33%和23.06%,無β-折疊。
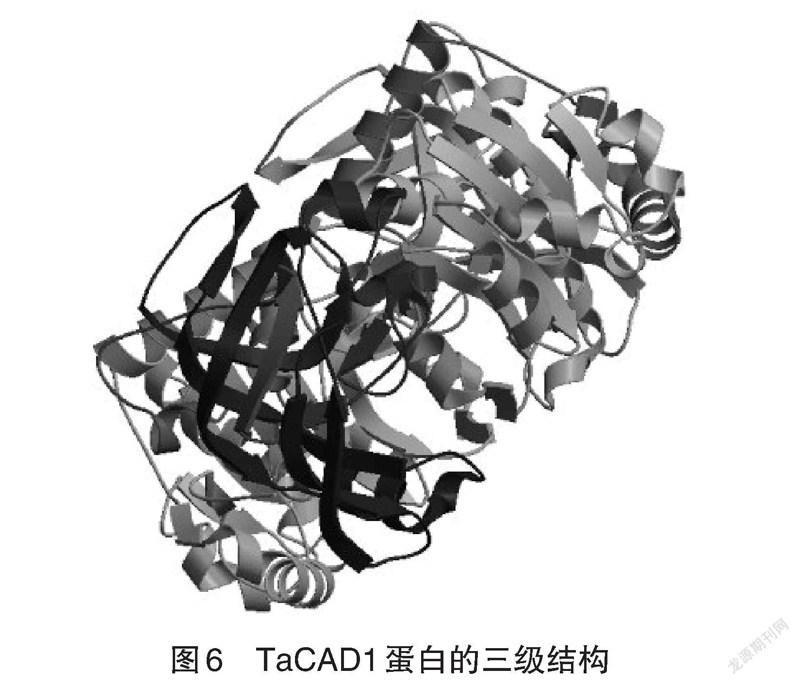
2.6 TaCAD1蛋白三維結構預測 使用SWISS-MODEL在線軟件工具對TaCAD1蛋白進行三級結構建模,在RasTop軟件下對以上模型做相應處理,構建蛋白質三維結構圖如圖6。由三維結構圖不難發現,TaCAD1蛋白主要由無規卷曲構成,這與前面對TaCAD1蛋白二級結構所預測的結果一致。
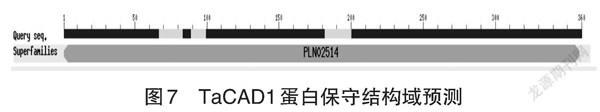
2.7 TaCAD1蛋白的保守結構域和功能預測 對TaCAD1蛋白進行Protein Blast分析以判斷其可能存在的保守結構域,結果見圖7。TaCAD1蛋白屬PLN02514超基因家族,含有PLN02514保守結構域。
2.8 TaCAD1蛋白同源性分析與系統進化樹構建 應用DNAMAN 8.0軟件,對TaCAD1與節節麥、大麥、高羊茅、黑麥草、短花藥野生稻CAD的蛋白序列進行同源性分析,結果(圖8)顯示,TaCAD1蛋白與節節麥、大麥、高羊茅、黑麥草、短花藥野生稻的CAD氨基酸序列一致性高達94.81%。
利用Blastp同源搜索,構建了TaCAD1與不同植物CAD蛋白序列的系統進化樹,TaCAD1與高羊茅、黑麥草CAD編碼蛋白的同源性最高(圖9)。
3 結論與討論
肉桂醇脫氫酶是小麥莖稈木質素生物合成途徑最后一步反應的關鍵酶之一。本研究通過NCBI搜索得到1個小麥肉桂醇脫氫酶基因(TaCAD1)cDNA序列,該序列全長1311 bp,包含1個長度為1083bp的開放閱讀框,編碼氨基酸個數為360個。木質素是一種通過β-O-4、β-β、β-5、5-5等共價鍵連接形成的網狀結構高分子聚合物,甲氧基是木質素結構中特征官能團之一,因此其理化性質比較穩定。小麥TaCAD1推導蛋白的不穩定系數為23.91,是穩定蛋白,符合木質素理化性質穩定這一特性。TaCAD1蛋白分子量為38625.45 Da,等電點(pI)為5.74。此外,TaCAD1蛋白在進化過程中較為保守,與節節麥、大麥、高羊茅、黑麥草、短花藥野生稻CAD蛋白的一致性達94.81%。系統進化樹結果表明,TaCAD1蛋白與高羊茅和黑麥草CAD蛋白同源性最高。本研究獲得小麥肉桂醇脫氫酶(TaCAD1)全長cDNA序列及蛋白功能預測結果,將為下一步驗證該基因功能奠定了理論基礎,也為進一步深入研究TaCAD1基因在小麥抗倒伏育種中的作用提供重要參考。
參考文獻
[1]Goicoechea M,Lacombe E,Legay S,et al. EgMYB2,a new transcriptional activator from Eucalyptus xylem,regulates secondary cell wall formation and lignin biosynthesis[J]. Plant Journal,2005,43(4):553-567.
[2]李崇德,許華.小麥穩產高產限制因素與主要技術措施[A].專題論壇,2010,9:50-52.
[3]Kim Y H,Bae J M,Huh G H. Transcriptional regulation of the cinnamyl alcohol dehydrogenase gene from sweetpotato in response to plant developmental stage and environmental stress[J]. Plant Cell Reports,2010,29(7):779-791.
[4]Tronchet M,C Balagué,Kroj T ,et al. Cinnamyl alcohol dehydrogenases-C and D,key enzymes in lignin biosynthesis,play an essential role in disease resistance in Arabidopsis[J]. Molecular Plant Pathology,2010,11(1):83-92.
[5]Welton F A. Lodging in oats and wheat. Bot Gaz,1928,85:121-151.
[6]Tripathi S C,Sayre K D,Kaul J N,et al. Growth and morphology of spring wheat (Triticum aestivum L.) culms and their association with lodging:effects of genotypes,N levels and ethephon[J]. Field Crops Research,2003,84(3):271-290.
[7]Donaldson L A. Lignification and lignin topochemistry - an ultrastructural view.[J]. Phytochemistry,2001,57(6):859-873.
[8]陳建中,葛水蓮,葉嘉,等.木質素生物合成的研究進展[J].邯鄲學院學報,2008,18(3):4.
[9]高原,陳信波,張志揚.木質素生物合成途徑及其基因調控的研究進展[J].生物技術通報,2007(2):5.
[10]Tobias C M,Chow E K.Structure of the cinnamyl-alcohol dehydrogenase gene family in rice and promoter activity of a member associated with lignification[J]. Planta,2005,220(5):678-688.
[11]Ma Q H. Functional analysis of a cinnamyl alcohol dehydrogenase involved in lignin biosynthesis in wheat[J]. Journal of Experimental Botany,2010,61(10):2735-2744.
[12]耿丹.小麥肉桂醇脫氫酶基因克隆和序列分析[D].合肥:安徽農業大學,2014.
基金項目:國家小麥產業技術體系漯河綜合試驗站建設項目(CARS-3-2-26)。
作者簡介:盧雯瑩(1994—),女,河南漯河人,研究實習員,研究方向:小麥遺傳育種。
通訊作者:廖平安(1964—),男,湖北鄂州人,研究員,研究方向:小麥育種。? 收稿日期:2021-12-14

