TLR5基因單核苷酸多態性與幽門螺桿菌感染、非賁門胃癌發病風險的關聯
高芳 衛星如 馬立聰 田旭陽 黨彤 賈彥彬,
(1包頭醫學院,內蒙古 包頭 014060;2包頭醫學院第二附屬醫院內蒙古消化病研究所)
幽門螺桿菌(H.pylori)是定植于胃黏膜、含有鞭毛蛋白特殊結構的需氧革蘭陰性菌。世界衛生組織將H.pylori確定為胃的Ⅰ類致癌原〔1〕。Toll樣受體(TLR)5是一種可識別細菌鞭毛蛋白〔2,3〕的模式識別受體,表達于胃上皮細胞膜的表面〔4〕。H.pylori感染機體后可引起慢性萎縮性胃炎、消化性潰瘍、胃癌等一系列胃腸道疾病〔5〕。而H.pylori感染人體后,宿主胃黏膜上皮中TLR5的表達水平上調〔6,7〕。研究證實,TLRs基因多態性在一定程度上可以影響宿主對H.pylori感染及相關疾病的遺傳易感性〔8〕。此外,TLR5可能參與了H.pylori感染后各種胃部疾病的發生發展〔9〕,甚至引起胃癌的發生。在我們前期研究TLR5基因多態性與H.pylori的感染、非賁門胃癌的發病風險中發現,包頭地區漢族人群中TLR5基因單核苷酸多態性(SNP)rs17163737與H.pylori、非賁門胃癌的發病風險相關聯〔10,11〕。由于單一基因多態性在癌變的過程中起微效效應,TLR5基因其余的單核苷酸多態性是否也和胃癌變有關還需進一步探索,本研究探討TLR5基因SNP rs5744138、rs2241096、rs160827 rs20961-41、rs160812基因多態性與H.pylori感染、非賁門胃癌的關系。
1 材料與方法
1.1研究樣本 選取2008年9月至2010年6月間包頭市腫瘤醫院經組織病理學診斷為非賁門胃癌的患者288例為病例組,均未接受過放療或化療。再選取281例同期在包頭醫學院第一附屬醫院無癌癥病史和明顯的胃部疾病的體檢者為正常對照組。兩組年齡、性別差異無統計學意義(t=0.40,P=0.69;χ2=0.06,P=0.80)。研究對象均為漢族,相互之間無血緣關系且在包頭居住5年以上,簽署了知情同意書,并提供血液標本2 ml。本研究已獲得包頭醫學院倫理學委員會的批準。
1.2H.pylori感染的檢測 采用酶聯免疫吸附試驗(ELISA)血清學方法,在正常對照組血清中檢測人抗H.pylori的抗體滴度。試劑盒購于(BIOHIT公司,芬蘭),嚴格按照說明書進行操作。并根據試劑盒推薦:抗體滴度≥30 EIU,判斷H.pylori感染為陽性。
1.3SNP位點的篩選和基因分型 采用Agorithm-Tagger-pairwise Tagging軟件,根據HapMap數據庫(http://hapmap.ncbi.nlm.nih.gov/)提供的中國漢族遺傳多態性數據,篩選研究基因的標記單核苷酸多態性(Tag-SNP)〔11〕。篩選范圍包括基因第一個外顯子上游20 kb和最后一個外顯子下游10 kb的序列。篩選過程中使用的參數為連鎖不平衡值r2>0.8,最小等位基因頻率(MAF)>5%。最終本研究篩選出TLR5 rs5744138、rs2241096、rs160827 rs2096141、rs160812的5個位點,見表1。對5個SNP采用TaqMan法在所有樣本中進行基因分型,成功率均大于98%。

表1 TLR5基因的Tag-SNPs的概況
1)頻率低的等位基因在前,頻率高的等位基因在后;2)基于GRCh37.p10
1.4統計學分析 數據分析使用SPSS 16.0統計軟件進行。在正常對照組中用χ2檢驗檢測樣本中的基因型分布是否遵循Hardy-Weinberg平衡;使用Haploview4.0軟件對SNP位點進行連鎖不平衡分析,并采用D′置信區間法劃分單體型塊(LD block)。TLR5單體型塊包括rs34270714-rs5744174-rs851139-rs2241097-rs17163737(Block 1)、rs574413-8-rs2241096-rs1640827-rs2096141-rs160812(Block 2)之間存在強連鎖不平衡,分別構成單體型區域。在我們先前的實驗中,已經對Block 1進行了研究〔11〕。本實驗我們重點研究了Block 2。
用非條件性Logistic回歸計算比值比(OR)及其95%可信區間(CI),評價各等位基因、基因型及單體型與H.pylori感染、非賁門胃癌發病風險的關聯性〔10,11〕。
2 結 果
2.1各SNP位點與H.pylori感染的關系 SNP rs5744138、rs2241096、rs160827、rs2096141、rs160812的基因型分布在H.pylori感染陰性組和陽性組均符合Hardy Weinberg平衡定律(P>0.05)。H.pylori的陽性率為43.3%。5個SNP位點的等位基因、基因型在H.pylori感染陽性和陰性人群中的分布差異無統計學意義(P>0.05),見表2。

表2 各SNP位點等位基因、基因型與H.pylori感染的關聯
1)數據有缺失,2)平衡了性別和年齡因素;表3同
2.2各SNP位點與非賁門胃癌發病風險的關系 所有檢測的SNP的等位基因、基因型頻率在兩組中的分布差異無統計學意義(P>0.05),見表3。
2.3TLR5基因的單體型與H.pylori感染的關系 以H.pylori感染為結局變量,使用Haploview軟件對TLR5基因的不同位點進行連鎖不平衡分析,單體型塊的劃分采用D′置信區間法,發現rs5744138-rs2241096-rs1640827-rs2096141(Block)之間存在強連鎖不平衡,構成單體型區域。分析結果顯示,各單體型在H.pylori感染陽性和陰性人群中的分布差異無統計學意義(P>0.05),見表4。
2.4TLR5基因的單體型與非賁門胃癌發生的關系 以非賁門胃癌為結局變量,使用Haploview軟件對TLR5基因的不同位點進行連鎖不平衡分析,單體型塊的劃分采用D′置信區間法,發現rs5744138-rs2241096-rs1640827-rs2096141-rs1640812(Block)之間存在強連鎖不平衡,分別構成單體型區域。分析結果顯示,各單體型在兩組中的分布差異無統計學意義(P>0.05),見表5。
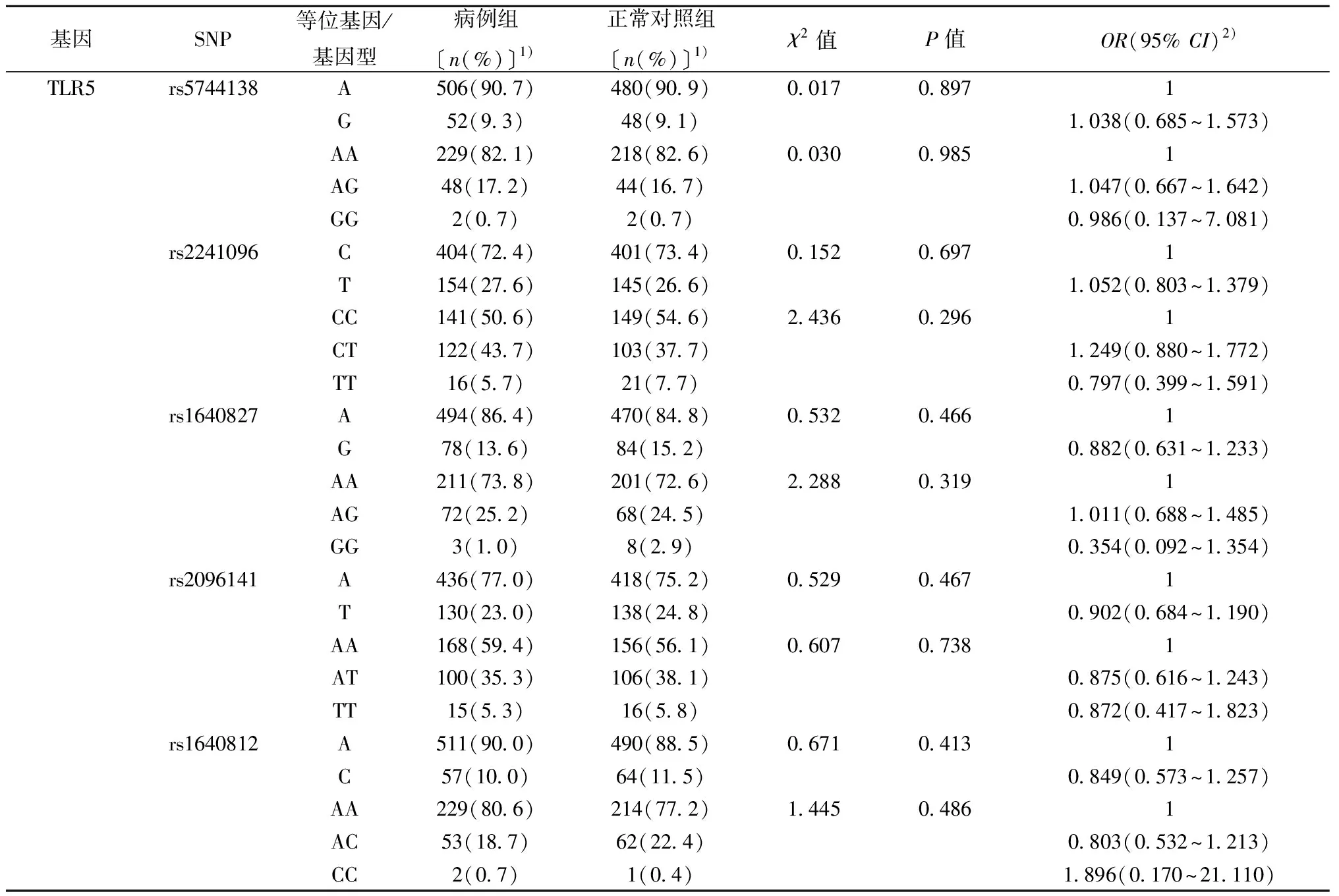
表3 各SNP位點等位基因、基因型與非賁門胃癌的關聯分析
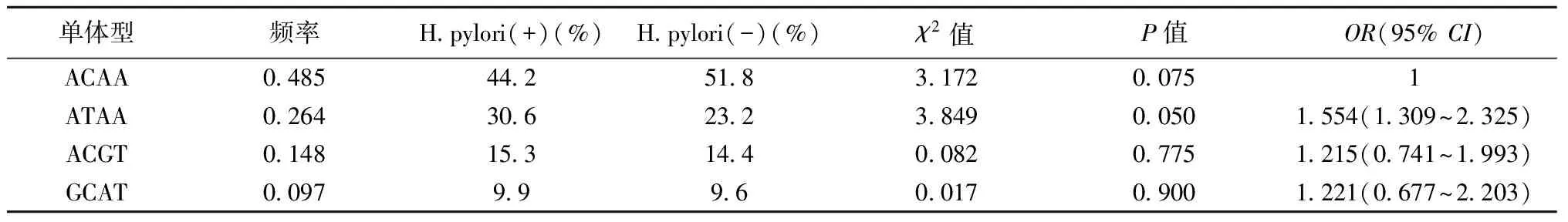
表4 TLR5基因單體型與H.pylor感染的關系
SNP順序為rs5744138-rs2241096-rs1640827-rs2096141

表5 TLR5基因單體型與非賁門胃癌的關系
SNP順序為rs5744138-rs2241096-rs1640827-rs2096141-rs1640812
3 討 論
胃癌是常見的惡性腫瘤之一,其發生發展過程受多種環境因素和遺傳因素的影響。H.pylori感染是引起胃黏膜病變的生物致病因子之一,可導致慢性胃炎尤其是慢性活動性胃炎的發生和進展,部分患者可進展為消化性潰瘍、萎縮性胃炎、胃癌等嚴重臨床結局〔12,13〕。
TLRs是一種模式識別受體,能夠特異性地識別細菌、病毒等多種病原微生物,進而激活機體產生非特異性免疫應答。TLRs的表達具有組織特異性,其中TLR2、TLR4、TLR5和TLR9在胃中有選擇性表達〔8〕。TLR5是TLRs中唯一可以識別細菌蛋白質的受體,其配體是細菌的鞭毛蛋白〔2,3〕。研究發現,在H.pylori感染者當中10%~20%的患者會發展成消化性潰瘍,發展成為胃癌的比例僅占1%~2%〔14〕,說明宿主的遺傳因素在胃癌的發生發展過程中發揮了重要作用。TLRs在H.pylori感染及其引起的相關疾病中起到了重要的作用,其編碼基因是研究H.pylori感染及其相關疾病分子遺傳學的重要候選基因〔15,16〕。
TLR5基因位于染色體1q41-q42,編碼TLR5蛋白質。Hayashi等〔17〕證實了先天免疫系統對H.pylori感染的識別主要是通過細菌鞭毛蛋白和TLR5之間的相互作用。Xu等〔16〕研究顯示,TLR5基因SNP rs1640827和rs17163737與H.pylori感染相關聯。衛星如等〔18〕研究表明,在共顯性遺傳模式下,TLR5基因SNP rs2072493與H.pylori的感染關聯,與AA基因型攜帶者相比,攜帶AG基因型者可使H.pylori感染的風險降低。而有研究表明〔10〕,TLR5 rs17163737與H.pylori感染關聯。H.pylori感染屬于多基因疾病,環境、遺傳因素都在疾病的發展中起一定的作用。單一SNP起微效效應。結果提示TLR5 rs5744138、rs2241096、rs160827 rs2096141、rs160812與H.pylori感染沒有關聯。目前,關于這5個SNP與H.pylori感染、胃癌的發病風險的研究報道不多,還需進一步研究證實。
本研究還探討了TLR5基因多態性與非賁門胃癌發病風險的關系。研究結果顯示,SNP rs5744138、rs2241096、rs160827 rs2096141、rs160812位點的等位基因、基因型頻率在非賁門胃癌病人和正常對照中的分布差異無統計學意義。Xu等〔16〕研究顯示,TLR5基因SNP rs5744174、rs1640827和rs17163737與GC發病風險上升。同時TLR5基因多態性可能增加萎縮性胃炎和胃癌的發病風險〔19〕。而有研究表明〔11〕,TLR5基因SNP rs17163737與非賁門胃癌的發病風險相關聯,與攜帶GG基因型的人群比較,GT基因型攜帶者非賁門胃癌的發病風險可提高1.605倍(GT vs GG:OR=1.605,95%CI=1.131~2.280),SNP rs34270714、rs5744174、rs851139與非賁門胃癌發病風險沒有關聯(P>0.05)。單個SNP在胃癌的發病過程作用是微小的。今后應進一步擴大樣本量,研究在胃癌變過程中,基因多態性的聯合效應。

