17 個常染色體STR 基因座在基諾族人群的遺傳多態性研究
趙 凱,張韻梅,王偉濤,李光興
(云南鑒識司法鑒定中心,云南 芒市 678400)
自1987 年首次用于刑事案件以來,對生物證據DNA 的分析徹底改變了法醫調查技術。在這三十年中,DNA 分析方法的辨別能力、速度和靈敏度以及分型越來越具有挑戰性的樣品的能力取得了重大進展。通過建立罪犯和犯罪現場概況以及群體等位基因頻率數據庫,可以從犯罪現場樣本中識別嫌疑人,并制定評估DNA 證據的統計框架。短串聯重復序列(STR)作為目前人類身份個體識別最高效的DNA 分子標記,廣泛地應用于法庭科學技術中,因STR 等位基因座在不同群體中存在比較明顯頻率分布差異,對不同民族人群中進行頻率調查,建立基礎數據庫,有很重要的意義。
基諾族古稱“攸樂”,在1979 年被確認為我國第56 個民族[1],是云南省特有的少數民族,主要聚居于云南省西雙版納傣族自治州景洪市基諾山基諾族民族鄉,根據2010 年全國第六次人口普查,基諾族總人口為2.3 萬余人。基諾族日常生活中使用基諾語,屬漢藏語系藏緬語族彝語支,基諾族沒有文字,有與其他彝語支民族相似的創世神話及葫蘆圖騰信仰。在漢文獻中,直到清朝初期才有關于“攸樂”人的零星記載。
此調查為云南省基諾族人群的法醫學個人識別和親子鑒定以及群體遺傳學研究提供基礎數據。
1 材料與方法
1.1 樣本
根據知情同意原則,收集云南省西雙版納傣族自治州景洪市基諾山基諾族民族鄉331 例基諾族無關個體的頰粘膜拭子樣本。
1.2 DNA 提取、擴增及檢測
使用chelex-100 螯合樹脂(伯樂生命醫學產品(上海)有限公司)熱裂解法提取拭子樣本DNA,使用AGCU 17+1 試劑盒(無錫中德美聯生物技術有限公司),擴增體系配制操同產品說明書,總反應體系 10 μL,在9700 型PCR 儀(美國Thermo Fisher Scientific 公司)上進行直接復合擴增,95 ℃2 min,94 ℃ 15 s、61 ℃ 40 s、65 ℃ 50 s、30 個循環,72 ℃ 10 min。取 1 μL 擴增產物與 0.5 μL 內標 LIZ500 和 9.5 μL 去離子甲酰胺混合,3130 基因分析儀(美國Thermo Fisher Scientific 公司)上進行全自動毛細管電泳,用 GeneMapper?ID-X 軟件進行等位基因分型。選擇F312(蘇州閱微基因技術有限公司)為陽性質控品,純化水為陰性質控品。
1.3 統計學處理
應用 Powerstates 軟件[2]對STR 分型數據進行處理,計算獲得17 個 STR 基因座的等位基因頻率和法醫學常用參數。使用Arlequin 3.5 軟件[3]進行基因座間連鎖不平衡檢驗(哈溫平衡檢驗)。應用dispan 軟件計算基諾族與其他10 個群體[4-13],利用MEGA 軟件[14]基于DA distances構建鄰接法進化樹。
2 結果
2.1 等位基因頻率分布及群體遺傳學參數
本研究在331 名基諾族無關個體共觀察到186 個等位基因和615 種基因型,等位基因頻率分布在0.001 5~0.561 9(表1)。經過Bonferroni 矯正,基因型分布均符合哈溫平衡(P> 0.002 9)。基諾族人群Ho 值分布在0.613 29~0.876 13,Penta E 和FGA 為高多態性基因座(PIC > 0.85),DP 值在0.770 5~0.968 9,累積個體識別率(CDP)達到0.999 999 999 999 999 999 999 9,PE 值 在0.307 1~0.747 0,三聯體累積非父排除率(CPE)達到0.999 999 932,見表2。
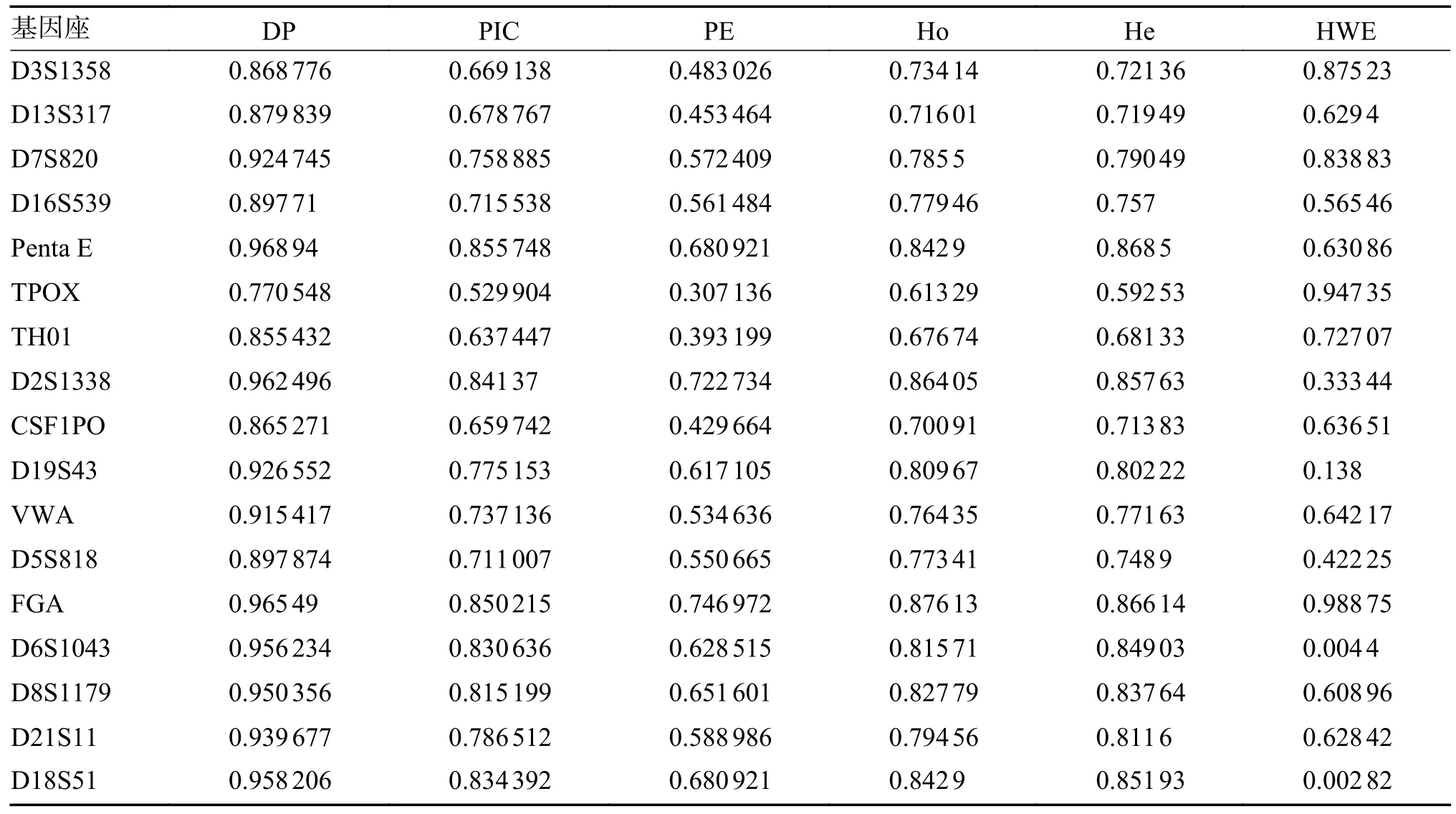
表2 基諾族人群17 個 STR 基因座法醫學參數(n=331)Tab.2 Forensic parameters of 17 STR loci in the Jino people(n=331)
2.2 群體遺傳結構分析
基諾族人群與白族、傣族、納西族等10 個人群常染色體STR 基因座鄰接法(Neighbor-Joining Algorithm)進化樹結果見圖1,遺傳距離參數見表3,以云南南北不同地勢、海拔為界分為兩組,基諾族與聚居在南邊的哈尼族和傣族聚類。
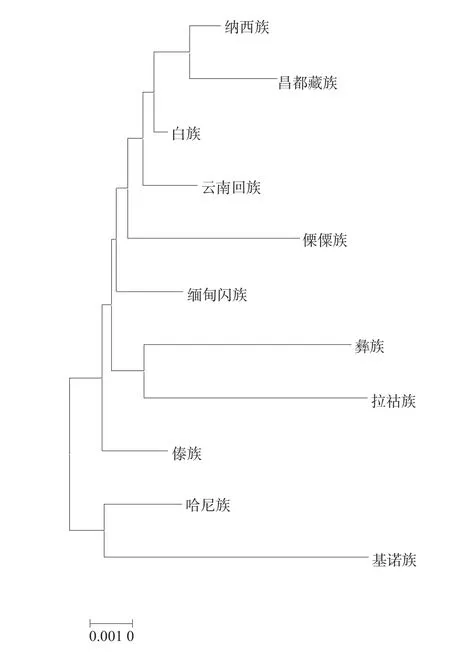
圖1 基于DA distances 構建鄰接法進化樹。Fig.1 Neighbor-Joining tree based on DA distances.
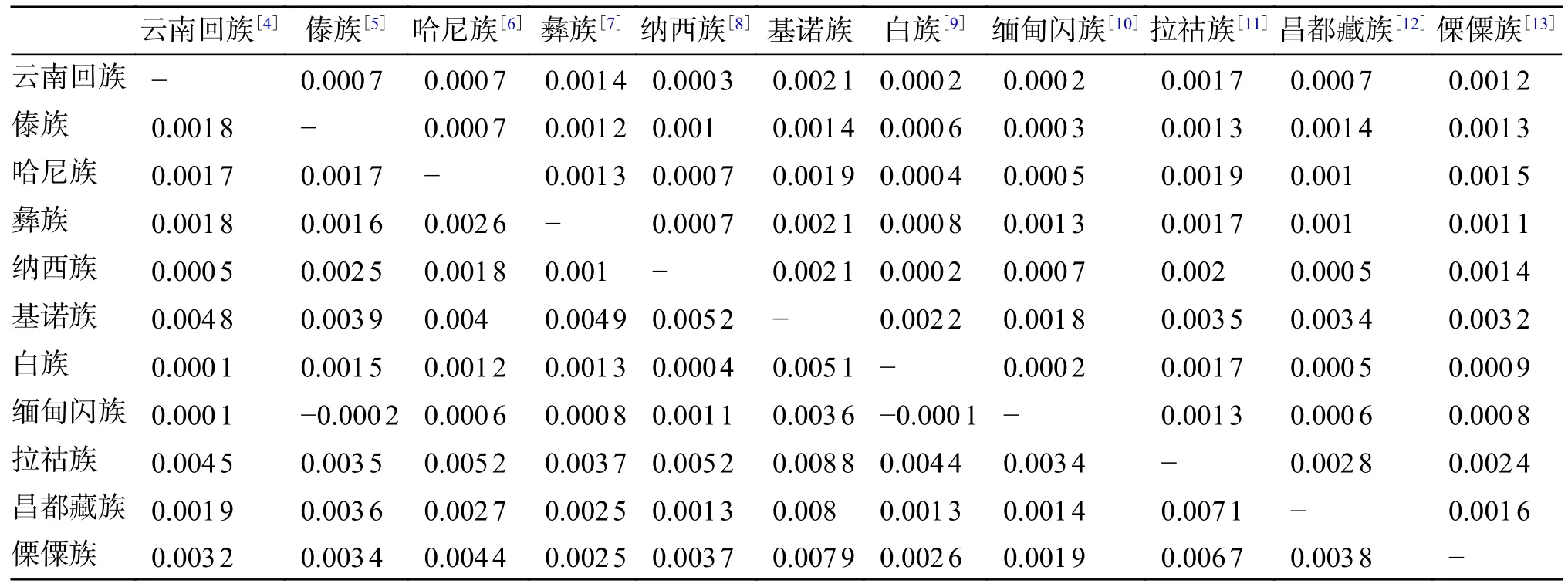
表3 基諾族人群與白族、傣族、納西族等10 個人群 Standard genetic distances(左下)和Standard error(右上)Tab.3 Standard genetic distances(bottom left)and Standard error(top right)of Jino people and 10 other groups
3 討論
在法醫DNA 分析中,隨機匹配概率(RMP)是普通人群中隨機的、無關的人表現出與嫌疑人匹配的DNA 圖譜的概率,DNA 圖譜越罕見,已知和質疑的證據來自同一個人的幾率就越大。基于毛細管電泳技術開發的STR 基因座檢測試劑盒通常以CODIS 位點為核心,增加性別、Y-STR 或SNP 位點的方式進行設計。CODIS 位點覆蓋了人基因組不同染色體的STR 基因座[15],且包含有序列復雜度較高的D8S1179、FGA、D21S11 基因座,在法醫物證學的實踐中,發現在不同的地區、人群中STR 基因座的識別效能存在比較大的差異,如TPOX 和TH01 基因座,在我國人群的識別效能都不算高,因此,對于不同群體進行基礎的人群頻率調查,有利于評估試劑盒的實用性。以本研究結果為例,按 DP≥ 0.9、H ≥ 0.7、PIC ≥0.7 的 STR 基因座屬高度多態性基因座的標準來衡量,基諾族17 個常染色體STR 基因座中,TPOX、TH01 基因座多態性和識別能力最低,D3S1358、D13S317、D16S539、CSF1PO、D5S818均有參數顯示多態性不佳。
所有基因座的基因型分布均符合哈溫平衡,說明采樣符合隨機性要求,同時各基因座間相互獨立。根據乘積原則,AGCU 17+1 試劑盒在基諾族人群中具有較好的個體識別和親權鑒定的系統效能(CDP > 0.999 999 999、CPE > 0.999 999),考慮到部分基因座多態性不佳的情況,可以適當增加試劑盒檢測的基因座個數,從而提高試劑盒的識別能力。CODIS 核心STR 數據庫于2015 年進行了拓展,核心CODIS 基因座變為19 個[15],增加了7 個基因座(D2S1338、D19S433、D1S1656、D12S391、D2S441、D10S1248 以及 DYS391),剔除了 TPOX 基因座。根據基諾族17 個常染色體STR 基因座人群頻率調查結果,TPOX、TH01 基因座多態性差,雖然17 個基因座聯合使用具有較高的識別能力,建議增多等位基因座檢測或選擇不含TPOX 和TH01 基因座試劑盒進行該人群法醫學個人識別和親權鑒定,尤其是特殊親權鑒定,如祖孫鑒定、同胞鑒定等情況[16]。在法醫學實踐中,應該以本國人群的STR 基因座數據,篩選合適的高識別度的基因座,形成核心基因座+高特異性基因座聯檢試劑盒。
本研究所調查的基諾族群體屬于云南人口較少的少數民族,主要聚居于西雙版納基諾鄉。從遺傳距離來看,基諾族與同屬于彝語支的哈尼族匯聚在一起,雖然基諾族與傣族在聚居地重疊,但是在遺傳距離數據看,用語系進行結構分類更具有接近度,提示基諾族由北方遷徙而來的觀點有一點的合理性。

