IBS 法和FSI 法在云南人群全同胞關系鑒定中的應用
易 娜 ,黃仁武 ,蔣佳梅 ,孟存溦 ,葉 好 ,李湘萍 ,劉林林 ,胡利平
(1)昆明醫科大學法醫學院法醫學系,云南 昆明 650500;2)昆明醫科大學司法鑒定中心,云南 昆明 650500)
近年來,隨著法醫學鑒定技術的發展,復雜親緣關系鑒定需求日益增加,全同胞關系作為復雜親緣關系的一種,指有相同生物學父親和生物學母親的多個子代個體之間的關系[1],目前在已在司法實踐的多個領域發揮著重要的作用[2]。因此,根據實際需要,司法部司法鑒定管理局于2014 年發布了《生物學全同胞關系鑒定實施規范》(SF/Z JD0105002—2014,后文簡稱“舊規范”),規范了基于狀態一致性評分(identity by state(IBS)score)的生物學全同胞關系鑒定的技術方法,但由于舊規范中過高的系統效能和對不同群體之間的群體差異考慮的欠缺,導致在實際應用過程中存在一定的誤判風險。隨后,司法部于2021 年發布并實施了《生物學全同胞關系鑒定技術規范》(SF/T 0117—2021,后文簡稱“新規范”)以代替舊規范。新規范相較于舊規范更新了累計狀態一致性評分(CIBS)的判定閾值并降低了對應檢測系統的系統效能,增加了基于等位基因頻率計算的全同胞指數(full sibling index,FSI)和累積全同胞指數(CFSI)的判定閾值及相應的系統效能[3]。本文基于新舊規范中相同的19 個必檢基因座檢測系統探討了2 份規范中3 種不同全同胞鑒定閾值劃分方法在系統效能上的差異以及新規范中兩種方法在云南人群生物學全同胞關系鑒定中的適用性。
1 材料與方法
1.1 樣本來源
在獲得知情同意的前提下,收集1 468 對來自昆明醫科大學司法鑒定中心日常案件積累的云南人群樣本(口腔拭子、血樣),其中全同胞有448 對,無關個體有1 020 對。全同胞對為已通過舊規范中IBS 法認定(包括增加20 個基因座后認定為全同胞的個體對)的兄弟姐妹組合,排除了同卵雙生子和STR 突變的家系。無關個體對為實驗室隨機抽取的無親緣關系的個體兩兩隨機組合。
1.2 STR 基因座選擇及分型檢測
案件鑒定用Chelex-100 法提取血樣 DNA,采用Promega 公司PowerPlex?21 System 試劑盒進行擴增,擴增產物經 ABI3130XL 型基因分析儀電泳分離,應用ABI GeneMapperID-1.5 軟件分析。每個樣本均得到D3S1358、D1S1656、D6S1043、D13S317、Penta E、D16S539、D18S51、D2S1338、CSF1PO、PentaD、TH01、vWA、D21S11、D7S820、D5S818、TPOX、D8S1179、D12S391、D19S433、FGA 和 Amelogenin 共21 個STR 基因座的分型結果,包含全同胞鑒定規范中的19 個必檢基因座。
1.3 計算累計狀態一致性評分
兩名有爭議個體在同一STR 基因座上出現相同等位基因的個數就是狀態一致性評分。當采用包含多個相互獨立的常染色體遺傳標記分型系統對兩名有爭議個體進行檢測時,各個遺傳標記上IBS 之和即稱為累計狀態一致性評分[3]。利用全同胞鑒定規范中狀態一致性評分方法,分別計算每對全同胞和無關個體之間19 個必檢基因座的CIBS 值,并對數據進行統計整理。
1.4 計算累積全同胞指數
對于每一個STR 基因座而言,兩名有爭議個體之間存在全同胞關系時其基因型出現的機率與兩名有爭議個體之間為無關個體時其基因型出現的機率之比值即為全同胞指數。當采用包含多個相互獨立的常染色體遺傳標記分型系統對兩名有爭議個體進行檢測時,各個遺傳標記上FSI 的乘積即稱為常染色體STR 基因座累積全同胞關系指數[3]。根據云南漢族人群中 19 個常染色體STR基因座的等位基因頻率資料[4]和新規范中全同胞指數和累積全同胞關系指數的算法[5],分別計算全同胞組和無關個體組的配對個體之間19 個必檢基因座的CFSI 值,并對數據進行統計整理。
1.5 3 種方法的閾值劃分
基于新舊規范,用IBS 法和FSI 法分別計算1 468對樣本的全同胞關系,并對計算結果按照以下三種方法進行劃分:方法一以舊規范中 IBS ≥ 22 傾向于認為兩名被鑒定人為全同胞,IBS ≤ 13 傾向于認為兩名被鑒定人為無關個體和13 < IBS < 22無法給出傾向性意見為標準進行閾值劃分;方法二以新規范中IBS>22 傾向于認為兩名有爭議個體為全同胞關系,IBS < 12 傾向于認為兩名有爭議個體為無關個體對和12 ≤ IBS ≤ 22 無法給出傾向性意見為標準進行閾值劃分;方法三以新規范中FSI>1.0×104傾向于認為兩名有爭議個體為全同胞關系,FSI < 1.0×10-4傾向于認為兩名有爭議個體為無關個體對和1.0×10-4≤ FSI ≤ 1.0×104無法給出傾向性意見為標準進行閾值劃分。
1.6 統計學處理
采用SPSS 25.0 軟件對全同胞組和無關個體組的lgCFSI 值和CIBS 值的分布進行正態檢驗后,統計不同組別lgCFSI 值和CIBS 值的算數均數和標準差,并對其進行獨立樣本t檢驗,對計數資料采用χ2檢驗,均以P< 0.05 為差異有統計學意義。
2 結果
2.1 CIBS 值和CFSI 值的分布
根據448 對全同胞和1 020 對無關個體的CIBS 值和對CFSI 值經對數轉換后的lgCFSI 值進行正態檢驗,結果表明全同胞組和無關個體組的CIBS 值及lgFSI 值均符合正態分布(圖1,圖2)。

圖1 全同胞組和無關個體組的CIBS 值擬合所得正態分布曲線Fig.1 The normal distribution curve obtained by fitting the CIBS of the full sibling group and the unrelated individual group
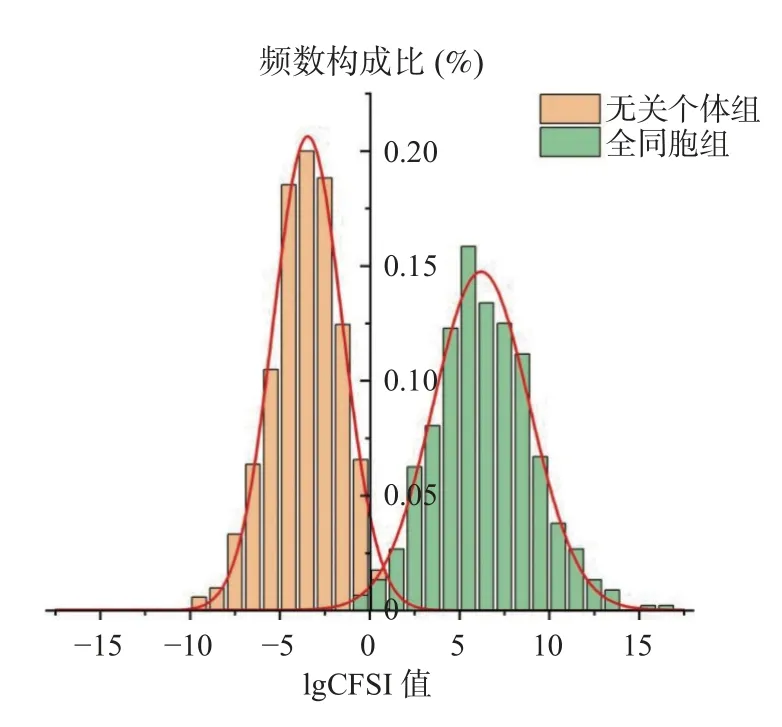
圖2 全同胞組和無關個體組的lgCFSI 值擬合所得正態分布曲線Fig.2 The normal distribution curve obtained by fitting the lgCFSI of the full sib group and the unrelated individual group
2.2 無關個體組與全同胞組的差異
統計并描述全同胞組和無關個體組CIBS 值和lgCFSI 值的算數均數、標準差、t值(表1),結果顯示無關個體組與全同胞組在CIBS 值與lgCFSI值的差異均有統計學意義,P< 0.001。
表1 全同胞組和無關個體組CIBS 值和lgCFSI 值的 和獨立樣本t 檢驗結果()Tab.1 The and independent sample t test results of the CIBS values and lgCFSI values between the full sibling group and the unrelated individual group()

表1 全同胞組和無關個體組CIBS 值和lgCFSI 值的 和獨立樣本t 檢驗結果()Tab.1 The and independent sample t test results of the CIBS values and lgCFSI values between the full sibling group and the unrelated individual group()
2.3 3 種方法的統計結果及差異
分別按照3 種方法對全同胞組和無關個體組的計算結果進行統計整理和χ2檢驗(表2),顯示方法一有1 087 對樣本可以得到傾向性意見,有381 對樣本無法得出傾向性意見,實際檢出率為74.05%;方法二有750 對樣本可以得到傾向性意見,有718 對樣本無法得出傾向性意見,實際檢出率為51.09%;方法三有972 對樣本可以得到傾向性意見,有496 對樣本無法得出傾向性意見,實際檢出率為66.21%。3 種方法在19 個常染色體STR 基因座檢測水平上的全同胞組檢測準確率和無關個體組檢測準確率均為100%。3 種方法的χ2檢驗結果顯示在全同胞組和無關個體組中,3 種方法的檢出率不完全相同,差異有統計學意義。對2 個組別中3 種方法的檢出率進行方法二與方法一和方法三的比較(表3),通過Bonferroni方法進行校正,結果顯示在2 個組別中方法二與方法一和方法三的差異均有統計學意義。

表2 3 種方法的統計結果(n)Tab.2 Statistical results of the three methods(n)

表3 3 種方法兩兩比較Tab.3 Pairwise comparison of three methods
3 討論
對于同樣的19 個常染色體STR 基因座檢測系統,以上不同全同胞鑒定閾值劃分方法的系統效能在兩份規范中分別為0.750 0,0.556 5 和0.662 5;在云南人群中的系統效能分別為0.740 5,0.510 9 和0.662 1。比較新舊規范中IBS 法的應用,因CIBS 閾值劃分標準不同,兩種方法的系統效能相差約0.193 5,在云南人群中2 種方法的系統效能相差約0.226 9。在新規范中減少的這部分系統效能是由于更新的規范中將個體對的得分為“CIBS=12”、“CIBS=13”、“CIBS=22”時判定為“無法給出傾向性意見”,同時靠近閾值附近的個體對數也明顯較多,從而導致兩種方法的系統效能相差較大。新規范降低了IBS 法判定全同胞關系的系統效能,降低了誤判率,使得全同胞鑒定時給出肯定意見的準確性有所提高[6]。
FSI 法對于全同胞關系指數的計算由ITO 法提供[7],用以判定兩名有爭議個體是否為全同胞關系的方法。該方法的計算過程較IBS 法稍顯復雜,但FSI 法考慮到了人群群體遺傳學差異,基于不同人群等位基因頻率在某一檢測系統下進行計算,因此能有效避免全同胞關系鑒定中的誤判,增加結果的可信度[8]。本研究基于新規范以及文獻報道的云南人群的等位基因頻率,在19 個常染色體STR 基因座檢測系統下,通過FSI 法計算的系統效能均高于IBS 法。同時,比較全同胞組和無關個體組的正態分布曲線,lgCFSI 值的重疊范圍和比例明顯小于CIBS 值的重疊范圍和比例,也說明FSI 法能對全同胞和無關個體進行更有效的區分。
本研究考慮到實際鑒定中,不同人群同一基因座上等位基因頻率分布的差異會使得同一基因座的個體識別能力有所不同[9],從而導致同一檢測系統在不同人群中的系統效能有一定差異的情況。應用新規范中IBS 法和FSI 法在19 個常染色體STR 檢測系統上對1 468 對云南人群樣本進行了全同胞關系鑒定,計算所得在云南人群中兩種方法的系統效能分別為0.510 9 和0.662 1 與新規范中同一檢測系統下對應方法的系統效能0.556 5和0.662 5 相近,說明新規范在云南人群中有很高的應用價值。對于云南地區少數民族聚集,不同的少數民族間或有部分等位基因頻率存在較大差異的情況[10],新規范規定,當某個基因座的個體識別能力與規范中存在較大差異時,可以在準確性不低于99.99%的前提下重新計算并制定全同胞關系鑒定的CIBS 閾值,這也能有效解決新規范在各地區人群中實際應用的局限。
對于無法給出傾向性意見可補充檢測基因座,以增加能給出傾向性意見的可能性[11]。新規范中也給出了基于19 個常染色體STR 基因座依次補充36 個與其不存在連鎖不平衡的常染色體基因座后相應的檢測系統效能,對于IBS 法和FSI 法其最高系統效能分別可達0.998 6 和0.996 5[3],能有效區分全同胞和無關個體。對于特殊的鑒定案件,還可補充檢驗X 染色體、Y 染色體或線粒體DNA遺傳標記的情況[12],但應該根據其遺傳規律采用文字描述的方式進行分析說明。
本研究發現,基于19 個常染色體STR 基因座檢測系統,新規范在云南人群全同胞關系鑒定中具有較高的法醫學應用價值;相較于舊規范,新規范中的IBS 法和FSI 法能有效降低全同胞關系鑒定的錯判風險。由于IBS 法和FSI 法在計算原理上的差異,本文建議實際應用中將IBS 法和FSI 法相結合,對結果進行更為科學和準確的判斷。需要注意的是,無論新規范還是舊規范均只適用于同父同母的全同胞關系鑒定,對于半同胞關系的判別并不適用。實際案件中如遇到不確定為全同胞的情況,可通過ITO 法對全同胞和半同胞關系進行判別[13-14],從而得出相應結論。

