eptA過表達介導的臨床分離的鮑曼不動桿菌多黏菌素耐藥研究
葉秀芬?俞容?周益琴

摘要:目的 為了確定鮑曼不動桿菌除PmrCAB以外的多黏菌素耐藥機制。方法 收集2020年金華市人民醫院住院患者痰液標本分離的3株鮑曼不動桿菌(AB1、AB2和AB3)。微量肉湯稀釋法測定3株鮑曼不動桿菌對亞胺培南、美羅培南和多黏菌素的最低抑菌濃度。通過PCR明確菌株是否攜帶eptA并進一步對操縱子pmrCAB進行分析,明確是否存在氨基酸突變。全基因組測序分析菌株的ST型、耐藥基因以及eptA的上游結構。使用熒光定量PCR技術檢測多黏菌素耐藥相關基因pmrB以及eptA基因的表達量。GraphPad Prism 8.0用于數據分析。結果 3株鮑曼不動桿菌對多黏菌素耐藥,MIC=16 μg/mL。根據Pasteur分型,它們都屬于ST2;根據Oxford分析,AB1和AB3屬于ST208,AB2屬于ST1806。3株鮑曼不動桿菌都攜帶blaOXA-23碳青霉烯酶耐藥基因,但都不存在PmrCAB氨基酸突變。表達量分析顯示3株菌存在eptA過表達(P<0.01)。ISAba1的插入位點分別在eptA起始密碼子之前第13和18個堿基處。然而,pmrCAB表達量改變差異無統計學意義(P>0.05)。結論 ISAba1插入介導的eptA的過表達可能是新的鮑曼不動桿菌多黏菌素耐藥機制之一。
關鍵詞:鮑曼不動桿菌;耐藥;多黏菌素;eptA
中圖分類號:R978.1文獻標志碼:A
Study on polymyxin resistance mediated by eptA overexpression in Acinetobacter baumannii clinical isolates
Ye Xiu-fen, Yu Rong, and Zhou Yi-qin
(Department of Clinical Laboratory, Jinhua Peoples Hospital, Jinhua 321000)
Abstract Objective To determine the polymyxin resistance mechanism in Acinetobacter baumannii (A. baumannii) other than PmrCAB. Methods Three A. baumannii clinical strains collected from sputum were selected in 2020. Minimal inhibitory concentrations (MICs) of imipenem, meropenem and colistin were tested by the broth micro-dilution method. PCR was conducted to test whether the strains carried eptA and further analyzed the pmrCAB mutation.? The ST type, drug resistance gene, and the upstream structure of eptA of the strain were analyzed using whole-genome sequencing. Real-time quantitative PCR (qRT-PCR) was used to detect the expression of polymyxin resistance-related genes pmrB and eptA. Experimental data were analyzed using GraphPad Prism 8.0. Results Three A. baumannii strains were resistant to polymyxin, and MICs were all 16 μg/mL. According to the Pasteur type, they all belonged to ST2; according to Oxford type, AB1 and AB3 belonged to ST208, and AB2 belonged to ST1806. Three strains all carried the blaOXA-23 carbapenemase resistance gene, but none of them had PmrCAB amino acid mutations. Expression analysis showed that the three strains had overexpression of eptA (P<0.01). The insertion sites of ISAba1 were 13 and 18 bases before the eptA start codon, respectively. However, there was no statistically significant difference in the expression of pmrCAB (P>0.05). Conclusion The overexpression of eptA mediated by the insertion of ISAba1 may be one of the new polymyxin resistance mechanisms in A. baumannii.
Key words Acinetobacter baumannii; Resistance; Polymyxin; eptA
鮑曼不動桿菌(Acinetobacter baumannii)可引起各種醫院相關感染。在過去的幾十年中,由于大多數可用抗菌藥物的耐藥菌株出現及擴散已經促使臨床醫生轉向多黏菌素作為治療的最終方案[1]。由于多黏菌素的使用量增加,導致其耐藥性逐漸增加,特別是碳青霉烯耐藥鮑曼不動桿菌(carbapenem-resistant A. baumannii, CRAB)[2]。因此,明確是否有除常見的PmrCAB氨基酸突變介導的多黏菌素耐藥外的新機制對臨床用藥和CRAB治療至關重要。
1 材料與方法
1.1 實驗材料
3株耐碳青霉烯鮑曼不動桿菌(CRAB)(AB1,AB2和AB3)分離自2020年金華市人民醫院住院患者痰液標本,質控菌株大腸埃希菌ATCC25922;亞胺培南(1.0 g,國藥準字H20084019,大連美侖生物技術有限公司)、美羅培南(0.5 g,國藥準字H20093466,上海上藥新亞藥業有限公司);多黏菌素(1.0 g,美國Sigma公司);MH肉湯(英國Oxoid公司);比濁儀(法國Merieux公司);熒光定量PCR儀(美國Thermo公司)。
1.2 碳青霉烯類藥物和多黏菌素敏感性試驗
采用微量肉湯稀釋法測定3株鮑曼不動桿菌對亞胺培南、美羅培南和多黏菌素的最低抑菌濃度。藥敏結果根據美國臨床和實驗室標準化協會CLSI 2020標準判讀。質控菌株為大腸埃希菌ATCC25922。
1.3 DNA擴增檢測耐藥相關基因突變情況
用DNA提取試劑盒提取細菌DNA。對eptA基因進行PCR,引物序列如表1所示[3-4],主要步驟如下:配置PCR反應體系(10×PCR Buffer,Mg2+,dNTP Mixture,上游引物,下游引物,rTaq酶,無菌雙蒸水,DNA模板)。PCR反應過程如下:預變性(94℃,5 min),變性(94℃,30 s),退火(55℃,30 s),延伸(根據引物長度,1000 bp/min),最后延伸(72℃,7 min),同時設置1個陽性對照、一個陰性對照以及不加DNA的空白對照。PCR產物瓊脂糖凝膠電泳及觀察。選取與目的大小片段相符的陽性產物送上海華大基因公司測序,測序結果再在NCBI數據庫進行BLAST序列比對分析。
1.4 全基因組測序以及eptA基因上游結構分析
對3株CRAB進行全基因組測序。首先,使用試劑盒提取細菌總DNA后送公司進行測序。通過使用CLC Genomics Workbench 10.1.1對序列進行拼接,并通過序列比對分析eptA基因的上游結構。
1.5 qRT-PCR檢測多黏菌素耐藥相關基因表達量的變化
使用熒光定量PCR技術檢測多黏菌素耐藥相關基因pmrB以及eptA基因的表達量。所用引物詳見表2[5]。結果采用2-ΔΔCt方法,菌株ATCC19606為參考菌株,rpoB為內參基因。以eptA基因為例,如下所示:ΔCt(試驗樣本)=Ct(試驗樣本eptA基因)-Ct(試驗樣本rpoB基因),ΔCt(校準樣本)=Ct(ATCC19606 eptA基因)-Ct(ATCC19606 rpoB基因),-ΔΔCt=ΔCt(校準樣本)-ΔCt(試驗樣本),2-ΔΔCt即為eptA的表達量。
1.6 統計學方法
GraphPad Prism 8.0對數據進行統計學分析及圖片繪制,P<0.05為差異有統計學意義。
2 結果
2.1 藥敏結果
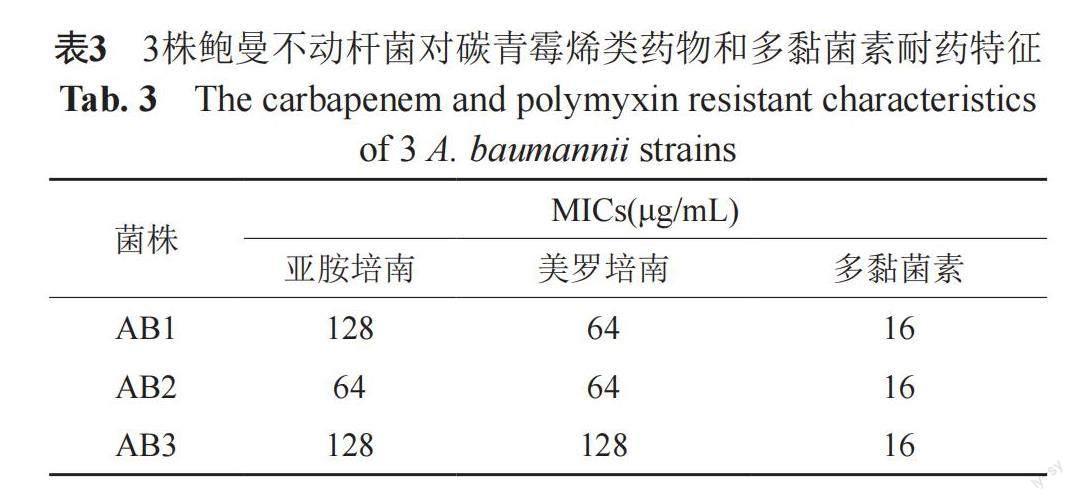
AB1,AB2,AB3對亞胺培南和美羅培南均耐藥,MIC均≥64 μg/mL,且均對多黏菌素耐藥,MIC=16 μg/mL,具體結果見表3。
2.2 ST型、耐藥基因以及常見多黏菌素耐藥基因突變情況
通過分析3株菌的耐藥基因,發現這3株菌都攜帶blaOXA-23碳青霉烯酶耐藥基因,此外還攜帶β-內酰胺類(blaADC-52,blaADC-176和blaOXA-51)、氨基糖苷類(armA,aph(3)-VI)以及大環內酯類[msr(E),mph(E)]耐藥基因。根據Pasteur分型,它們都屬于ST2;根據Oxford分析,AB1和AB3屬于ST208,AB2屬于ST1806。此外,與多黏菌素耐藥相關的基因pmrA、pmrB和pmrC沒有氨基酸突變(表4)。
2.3 eptA基因上游結構分析
通過全基因組測序進一步分析eptA基因上游結構。結果顯示:AB1和AB2在上游13個堿基處有一個ISAba1,而AB3在上游18個堿基處有一個ISAba1。其余的結構相同。具體結果見圖1。
2.4 多黏菌素耐藥相關基因表達量變化
進一步對鮑曼不動桿菌多黏菌素耐藥相關基因的表達量進行了分析。結果顯示,eptA的表達量可增加4~7倍,差異具有非常統計學意義(P<0.01)。然而,染色體上的其他多黏菌素耐藥相關基因pmrB和pmrC的表達量無明顯變化,差異無統計學意義(P>0.05)。具體結果詳見圖2。
3討論
CRAB對多黏菌素的耐藥率越來越高,已成為許多臨床醫生面臨的主要挑戰[6-7]。研究表明,鮑曼不動桿菌對多黏菌素的耐藥性主要依賴于LOS促修飾酶,從而使抗菌藥物與細菌初次接觸后,細菌表面的凈負電荷減少,雙組分系統PmrAB的突變激活是主要的多黏菌素耐藥機制,其可導致磷酸乙醇胺(pEtN)轉移酶PmrC過表達進而導致多黏菌素耐藥[8-9]。PmrCAB介導的多黏菌素耐藥機制已經完全明確,而是否存在新的耐藥機制有待進一步研究。
最近有研究發現在編碼H-NS相關轉錄調節因子的基因中插入ISAba125元件有助于鮑曼不動桿菌臨床菌株產生高水平多黏菌素耐藥性[10]。這種插入觸發了遠端基因的表達,該基因為eptA,其產物與PmrC具有93%的同源性,且與PmrC一樣可介導pEtN轉移酶活性[11]。因此,研究eptA基因是否可以介導多黏菌素耐藥意義重大。
eptA基因在鮑曼不動桿菌分離株中很常見。在法國NRC-AR分析的32株黏菌素敏感菌株中,27株(84%)分離株eptA陽性,但它們都沒有攜帶基因上游的ISAba1序列[12-13]。Lesho等[10]在116株鮑曼不動桿菌菌株中檢測到20%菌株除pmrC外還存在eptA同源物。本研究中,發現3株多黏菌素耐藥鮑曼不動桿菌存在pmrC同系物eptA過表達,這可能是由于上游插入ISAba1導致。ISAba1是一種在鮑曼不動桿菌基因組中普遍存在的遺傳元件[14-15],它可以為下游基因提供一個強的啟動子,進而使下游基因表達量增加,從而導致多黏菌素耐藥,對eptA表達量的分析結果也進一步證實了這一點。在研究的分離株中,通過不同的遺傳事件,ISAba1的插入發生在eptA上游的不同位置(13和18 bp)。然而,eptA的表達水平與ISAba1和eptA轉錄起始之間的距離無關。在此次研究菌株中,在eptA上游插入ISAba1構成了一種更直接的eptA激活方式,其導致的多黏菌素MIC可以直接達到耐藥水平。由于鮑曼不動桿菌中ISAba1和eptA基因相當普遍,因此在反復進行多黏菌素治療的壓力下,由隨機插入事件引起多黏菌素耐藥的現象時常發生。
總之,本研究表明CRAB對多黏菌素耐藥機制的復雜性和多樣性,提出了ISAba1和eptA共同作用下介導的多黏菌素耐藥機制,為進一步研究CRAB對多黏菌素耐藥提供依據。
參 考 文 獻
Carrasco L D M, Dabul A N G, Boralli C, et al. Polymyxin resistance among XDR ST1 carbapenem-resistant Acinetobacter baumannii clone expanding in a teaching hospital[J]. Front Microbiol, 2021, 12: 622704.
Trebosc V, Gartenmann S, Totzl M, et al. Dissecting colistin resistance mechanisms in extensively drug-resistant Acinetobacter baumannii clinical isolates[J]. mBio, 2019, 10(4): e01083-19.
Xu F, Hinenoya A, Zeng X, et al. Critical role of 3'-downstream region of pmrB in polymyxin resistance in Escherichia coli BL21(DE3)[J]. Microorganisms, 2021, 9(3): 655.
Ilsan N A, Lee Y J, Kuo S C, et al. Antimicrobial resistance mechanisms and virulence of colistin-and carbapenem-resistant Acinetobacter baumannii isolated from a teaching hospital in Taiwan[J]. Microorganisms, 2021, 9(6):1295.
Kim M, Park J, Park W. Genomic and phenotypic analyses of multidrug-resistant Acinetobacter baumannii NCCP 16007 isolated from a patient with a urinary tract infection[J]. Virulence 2021, 12(1): 150-164.
Yoon E J, Kim H S, Woo H, et al. Trajectory of genetic alterations associated with colistin resistance in Acinetobacter baumannii during an in-hospital outbreak of infection[J]. J Antimicrob Chemother, 2021, 77(1): 69-73.
Piperaki E T, Tzouvelekis L S, Miriagou V, et al. Carbapenem-resistant Acinetobacter baumannii: In pursuit of an effective treatment[J]. Clin Microbiol Infect, 2019, 25(8):951-957.
Nodari C S, Fuchs S A, Xanthopoulou K, et al. pmrCAB recombination events among colistin-susceptible and -resistant Acinetobacter baumannii clinical isolates belonging to international clone 7[J]. mSphere, 2021: e0074621.
Lima W G, Alves M C, Cruz W S, et al. Chromosomally encoded and plasmid-mediated polymyxins resistance in Acinetobacter baumannii: A huge public health threat[J]. Eur J Clin Microbiol Infect Dis, 2018, 37(6):1009-1019.
Lesho E, Yoon E J, McGann P, et al. Emergence of colistin-resistance in extremely drug-resistant Acinetobacter baumannii containing a novel pmrCAB operon during colistin therapy of wound infections[J]. J Infect Dis, 2013, 208(7): 1142-1151.
Deveson L D, Crane B, Wright A, et al. Emergence of high-level colistin resistance in an Acinetobacter baumannii clinical isolate mediated by inactivation of the global regulator H-NS[J]. Antimicrob Agents Chemother, 2018, 62(7): e02442-17.
Gerson S, Lucassen K, Wille J, et al. Diversity of amino acid substitutions in PmrCAB associated with colistin resistance in clinical isolates of Acinetobacter baumannii[J]. Int J Antimicrob Agents, 2020, 55(3): 105862.
Gerson S, Betts JW, Luca?en K, et al. Investigation of novel pmrB and eptA mutations in isogenic Acinetobacter baumannii isolates associated with colistin resistance and increased virulence In vivo[J]. Antimicrob Agents Chemother, 2019, 63(3): e01586-18.
Agoba E E, Govinden U, Peer A K C, et al. ISAba1 regulated OXA-23 carbapenem resistance in Acinetobacter baumannii strains in durban, South Africa[J]. Microb Drug Resis, 2018, 24(9): 1289-1295.
付啟云, 鄭紹同, 連建春, 等. 耐藥鮑曼不動桿菌不同年份分離株耐藥元件基因比較研究[J]. 中國抗生素雜志, 2019, 44(5): 595-599.

