肉牛剩余采食量相關下丘腦差異lncRNA篩選及特征分析
蘇總華,周小南,王曉薇,楊朝云,丁燕玲,張巖峰,李承隆,曾 靈,明文軒,史遠剛,康曉龍
(寧夏大學 動物科技學院,寧夏 銀川 750021)
在家畜飼養過程中,飼料利用率是評價生產成本最重要的指標之一。在肉牛養殖過程中,飼料成本占總成本支出的70%以上[1]。因此,提高動物飼料利用率能夠直接降低飼料成本,減少甲烷排放[2]。使用剩余采食量(Residual feed intake,RFI)不僅能夠精準測定家畜的飼料利用效率[3],而且能夠排除動物生長性狀以及生長速率的影響[4];RFI具有中等遺傳力(h2=0.29~0.46)[5-8],并在眾多畜禽飼料效率研究中得到開展,通過獲得RFI相關的SNP位點[9]、表達調控相關基因[10]、QTL[11]、GWAS[12]等數據,為從遺傳角度進行RFI解析提供依據。
長鏈非編碼RNA(Long non-coding RNA)在各種生物過程中發揮著關鍵調節作用,與其相關染色質修飾、基因組印記、轉錄調控等眾多生物學領域得到廣泛關注[13-14]。越來越多的證據表明,lncRNA參與調控牛的多種生產性能,例如奶牛泌乳性能[15]、乳腺炎[16]、骨骼肌發育[17]、熱應激[18]、酸中毒[19]和精子發生等[20]。飼料效率屬于復雜性狀,涉及飼料消化代謝、脂肪代謝、運動等多個層面。下丘腦作為重要的調節中樞,能夠整合機體代謝、神經內分泌信號協調動物的攝食行為及能量平衡。在下丘腦相關研究中,已經確定弓狀核在調節動物食欲方面具有關鍵作用,其通過2個神經元群調節采食量[21]。如促食欲神經肽NPY(Neuropeptide Y)和AGRP(Agouti-related peptide),能夠促進動物采食[22];相反的,厭食神經肽如α-MSH(α-Melanocyte-stimulating hormone),通過產生飽腹感抑制動物進食[22]。除了神經肽,其他調節因子,如激素、受體、酶類、轉錄因子等多種分子能將外周組織與中樞神經系統形成相互關聯的神經內分泌和代謝途徑網絡[23],從而對下丘腦參與調控機體食物攝入發揮關鍵作用。目前,已有研究分別從某一組織器官的分子功能予以揭示lncRNA相關調控分子的功能作用:骨骼肌[24]、肝臟[25-26]、腎上腺[27]、瘤胃[28],但采食中樞相關的lncRNA是否參與RFI表型變化尚不明確。本研究選取不同RFI表型安格斯肉牛,采集下丘腦組織,篩選肉牛下丘腦中與RFI相關差異表達lncRNA,預測其靶基因、相關生物學通路及其在肉牛能量平衡、飼料效率的潛在作用,研究結果將為提高動物飼料利用率及開展肉牛RFI研究提供理論支持。
1 材料和方法
1.1 研究動物與數據收集
本試驗個體選自寧夏某養殖場22月齡、初生體質量一致、健康狀態良好的安格斯公牛。試驗群體(30頭)按照NRC(2000)動物營養標準進行飼養,個體限欄飼喂,每天喂食2次,每次采食1.5 h,自由飲水。每周測量收集采食量(Feed intake,FI)數據,測定當天早晚各1次,利用試驗期內(81 d)每個個體采食量觀測值和預期采食量之差表示RFI。使用FI對中期代謝體質量 (Midpoint metabolic body weight,MMBW0.75)和平均日增質量 (Average daily gain,ADG)進行多元線性回歸估算個體RFI,RFI統計分析模型如下[3,29]:
RFI=FI-(b0+b1ADG+b2MMBW0.75)
式中b1和b2分別為MMBW0.75和ADG的偏回歸相關系數,b0為表型殘差。依據計算結果,RFI大于0的個體定義為高剩余采食量組,小于0的個體定義為低剩余采食量組。
1.2 下丘腦樣本采集
根據所收集數據,選取剩余采食量極端差異個體各5頭,分為高剩余采食量(H組)和低剩余采食量(L組)2組,依次編號為H1~H5和L1~L5。人道方法屠宰試驗牛后立即打開腦腔,采集下丘腦PBS清洗后放入無菌、無酶的凍存管,凍存于液氮中保存。
1.3 RNA提取與檢測
根據制造商提供的說明書,使用TRIzol試劑對采集樣本提取總RNA。對提取的總RNA分別使用1%瓊脂糖凝膠電泳、Nanodrop、Agilent 2100驗證RNA降解程度與是否被污染。保證樣本濃度≥500 ng/μL,從而滿足后續測序試驗條件。
1.4 cDNA文庫構建和轉錄組測序
樣本提取合格總RNA后,使用去核糖體RNA的方法構建鏈特異性cDNA文庫。按照Illumina TruseqTM樣品試劑盒說明書進行文庫構建。文庫先使用Qubitu 2.0初步定量,稀釋至1 ng/μL,之后使用Agilent 2100對文庫的插入片段大小進行檢測,隨后使用Q-PCR對文庫的有效濃度進行準確定量(文庫有效濃度>2 nmol/L),最后利用Illumina平臺測序,產生150 bp的雙端原始測序數據(Raw data)。
1.5 質控、比對和轉錄本組裝
為確保比對過程中提供高質量的數據,使用FastQC軟件對Raw data進行質量評估,再利用軟件Trim Galore并使用默認參數進行質控,去除接頭序列(adapter)和低質量片段序列(reads)及Poly-N等[30]。將Clean data通過HISAT2[31]軟件比對到牛參考基因組(ARS-UCD1.2),得到sam格式比對文件。利用samtools將sam文件轉化為bam格式并排序,之后利用StringTie進行轉錄本拼接與定量[32],采用FPKM(Fragments Per Kilobase of exon model per Million mapped fragments)表示轉錄本表達量。
1.6 lncRNA的篩選
在篩選之前,先使用cuffmerge軟件,將各樣本拼接得到的轉錄本文件進行合并,去掉鏈方向不確定的轉錄本,得到完整的轉錄樣信息。根據lncRNA的結構和編碼能力特征,對合并的轉錄本集合設置嚴格的5步篩選條件[33]:①轉錄本exon個數篩選(篩選exon≥2的轉錄本);②轉錄本長度篩選(篩選轉錄本長度≥200 bp的轉錄本);③通過Cuffcompare軟件對轉錄本已知注釋篩選(篩選去除與數據庫注釋exon區域有重疊的轉錄本);④通過Cuffquant軟件對轉錄本表達量篩選(篩選FPKM≥0.5的轉錄本);⑤使用CNCI[34]、CPC2[35]、Pfam-scan[36]3種軟件判斷轉錄本是否具有編碼潛能,取3種軟件的分析結果中沒有編碼潛能的轉錄本的交集用于后續分析。
1.7 主成分分析
本研究對測序后轉錄本使用R包factoextra進行主成分分析(Principal component analysis,PCA),用方差來衡量數據的差異性和相似性。根據PCA結果,剔除掉異常樣本,平衡在試驗和測序過程產生的誤差,同時保證在轉錄組測序中至少3個生物學重復。
1.8 差異lncRNA篩選及潛在靶基因的識別
使用R包edgeR,在lncRNA轉錄本表達矩陣中篩選H組和L組差異表達lncRNA,設置參數為|log2(Fold Change)|>1和padj<0.05,使用層次聚類的方法對差異lncRNA表達值進行聚類。接著對篩選測得的差異lncRNA潛在靶基因進行預測,設置co-location閾值為lncRNA上下游100 kb篩選順式靶基因。再根據樣本間lncRNA與mRNA表達量相關性(pearson,r= 0.95)篩選反式靶基因。
1.9 功能富集分析及蛋白互作網絡分析
使用R包clusterProfiler和enrichplot對篩選得到的順式靶基因和反式靶基因進行GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genes and Genomes)富集分析[37],設置篩選參數P<0.05。GO富集可以進一步明確lncRNA靶基因的主要生物學功能。KEGG通路富集可用于了解差異lncRNA通過靶基因參與調節的信號通路。根據GO和KEGG結果使用string(https://cn.string-db.org/)數據庫將相關基因進行蛋白互作分析,分析靶基因蛋白在生物系統中的相互作用關系。接著將分析結果導入Cytoscape軟件,使用插件MCODE設置參數(Degree Cutoff=2,Node Score Cutoff=0.2,K-core=2,max depth=100)篩選得到核心基因簇。
1.10 差異表達lncRNA的qRT-PCR驗證
為了驗證測序數據的準確性及可靠性,隨機選取差異表達上調lncRNA:LNC_001283、LNC_014918、LNC_007183、LNC_002434;差異表達下調lncRNA:LNC_015216、LNC_015680、LNC_017103、LNC_001739進行qRT-PCR驗證,選擇GAPDH作為內參基因,引物信息見表1。反應體系:TB Green Taq Ⅱ 10 μL,Rox Dye Ⅱ 0.4 μL,200 ng/μL cDNA 2 μL,10 nmol/L上下游引物各0.8 μL,加ddH2O至總體積20 μL。qRT-PCR反應條件:95 ℃ 30 s;95 ℃ 10 s,55 ℃ 30 s,循環39次;65~95 ℃ 5 s,每個基因3個重復。
1.11 統計分析
相關數據使用非參數檢驗或t檢驗分析處理組與對照組間的統計學顯著性,所有分析均使用R軟件進行。
2 結果與分析
2.1 安格斯牛剩余采食量表型值
在飼養的30頭公牛中,去除一頭公牛意外死亡,剩余29頭公牛測量數據如表2所示,利用采食量構建多元線性回歸計算模型,計算公式如下:
RFI=FI-(6.343 5+0.070 7×MMBW0.75+0.745 4×ADG)
式中RFI(剩余采食量)、FI(采食量)、MMBW0.75(中期代謝體質量)、ADG(平均日增質量)。
根據上述公式,測序樣本剩余采食量表型值如表3。
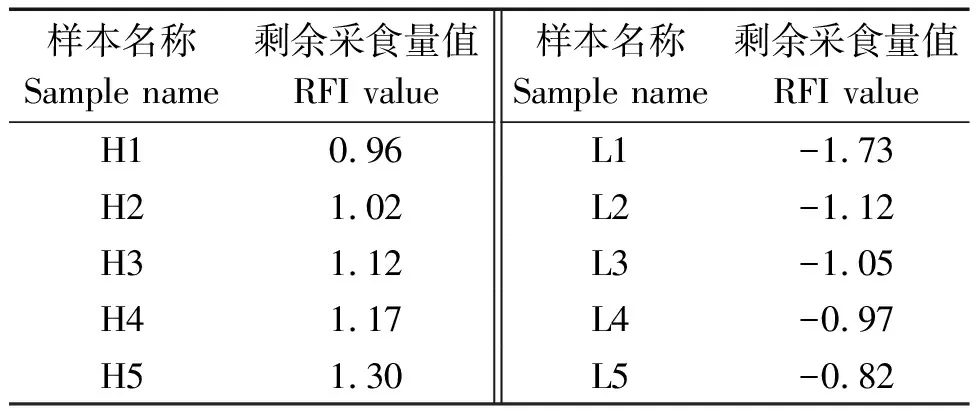
表3 測序樣本RFI值
2.2 lncRNA測序數據質量及比對情況
原始數據下機后經數據質控得到Clean reads(表4)。Clean data中Q20≥95.00%,Q30≥89.00%,GC含量占比均衡、穩定,說明測序數據質量較好。之后將Clean data比對到牛參考基因組后,其中能比對到參考基因組的reads占比在93.00%以上,有唯一比對位置的reads占比在79.00%以上,且在H組和L組中比對率平均值較為接近,比對結果良好(表5)。
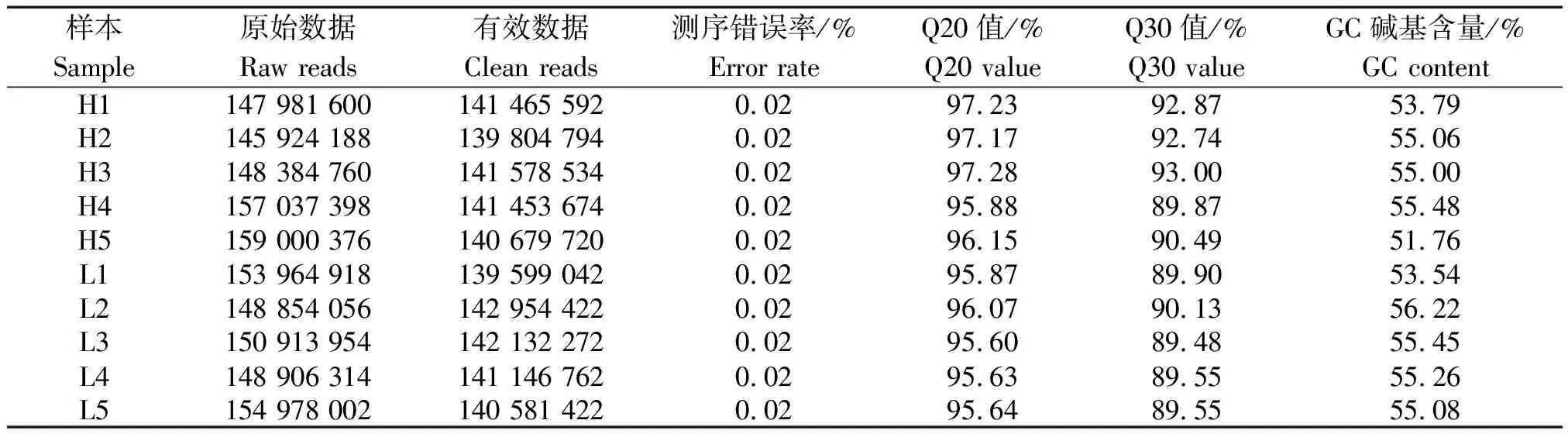
表4 樣本測序數據質量控制
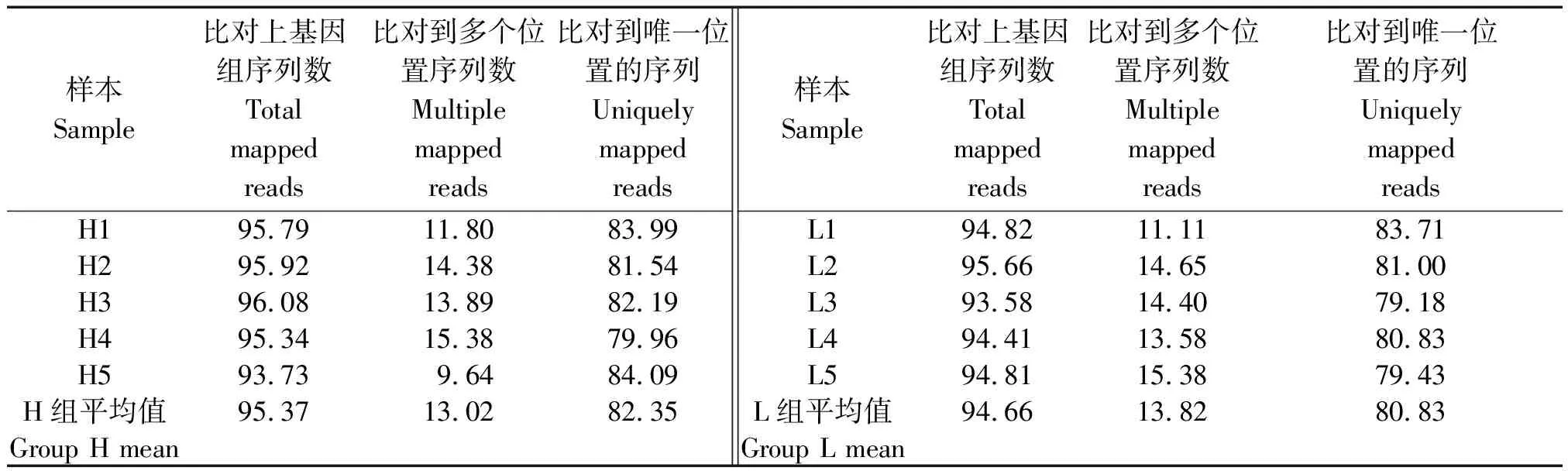
表5 測序reads基因組比對統計
2.3 lncRNA篩選
基于轉錄組拼接結果,對lncRNA分步篩選(圖1-A),并將各軟件識別出的非編碼轉錄本進行統計(圖1-B),最終得到20 996個lncRNA轉錄本集進行后續分析。
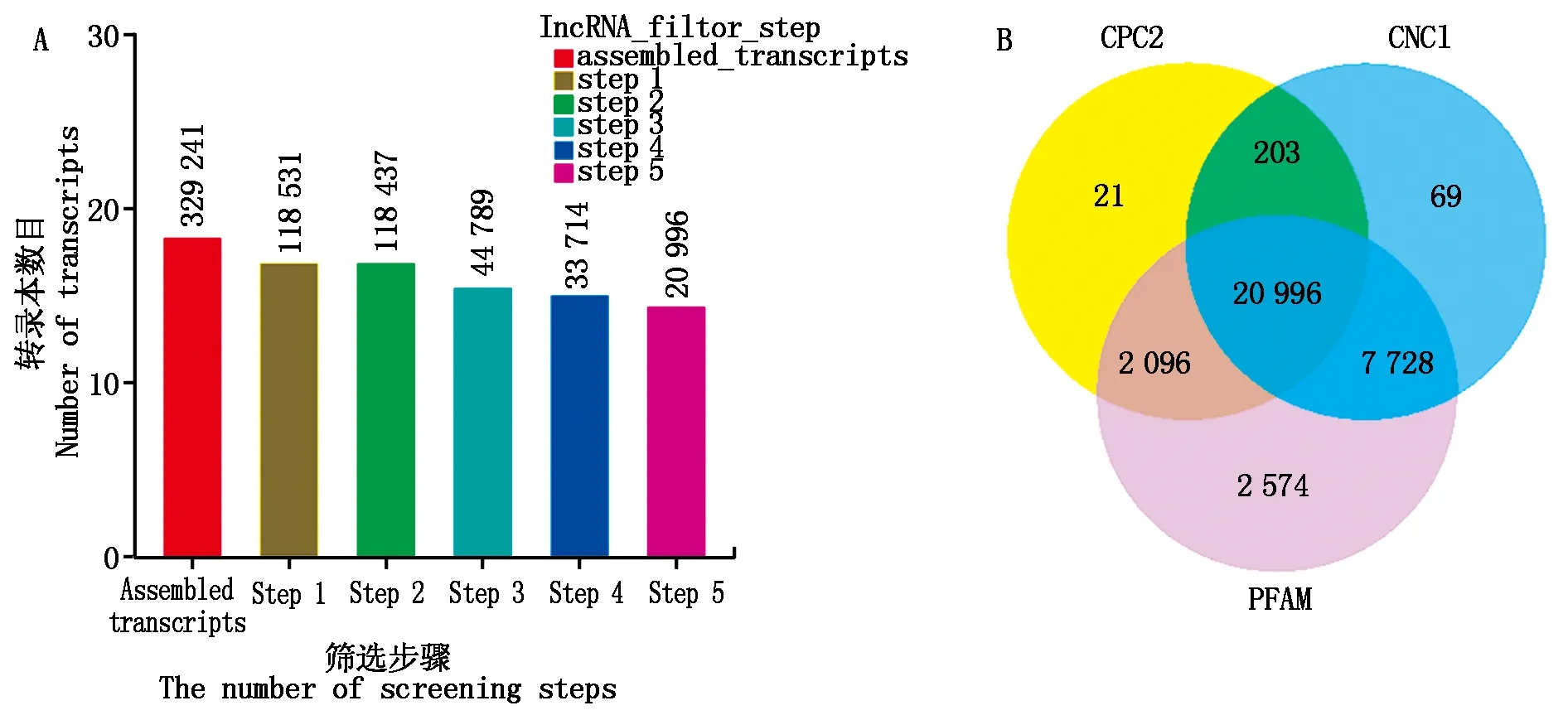
A.lncRNA篩選步驟統計圖;B.lncRNA篩選結果韋恩圖,表示3種軟件分析到的lncRNA數目。
2.4 牛RFI相關的下丘腦lncRNA表達分布及與mRNA結構特征分析
對lncRNA進行定量分析后,共得到20 996個lncRNA,后續分析lncRNA均基于此數據集,其中antisense_lncRNA(反義lncRNA)占比9.5%,主要產生于編碼鏈的反義鏈;intronic_lncRNA(內含子lncRNA)占比47.5%,主要產生于編碼基因的內含子區域;lincRNA(基因間lncRNA)占比43.0%,主要產生于2個編碼基因的中間區域(圖2-A)。
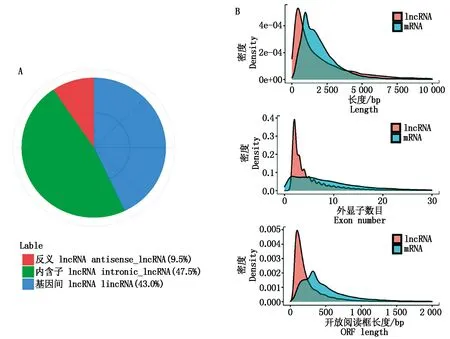
A.lncRNA類型分布;B.lncRNA與mRNA結構特征分析(lncRNA與mRNA的轉錄本長度、lncRNA與mRNA外顯子數目、lncRNA與mRNA ORF長度)。
將篩選得到的lncRNA及比對組裝的mRNA對轉錄本長度、外顯子個數、開放閱讀框(Open reading frames,ORF)長度進行比較分析,具體分析情況以核密度圖展示(圖2-B)。統計分析顯示,lncRNA轉錄本的平均長度為2 423 bp,比mRNA轉錄本的平均長度2 040 bp長383 bp,但根據圖2-B發現lncRNA峰值轉錄本長度小于mRNA。lncRNA平均外顯子數目為5.5個,mRNA平均外顯子為10.0個,但lncRNA與mRNA外顯子數目均集中于2~5個。統計分析ORF發現lncRNA平均ORF長度為255 bp,mRNA平均ORF長度為534 bp。
2.5 樣本相關性和差異性
使用PCA衡量樣本總體的相關性及差異性,剔除掉異常樣本。結果發現,在HRFI組中H1和H5明顯偏離總體水平,在LRFI組中L5偏離總體水平(圖3)。為消除組內誤差和增強結果可靠性,從HRFI中剔除掉H1和H5 2個樣本,從LRFI組中剔除掉L3和L5 2個樣本,剩余樣本進行后續分析。最后對剩余表型H2、H3、H4、L1、L2、L4對應重新分組命名為HRFI組:HRFI1、HRFI2、HRFI3;LRFI組:LRFI1、LRFI2、LRFI3。
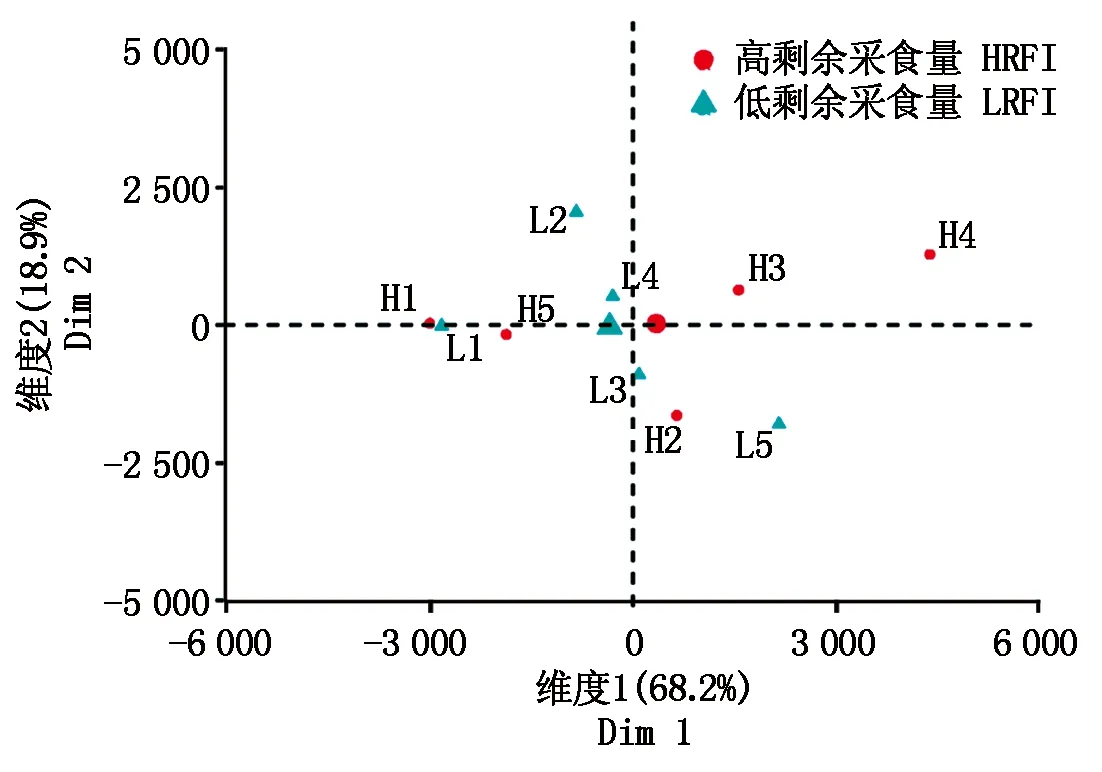
圖3 測序樣本PCA分析
2.6 差異lncRNA篩選及聚類分析
根據轉錄本表達量篩選RFI肉牛下丘腦差異lncRNA,在HRFI和LRFI組中共有105個顯著差異的lncRNA轉錄本,其中46個lncRNA在LRFI組下調,59個lncRNA上調(圖4-A),上下調中高表達lncRNA包括LNC_001285、LNC_007644、LNC_020193、LNC_013521等(表6)。
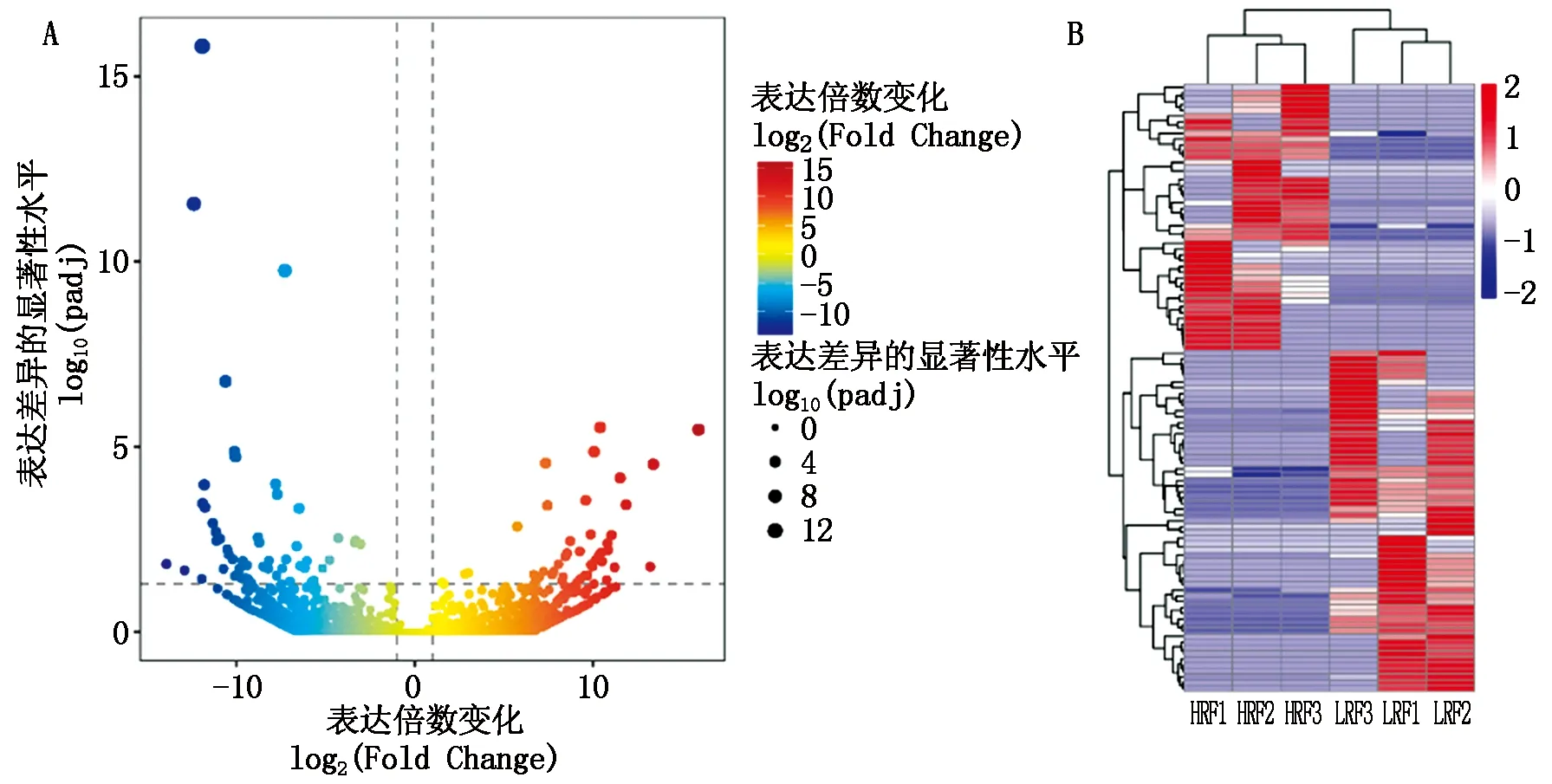
A.差異表達lncRNA火山圖;B.差異表達lncRNA聚類分析,每一列代表一個樣本,每一行代表一個差異lncRNA。
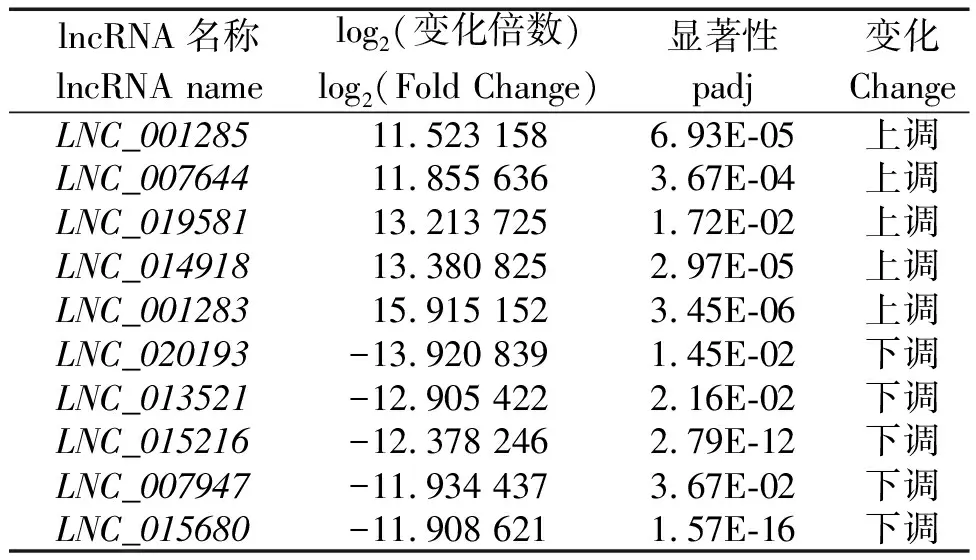
表6 差異上調或下調的lncRNA(Top5)
聚類分析用于判斷HRFI和LRFI組中lncRNA的表達模式,根據差異lncRNA的FPKM值為表達水平做層次聚類分析,將表達模式相同或者相近的lncRNA聚集成類(圖4-B)。聚類分析越接近的lncRNA,其表達規律越接近,表達模式相近的lncRNA可能有相同或者相關的功能。對差異lncRNA進行聚類分析表明,同一組樣本之間的差異很小,同一基因的表達水平在相同組內具有類似表達模式。
2.7 差異lncRNA與mRNA靶向關系
根據lncRNA與mRNA的位置關系及表達量預測差異lncRNA靶向關系。結果顯示,差異表達lncRNA共預測得到2 640個靶向mRNA,其中共表達預測到2 163個靶向mRNA,共定位預測到477個靶向mRNA。分析發現:顯著差異的lncRNA中LNC_007569及LNC_004079可能調節促腎上腺皮質激素釋放激素結合蛋白(Corticotropin releasing hormone-binding protein,CRH-BP),該蛋白是促腎上腺皮質激素釋放激素(Corticotropin releasing hormone,CRH)的重要調節劑,能夠抑制垂體分泌促腎上腺皮質激素(Adrenocorticotropic hormone,ACTH)[38]。同時表達變化最明顯的上調和下調的前5個lncRNA中,其中共有7個lncRNA顯著富集到靶向mRNA(圖5)。這些潛在調控關系為研究lncRNA調控基因表達進而影響RFI提供新的素材。
2.8 差異表達lncRNA靶基因富集分析及蛋白互作分析
本研究選取差異lncRNA預測共表達及共定位靶基因進行GO富集分析,將所有基因進行富集分析,共挑選顯著富集的前5條GO富集條目并展示相關靶基因(圖6-A),結果發現前5條GO條目中,有4條GO條目為細胞組分(CC):線粒體被膜(Mitochondrial envelope)、線粒體內膜(Mitochondrial inner membrane)、線粒體膜(Mitochondrial membrane)、含蛋白線粒體復合物(Mitochondrial protein-containing complex),一條生物過程(BP)的GO條目:TOR信號的正向調節(Positive regulation of TOR signaling)。這些GO條目表明,差異表達lncRNA靶基因mRNA主要與線粒體功能及TOR信號通路有關。

A.靶向mRNA的GO富集分析;B.靶向mRNA的KEGG富集分析。
將差異表達lncRNA靶基因進行KEGG通路富集分析,挑選富集最顯著的20個通路進行分析(圖6-B)。圖中橫坐標表示基因比率,縱坐標表示富集到的信號通路,氣泡大小表示富集到此信號通路的差異lncRNA靶基因的數量。在這些信號通路中發現生熱作用(Thermogenesis),氧化磷酸化(Oxidative phosphorylation)與動物的能量利用聯系密切,且分析發現,上述通路中相關基因(ATP酶、呼吸鏈復合物)主要與差異表達lncRNA(LNC_006637、LNC_007569、LNC_012079、LNC_005449)相關。蛋白互作分析共得到46個核心基因(Hub gene),有595條相互作用關系(圖7),主要為調控線粒體活性及電子呼吸鏈功能的線粒體核糖蛋白及泛醌氧化還原酶亞基等。例如MRPL12基因,該基因不僅是線粒體核糖體的關鍵成分,而且能夠增強線粒體DNA編碼RNA的能力[39]。NDUFA4、SDHD、UQCRH被提出在LRFI牛中表達量更高[40]。
2.9 差異表達lncRNA的qRT-PCR驗證
隨機選擇的8個差異表達的lncRNA進行qRT-PCR驗證(圖8)。結果表明,這些lncRNA驗證結果與測序結果表達趨勢一致,說明測序數據較為可靠,為差異lncRNA的后續功能驗證奠定基礎。
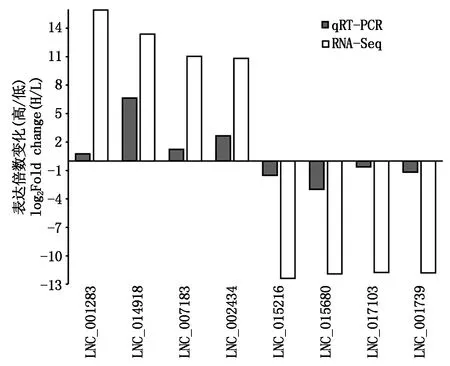
圖8 差異表達lncRNA的qRT-PCR驗證
3 結論與討論
肉牛生產成本一直是養殖業最關注的問題,而動物飼料效率是影響動物生長發育速率的關鍵因素,提高飼料效率是集約化肉牛生產最有效的方法之一。除了研發優質飼料,深入挖掘影響肉牛飼料效率的遺傳因素仍是育種工作者重要的科研方向之一。長鏈非編碼RNA通常缺乏蛋白編碼能力,通過轉錄和轉錄后調節蛋白質編碼基因的表達來發揮生物學調控功能[41],目前,lncRNA如何影響靶基因轉錄與翻譯水平,并影響肉牛剩余采食量的分子機制相對缺乏。因此,對其功能的研究仍然需要結合其他表達信息才能解釋。迄今為止,許多研究表明,非編碼RNA在下丘腦-垂體-腎上腺素軸[27]、脂質代謝[26]、神經元信號轉導[42]中起著重要的調節作用。如在豬脂肪組織中進行轉錄組分析確定了在高飼料效率和低飼料效率中17個差異表達的基因間長鏈非編碼RNA與RFI相關[43];在雞的肝組織中,應用RNA-Seq技術也發現了影響飼料效率的差異lncRNA[26]。這些研究表明,目前主要采用測序方法對不同組織差異lncRNA進行表征,從而為解釋家畜采食量差異的遺傳因素提供依據,但lncRNA的調節機制復雜,對其功能的探究需要結合其他調控分子才能闡明其調控作用原理。
在本研究中,在高、低RFI肉牛中發現了20 996個lncRNA,將得到的lncRNA與編碼基因比較發現,lncRNA具有更短的轉錄本、更少的外顯子數目、更短的ORF長度,這說明lncRNA編碼蛋白能力弱,符合lncRNA的表達特征[44]。在lncRNA差異表達分析中,共鑒定出差異表達lncRNA 105個,其中46個lncRNA在LRFI組下調,59個lncRNA上調。多數lncRNA不具備蛋白編碼能力,主要通過結合一種或多種蛋白來實現相應的調控機能[45]。lncRNA能夠與順式或反式靶基因互作調節生物表型變異,為了明確RFI和差異表達lncRNA之間的關系,本研究選擇差異表達lncRNA,預測其靶基因,其中預測到共表達靶向靶基因2 163個,共定位靶基因477個。對靶基因進行GO功能注釋和KEGG通路富集分析發現,GO條目主要富集到線粒體相關細胞組分,在線粒體被膜、線粒體內膜、線粒體膜等被顯著富集。在KEGG富集信號通路中,相關通路主要富集于與能量代謝有關,包括生熱作用及氧化磷酸化信號通路,這與GO條目主要富集在線粒體功能上相對應。線粒體功能及產熱效率方面的研究已經被證明其與家畜的生產性能及飼料效率表型差異有關[46]。富集在這些通路上的靶基因主要是ATP酶基因(ATP5F1B、ATP5IF1、ATP5MC2、ATP5MF、ATP5PO等)和呼吸鏈復合物(COXA3、COX7A1、NDUFB7、NDUFS3等),而這些靶基因主要由差異表達lncRNALNC_006637、LNC_007569、LNC_012079、LNC_005449調節。下丘腦是協調信號傳輸的重要中樞系統,能夠調節機體能量穩態及形成厭食和飽食信號調控采食量。根據前人關于RFI研究,本試驗進一步在下丘腦差異lncRNA靶向關系分析發現顯著差異的lncRNA中LNC_007569及LNC_004079可能調節促腎上腺皮質激素釋放激素結合蛋白,該蛋白是促腎上腺皮質激素釋放激素的重要調節劑,能夠抑制垂體分泌促腎上腺皮質激素[38]。研究表明,下丘腦-垂體-腎上腺素(Hypothalamic-pituitary-adrenal,HPA)軸是影響RFI變異的重要因素之一[47],其通過調控動物的應激反應對動物的采食量及熱量消耗產生影響;HPA軸激活會導致腎上腺皮質分泌皮質醇,參與分解代謝,產生葡萄糖,激活免疫系統并降低食欲[48]。線粒體產生大約90%的細胞能量,大量存在于代謝活躍的細胞,如肝、腎、肌肉和腦細胞。線粒體功能及產熱效率方面的變化已經被證明與許多畜牧業動物的生產速率和飼料效率的表型差異有關[46,49],其主要通過氧化磷酸化過程產生能量。氧化磷酸化在新陳代謝中起著核心作用,它將呼吸作用與ATP的產生聯系起來,但是這種偶聯不是完全緊密的,偶爾質子可以回到線粒體基質中產生熱量而不是ATP,這將降低氧化磷酸化的效率[50]。先前研究表明,LRFI動物的線粒體呼吸的速率增加[51],電子傳遞鏈的偶聯得到提升[47],呼吸鏈復合物(Ⅰ~Ⅴ)的活性更高[52],每千克代謝體質量 (BMW,Metabolic body weight)的產熱量更低[2]。另外,電子傳遞鏈也被公認為ROS(Reactive oxygen species)的產生部位,升高的ROS通過增加各種細胞成分氧化損傷的敏感性,對細胞抗氧化防御系統構成嚴重威脅。在家禽的其他研究中發現高飼料效率的動物中,其氧化應激現象也相應較低[46,52-53]。在富集到的靶基因中,細胞色素c氧化酶(COX Ⅱ;復合物Ⅳ)在調節細胞能量產生過程中起著關鍵作用,催化電子從還原的細胞色素c轉移到分子氧中,并參與質子的跨膜轉運[54]。Kelly等[10]提出在LRFI動物中COX Ⅱ的轉錄豐度更高;Kong等[55]發現在高飼料效率肉牛中線粒體基因表達量更高及線粒體基因拷貝數更低,這也說明了LRFI肉牛線粒體活性可能更強、轉錄率更高。此外,本研究GO富集到TOR信號的正向調節(Positive regulation of TOR signaling),Cota等[56]提出mTOR(Mammalian TOR)蛋白能夠通過感知能量狀態的變化來調節細胞的生長周期,也證明了mTOR信號在響應營養供應、調節能量平衡的大腦機制中發揮重要作用,這也可以作為后續研究依據進行分析。
挖掘影響動物采食效率的生物機制對集約化畜牧業發展意義重大,飼料效率的提高不僅可以提高生產效益,還可以減少肉牛生產過程中產生的環境污染。RFI作為飼料效率研究的主要經濟性狀,對家畜RFI的遺傳調控研究將為深入解析家畜RFI變異提供堅實基礎,也為節糧型家畜的培育提供依據。
本研究表征了不同RFI肉牛下丘腦組織中lncRNA轉錄組表達譜,差異lncRNA及其主要參與生熱作用和氧化磷酸化信號通路對線粒體功能調節產生重要作用,這對于理解下丘腦神經中樞對家畜采食行為的調控提供了參考依據,而這一調節過程是否通過HPA軸發揮作用,仍然需要細胞及分子試驗才能明確。

