日本囊對蝦群體遺傳多樣性的線粒體序列分析
孟繁星,徐田軍,王日昕
(浙江海洋學院海洋科學學院,海洋生物資源與分子工程實驗室,浙江舟山 316004)
日本囊對蝦Marsupenaeus japonicus屬甲殼綱、十足目、游泳亞目、對蝦科、囊對蝦屬,從紅海、非洲東部到朝鮮、日本一帶沿海都有分布,在我國東南沿海也有分布[1]。自20世紀90年代我國北方養殖的中國明對蝦Fenneropenaeus chinensis發生白斑綜合癥病毒(WSSV)病后,日本囊對蝦因肉質鮮嫩,營養豐富,耐低溫、耐干能力強,逐漸成為我國的主要養殖品種[2]。日本囊對蝦已成為黃渤海沿岸的重點養殖對象,并成功地開展了大規模的人工增殖放流,獲得較高的回捕率[3,4]。許多研究表明遺傳變異水平與生物的生長速度、抗病能力等生產性狀密切相關[5],隨著日本囊對蝦養殖面積的擴大和養殖苗種的推廣,其遺傳多樣性和遺傳結構必定會受到影響。因此,對我國現有的各養殖場的日本囊對蝦進行科學的遺傳評估,分析其遺傳背景,從而選出優良的品種對促進我國淡水養蝦業的可持續發展具有重要意義。
動物線粒體DNA(mtDNA)由于具有分子量小、結構簡單、母性遺傳、進化速度快等特征而被作為一種優良的分子標記廣泛應用于動物的群體遺傳學和系統進化研究中[6]。動物線粒體基因組中含細胞色素氧化酶(cytochrome c oxidase)三個亞基基因(COI,COII,COⅢ)、Cytb、ATPase6、ATPase8等 13個結構蛋白基因,它們都是線粒體內膜呼吸鏈的重要組分,是研究遠緣物種分子系統演化和分類的有效基因[7]。本研究以浙江、福建、臺灣的日本囊對蝦為研究對象,對其線粒體COⅢ基因進行了分析,旨為進一步研究日本囊對蝦分子系統學及其種群遺傳學和自然種質資源保護提供理論依據。
1 材料與方法
1.1 實驗材料
實驗所用的日本囊對蝦3個群體的樣本分別采于我國東南沿海的浙江(ZJ)、福建(FJ)、臺灣(TW)沿海,新鮮樣品低溫冷凍后長期保存。
1.2 實驗方法
1.2.1 基因組 DNA 的提取
采取常規苯酚氯仿提取法,并略有改進,每個樣本取100 mg左右的尾部肌肉,剪碎后,加入500 μL組織勻漿緩沖液 (10 mmol/L Tris-HCl,pH 8.0;100 mmol/L EDTA,pH 8.0),混勻后加入終濃度為 1%的SDS和100 μg/mL的蛋白酶K,55℃消化3 h,然后分別用等體積的 Tris飽和酚、Tris飽和酚∶氯仿∶異戊醇(25∶24∶1)、氯仿∶異戊醇(24∶1)各抽提1次。無水乙醇沉淀,70%乙醇洗滌2次,TE溶解。 紫外分光光度計測定樣品DNA溶液的濃度和純度,4℃保存備用。
1.2.2 PCR 擴增和產物鑒定
用于PCR擴增的引物分別為4750F:GACACCACGCCTTCCACCTA和5523R:GCAGCAGCTTCAAATCCAAA,引物由上海生工生物技術有限公司合成。反應在Bio-Rad Mycycler型PCR儀上進行。反應體系為 50 μl,其中含 10×Buffer 5 μL,dNTP 各 0.2 mmol/L ,上、下游引物各 0.2 μmol/L,模板 DNA 50~100 ng,Taq plus DNA聚合酶2 U(Tiangen),MgCl21.5 mmol/L。反應條件如下:94℃預變性3 min;94℃變性1 min,52℃退火1 min,72℃延伸1 min,35個循環;72℃延伸10 min。每次反應都設不含模板的空白對照。PCR擴增產物經1.5%瓊脂糖凝膠電泳檢測,電泳緩沖液為0.5×TBE(pH 8.0),電壓為5 V/cm,常溫電泳,EB染色,用Bio-Rad GD2000型凝膠成像系統拍照記錄。
1.2.3 DNA 序列測定
將PCR產物送往上海生工生物技術有限公司,純化后直接進行測序反應,測序引物為4750F:GACACCACGCCTTCCACCTA。
1.2.4 序列分析
獲得的序列經人工校正后在NCBI中進行同源序列BLAST,確認其為日本囊對蝦COⅢ基因的部分DNA序列,并從GenBank數據庫中下載了加州美對蝦(Farfantepenaeus californiensis,EU497054)、斑節對蝦(Penaeus monodon,AF217843)、凡納濱對蝦(Litopenaeus vannamei,NC_009626)、細角濱對蝦(Litopenaeus stylirostris,EU517503)、中國明對蝦(Fenneropenaeus chinensis,DQ518969)等 5 種對蝦的 COⅢ 基因的DNA序列用于系統發生關系的分析。利用MEGA4.0軟件分析DNA序列,計算堿基組成比例和遺傳距離并構建系統進化樹;利用DnaSP4.0軟件進行多態性指標。
2 結果
2.1 日本囊對蝦COⅢ序列特征及突變分析
對所有測得的日本囊對蝦3群體的COⅢ序列進行比對后,共得到了577 bp用于進行分析的基因序列,與GenBank數據庫中的序列對比后發現與對蝦科的其他蝦類具有很高的相似性,適合進行分析。堿基組成見表1,T、C、A、G四種堿基的含量分別為27.2%、16.5%、33.8%、22.6%,在所有試驗個體中沒有明顯差異,A+T平均含量為61%,明顯高于G+C的39%。在所測序列中共有51個變異位點,占位點總數的8.8%,共有簡約信息位點18個,占位點總數的3.1%,無插入或缺失位點,轉換與顛換比值為4.6;臺灣群體中包含變異位點11個,簡約信息位點7個,浙江群體中包含變異位點25個,簡約信息位點5個,福建群體中包含變異位點29個,簡約信息位點3個。這3個群體的平均核苷酸變異數為4.537。
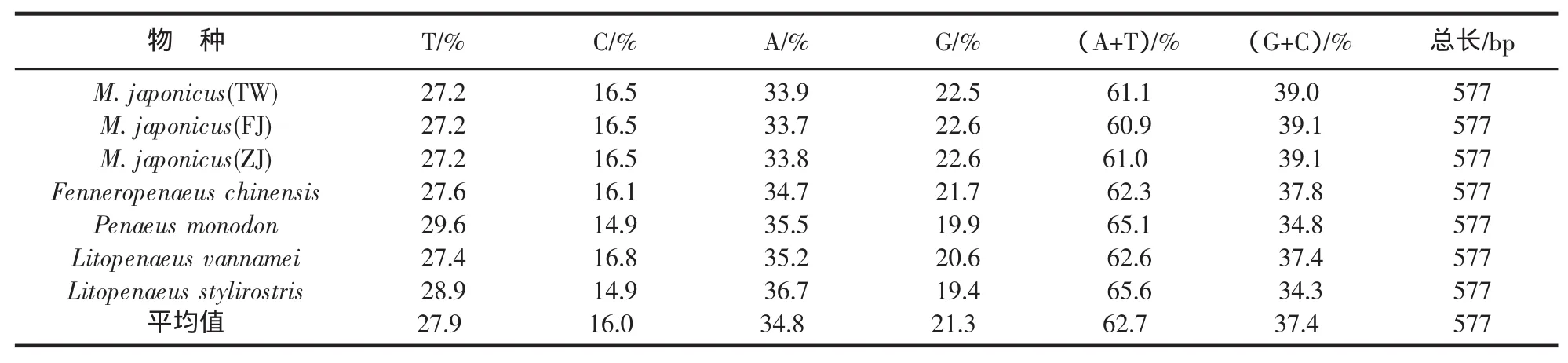
表1 5種對蝦mt DNA COⅢ基因部分序列的堿基組成Tab.1 Base composition in partial mtDNA COⅢgene sequences of five species of penaeidae
從51個COⅢ堿基序列變異位點中,共定義了31個單倍型,用H1~H31表示,單倍型及其位點見表2。臺灣、福建、浙江群體中分別有9、13、11個單倍型。日本囊對蝦3個群體的遺傳多樣性參數見表3,福建群體的單倍型多樣性和核苷酸多樣性均最高,分別為0.989和0.009 2;而臺灣群體則最低,分別為0.964和0.005 8,浙江群體居中,分別為0.989和0.007 8。

表2 日本囊對蝦COⅢ序列核苷酸多態位點及其各單倍型Tab.2 Nucleotide polymorphic sites and haplotypes in COⅢgene sequences of M.japonicus

表3 日本囊對蝦3個群體的遺傳多樣性參數Tab.3 Parameters summary of genetic diversity of M.japonicus from the three stocks
2.2 日本囊對蝦的遺傳多樣性分析
基于所測得的3種日本囊對蝦和對蝦科中另外5種對蝦的COⅢ同源序列,采用Neighbor-Joining法[8]構建系統發生樹,用Kimura雙參數法(Kimura 2-parameter)計算遺傳距離,用MEGA4.0軟件[9]進行系統發生的分析。發現日本囊對蝦的三個群體先聚成一小枝,再與中國明對蝦聚成一大枝;凡納濱對蝦和細角濱對蝦聚成一小枝,再分別于加州美對蝦和斑節對蝦聚成另一大枝。
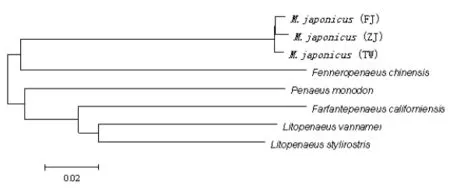
圖1 NJ法構建的6種對蝦的分子系統樹Fig.1 Molecular phylogenetic tree of six species of penaeidae shrimp
3 討論
ZARDOYA等[10]分析了脊椎動物mtDNA基因上13個蛋白編碼基因包含的系統發育信息,不考慮數據處理和加權的方法,將13個基因分為好、中、差3組。好的一組基因含有良好的系統發育信息,它們是:ND4、ND5、ND2、Cytb 和 CO I;中等的一組基因含有一定的系統發育信息,它們是:COII、COⅢ 、ND1 和ND6;差的一組基因不能很好地提供系統發育信息,它們是:ATPase6、ND3、ATPase8和ND4L。而日本學者MIYA等[11]對已知分類關系的8種硬骨魚類的線粒體不同蛋白質編碼基因所含有的系統發育信息進行了研究后表明,13個蛋白編碼基因含有的系統發育信息明顯不同,大致分為非常好、好、中等、差和非常差5個類別。 非常好的一組包括ND5、ND4、COⅢ 和COI,好的一組包括COII和Cytb,中等的一組包括ND3和 ND2,差的一組包括ND1和ATPase 6,非常差的一組包括ND4L、ND6和 ATPase 8。這與ZARDOYA的觀點有所差異,MIYA認為觀察的普遍性還不能確認,需要進一步研究。一些研究學者認為,對屬內不同種、種內不同亞種或不同地理型之間的物種鑒定,COⅢ基因不失為一種較為有效的分子標記[12-14]。
本研究測得了日本囊對蝦COⅢ基因的部分堿基序列,其中AT含量為61%,明顯高于GC含量,這和在其他甲殼類中觀察到的mtDNA的堿基分布情況相似[15-18]。序列的堿基組成也符合AT含量高,堿基G的相對缺乏是動物mtDNA堿基組成的特點。在線粒體中,變異位點的轉換較易在近親種間頻繁地發生,而顛換在較遠緣種間逐漸明顯;在同一物種中,轉換往往在數量上遠超過顛換[19]。本研究中日本囊對蝦mtDNA COⅢ堿基序列轉換與顛換比為4.6,這與核苷酸的替換主要以轉換為主,轉換多于顛換,表現較高的轉換偏向[20]的規律相符。這也說明本文所研究的3個日本囊對蝦群體線粒體的變異仍然是種內變異。
用NJ法構建的系統樹顯示日本囊對蝦的3個群體先聚成一小枝,再與中國明對蝦聚成一大枝;凡納濱對蝦和細角濱對蝦聚成一小枝,再分別于加州美對蝦和斑節對蝦聚成另一大枝。目前對于對蝦科種類的mtDNA COⅢ基因的研究較少,本次實驗得到了日本囊對蝦mtDNA COⅢ基因序列,雖然發現這3個群體間存在一定的差異,但是由于變異位點較少,不足以準確地區分日本囊對蝦這3個群體的遺傳特性。本研究接下來會對線粒體其他變異較大的基因(如COI、Cytb、D-loop)進一步分析,以得到更多的信息,從而更全面、更客觀地了解日本囊對蝦的遺傳背景。
[1]劉瑞玉,鐘振如.南海對蝦類[M].北京:農業出版社,1988:184-187.
[2]劉 萍,孟憲紅,莊志猛,等.3種對蝦種間RAPD遺傳標記[J].海洋水產研究,2002,23(2):1-6.
[3]朱金聲,莊志猛,鄧景耀,等.萊州灣日本對蝦放流移植的研究[J].中國水產科學,1998,5(1):56-61.
[4]蘇振明,王克行,張存義,等.黃海增殖日本對蝦的生長特性[J].水產學報,1996,20(1):25-29.
[5]GARTON D W,KOEHN R K,SCOTT T M.Multiple locus heterozygosity and physiological energetics of growth in the coot clam,Muluva laterals,from a natural population[J].Genetics,108:445-455.
[6]KAPPNER I,BIELER R.Phylogeny of venus clams(Bivalvia:Venerinae)as inferred from nuclear and mitochondrial gene sequences[J].Molecular Phylogenetics and Evolution,2006,40:317-331.
[7]MEYER A.Evolution of mitochondrial DNA in fishes[M]//MOCHACHKA P K,MOMMSEN T P,eds.Biochemistry and Molecular Biology of Fishes.Amsterdam:Elsevier,1993:1-38.
[8]SAITOU N,NEI M.The neighbor-joining method:A new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution,4:406-425.
[9]TAMURA K,DUDLEY J,NEI M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(18):1 596-1 599.
[10]ZARDOYA R,MEYER A.Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates[J].Molecular Biological Evolution,1996,13(7):933-942.
[11]MIYA M,NISHIDA M.Use of mitogenomic information in teleostean molecular phylogenetics:a tree-based exploration under the maximum-parsimony optimality criterion[J].Molecular Phylogenetic Evolution,2000,17:437-455.
[12]魏兆軍,趙巧玲,張志芳,等.蓖麻蠶線粒體基因組細胞色素氧化酶亞基Ⅲ的序列及其分子進化分[J].昆蟲學報,2002,45(2):193-197.
[13]邵愛華,朱 江,史全良,等.暗紋東方鲀線粒體COⅢ克隆及序列分析[J].水產科學,2006,25(8):391-396.
[14]陳姝君,赫崇波,木云雷,等.硬骨魚類線粒體基因系統發育信息效率分析[J].中國水產科學,2007,14(5):12-21.
[15]高天翔,李 健,王清印,等.中國對蝦線粒體16S rRNA基因序列分析[J].中國水產科學,2003,10(5):359-364.
[16]QUAN J X,LU X M,ZHUANG Z M,et al.Low genetic variation of Penaeus chinensis as revealed by mitochondrial COI and 16S rRNA gene sequences[J].Biochemical Genetics,2001,39:279-284.
[17]郭天慧,孔曉瑜,陳四清,等.三疣梭子蟹線粒體DNA 16S rRNA和COⅠ基因片段序列的比較研究 [J].中國海洋大學學報:自然科學版,2004,34(1):230-236.
[18]LIU M Y,CAI Y X,TZENG C S.Molecular systematic of the freshwater prawn genus Macrobrachium Bate,1868 (Crustacea:Decapoda:Palaemonidae)inferred from mtDNA sequences,with emphasis on east asian species[J].Zool Stu,2007,46(3):272-289.
[19]KOCHER T D,THOMAS W K,MEYER A,et al.Dynamics of mitochondrial DNA evolution in animals:Amplification and sequencing with conserved primers[J].PNAS,1989,86:619-620.
[20]STEPHEN A K,DAWN S W.Phylogeography and systematics of the mud turtle,Kinosternon baurii[J].Copeia,2001,3:797-801.

