SLC30A9基因在乳腺癌組織中的差異表達及臨床意義
廖 燕 林書瀚 黎丹戎 利基林 唐東平 唐 凱 張力圖
SLC30A9(solute carrier family 30 member 9)又名GAC63,是一個與DNA修復相關的基因[1],其大部分功能未知。SLC30A9定位于4p13,在以往的研究中,大約50%膀胱癌被證實在4p13的4個區域存在雜合性缺失[2],在其它的人類腫瘤包括子宮頸癌[3]、胃腺癌[4]和 BRCA1突變的腫瘤中[5],通過掃描腫瘤組織染色體區域發現,4號染色體短臂存在頻繁的雜合性缺失,提示該區域的基因可能與腫瘤的發病有關。近年來發現,SLC30A9可以與β-catenin相互作用,影響Wnt信號通路的激活[6],而Wnt信號通路與多種腫瘤的發展相關[7]。我們從生物信息學分析得到SLC30A9在乳腺癌組織和正常乳腺組織中存在差異性表達,因此,本研究以RT-PCR技術探討SLC30A9在乳腺癌組織中的表達情況,為進一步分析SLC30A9與腫瘤發生的關系奠定基礎。
1 材料與方法
1.1 研究對象與標本保存
30例乳腺癌標本取自2004年12月至2005年6月在廣西醫科大學附屬腫瘤醫院住院的手術病例。患者均為女性,年齡30~74歲,中位47歲。其中浸潤性導管癌25例,低分化腺癌2例,乳頭狀癌1例,黏液腺癌1例,導管內癌1例。有淋巴結轉移19例,無淋巴結轉移11例。所有病例均經病理檢查確診,分別收集30例乳腺癌癌灶組織、30例癌旁組織和10例相應的正常乳腺黏膜組織(距離病灶5cm以上且無癌浸潤者)。期間還收集了20例乳腺良性病變組織。這些組織分別離體后,10min內投入液氮速凍,然后轉移至-80℃超低溫冰箱凍存備用。
1.2 主要試劑和儀器
Trizol總RNA抽提試劑由Invitrogen公司提供,逆轉錄試劑盒及Taq酶、RNAse-free水等試劑由Promega公司提供。引物設計采用Primer 5軟件設計,引物合成由上海博亞生物技術有限公司完成。儀器主要為美國PTC2000型多通道PCR儀、美國PE公司PX2型高性能PCR儀,美國Bio-Rad Gel DOC2000型凝膠成像分析系統。
1.3 RNA的提取
1.3.1 物品和試劑的特殊準備:將清潔的金屬器械和泡酸晾干的玻璃器皿放入烤箱,180℃以上高溫烘烤5h,以滅活RNA酶,不耐高溫的塑料和橡膠制品用配好的1‰DEPC液浸泡12h以上,撈起裝盒、打包,高壓蒸汽滅菌后烘干備用。所用的溶液均用RNAsefree DEPC水配制。
1.3.2 抽提總RNA的步驟 利用TROZOL一步法提取。
1.3.3 總RNA的鑒定 測定RNA的濃度和純度:取5μl RNA液加入195μl Rnase-free DEPC水稀釋40倍,用雙光束紫外分光光度儀(超薄石英杯)測定λ260nm和λ280nm的吸光度值(OD值),根據公式計算濃度CRNA(μg/ml)=OD260×40×40÷1 000,根據OD260/OD280比值反映RNA純度,所有樣本的OD260/OD280比值均在1.8~2.0之間,說明提出的RNA純度較高,符合標準。
總RNA的電泳鑒定:用1%瓊脂糖凝膠電泳1h,瓊脂糖凝膠用凝膠成像分析系統掃描,觀察若有28S、18S兩條帶出現,5S條帶很微弱,加樣孔內無明顯DNA殘留,說明所提取的總RNA符合要求。
1.3.4 逆轉錄-多聚酶鏈式反應(RT-PCR) 按照A3500型逆轉錄試劑盒(美國Promega公司)實驗操作步驟進行。在進行逆轉錄前先去除RNA中可能存在的痕量DNA。
1.3.5 多聚酶鏈式反應(PCR) 所用引物為:F-5'GCC AAT TGC ATG GGC CTT AGT TC 3'R-5'TGC CCT TAC TGA TCG GTC ATT CTC C 3'10μl反應體系。PCR反應條件為94℃變性30s、65℃復性30s、72℃延長1min,30次循環。
1.3.6 RT-PCR結果判定 1.6%的瓊脂糖凝膠電泳驗證,凝膠電泳成像系統成像,測算出相應的光密度比值(SLC30A9 mRNA與GAPDH mRNA之比)。凝膠電泳分析軟件(BIORAD公司Quantity one軟件)分析測定其光密度,各電泳條帶凈密度值按下列公式計算:凈密度值=(平均密度-背景密度)×面積。以所擴GAPDH密度作為內參照,以目標基因凈密度與GADPH凈密度的比值進行半定量比較。
1.4 統計學處理
用SPSS10.0 for windows軟件對數據進行統計學分析。計數、計量資料分別做t檢驗和χ2檢驗,P<0.05為差異有統計學意義。
2 結果
2.1 SLC30A9 mRNA的表達水平
全組90例乳腺組織樣品中均有SLC30A9 mRNA表達。在30例乳腺癌患者中,5例(4、9、10、12號和13號)患者的癌組織、癌旁組織及正常組織中均存在SLC30A9基因mRNA差異性表達,占16.7%(5/30)。4、9號和12號患者的癌組織中,SLC30A9的表達比相應的正常組織低,而10號和13號患者則表現出在癌組織中表達上調。SLC30A9 mRNA在乳腺癌組織、癌旁組織、正常乳腺組織和乳腺良性病變組織的表達情況。見圖1-3。
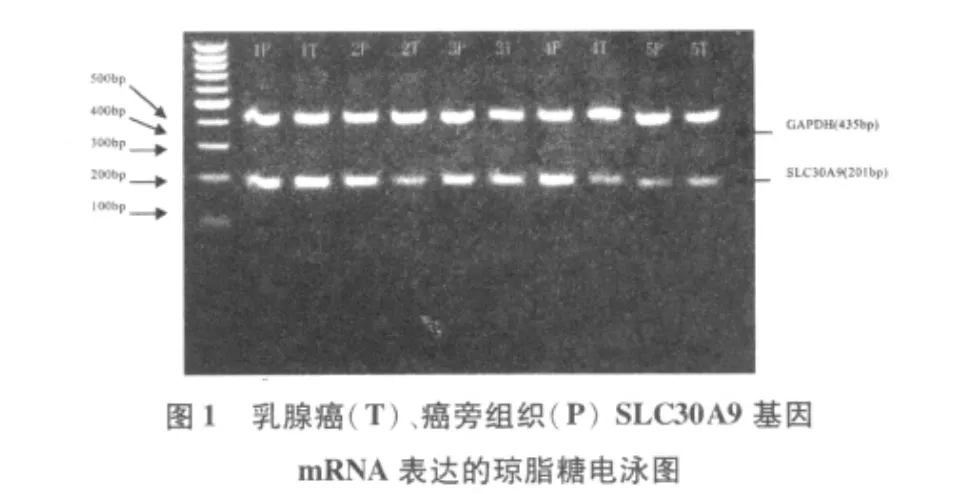


圖3 乳腺癌(T)、乳腺良性病變組織(B)SLC30A9基因mRNA表達的瓊脂糖電泳圖
從組間分析來看,SLC30A9基因mRNA在30例乳腺癌患者的癌組織、癌旁組織和10例正常乳腺組織及20例乳腺良性病變組織中均有表達。其平均光密度比值分別為0.443±0.247、0.427±0.253、0.405±0.209、0.547±0.190。上述4組分別兩兩比較,SLC30A9 mRNA表達水平的差異均無統計學意義(P>0.05)。見表1。
表1 乳腺癌組織、癌旁組織和正常乳腺組織SLC30A9 mRNA表達水平比較)

表1 乳腺癌組織、癌旁組織和正常乳腺組織SLC30A9 mRNA表達水平比較)
組織類型 n SLC30A9 mRNA P乳腺癌組織 30 0.443±0.247乳腺癌癌旁組織 30 0.427±0.253正常乳腺組織 10 0.405±0.209乳腺良性病變組織 20 0.547±0.190>0.05
2.2 乳腺癌組織SLC30A9 mRNA的表達與各臨床病理因素的關系
乳腺癌組織中SLC30A9 mRNA的平均光密度比值在不同年齡、臨床分期、病理類型、淋巴結轉移及遠處轉移的組間比較,差異均無統計學意義(P>0.05)。見表2。
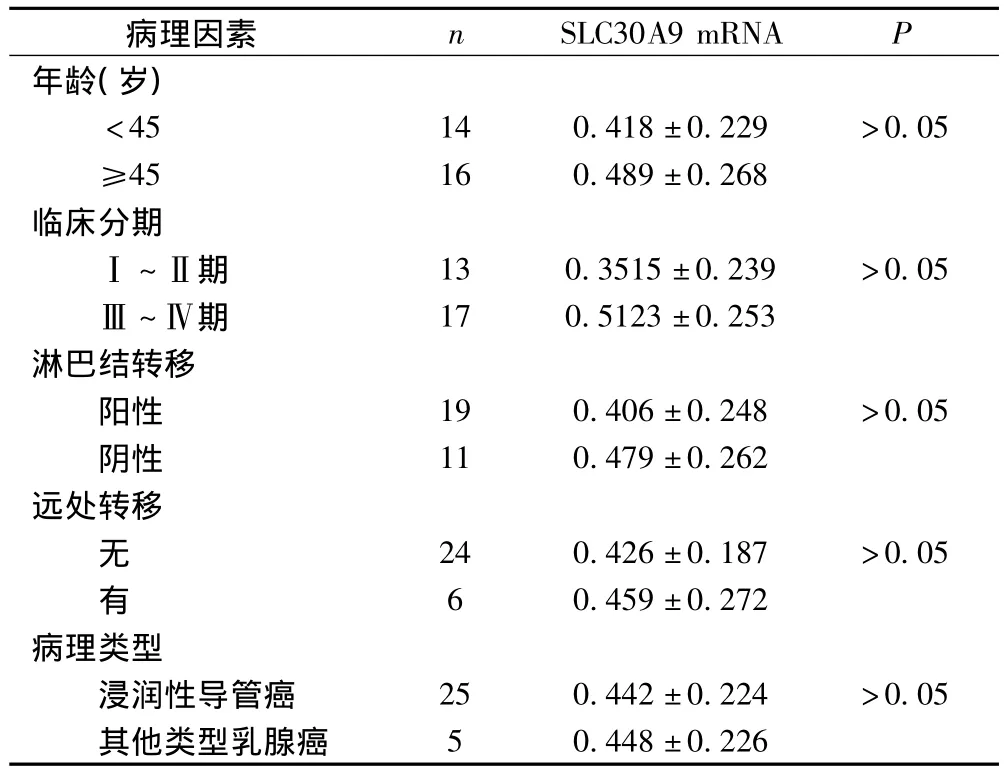
表2 SLC30A9 mRNA在癌組織中的表達與各臨床病理因素的關系
3 討論
本研究利用半定量RT-PCR法對30例乳腺癌的癌組織及其相應的癌旁組織、10例正常乳腺組織和20例乳腺良性病變組織進行SLC30A9 mRNA表達水平的檢測。結果發現SLC30A9基因mRNA在90例乳腺組織樣品中均有表達。對其平均光密度值進行比較,組間差異均無統計學意義(P>0.05)。即從整體組間的比較來看,沒有差異性表達,提示SLC30A9對乳腺癌的發生不是一個主要的因素。但研究發現,腫瘤的發生是多個基因相互作用的過程,大多數涉及的基因只起著部分作用。在突變掃描的研究中,往往只在大量的目的標本中發現極少部分的突變。因此,在篩選疾病基因的過程中我們更應注重個體的差異。在本實驗中,我們發現5例標本在癌組織、癌旁組織及正常組織中存在表達異常,占16.7%(5/30),其中,有3例癌組織SLC30A9的表達比相應的正常乳腺組織低,而其他2例則表現出在癌組織中表達上調。實驗結果提示在這些標本中存在有基因突變的可能,這有待于突變掃描的結果證實。也可能該基因的表達調控出現誤差,造成表達的不穩定。提示SLC30A9基因可能不是乳腺癌主導基因,但有可能起部分作用或與其他因素共同作用。
將30例乳腺癌患者按不同年齡(是否≥45歲)、臨床分期的早晚(Ⅰ/Ⅱ期或Ⅲ/Ⅳ期)、病理類型(浸潤性導管癌、黏液腺癌、低分化腺癌、乳頭狀癌、導管內癌)、有無淋巴結轉移和遠處轉移進行分組后,分別在不同組別間比較癌組織SLC30A9 mRNA表達水平,差異均無統計學意義。提示SLC30A9 mRNA與乳腺癌患者不同年齡、臨床分期、病理類型、有無淋巴結轉移和遠處轉移等無關。
由于我們發現在一些標本中SLC30A9的表達有上調,而一些標本中有下調,因此我們用生物信息學的方法分析基因的microRNA(miRNA)調控情況,發現在SLC30A9的3'端非翻譯區存在有miRNA的結合位點,這說明該基因可能受mRNA的調控。這提示我們需要做該基因的miRNA結合位點的掃描分析,觀察是否存在基因突變從而影響表達。目前發現,大約有30%的人類基因表達可以被miRNA調控,而miRNA的調控與腫瘤的發生相關[8~10]。有關方面值得深入研究。
[1] 張忠清.2001年美國癌癥統計報告介紹[J].中國腫瘤,2001,10(9)∶520-522.
[2] Walker RA,Jones JL,Chappell SA,et al.Molecular pathology of breast cancer and its applocation to clinical management[J].Cancer and Metastasis Reviews,1997,16(1)∶5-27.
[3] Strausberg RL,Feingold EA,Grouse LH,et al.Generation and initial analysis of more than 15 000 full-length human and mouse cDNA sequences[J].Proc Natl Acad Sci USA,2002,99(26)∶16899-16903.
[4] Sim DL,Chow VT.The novel human HUEL(C4orf1)gene maps to chromosome 4p12~p13 and encodes a nuclear protein containing the nuclear receptor interaction motif[J].Genomics,1999,59∶224-233.
[5] Sim DL,Yeo WM,Chow VT.The novel human HUEL(C4orf1)protein shares homology with the DNA-binding domain of the XPA DNA repair protein and displays nuclear translocation in a cell cycle-dependent manner[J].The International Journal of Biochemistry & Cell Biology,2002,34∶487-504.
[6] Chen YH,Yang CK,Xia M,et al.Role of GAC63 in transcriptional activation mediated by beta-catenin[J].Nucleic Acids Res,2007,35(6)∶2084-2092.
[7] 杜彥艷,劉 鑫,單保恩.Wnt/β-catenin信號轉導通路與腫瘤關系的研究進展[J].腫瘤,2009,29(8)∶803-805.
[8] Denli AM,Tops BB,Plasterk RH,et al.Processing of primary microRNAs by the microprocessor complex[J].Nature,2004,432∶231-235.
[9] Gregory RI,Yan KP,Amuthan G,et al.The Microprocessor complex mediates the genesis of microRNAs[J].Nature,2004,432∶235-240.
[10] Lund E,Guttinger S,Calado A,et al.Nuclear export of microRNA precursors[J].Science,2004,303∶95-98.

