番茄青枯病罹病植株和健康植株根際土壤細菌群落結構的初步分析
楊尚東 趙久成 郭伊娟 吳 俊 龍明華
(1 廣西大學農學院園藝系,廣西南寧 530004;2 廣西作物遺傳改良生物技術重點開放實驗室,廣西農業科學院,廣西南寧 530007)
番茄青枯病是一種典型的土傳病害,是由青枯雷爾氏菌(Ralstonia solanacearum)引起的細菌性維管束病害。國內外針對該病害的防治方法開展了大量的研究(Minuto et al.,2006;譚兆贊 等,2009),但至今仍未找到理想的防治措施。
生物防治是一項有望于預防和解決包括青枯病等土傳病害的技術,但傳統的生物防治技術將拮抗菌導入土壤后成功的例子很少。究其原因,就是因為接種的拮抗菌與土著微生物之間存在著激烈的競爭關系,拮抗菌進入土壤后,其生存競爭能力往往低于土著微生物而難以大量繁殖,從而難以發揮出應有的拮抗能力(郭堅華 等,1997)。已有的研究表明:當土壤微生物群落結構越豐富,物種越均勻,多樣性越高時,對抗病原菌的綜合能力越強(Nishiyama et al.,1999;陸合和張碧波,2009)。但番茄植株罹患青枯病是否與根際土壤微生物多樣性下降有關還鮮見報道。
本試驗采用聚合酶鏈式反應和變性梯度凝膠電泳(PCR-DGGE)技術,在比較分析番茄青枯病罹病植株和健康植株根際土壤理化性狀、生物學特征的基礎上(楊尚東 等,2013),進一步分析番茄青枯病罹病植株和健康植株根際微生物群落結構之間的差異,擬為預防和提高番茄青枯病生物防治技術的應用提供參考。
1 材料與方法
1.1 材料
供試番茄(Solanum lycopersicumL.)品種為中果型番茄品種0626。
2012年3月下旬在廣西武鳴縣太平鎮文溪村番茄基地隨機選取番茄健康植株和青枯病罹病植株各15株。青枯病病原菌及病株的鑒定與診斷由武鳴縣植保站技術服務部完成。其中,罹病植株分別選取重度萎蔫(4片葉以上至全株枯萎)和輕度萎蔫(1~2片葉萎蔫)的病株。挖出整個根系,采用抖根分離法(云鵬 等,2010)取根系所粘土壤,即為根際土壤。
1.2 儀器設備
變性梯度凝膠電泳(DGGE)所用儀器為DCodeTM突變檢測儀(Univertion Mutantion Detection System,Bio-Rad Pacific limited,USA)。凝膠成像儀和梯度PCR 儀均為美國Bio-Rad 生產。
1.3 測定項目
1.3.1 土壤基因組總DNA 的提取 參照Krsek 和Welingto(1999)的方法進行。
1.3.2 基因組總DNA 的純化 采用生工生物工程(上海)股份有限公司的DNA 膠回收試劑盒 (Biospin gel extraction kit,Bioflux,產 品號:bsc02m1)對DNA 粗提取液進行純化;并采用核酸蛋白測定儀(Eppendorf AG,德國)進行檢測。純化后樣品于-20℃冰箱保存備用。
1.3.3 土壤細菌16S rDNA基因V3可變區的擴增
16S rDNA基因V3可變區的擴增以及PCR 反應分別參照Li 等(2008)和劉瑋等(2010)的方法進行。其中,特異性引物對F338GC 和R518 的序列分別為5′-CGCCCGCCGCGCGCGGCGGGCGGGG CGGGGGCACGGGGGGACTCCTACGGGAGGCAGC AG-3′和5′-ATTACCGCGGCTGCTGG-3′,擴增產物片段長約230 bp;50 μL PCR 反應體系:10×PCR buffer(加 MgCl2)5.0 μL,dNTPs(10 mmol·L-1)1.0 μL,上游引物(10 μmol·L-1)1.0 μL,下游引物(10 μmol·L-1)1.0 μL,TaqDNA 聚合酶(5 U·μL-1)0.5 μL,模 板DNA(50 ng·μL-1)1.0 μL,最后用ddH2O 補齊50 μL。PCR 反應按照van Hannen 等(1999)的方法進行,PCR 產物用1.5%(m/V)瓊脂糖凝膠電泳檢測。
1.3.4 PCR 反應產物的變性梯度 凝膠電泳分析采用DCodeTM基因突變檢測系統對擴增得到的PCR產物進行變性梯度凝膠電泳分析。其中,變性膠的制備參照Sambrook 等(2002)的方法進行;待膠完全凝固后,將膠板放入裝有1×TAE 電泳緩沖液的裝置中,在每個加樣孔中加入含有3∶1 的6×溴酚藍二甲苯氰溶液的PCR 樣品20~30 μL。電泳及染色參照Yu 和Morrison(2004)的方法進行。
1.4 數據處理
細菌群落結構的豐富度(Richness,S)測定采用數條帶的方法,多樣性指數(Shannon-Wiener index,H)和均勻度(Evenness,EH)的計算方法采用羅海峰等(2004)的方法。聚類分析和相似性系數分析采用Quantity one 軟件。
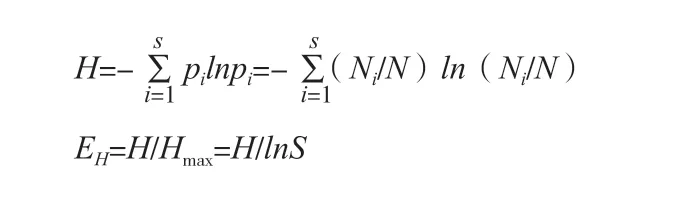
式中,Ni表示某一泳道中某一條帶的OD值,N是某一泳道中所有條帶OD值的總和,pi是某個土壤樣品中單一條帶的強度在該樣品所有條帶總強度中所占的比率;S是某個土壤樣品中所有條帶數目的總和。
2 結果與分析
2.1 根際土壤細菌總DNA 的提取及PCR 擴增
分別從番茄健康植株和青枯病罹病植株根際土壤中提取總DNA,取4 μL DNA 樣品,用1%瓊脂糖凝膠電泳檢測。從圖1可以看出,提取的總DNA亮度較好,而且無明顯拖帶現象,大小均為2 310 bp 左右。另外,在核酸蛋白測定儀上測定OD260和OD280的值,OD260/OD280值介于1.8~2.0 之間,說明所得到的總DNA 質量符合試驗要求(徐曉宇 等,2005)。
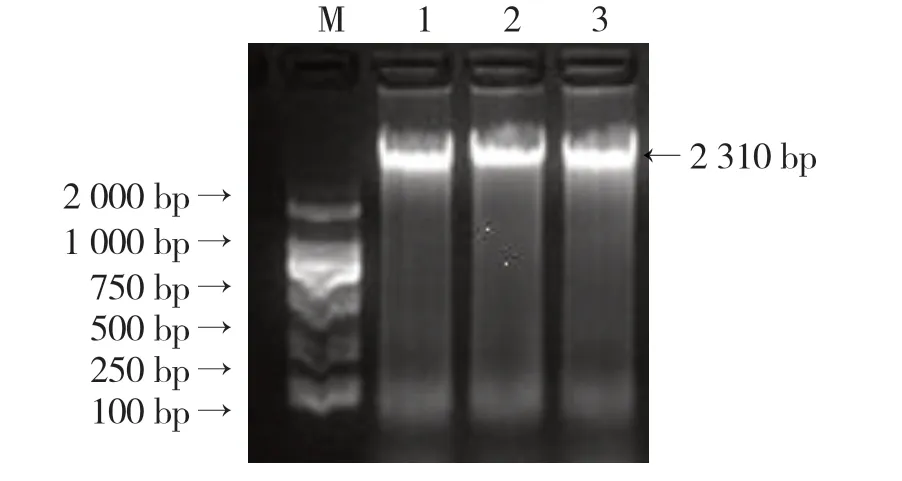
圖1 番茄青枯病罹病植株和健康植株根際土壤總DNA電泳圖譜
如圖2所示,番茄健康植株和青枯病罹病植株根際土壤細菌16S rDNA 經PCR 擴增后的DNA片段長度為250 bp 左右,特異性好、無雜帶,與理論值(230 bp)相符。說明該PCR 程序適用于16S rDNA 的擴增,并且能夠得到較好的產物。
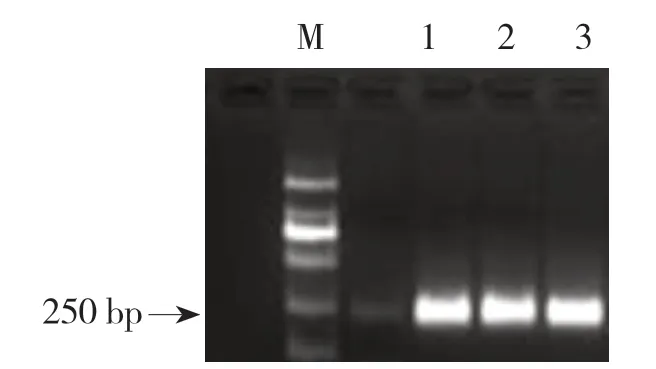
圖2 番茄青枯病罹病植株和健康植株根際土壤細菌16S rDNA基因V3可變區PCR 擴增圖譜
2.2 根際土壤細菌DNA 的PCR-DGGE 分析
應用DGGE 技術分離16S rDNA基因V3片段PCR 產物,可分離到數目不等、位置各異的電泳條帶(圖3)。根據DGGE 能分離長度相同而序列不同DNA 的原理,每一個條帶大致與群落中的一個優勢菌群或操作分類單元(operational taxonomic unit,OTU)相對應,條帶數越多,說明生物多樣性越豐富;條帶染色后的熒光強度越亮,表示該種屬的數目越多。從而反映土壤中的微生物種類和數量(Krsek & Welington,1999)。
采用凝膠成像分析系統對DGGE 圖譜進行分析,結果表明(圖3):與番茄健康植株相比,青枯病罹病植株根際土壤DGGE 圖譜中條帶的位置和數目均存在較大的差異;同時,輕度萎蔫和重度萎蔫的番茄青枯病罹病植株根際土壤的DGGE 條帶數目和位置亦存在較大差異。
從圖3還可以看出,番茄青枯病罹病植株與健康植株根際土壤細菌DGGE 圖譜的條帶數量大小順序為:健康植株(S為14)>青枯病罹病植株(輕度萎蔫)(S為10)>青枯病罹病植株(重度萎蔫)(S為7),表明番茄植株根際土壤細菌豐度以健康植株為最大,并隨著青枯病罹患程度的加重而降低,這可能與番茄植株罹患青枯病后根際土壤的理化性狀及生物學特性均隨著感病程度的加劇而下降有關。另一方面,DGGE 圖譜中各泳道的條帶粗細不一,對應其在DGGE 膠上的密度大小不同,密度大,則條帶比較粗黑;密度小,則條帶比較細。其中1、2、4、6、7、8、9、12、13、14、15號條帶是健康植株和罹病植株根際土壤共有的條帶,而5、10、11號條帶是健康植株根際土壤的特有條帶(圖3)。
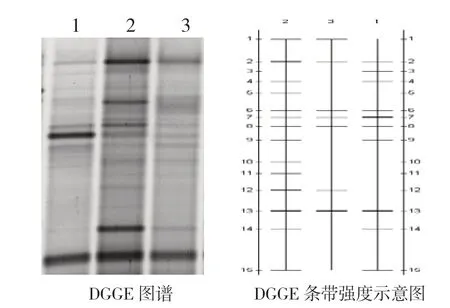
圖3 番茄青枯病罹病植株和健康植株根際土壤細菌的DGGE 圖譜和DGGE 條帶強度示意圖
根據細菌16S rDNA 的PCR-DGGE 圖譜中條帶的位置和亮度的數值化結果計算細菌群落結構指標Shannon-Wiener 指數。薛冬等(2007)的研究表明:Shannon-Wiener 指數越大,表明細菌群落多樣性越高。分析番茄青枯病罹病植株和健康植株根際土壤細菌的多樣性指數,結果表明(表1):番茄根際土壤細菌豐度及多樣性指數的大小順序為:健康植株>青枯病罹病植株(輕度萎蔫)>青枯病罹病植株(重度萎蔫)。表明番茄植株罹患青枯病并出現萎蔫癥狀后,根際土壤細菌豐度和多樣性指數下降,并隨著萎蔫程度的加劇豐度和多樣性指數下降更為明顯。
均勻度是表示物種在環境中的分布狀況,各物種數目越接近,均勻度數值越高(吳展才 等,2005)。從表1可以看出,番茄植株罹患青枯病后亦導致了根際土壤細菌均勻度的降低,但隨著萎蔫程度的加劇均勻度呈現回升的趨勢。表明番茄植株罹患青枯病后,導致部分種屬細菌數量變多,部分種屬細菌數量變少。
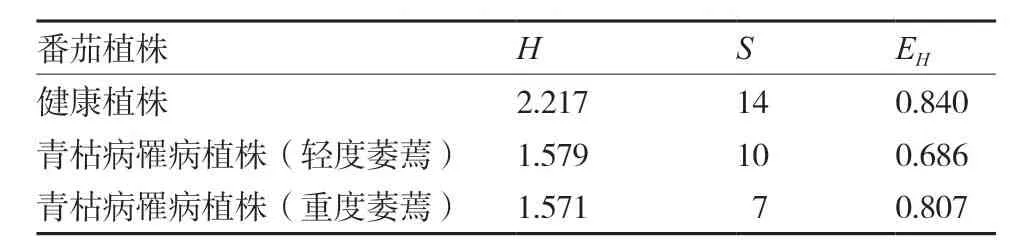
表1 番茄青枯病罹病植株和健康植株根際土壤細菌種群多樣性指數、豐度和均勻度
對所得DGGE 圖像采用非加權平均法(UPGMA)進行聚類分析。結果表明(圖4):番茄根際土壤細菌基本上分為兩大簇群,其中青枯病罹病植株分為一大簇群,健康植株分為另一大簇群。表明番茄植株罹患青枯病后明顯改變了根際土壤細菌的群落多樣性。
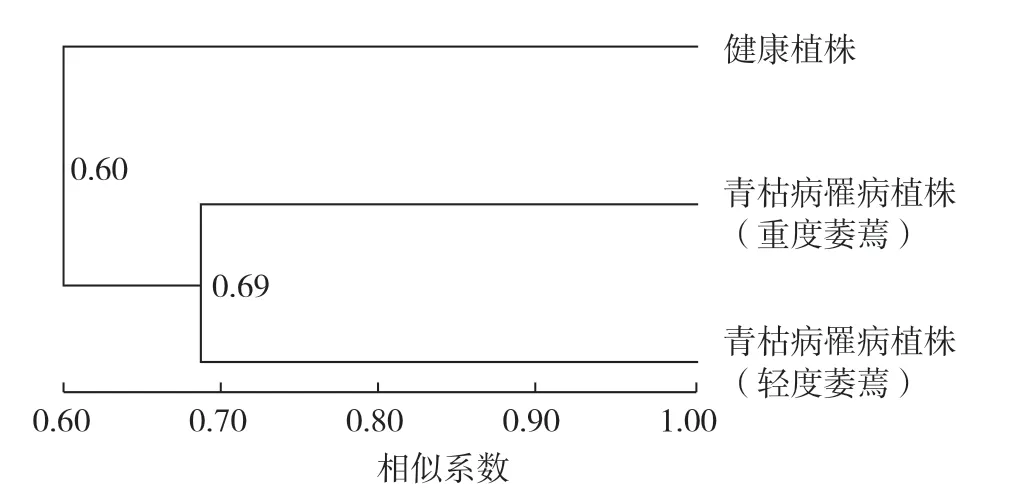
圖4 番茄青枯病罹病植株和健康植株根際土壤細菌16S rDNA 的聚類分析結果
對番茄青枯病罹病植株與健康植株根際土壤細菌群落結構進行相似性分析。結果顯示(表2),番茄青枯病罹病植株中輕度萎蔫植株、重度萎蔫植株與健康植株之間根際土壤細菌群落結構的相似性系數分別為55.5%和65.5%,而輕度萎蔫植株與重度萎蔫植株根際土壤細菌群落結構相似性系數為69.1%。表明番茄植株罹患青枯病后導致根際土壤細菌群落發生顯著變化。
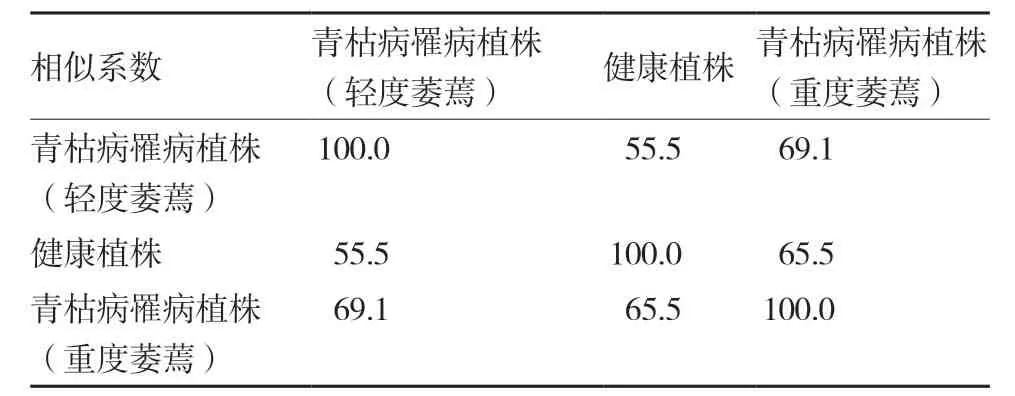
表2 番茄青枯病罹病植株和健康植株根際土壤細菌群落的相似性系數
3 結論與討論
土壤微生物是土壤生態系統變化的敏感指標之一,其活性和群落結構變化能反映出土壤生態系統的質量和健康狀況(鐘文輝和蔡祖聰,2004)。傳統研究土壤微生物群落結構變化的方法多采用培養法,但由于土壤中只有極小部分微生物是可培養的(Amann et al.,1995),因此采用傳統方法所獲得的土壤微生物群落結構信息是極其有限的。自從Muyzer 等(1993)首次將PCR-DGGE 技術應用于微生物生態學研究以來,近年來已有很多學者將其作為研究微生物群落結構動態變化的分子工具之一(Girvan et al.,2004;秦華 等,2010;顧美英 等,2012)。例如:張鵬等(2013)采用實時熒光定量RT-PCR、PCR-DGGE 技術研究生物有機肥對連作番茄根際土壤微生物區系及茄科雷爾氏菌的影響,結果表明施用生物有機肥提高了番茄和辣椒根際土壤微生物的多樣性指數、豐度和均勻度,其減輕青枯病為害的原因與調控根際土壤微生物的組成有關。此外,朱紅惠等(2003)研究發現,青枯病抗性不同的番茄品種根際拮抗菌存在差異,從抗病品種根際土壤中獲得的拮抗菌較多。張海利等 (2008)的研究亦指出,番茄青枯病發病嚴重的品種,其植株根際的拮抗菌群落多樣性低且拮抗能力較弱,輕微發病品種和未發病品種植株根際中篩選到的拮抗菌株數較多且拮抗能力較強。
本試驗采用PCR-DGGE 技術比較分析了番茄青枯病罹病植株和健康植株根際細菌群落結構的變化,同樣發現健康植株根際土壤細菌DGGE 圖譜的條帶數量(豐度)不僅明顯高于青枯病罹病植株,而且條帶獨立且亮度高于后者。這一結果與朱紅惠等(2003)和張海利等(2008)的研究結果相似,說明番茄罹病植株根際土壤細菌多樣性指數、豐度和均勻度下降或許就是導致植株出現青枯病為害癥狀的重要原因,而具有較高多樣性指數、豐度和均勻度的根際土壤可能也是健康植株尚未出現青枯病為害癥狀的重要原因之一。
顧美英,徐萬里,茆軍,張志東,唐光木,葛春輝.2012.新疆綠洲農田不同連作年限棉花根際土壤微生物群落多樣性.生態學報,32(10):3031-3040.
郭堅華,孫平華,吳云波,方中達.1997.植物細菌性青枯病的生物防治機制和途徑.中國生物防治,13(1):42-46.
劉瑋,張嘉超,鄧光華.2010.不同栽培時間三葉赤楠根際微生物多樣性及其PCR-DGGE 分析.植物研究,30(5):582-587.
陸合,張碧波.2009.有機肥防治生姜青枯病及其RAPD 分析.微生物學雜志,29(1):74-78.
羅海峰,齊鴻雁,張洪勛.2004.乙草胺對農田細菌多樣性的影 響.微生物學報,44(4):519-522.
秦華,李國棟,葉正錢,徐秋芳,曹志洪.2010.集約種植雷竹林土壤細菌群落結構的演變及其影響因素.應用生態學報,21(10):2645-2651.
譚兆贊,徐廣美,劉可星,廖宗文.2009.不同堆肥對番茄青枯病的防病效果及土壤微生物群落功能多樣性的影響.華南農業大學學報,30(2):10-14.
吳展才,余旭勝,徐源泰.2005.采用分子生物學技術分析不同施肥土壤中細菌多樣性.中國農業科學,38(12):2474-2480.
徐曉宇,閔航,劉和,王遠鵬.2005.土壤微生物總DNA 提取方法的比較.農業生物技術學報,13(3):377-381.
薛冬,姚槐應,黃昌勇.2007.茶園土壤微生物群落基因多樣性.應用生態學報,18(4):843-847.
楊尚東,吳俊,趙久成,郭伊娟,龍明華.2013.番茄青枯病罹病植株和健康植株根際土壤理化性狀及生物學特性的比較.中國蔬菜,(22):64-69.
云鵬,高翔,陳磊,盧昌艾,張金濤,劉榮樂,汪洪,劉國林.2010.冬小麥—夏玉米輪作體系中不同施氮水平對玉米生長及其根際土壤氮的影響.植物營養與肥料學報,16(3):567-574.
張海利,陳永兵,徐堅.2008.番茄青枯病生物防治研究進展.農業科技通訊,(8):98-101.
張鵬,韋中,朱震,高雪蓮,鄧開英,冉煒,沈其榮.2013.生物有機肥對連作番茄和辣椒根際土壤微生物區系及茄科雷爾氏菌的影響.南京農業大學學報,36(4):77-82.
鐘文輝,蔡祖聰.2004.土壤管理措施及環境因素對土壤微生物多樣性影響研究進展.生物多樣性,12(4):456-465.
朱紅惠,姚青,李浩華.2003.青枯病抗性不同的番茄品種根際拮抗菌拮抗能力差異研究.微生物學雜志,23(4):4-7.
Amann R I,Ludwig W,Schleifer K H.1995.Phylogenetic identification and in situ delection of individual microbial cells without cultivation.Microbiological Review,59:143-169.
Girvan M S,Bullimore J,Ball A S,Pretty J N,Osborn A M.2004.Responses of active bacterial and fungal communities in soils under winter wheat to different fertilizer and pesticide regimens.Applied and Environmental Microbiology,70(5):2692-2701.
Krsek M,Welington E M H.1999.Comparison of different methods for the isolation and purification of total community DNA from soil.Micrbiol Methods,39:1-16.
Li A J,Yang S F,Li X Y,Gu J D.2008.Microbial population dynamics during aerobic sludge granulation at different organic loading rates.Water Research,42(13):3552-3560.
Minuto A,Spadaro D,Garibaldi A,Gullino M L.2006.Control of soilborne pathogens of tomato using a commercial formulation ofStreptomyces griseoviridisand solarization.Crop Protection,25:468-475.
Muyzer G,Wall E C D,Uitterlinden A G.1993.Profiling of complex microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction amplified genes coding for 16S rDNA.Appl Environ Microbiol,59(3):695-700.
Nishiyama M,Yoshitaka S,Suzuki S.1999.Suppression of growth ofRalstonia solancearum,tomato bacterial wilt agent,on/in tomato seedling cultivated in a suppressive soil.Soil Sci Plant Nutr,45 (1):79-87.
Sambrook J,Fritsch E F,Maniatis T.2002.Molecular cloning:a laboratory manual.3rd.Beijing:Science Press.
Yu Z,Morrison M.2004.Comparison of different hypervariabl regions of ITS genes for use in fingerprint of microbial communities by PCRDenaturing gradient gel electrophoresis.Appl Environ Microbiol,70(4):4800-4806.
van Hannen E J,Zwart G,van Agterveld M P,Gons H J,Ebert J,Laanbroek H J.1999.Changes in bacterial and ewukaryotic community structure after mass lysis of filamentous cyanobacteria associated with virus.Appl Environ Microbiol,65:795-801.

