阿爾茨海默病的基因組學研究進展①
王美琴,楊鎧冰,冀燃,龐清華,張大保,2,張敏,2
阿爾茨海默病的基因組學研究進展①
王美琴1,楊鎧冰1,冀燃1,龐清華1,張大保1,2,張敏1,2
本文主要介紹遺傳因素參與阿爾茨海默病(AD)發病機制的研究進展,闡述近年來國內外研究較多的與AD發病機制相關的易感基因,通過對易感位點的進一步研究,尤其是通過旁路分析可以更好地認識遲發性阿爾茨海默病(LOAD)的發生、進展,從而發現重要的樞紐基因,為臨床預防、診斷和治療提供靶點。由于遺傳變異信息的分散性,需要借助詳細的文獻、網上搜索和一些生物信息學分析方法,歸納LOAD的致病基因和信號轉導通路。本文以3個常用數據庫為來源,采用多手段、多途徑的分析和挖掘數據方法,充分利用已有AD數據資源,為AD基因學的進一步研究和臨床治療方面的應用提供理論依據。
阿爾茨海默病;單核苷酸多肽性;通路;數據庫;綜述
[本文著錄格式]王美琴,楊鎧冰,冀燃,等.阿爾茨海默病的基因組學研究進展[J].中國康復理論與實踐,2015,21(12): 1365-1369.
CITED AS:Wang MQ,Yang KB,JiR,etal.Research progress of genetics of Alzheimer's disease(review)[J].Zhongguo Kangfu Lilun Yu Shijian,2015,21(12):1365-1369.
阿爾茨海默病(A lzheimer's disease,AD)是以進行性認知功能障礙和記憶損害為特征的原發性中樞神經系統退行性疾病[1]。臨床上以遲發性阿爾茨海默病(late onset-AD,LOAD)為主[2]。目前關于AD的發病機制尚不清楚,其可能的發病機制包括自由基學說、淀粉樣蛋白斑塊假說、神經原纖維纏結假說、炎性機制、膽堿能機制等,但都不能很好地解釋該病的全部病變。越來越多的證據表明基因變異參與AD的發病過程,AD相關基因的陸續發現為其基因治療帶來希望。由于AD相關基因在發病機理及診治上的重要性,闡明已知基因的作用與發現新的相關基因將是國際學術界的長期目標[3]。對于LOAD遺傳相關性的研究方式有兩種:候選基因模式[4]和高通量全基因組相關性研究[5]。近期高通量全基因組相關性研究已取代傳統的候選基因模式[6],該方法可同時檢測百萬以上的單核苷酸多肽性(single nucleotide polymorphism,SNP),使研究者可以全面、無偏倚地進行全基因組相關性分析。從實驗操作來看,通過SNP發現疾病相關基因突變要比通過家系來得容易;有些SNP并不直接導致疾病基因的表達,但由于它與某些疾病基因相鄰,而成為重要的標記[7];SNP在基礎研究中也發揮巨大的作用。因此,本文以3個常用數據庫為來源,采用多手段、多途徑的分析和挖掘數據方法,探尋LOAD與相關基因的關聯性,以期為AD基因學的進一步研究及臨床和治療方面的應用提供依據。
1 SNP數據庫
美國國立生物技術信息中心(NationalCenter for Biotechnology Information,NCBI)與人類基因組研究所(National Human Genome Research Institute,NHGRI)合作建立的SNP數據庫[8](Single-Nucleotide Polymorphism database,dbSNP,http://www. ncbi.nlm.nih.gov/projects/SNP/)是一個包含來自任何生物體的核苷酸序列數據庫。它是一個提交的核苷酸變異的開放數據庫,并且是NCBI搜索和檢索系統Entrez的一部分[9]。有兩種基本檢索方案:基于文本的和基于序列的搜索。結果展示格式為GeneView,可獲得特定基因所有提交的SNPs的信息。
由于現有遺傳變異信息也不是來自單一的數據庫,它需要的信息需要從多個數據庫中收集,甚至所有數據庫中仍無法獲得,這樣就需借助詳細的文獻、網上搜索或一些生物信息學分析方法。本研究通過dbSNP數據庫以及文獻信息數據挖掘技術,獲取到354個與AD相關的SNPs,見表1。所得數據與傳統的技術相比,由于其經過實驗的多次論證,更具說服力。
2 AD相關基因數據庫
高通量全基因組相關性研究應用于AD易感基因的發現,這些AD相關基因統一整理在一個公共信息數據庫即AlzGene數據庫[10](http://www.alzgene.org)。A lzGene數據庫收集、概括和Meta分析了所有關于AD遺傳學的相關研究,包括全基因組相關性研究,可以檢索到以英文發表于同類綜述類雜志可引用的文獻和研究[11];并從原文中提取關鍵詞,如家系、AD診斷類型、樣本量、發病年齡和基因型分類等;進行易感基因的隨機效應模型Meta分析[12]。這種方法綜合了來自不同人群或實驗室對同一位點多態性的研究,從而提供了一項概括不同研究之間AD患病風險的評價[13]。
根據A lzGene數據庫的最近更新顯示,目前研究與AD相關的基因有695個,基因多態性位點有2973個,研究組織有1395個,為AD的研究提供新的方向和研究熱點目標。表2列出了A lzGene數據庫中基因變異位點。

表1 dbSNP數據庫SNPs信息
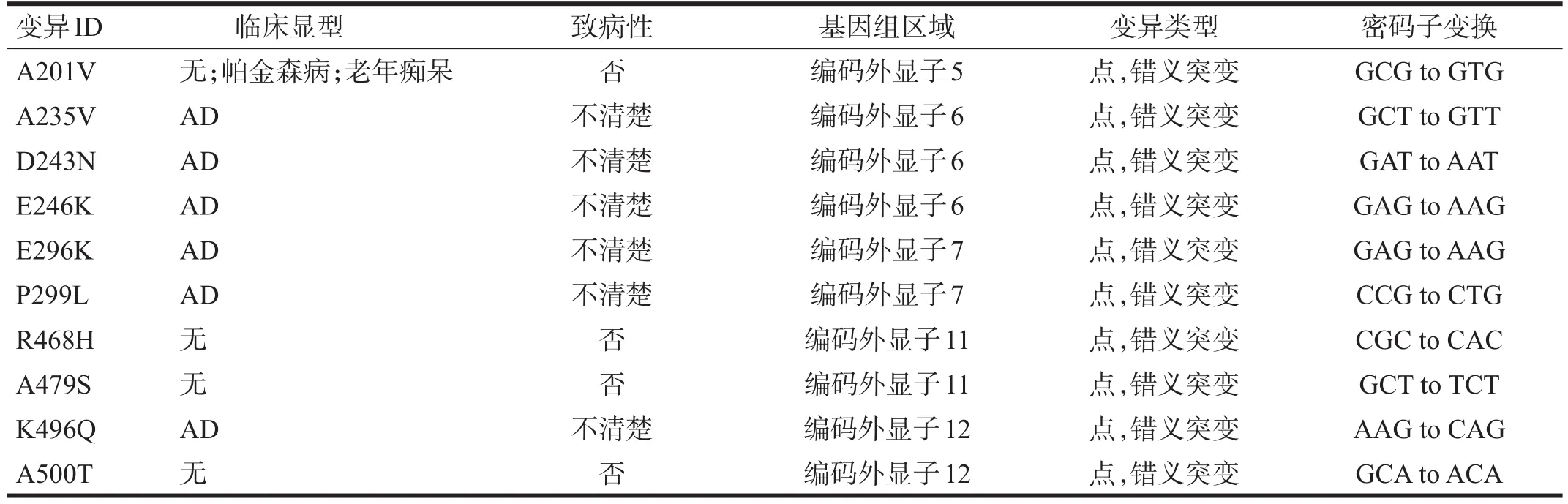
表2 AlzGene數據庫中給出的AD基因變異位點
2.1APOE基因
APOE編碼的蛋白與Aβ的沉積和清除有關[14],還參與突觸發生、Tau蛋白磷酸化、神經元死亡和脂質代謝。APOE編碼蛋白有3種同型異構體(ε2,ε3,ε4)。其中ε3在人群中最常見。研究發現,APOEε3/ε4攜帶者發生AD的相對危險度是APOEε3/ε3攜帶者的2~4倍,而APOEε4/ε4攜帶者發生AD的相對危險度是6~30倍[15]。APOEε4有明顯的年齡相關性,對于70歲之前發病的患者影響更明顯[16]。
2.2CLU基因
CLU基因編碼蛋白為聚集素,與Aβ以可逆方式特異性結合,使可溶性Aβ轉化為不可溶性Aβ,通過血腦屏障后將其清除[17]。聚集素與APOE具有互補作用[18]。最新研究發現,CLU基因變異引起對神經元起保護作用的髓鞘損傷[19]。在青少年時期神經元可以自我修復損傷的髓鞘、不會出現認知能力減退;隨著年齡增長,髓鞘自我修復能力減弱,認知能力逐漸減退,最終導致LOAD[20]。
2.3CR1基因
研究發現LOAD和免疫炎癥反應密切相關,而CR1作為補體系統的重要成分在全基因組關聯分析(genome-wide association study,GWAS)研究中已經被確認為LOAD的易感位點。其編碼蛋白可以將Aβ作為外源物質通過補體系統給予清除[21]。一項研究顯示,CR1與AD病理改變(淀粉樣斑塊沉積)、認知功能減退密切相關[22]。
2.4PICALM基因
PICALM編碼的蛋白是網格蛋白介導內吞作用的重要成分。它能夠募集網格蛋白和銜接蛋白AP-2到細胞膜[23],后者能識別目標蛋白。三者形成網格蛋白三角復合體,通過內吞作用進入細胞內。研究發現,PICALM可能在LOAD中參與Aβ的運輸和清除,但具體機制尚不清楚[24-25]。
2.5其他易感性相關基因
B1N1也是一個顯著的LOAD的易感位點,研究發現B1N1和動粒蛋白的細胞內吞相關;而另一個重要的易感基因PICALM也在細胞內吞中發揮作用。此外,CD33也是LOAD的易感位點[26],并且也得到了復制[27]。CD33在調節細胞內免疫應答中發揮作用,可以通過網格蛋白調控細胞內吞[28-29]。
根據AlzGene數據庫,LOAD的易感位點中APOE的相對危險度最高,其他主要的易感位點與之相比相對危險度差距很大[30]。此推論與大量的文獻報道相一致。
3 京都基因與基因組百科全書
該系統主要通過對基因功能和信號轉導通路的詮釋,有助于發現AD和其他神經變性疾病的發病機制,為鑒定復雜疾病的遺傳因素,理解慢性復雜疾病潛在的病因學、生物學、發病機制,以及提供恰當的基因學檢測和遺傳學咨詢提供幫助。
京都基因與基因組百科全書(Kyoto Encyclopedia of Genes and Genomes,KEGG,京都遺伝子ゲノム百科事典)是系統分析基因功能、基因組信息的數據庫,它有助于研究者把基因及表達信息作為一個整體網絡進行研究。基因組信息存儲在GENES數據庫里,包括完整和部分測序的基因組序列;更高級的功能信息存儲在通路(PATHWAY)數據庫里,包括圖解的細胞生化過程,如代謝、膜轉運、信號傳遞、細胞周期,還包括同系保守的子通路等信息[31]。KEGG提供的查詢整合代謝途徑完善,其中包括碳水化合物、核苷、氨基酸等的代謝及有機物的生物降解[32],不僅提供所有可能的代謝途徑,而且對催化各步反應的酶進行全面的注解,包含所有氨基酸序列、蛋白質資料銀行(protein databank,PDB)庫的鏈接等。KEGG是進行生物體內代謝分析、代謝網絡研究的強有力工具[33]。目前在PATHWAY數據庫中的代謝通路是已建立的最全面的網絡體系,每個參考代謝途徑是一個由酶或EC號(酶的識別符)組成的網絡。
如圖1所示,利用基因和表達信息可通過計算機構建出生物體特有的代謝通路:先根據基因的序列相似性和位置相關性確定基因組中酶的基因,然后合理地安排酶的識別符號,最后將基因組中的基因和參照通路中用酶的識別符號編號的基因產物結合起來,對可能的代謝途徑進行推測和驗證[34],為科研工作者提供指導作用。

圖1 map05010
4個主要功能蛋白包括:淀粉樣前體蛋白(APP)、presenilin 1(PS1)、presenilin 2(PS2)和載脂蛋白E(ApoE)。與APP和PS蛋白質相關的所有突變會導致β淀粉狀蛋白質多肽產物的增加,尤其是增多淀粉樣形式Aβ42。
4 分析與展望
遺傳變異的范圍極為廣泛,還有很多未知領域。目前,全基因組相關性研究已逐漸成為探索AD等復雜疾病的有效工具,代表未來研究慢性復雜性疾病病因學、生理病理學機制的方向標[35]。雖然許多生物學數據庫是在出現高通量數據后應運而生,但缺乏有效的數據分析方法。基因測序、基因組、SNP、基因芯片、小分子化合物和GWAS數據等數據庫信息的共享,促進了生物信息學中多手段、多途徑的分析和挖掘數據的飛躍發展。現有遺傳變異信息并不是來自單一的數據庫,需要的信息要從幾個數據庫中收集,甚至所有數據庫中仍無法獲得全部的信息,這樣就更加需要借助詳細的文獻、網上搜索和一些生物信息學分析方法挖掘資料獲得信息。
上述與發病機制和途徑有關的基因均需要更多的證據來證實它們作為AD重要標志的臨床實用性,揭示AD的神經病理學機制及其與發病機制間的密切關系。
總之,該領域的研究仍相對薄弱,有待于國內外學者的進一步闡述,以便為AD的早期診斷和基因治療提出新的方向。
[1]Sassi C,Guerreiro R,Gibbs R,et al.Investigating the role of rare coding variability in Mendelian dementia genes(APP, PSEN1,PSEN2,GRN,MAPT,and PRNP)in late-onset A lzheimer'sdisease[J].Neurobiol Aging,2014,35(12): 2881-2886.
[2]Floudas CS,Um N,Kamboh M I,etal.Identifying genetic interactions associated with late-onsetAlzheimer's disease[J].Bio-DataMin,2014,7(1):35-54.
[3]Zou Z,Liu C,Che C,etal.Clinicalgeneticsof Alzheimer'sdisease[J].Biomed Res Int,2014,2014(1):862-872.
[4]D'Aoust LN,CummingsAC,Laux R,etal.Examination of candidate exonic variants for association to Alzheimer disease intheAmish[J].PLoSOne,2015,10(2):43-59.
[5]Reitz C.Genetic loci associated with Alzheimer's disease[J]. Future Neurol,2014,9(2):119-122.
[6]Kauwe JS,Bailey MH,Ridge PG,etal.Genome-wide association study of CSF levels of 59 Alzheimer's disease candidate proteins:significantassociationswith proteins involved in amyloid processing and inflammation[J].PLoS Genet,2014,10 (10):e1004758.
[7]Nguyen TT,Huang J,Wu Q,et al.Genome-wide association data classification and SNPs selection using two-stage quality-based Random Forests[J].BMCGenom ics,2015,16(2):S5.
[8]NCBIResource Coordinators.Database resources of the national center for biotechnology information[J].Nucleic Acids Res, 2015,43(1):6-17.
[9]Bhagwat M.Searching NCBI's dbSNP database[J].Curr Protoc Bioinformatics,2010,32(1):19-47.
[10]Bertram L,McQueen MB,Mullin K,et al.Systematic meta-analyses of Alzheimer disease genetic association studies: theAlzGene database[J].NatGenet,2007,39(1):17-23.
[11]Olgiati P,Politis AM,Papadim itriou GN,et al.Genetics of late-onset Alzheimer's disease:update from the alzgene database and analysisof shared pathways[J].Int JAlzheimers Dis, 2011,10(1):4061-4075.
[12]Wang XB,CuiNH,Yang J,et al.Angiotensin-converting enzyme insertion/deletion polymorphism is notamajor determ ining factor in the development of sporadic A lzheimer disease: evidence from an updatedmeta-analysis[J].PLoSOne,2014,9 (10):406-419.
[13]Potkin SG,Turner JA,GuffantiG,etal.Genome-wide strategies for discovering genetic influences on cognition and cognitive disorders:methodological considerations[J].Cogn Neuropsychiatry,2009,14(4):391-418.
[14]LiX,Song D,Leng SX.Link between type 2 diabetesand Alzheimer's disease:from epidemiology tomechanism and treatment[J].Clin Interv Aging,2015,10(1):549-560.
[15]Lyall DM,Harris SE,Bastin ME,etal.Are APOE varepsilon genotype and TOMM 40 poly-T repeat length associationswith cognitive ageing mediated by brain whitematter tract integrity[J].Transl Psychiatry,2014,4(1):449-456.
[16]Manning EN,Barnes J,Cash DM,etal.APOE epsilon4 is associated with disproportionate progressive hippocampal atrophy in AD[J].PLoSOne,2014,9(5):608-616.
[17]DiBattista AM,Stevens BW,Rebeck GW,etal.Two A lzheimer's disease risk genes increase entorhinal cortex volume in young adults[J].FrontHum Neurosci,2014,8(1):779-786.
[18]Khoury MJ,Bertram L,Boffetta P,etal.Genome-wide association studies,field synopses,and the development of the know ledge base on genetic variation and human diseases[J]. Am JEpidemiol,2009,170(3):269-279.
[19]Sekar S,M cDonald J,Cuyugan L,et al.Alzheimer's disease is associated with altered expression of genes involved in immune response and mitochondrial processes in astrocytes[J]. NeurobiolAging,2015,36(2):583-591.
[20]Braskie MN.Common Alzheimer's disease risk variantwithin the CLU gene affects white matter microstructure in young adults[J].JNeurosci,2011,31(1):6764-6770.
[21]Sleegers K.The pursuit of susceptibility genes for Alzheimer's disease:progress and prospects[J].Trends Genet,2010, 2010(2):84-93.
[22]Rosenthal SL,Kamboh M I.Late-onset A lzheimer's disease genes and the potentially implicated pathways[J].Curr Genet Med Rep,2014,2:85-101.
[23]Moreau K,Flem ing A,Imarisio S,et al.PICALM modulates autophagy activity and tau accumulation[J].Nat Commun, 2014,5:4998.
[24]Bohm C,Chen F,Sevalle J,etal.Currentand future implications of basic and translational research on amyloid-beta peptide production and removal pathways[J].Mol Cell Neurosci, 2015,2(1):1-9.
[25]Baig S,Joseph SA,Tayler H,et al.Distribution and expression of picalm in Alzheimer disease[J].J Neuropathol Exp Neurol,2010,69(10):1071-1077.
[26]Hollingworth P,Harold D,Sims R,et al.Common variants at ABCA7,MS4A6A/MS4A4E,EPHA1,CD33 and CD2AP are associated with A lzheimer's disease[J].Nat Genet,2011,5(1): 429-435.
[27]Carrasquillo MM,Belbin O,Hunter TA,et al.Replication Of EPHA 1 and CD33 associationswith late-onsetA lzheimer's disease:amulti-centre case-control study[J].Mol Neurodegener, 2011,6(1):54.
[28]Malik M,Simpson JF,Parikh I,et al.CD33 A lzheimer's risk-altering polymorphism,CD33 expression,and exon 2 splicing[J].JNeurosci,2013,33(33):13320-13325.
[29]Beecham GW,Hamilton K,NajAC,etal.Genome-wideassociation meta-analysis of neuropathologic features of Alzheimer's disease and related dementias[J].PLoS Genet,2014,10 (9):606-621.
[30]張博,金錚,別黎,等.晚發性阿爾茨海默病全基因組關聯研究進展[J].中國臨床醫師雜志,2012,6(5):1278-1280.
[31]Kanehisa M,Goto S.KEGG:kyoto encyclopedia of genes and genomes[J].Nucleic Acids Res,2000,28(1):27-30.
[32]Anand Brown A,Ding Z,Vi?uela A,etal.Pathway based factor analysis of gene expression data produces highly heritable phenotypes that associate with age[J].G3(Bethesda),2015,5 (5):839-847.
[33]Kanehisa M,Goto S,Sato Y,et al.Data,information,know ledge and principle:back to metabolism in KEGG[J].Nucleic Acids Res,2014,42(Database issue):D199-D205.
[34]Nersisyan L,Samsonyan R,Arakelyan A.CyKEGGParser: tailoring KEGG pathways to fit into systems biology analysis workflows[J].F1000Res,2014,3(1):145-157.
[35]Bertram L,TanziRE.Genome-wide association studies in Alzheimer'sdisease[J].Hum MolGenet,2009,18(2):137-145.
Research Progressof Geneticsof A lzheim er's Disease(review)
WANGMei-qin1,YANG Kai-bing1,JIRan1,PANGQing-hua1,ZHANG Da-bao1,2,ZHANGMin1,2
1.Departmentof Bioinformatics,School of Biomedical Engineering,Capital Medical University,Beijing 100069, China;2.A lzheimer's Disease Center,Beijing Institute for Brain Disorders,CapitalMedicalUniversity,Beijing 100069, China
Genetic factors play an important role in the developmentof A lzheimer's disease(AD).This articlemainly introduced some genes,which have been reported in recentyears,predisposing to differentaspects of AD.The occurrence and progress of the late onsetAlzheimer's disease(LOAD)can be better understood through further study of the susceptibility loci,especially by using the pathway analysis, and the importanthub genes can be found so as to provide targets for clinical prevention,diagnosis and treatment.Due to the dispersion of genetic variant information,itneeds detailed literature,online search and some bioinformaticsmethods to analyze the causative genes and signaling pathways related to LOAD.3 databaseswere took as recourses,and they were analyzed withmulti-meansandmulti-waysof analysisand dataminingmethods,in order to provide theoreticalbasis for furtherstudy and clinical treatmentof AD genetics.
A lzheimer's disease;single nucleotide polymorphism;pathway;database;review
10.3969/j.issn.1006-9771.2015.12.001
R749.1
A
1006-9771(2015)12-1365-05
1.科技部 “973”項目(No.2014CB744604);2.北京市教委科技計劃面上項目(No.KM 201010025004;No.KM 201410025013);3.北京市腦重大疾病研究院基金項目(No.BIBDPXM 2014_014226_000016)。
1.首都醫科大學生物醫學工程學院生物信息學系,北京市100069;2.北京腦重大疾病研究院阿爾茨海默病研究所,北京市100069。作者簡介:王美琴(1990-),女,漢族,江蘇徐州市人,碩士研究生,主要研究方向:生物信息學。通訊作者:張敏(1970-),女,河北保定市人,博士,教授,博士研究生導師,長期從事生物信息大數據分析。E-mail:zhangmin@ccmu.edu.cn。
2015-02-09
2015-03-30)

